黄瓜DVR基因的生物信息学分析
洪蓓蓓
摘 要:联乙烯还原酶(divinyl reductase,DVR)将各种叶绿素中间物质的8-乙烯基转化为乙基,是生物合成叶绿素必不可少的一个关键酶。本文用生物信息分析方法对黄瓜DVR基因编码的氨基酸序列与组成、疏水性/亲水性、理化性质等进行分析与预测。结果表明DVR基因全长1260 bp,编码419个氨基酸,推测为亲水性的非跨膜类蛋白。根据同源序列的遗传距离得知黄瓜DVR与黄瓜(JX239753.1)同源基因关系最近,其次为香瓜,与鹰嘴豆基因关系最远。
关键词:黄瓜;联乙烯还原酶;生物信息学分析
黄瓜(Cucumissativus L. var.sativus)又称胡瓜,是葫芦科、甜瓜属植物,起源于喜马拉雅山南麓的热带雨林地区,是葫芦科甜瓜属一年生草本蔓生攀缘植物[1]。黄瓜雌雄同株,雄花:常数朵在叶腋簇生;花梗纤细;花冠黄白色,花冠裂片长圆状披针形。雌花:单生或簇生;花梗粗壮;子房粗糙。果实长圆形或圆柱形,熟时黄绿色,表面粗糙,花果期夏季[2]。黄瓜味甘性凉,具有清热利水,解毒的功效。黄瓜含水分为98%,并含有少量的维生素C、胡萝卜素、蛋白质、钙、磷、铁、维生素(VA、VB1、VB2、VC、VE)等人体必需的营养素[3]。食用方便,可生食或烹调用。现代药理学研究认为,黄瓜中含有的丙醇二酸能抑制糖类物质转化为脂肪,葫芦索C可抗癌,黄瓜汁有美容功效[4]。
联乙烯还原酶[5]是一种膜结合蛋白,它嵌入质体和叶绿体膜中,催化卟啉环上的C-8位乙烯基还原为乙基,是植物体内合成正常的MV-Chl必不可少的一个关键酶。在高等植物中各种DVR的基因活性是由一个基因编码的具有广谱底物专化性的DVR蛋白所催化,来源于不同物种的DVR蛋白的催化活性具有极显著的差异,并且即使是同一个DVR蛋白,对不同的联乙烯底物也可能具有显著不同的催化活性[6]。可见,对黄瓜联乙烯还原酶的结构和功能进行推断和预测具有一定的生物学意义,故本文将对黄瓜联乙烯还原酶的生物信息学进行分析。
1 材料与方法
1.1 实验材料
黄瓜DVR基因序列来源于实验获得,从在美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)核酸及蛋白质数据库中检索得到。同时通过检索与黄瓜DVR基因具有较为相近同源序列的基因:黄瓜(登录号JX239753.1)、香瓜Cucumismelo(登录号XM_008440580.1)、野草莓Fragariavesca subsp. vesca(登录号XM_004296747.2)、白梨Pyrus xbretschneideri(登录号XM_009347536.1)、白梨Pyrusx bretschneideri(登录号XM_009347528.1)、白梨Pyrus x bretschneideri(登錄号XM_009370831.1)、白梨Pyrus xbretschneideri(登录号XM_009353937.1)、鹰嘴豆Cicerarietinum(登录号XM_004486002.2)。
1.2 实验方法
通过软件primer 5.0获得相应的氨基酸序列[7];MEGA3.1软件采用Neighbor-Joining的方法进行进化树分析
[8];通过npsa-prabi在线工具(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)进行二级结构预测;通过Protscale(http://expasy.org/tools/protscale.html)预测分析蛋白质功能和疏水性/亲水性[9];利用CBS网站TMHMM Serverv.2.0(http://www.cbs.dtu.dk/services/TMHMM/)在线工具对氨基酸序列进行跨膜分析预测[10]。
2 结果与分析
2.1 黄瓜DVR基因序列的分析
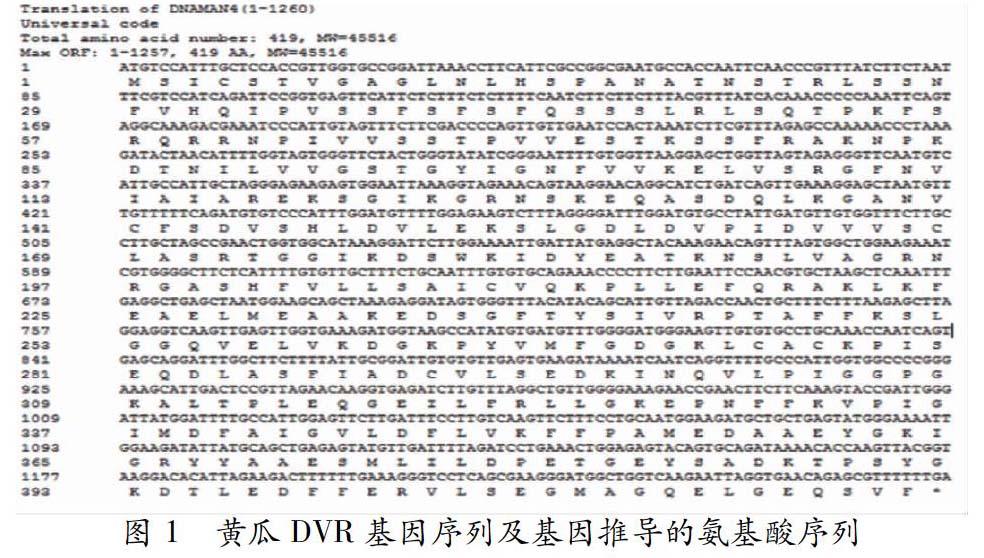
根据DNAMAN软件分析可知,DVR基因序列全长1260pb,分别以ATG和TGA为起始密码子和终止密码子,共编码419个残基(图1),编码的蛋白质分子质量为45516 Da。
2.2黄瓜DVR及其同源序列的分析进化树
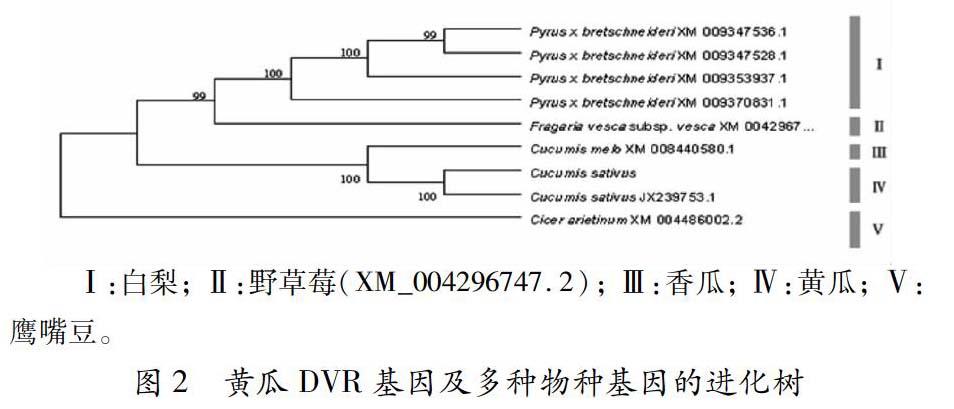
根据MEGA软件分析,将进化树分为五段,分别是四种白梨、野草莓、香瓜、两种黄瓜和鹰嘴豆(图2)。黄瓜DVR与黄瓜(JX239753.1)的同源基因关系最为接近,几乎完全相同,其次为香瓜,测得黄瓜与香瓜的同源距离约为0.029,与鹰嘴豆基因关系最远,测得它们之间的距离达到0.357。
2.3 黄瓜DVR蛋白二级结构预测
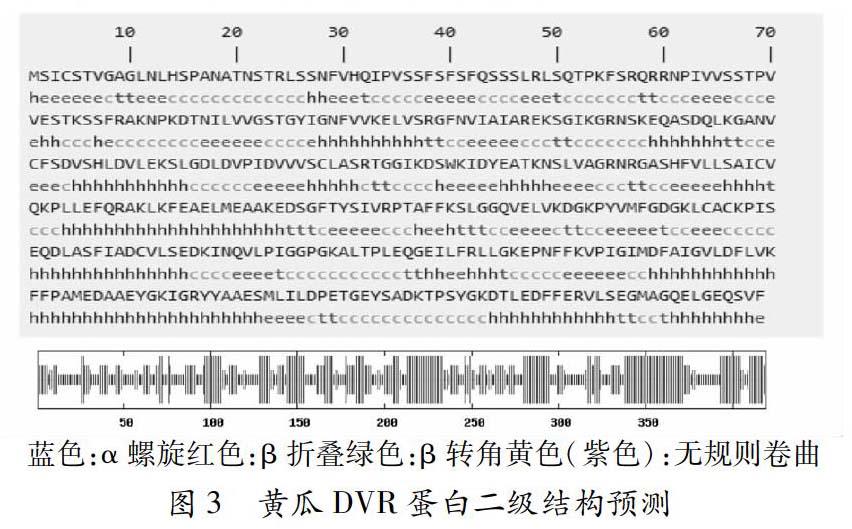
通过sopma在线软件预测可知,该蛋白由419个氨基酸组成,其中α螺旋和无规则卷曲所占比例最高,α螺旋有146个,占总数的34.84%,无规则卷曲有140个,占总数的33.41%。β折叠数量略少于无规则卷曲,但远多于β转角(图3)。
2.4 黄瓜DVR蛋白跨膜结构分析
跨膜结构域一般富含疏水性氨基酸残基,起着固系于细胞膜中的“抛锚”作用,具有跨膜结构域的蛋白属于跨膜蛋白类。通过TMHMM在线软件预测可知,横坐标表示氨基酸残基位置,纵坐标表示残基具有相应结构的可能性, 结果显示,联乙烯还原酶蛋白没有检测到跨膜区,可能不是跨膜蛋白,联乙烯还原酶蛋白极可能为覆盖蛋白(图4)。
2.5 黄瓜DVR蛋白的亲疏水性预测
用protscale软件分析可知,图中大于零的氨基酸为疏水性氨基酸,小于零的氨基酸为亲水性的氨基酸。通过预测可知,组成联乙烯还原酶蛋白的氨基酸中高亲水性的氨基酸的位点有两个(图5),分别是57和58,分值都是-2.667;组成联乙烯还原酶蛋白的氨基酸中高疏水性的氨基酸的位点206,分值是2.978。
3 讨论
本实验预测得黄瓜联乙烯还原酶蛋白为亲水性的非跨膜类蛋白,通过对蛋白质二级结构的预测可知,黄瓜联乙烯还原酶蛋白二级结构以α螺旋和无规则卷曲为主;根据同源序列的遗传距离得知黄瓜DVR与黄瓜(JX239753.1)同源基因关系最近,与鹰嘴豆基因关系最远。
绝大多数绿色植物进行光合作用离不开叶绿体的存在[11],而联乙烯还原酶是生物合成叶绿素必不可少的一个关键酶,迄今已在高等植物中检测到5种DVR活性[12]。联乙烯还原酶具有活性,对叶绿素的生物合成途径具有十分重要的意义
[13]。联乙烯蛋白酶基因的突变可能会引起联乙烯还原酶失活,对植物的生存、生长和繁殖造成较大的影响
[15]。应用生物信息学方法对已知黄瓜DVR序列进行比对、分析,从而对其结构和功能进行推断和预测,这可以为我们在开展试验研究前提供尽可能多的信息,从而为选择合适的试验方法提供理论参考,为进一步对该基因的功能研究提供线索。
[参考文献]
[1]闫世江, 张继宁, 刘洁. 低温对黄瓜伤害的研究进展[J]. 蔬菜, 2010, 5(10):31-34.
[2] 陈惠明, 刘晓虹. 黄瓜性型遗传规律的研究[J]. 湖南农业大学学报:自然科学版, 1999, 25(1):40-43.
[3] 孙洪涛. 黄瓜果实横径遗传分析及分子标记[D]. 哈尔滨: 东北农业大学, 2010.
[4] 曾维华. “黄瓜”始名考[J]. 上海师范大学学报:哲学社会科学版, 2000, 29(4): 313-315.
[5] Nielsen J G, Newman M, Nielsen H. Control and testing of a dynamic voltage restorer (DVR) at medium voltage level [J]. IEEE Transactions on Power Electronics, 2004, 19(3):806-813.
[6] 王平榮, 邓晓建. 高等植物叶绿素生物合成的联乙烯还原酶及编码基因研究进展 [J]. 西北植物学报, 2013, 33(4):843-849.
[7] 王江波. 中国水仙LTR反转录转座子研究及IRAP、REMAP分子标记的开发 [D]. 福州: 福建农林大学, 2012.
[8] 李凤梅, 盖雪梅. 半胱氨酸蛋白酶抑制剂的系统发生分析(英文) [J]. 农业科学与技术, 2010, 14(2): 44-54.
[9] 廖文彬, 崔百明, 温玮. 赤霉素负调控因子GhRGL(RGL-LIKE)基因序列与功能预测分析 [J]. 热带作物学报, 2009, 30(2): 773-781.
[10] 张繁. 结核分枝杆菌哺乳动物细胞入侵因子mce家族Rv0590A基因的性质及功能研究 [D]. 重庆: 西南大学, 2011.
[11] 兰金苹, 李莉云, 贾霖, 曹英豪, 白辉. 叶绿体基因编码蛋白质在水稻叶片生长过程中的表达研究[D]. 保定: 河北农业大学, 2011.
[12] 王平荣. 水稻824ys黄绿叶突变基因的图位克隆及功能分析[D]. 雅安: 四川农业大学, 2010.
[13] 王平荣, 马晓智, 李春梅, 孙昌辉, 邓晓建. 三个水稻联乙烯还原酶基因突变体的比较研究 [J]. 中国农业科学, 2013, 46(7):1305-1313.
(作者单位:台州学院 英语系,浙江 临海 317000)

