澳洲坚果SSR体系的建立及F1代的鉴定
李玉宏 倪书邦 贺熙勇 马静

摘 要 为了探索澳洲坚果SSR-PCR的可行性,采用单因素分析法对影响PCR的各个因素进行分析,建立SSR-最佳反应体系。结果表明:在25 μL PCR反应体系中,当Mg2+浓度、dNTPs浓度、引物浓度、Taq DNA聚合酶用量分别为2.50 mmol/L、0.10 mmol/L、0.40 μmol/L、1.25 U时,PCR扩增效果最好。筛选到19对可得到扩增结果的SSR引物,其中有2对在以900为母本、294为父本的组合中PCR扩增结果存在差异,可以应用于900×294组合的F1代鉴定。
关键词 澳洲坚果 ;分子标记 ;SSR
中图分类号 S664.9 文献标识码 A Doi:10.12008/j.issn.1009-2196.2016.09.007
Abstract In order to explore the feasibility of SSR-PCR in Macadamia, a singular factor test was used to optimize SSR-PCR amplification system for macadamia. The results showed the optimal PCR reaction system was 2.50 mmol/L Mg2+, 0.10 mmol/L dNTPs, 0.40 μmol/L primers, and 1.25 U Taq DNA polymerase in a volume of 25 μL. Nineteen pairs of SSR primers with amplifications were obtained in SSR-PCR, but only two pairs of primers had different results in PCR amplification in the combination of 900 as female and 294 as male parents. These two pairs of primers can hence be used to identify the F1 generation of the 900×294 combination. Therefore the SSR-PCR could be used for identification of hybrid progeny in macadamia.
Keywords Macadamia ; molecular marker ; SSR
澳洲坚果(Macadamia spp.)属山龙眼科(Proteacase)澳洲坚果属(Macadamia)常绿乔木。原产于澳大利亚昆士兰州东南部和新南威尔士州北部、南纬25°~32°之间的沿海亚热带雨林[1]。素有“干果之王”的誉称。中国于20世纪70 年代引种试种澳洲坚果,在云南、海南、广东、广西、福建、浙江等省(区)引种试种,种植面积近5 400 hm2,产量约600 t,其中种植规模最大的是云南,其他省(区)还处于零星试种阶段[2]。
目前,有关澳洲坚果性状的研究主要集中在形态学的特征性描述与遗传多样性研究。形态标记和生化标记也已应用于澳洲坚果的品种鉴定、分类和亲缘关系研究等方面[3-4]。由各种DNA分子标记衍生的DNA指纹技术在鉴定品种等方面具有高效、准确的特点[5-6]。Steiger等[7-9]利用AFLP技术在澳洲坚果上检测到了丰富的多态性,RAF标记表明澳洲坚果种质资源具有丰富的遗传多样性[10];Peace等[8]利用PAPD与SSR对澳洲坚果种质进行了遗传多样性研究,与其后Vithanage等[9]采用同工酶研究结果一致,表明DNA标记对澳洲坚果的杂交育种有辅助作用。近年来,一些研究显示DNA分子标记应用于澳洲坚果中具有高度的一致性和可信度[11]。虽然分子标记在澳洲坚果种质资源鉴定上的应用还处于初步阶段,但杂交育种已广泛应用在澳洲坚果选育种中。本课题虽在澳洲坚果杂交育种方面做了大量工作,也得到了大量的杂交子代,但其子代只能使用传统的形态学鉴定方法,完成鉴定必须等到开花结实,周期长达8 a,耗费大量的人力和财力。为了提高育种效率,迫切需要一种快捷简易的鉴定方法。而SSR标记具有共显性的特征,原则上子代将同时含有父母本的特征性条带。为此,本实验选择SSR标记方法,研究SSR标记在澳洲坚果子代鉴定中的可行性。
本研究采用SSR分子标记技术,以900×294组合及其子代为研究对象,采用单因素分析法对影响SSR-PCR的各个因素进行分析,建立SSR-PCR反应体系,并初步探讨SSR-PCR标记在澳洲坚果亲本标记和杂交子代鉴定方面的可行性,为澳洲坚果分子标记辅助育种提供理论依据。
1 材料和方法
1.1 材料
采集母本900、父本294及杂交子一代幼叶于液氮中保存。主要试剂有:引物(上海生工合成);十六烷基三甲基溴化铵(CTAB)、十二烷基磺酸钠(SDS)、三羟甲基氨基甲烷(Tris)、乙二胺四乙酸(EDTA)、琼脂糖、硝酸银等从上海生工购买;Taq DNA聚合酶等PCR试剂为TAKARA产品;氯仿、异丙醇、无水乙醇、丙三醇、饱和酚等为国产分析纯。
1.2 方法
1.2.1 DNA提取
采用改良CTAB法提取基因组DNA[12]。
1.2.2 琼脂糖凝胶电泳检测
配置1%琼脂糖胶,核酸染料采用goldviewⅡ,每100 mL加5 μL染料,电泳条件为150 V,30 min,结束后置于紫外灯下观察提取的DNA质量。
1.2.3 SSR引物的筛选
从NCBI里查找澳洲坚果DNA系列,用SSRHunter 1.3查找到可能的SSR系列,再用primer 5设计100对引物,送上海生工合成。将合成的引物用灭菌的去离子水溶解至10 μmol/L。采用25 μL PCR反应体系,包括10×PCR buffer(Mg2+ free)2.5 μL、dNTPs(10 mmol/L)0.5 μL、MgCl2(25 mmol/L)2 μL、Primer(10 μmol/L)1 μL、Taq(5 U/μL)0.5 μL、DNA1 μL,用ddH2O补充到25 μL。在确定最适反应体系时,在不改变其他组分的条件下,分别对反应体系各组分浓度进行调整。将最终确定的最佳反应体系用于SSR引物的筛选。PCR 反应为:94℃预变性3 min,94℃变性30 s,最适Tm退火值 30 s;72℃延伸60 s,再重复2~4步,30个循环,后于72℃延伸5 min。PCR 反应产物用琼脂糖凝胶预检测其扩增效果,并用梯度PCR确定每对引物的最适退火温度。调整好最适退火温度,分别以父本、母本为模板再次进行PCR 扩增,PCR产物用聚丙烯酰胺凝胶电泳检测,找出能区分父本和母本的特异性引物和扩增结果良好的引物(聚丙烯酰胺凝胶电泳分辨率高)。
1.2.4 F1代鉴定
子代F1和母本900、父本294同时进行PCR扩增(反应体系和条件以上面确定的一致),将扩增后的PCR 产物先用琼脂糖凝胶检测,以确定PCR扩增是否成功,然后进行聚丙烯酰胺凝胶电泳检测,硝酸银染色后拍照。
2 结果与分析
2.1 澳洲坚果DNA提取
DNA作为本研究的直接材料,其纯度和完整性是进行SSR-PCR试验的前提条件。本研究采用CTAB法提取DNA,采用琼脂糖凝胶电泳检测(图1)。从图1可看出,所得DNA样本条带清晰,完整度好,且没有RNA污染和降解。说明DNA质量达到PCR的基本要求。
2.2 确定SSR-PCR扩增的最适Mg2+浓度
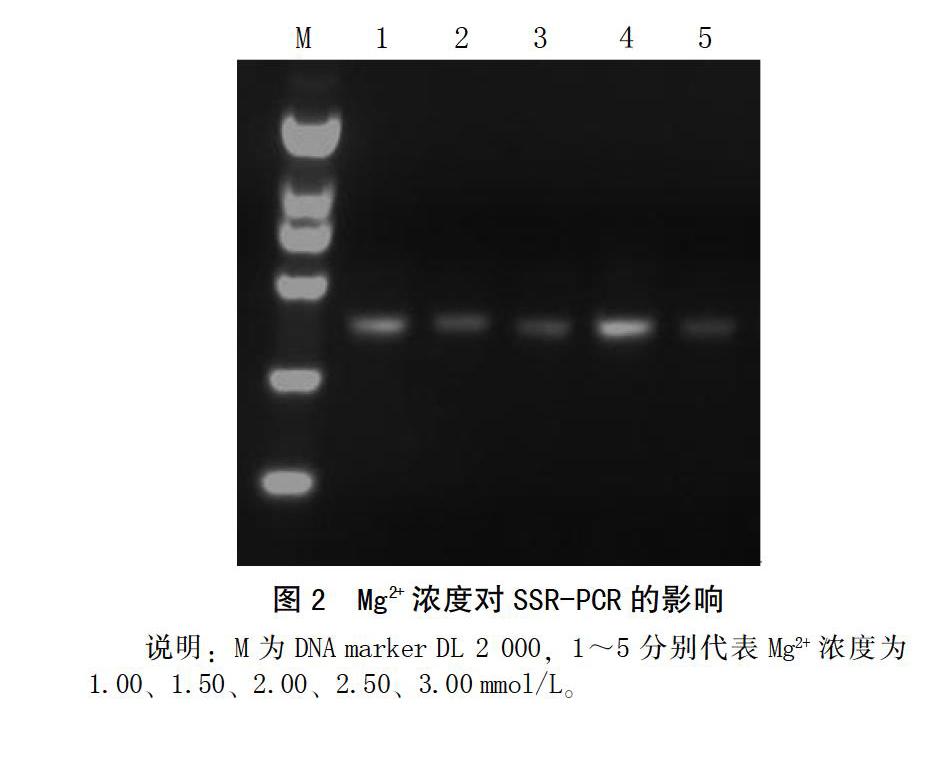
由于Taq DNA聚合酶是镁离子(Mg2+)依赖性酶,因此Mg2+浓度对PCR扩增有显著影响,在一般的PCR反应中,Mg2+的工作浓度为1.5~2.0mmol/L。Mg2+浓度过高,反应特异性降低,出现非特异性扩增,但Mg2+浓度过低又会降低Taq DNA聚合酶的活性,得不到扩增产物。本实验设计了1.00、1.50、2.00、2.50、3.00 mmol/L共5个浓度梯度。从图2可看出,各个Mg2+浓度下都可得到清晰的扩增条带,但Mg2+浓度为2.50 mmol/L时条带最亮,因此本实验中最适Mg2+浓度为2.50 mmol/L。
2.3 确定SSR-PCR扩增的最适dNTPs浓度
dNTPs浓度直接影响PCR产物的量,dNTPs浓度过高,会导致聚合酶错配,过低又会降低PCR扩增效率。在一般PCR体系中,dNTPs浓度为0.1 mmol/L。本实验选取0.050、0.075、0.100、0.125、0.150 mmol/L 5个dNTPs浓度进行PCR扩增。从图3可看出,随着dNTPs浓度的增加,得到的PCR产物量也增加,但当dNTPs浓度超过0.1 mmol/L时,PCR产物量的增加不明显,因此本实验中最适dNTPs浓度为0.1mmol/L。
2.4 确定SSR-PCR扩增的最适引物浓度
引物浓度直接影响PCR扩增结果,引物的浓度太低时,扩增产物量太少,但引物浓度太高时,比较容易出现非特异性扩增反应。PCR中引物的浓度一般在0.1~1.0 μmol/L。因此本实验选择0.1、0.2、0.4、0.6、0.8 μmol/L 5个浓度。从图4可看出,当引物浓度为0.4 μmol/L时,SSR-PCR的效果最好。
2.5 确定SSR-PCR扩增的最适Taq DNA聚合酶浓度
Taq DNA聚合酶是PCR反应中的重要因素,用量过多会引起非特异性扩增,过少又使产物量减少。本实验选择0.5、0.75、1.00、1.25、1.50 U 5个浓度。从图5可看出,随着Taq DNA聚合酶量的增加,PCR扩增条带亮度呈上升的趋势,但当Taq DNA聚合酶用量达到1.00 U后,扩增条带亮度上升不明显,从节约的角度出发,选择1.00 U为本次实验中的使用浓度。
2.6 SSR引物的筛选
由上述SSR-PCR体系优化实验,确定最适PCR体系,即10×PCR Buffer(Mg2+ free)2.5 μL、dNTPs(10 mmol/L)0.25 μL、MgCl2(25mmol/L)2.5 μL、Primer F(10 μmol/L)1 μL、Primer R(10 μmol/L)1 μL、Taq(5 U/μL)0.25 μL、DNA 1 μL、ddH2O 16.5 μL。由这一PCR反应体系,以900和294植株的DNA为模板,从100对SSR引物中筛选到具有扩增产物的引物19对,再通过梯度PCR确定每对引物的最佳退火温度,筛选结果见表1。
2.7 澳洲坚果杂交F1代的鉴定
确定退火温度后,再一次用900(母本)、294(父本)及其杂交子代F1为DNA模板进行PCR扩增,其PCR产物用10%聚丙烯酰胺凝胶电泳检测,以硝酸银染色,结果显示只有引物15和引物17在此组合中存在特异性条带(图6)。从图6可看出,两对引物所得结果具有同一性,因此SSR标记可以作为澳洲坚果杂交子代鉴定的方法。
3 讨论
虽然SSR标记技术已经广泛应用于作物的辅助育种,但其原理是基于PCR,会受到模板DNA质量、dNTPs浓度、Mg2+浓度、Taq DNA聚合酶浓度以及引物浓度等因素的影响。在本实验中,当各个因素水平较低或较高时,PCR扩增效果都不好,因此对SSR体系的优化很有必要。
传统的澳洲坚果资源鉴定方法是形态性和农艺学性状,即进行性状调查,通过株型、叶型、果型、花色、果色等表型性状对其进行鉴定,费时费力且易受环境因素的干扰,其他作物上研究表明,分子标记是行之有效的遗传多样性的鉴定方法。本实验利用SSR标记技术成功进行了澳洲坚果杂交F1代的鉴定,说明SSR标记技术可以代替传统的鉴定,克服田间调查周期长、易受环境干扰等问题。此外本实验从100对SSR 引物中仅筛选出2对有效的引物,其利用率低。SSR引物的开发主要决定于遗传基因的多态性,但澳洲坚果的不同品种间由于亲缘关系较近,SSR表现出来的多态性都较差,因此澳洲坚果SSR 引物开发难度比较大。但是随着DNA测序技术的发展,更多SSR位点被发现,开发引物的难度将会大大降低,相信在将来SSR标记技术将会广泛应用于澳洲坚果子代鉴定上,成为育种工作的重要辅助方法。
参考文献
[1] 郭凌飞,邹明宏,曾 辉,等. 澳洲坚果ISSR-PCR反应体系的建立与优化[J]. 林业科学,2008,5:160-164.
[2] 云南澳洲坚果产业调查组. 云南省澳洲坚果产业发展现状、存在问题及建议[J]. 热带农业科技,2007,30(1):10-14.
[3] 岳 海,陈丽兰,陈国云,等. 云南省澳洲坚果品种鉴别分析研究[J]. 西南农业学报,2008,21(5):1 395-1 400.
[4] 姚宏伟,张立冬,孙金阳,等. DNA分子标记技术概述[J]. 河北渔业,2010,07:42-46.
[5] 刘爱群,赵丽丽,王国政,等. 分子标记鉴定葫芦科瓜类作物种子纯度的研究进展[J]. 长江蔬菜,2012(8):6-10.
[6] 梁 勇,李焕秀,何 艳,等. 几种DNA分子标记在种子纯度鉴定上的应用进展[J]. 广西农业科学,2008,39(1):17-20.
[7] Steiger D L, Moore P H, Zee F, et al.Genetic relationships of macadamia cuhivars and species revealed by AFLP marker[J]. Euphytica, 2003, 132:269-277.
[8] Peace C P, Allan P, Vithanage V, et al.Progress made with molecular markers for genetic improvement of Macadamia[J]. Acta horticulturae, 1998, 461: 199-207.
[9] Vithange V, Peace C P, Carroll B J, et al.Progress made with molecular markers for genetic improvement of Macadamia[J]. Acta horticulturae, 1998, 461: 199-207.
[10] 郭凌飞,邹明宏,杜丽清,等. 利用ISSR和分析澳洲坚果的亲缘关系[J]. 园艺学报,2011,38(9):1 741-1 746.
[11] 郭凌飞,邹明宏,曾 辉,等. 利用改良CTAB法提取澳洲坚果成熟叶片高质量DNA[J]. 分子植物育种, 2007,5(6):187-190.

