灵芝DNA甲基转移酶(GlMET1)基因克隆及原核表达诱导分析
查良平 王亚君 刘爽 赵玉洋 袁媛 黄璐琦


[摘要]DNA甲基转移酶是DNA甲基化过程中最关键的酶,对生物体内的基因表达调节起着重要作用。研究根据灵芝转录组数据,通过全基因合成首次克隆得到灵芝DNA甲基转移酶GlMET1,Genbank注册号为KU239998,并对其基因特性和空间结构进行全面的生物信息学分析。原核表达诱导分析表明pET28a(+)GlMET1重组质粒在BL21(DE3)中能成功表达出目的蛋白,对其诱导条件进行优化,得出蛋白最合适的表达条件为:诱导温度为16 ℃,重组菌生长25 h(A600为08),IPTG浓度为02 mmol·L-1,诱导时间为12 h。实时荧光定量PCR结果表明不同品种灵芝GlMET1的基因表达水平均有明显差异,且GlMET1在成熟期的表达水平均低于幼年期,说明GlMET1表达量随着灵芝的生长发育呈下降趋势。该研究结果为深入研究DNA甲基转移酶蛋白的作用机制奠定了基础。
[关键词]灵芝;甲基化;甲基转移酶;基因克隆;原核表达
表观遗传修饰是指不改变DNA序列的可遗传的对碱基和组蛋白的化学修饰,主要包括DNA甲基化、组蛋白修饰、染色质重塑以及非编码RNA等[1]。DNA甲基化作为一种重要的表观遗传修饰,在生物体内的基因表达调节方面起着重要作用,如参与转座子沉默,基因印迹和X染色体失活等生物学过程。DNA甲基化是在DNA甲基转移酶的催化下完成的,甲基转移酶将SAM的甲基基团转移到胞嘧啶的5位碳原子上从而完成甲基化过程[2]。DNA 甲基化反应的催化剂DNA 甲基转移酶(DNA methyltransferase,Dnmt) 在染色质重塑和基因表达调控中起着关键作用。目前在植物中至少已经发现3种DNA甲基转移酶,即甲基转移酶l (methyltransferas,METl)、结构域重排甲基转移酶(domains rearranged methyltransferas,DRM)和染色质甲基化酶(chromomethylase 3,CMT3) [35]。MET1被认为是具有统治地位的 DNA 甲基转移酶,它具有使核苷酸序列中CG碱基的胞嘧啶的甲基化的作用,同时METl能够影响植物的形态特征、调控开花的时间等[6]。
中药灵芝G lucidum是食药兼用的一种真菌植物,含有多糖、萜类化合物、生物碱、核苷、氨基酸多肽和微量元素等多种活性成分,其中灵芝多糖和三萜类化合物是灵芝的主要活性成分,研究表明它们具有抗肿瘤、抗氧化、抗衰老和降血糖等多种生物活性[79]。自20世纪80年代以来,对灵芝的分子生物学、次生代谢产物和药理等均进行了深入的研究,尤其是围绕灵芝三萜酸生物合成相关基因的筛选、克隆和功能分析已有大量报道,转录组和基因组信息为研究灵芝基因组多样性奠定了基础,灵芝被认为是研究真菌次生代谢的理想模式物种[1011]。目前已从多种植物中分离克隆出METl酶基因,如拟南芥[12]、玉米[13]、草莓[14]、马铃薯[15]等,但药用植物的MET1酶基因报道较少。本研究首次从灵芝转录组中筛选得到灵芝DNA甲基转移酶基因GlMET1,采用全基因合成克隆得到其cDNA序列,对其基因特性和空间结构进行全面的生物信息学分析,并构建pET28a(+)GlMET1重组质粒,转换至表达菌BL21( DE3)中,对影响蛋白表达的诱导温度、诱导时间、IPTG浓度及宿主菌吸光度等4个因素进行了优化,同时采用实时荧光定量PCR检测不同品种和不同生长时期的GlMET1表达水平,为进一步研究DNA甲基转移酶基因的功能及其在灵芝发育过程中的调控功能奠定基础。
1材料与方法
11药材
霍芝1号采自于安徽省六安市霍山县衡济堂公司,赤芝10号采自于安徽省六安市霍山县一品堂公司,金芝1号采自于安徽省六安市金寨县乔康药业,野生灵芝采自于安徽省金寨县铁冲乡。经安徽中医药大学药学院彭华胜教授鉴定,样品经鉴定后保存于-80 ℃冰箱。
12菌株、载体和试剂
大肠杆菌BL21(DE3)为本实验室保存。原核表达载体pET28a(+)为本实验室保存;DNA测序由北京睿博生物科技公司完成,寡核苷酸引物由上海生工生物工程公司合成。限制性内切酶购自NEB有限公司。
13总RNA 提取及cDNA的合成
称取100 mg左右灵芝新鲜的子实体,加液氮置于球磨机上研磨,采用CTAB法从新鲜的子实体中提取总RNA,1%的琼脂糖凝胶电泳用于评价RNA的质量。ND2000测定总RNA的A260,A280,选择A260/A280为18~20的总RNA反转录成cDNA。
14GlMET1全基因合成及原核表达载体构建
灵芝DNA甲基转移酶1(GlMET1)的基因来源于灵芝转录组数据库。选择整条序列的3 554个碱基作为克隆的对象,在引物的两端各设计了BamHI和SalI酶切位点和保护性碱基,序列总全长为3554 bp,全基因合成由江苏苏州泓迅生物科技有限公司完成。对带有酶切位点的GlMET1和pET28a(+)载体分别进行双酶切,利用T4 DNA连接酶进行连接,25 ℃连接3 h。将连接产物转化至DH5α感受态细胞,在含有卡那霉素的LB培养板上37 ℃。挑单克隆菌液PCR,双酶切和测序鉴定。
15GlMET1的生物信息学分析
将测序获得的序列结果使用ORF Finder (http://wwwncbinlmnihgov/gorf/gorfhtml)查找开放阅读框(ORF)。利用在线工具Protparam (http://wwwexpasych/tools/protparamhtml)预测基因编码蛋白的相对分子量、氨基酸数目、等电点、不稳定系数、脂肪指数、亲水性/疏水性和编码区全长等理化性质;采用CDD(http://wwwncbimlmnihgov/Structure/cdd/wrpsbcgi)进行蛋白质结构域分析;采用CFSSP(http://wwwbiogemorg/tool/choufasman/)进行蛋白质二级结构分析;利用Swiss Model(http://swissmodelexpasyorg/)程序,根据基因氨基酸序列进行建模,预测蛋白质的三级结构。用DNAMAN软件对序列进行多重比对以及限制性酶切位点的预测,用ClustalW软件与其他植物的氨基酸序列进行比较,用MEGA 606软件构建Neighborjoining系统进化树,bootstrap 重复次数为1 000次。
16pET28a(+)GlMET1的原核表达及条件优化
将构建好的pET28a(+)GlMET1质粒转化到BL21(DE3)表达感受态中,进行转化表达宿主菌。取转化表达菌液按照1∶100加到含卡那霉素抗性的LB培养液中,37 ℃ 250 r·min-1振荡培养约25 h至A600 04~06,加IPTG于16 ℃低温诱导12 h,收集菌体,加入等体积的2×loading buffer 煮沸,上样,用10%的聚丙烯酰胺凝胶进行SDSPAGE分析。针对诱导温度、诱导时间、IPTG浓度,及IPTG添加时间( 即诱导起始菌液浓度A600) 等4个因素对原核表达产物的积累有一定影响,分析了这4个因素对灵芝DNMT1重组蛋白表达的影响。
17Realtime PCR
从转录组中数据中获取灵芝内参照GlGPD和GlMET1的核苷酸序列,利用Primer Premier 50设计实时荧光定量引物,扩增产物长度在100~250 bp,送由生工生物工程(上海)公司合成,其中GPD上游引物为5′CGCTCAACAAGAACTTCGTCAA3′,GPD下游引物为5′CGTAGACAAGGAGGTCACAGA3′,GlMET1上游引物为5′ATCGATCCAACGGCAAAG3′,GlMET1下游引物为5′CACAGAAGACGAACGAATCC3。反应体系: 5 μL 2×SYBR green,02 μL ROX Reference Dye,02 μL引物1,02 μL引物2,10 μL cDNA,34 μL H2O。PCR 反应条件为: 95 ℃ 30 s; 95 ℃ 5 s,60 ℃ 34 s,40 个扩增循环;dissociation stage。在ABI 7500荧光定量PCR仪上进行荧光定量,结束后分析熔解曲线。各基因表达量以内参基因作为标准进行相对定量,每个反应重复3次,相对定量方法采用2-ΔCT法分析结果。
2结果
21GlMET1基因的全基因合成及生物信息学分析
依据灵芝转录组数据获得灵芝GlMET1的cDNA,利用DNAMAN软件结合ORF Finder在线软件对GlMET1基因全长cDNA 序列进行分析, 预测其含3 554 bp完整的开放阅读框,通过全基因合成技术完成GlMET1的克隆,其测序结果与预测结果相一致。NCBI BLASTX 显示GlMET1与污叉丝孔菌Dichomitus squalens 65%相似,云芝Trametes versicolor 46%相似,粉孢革菌Coniophoraputeana 42%相似。比对表明所获基因属于MET1家族,将该基因命名为GlMET1,GenBank注册号为KU239998。
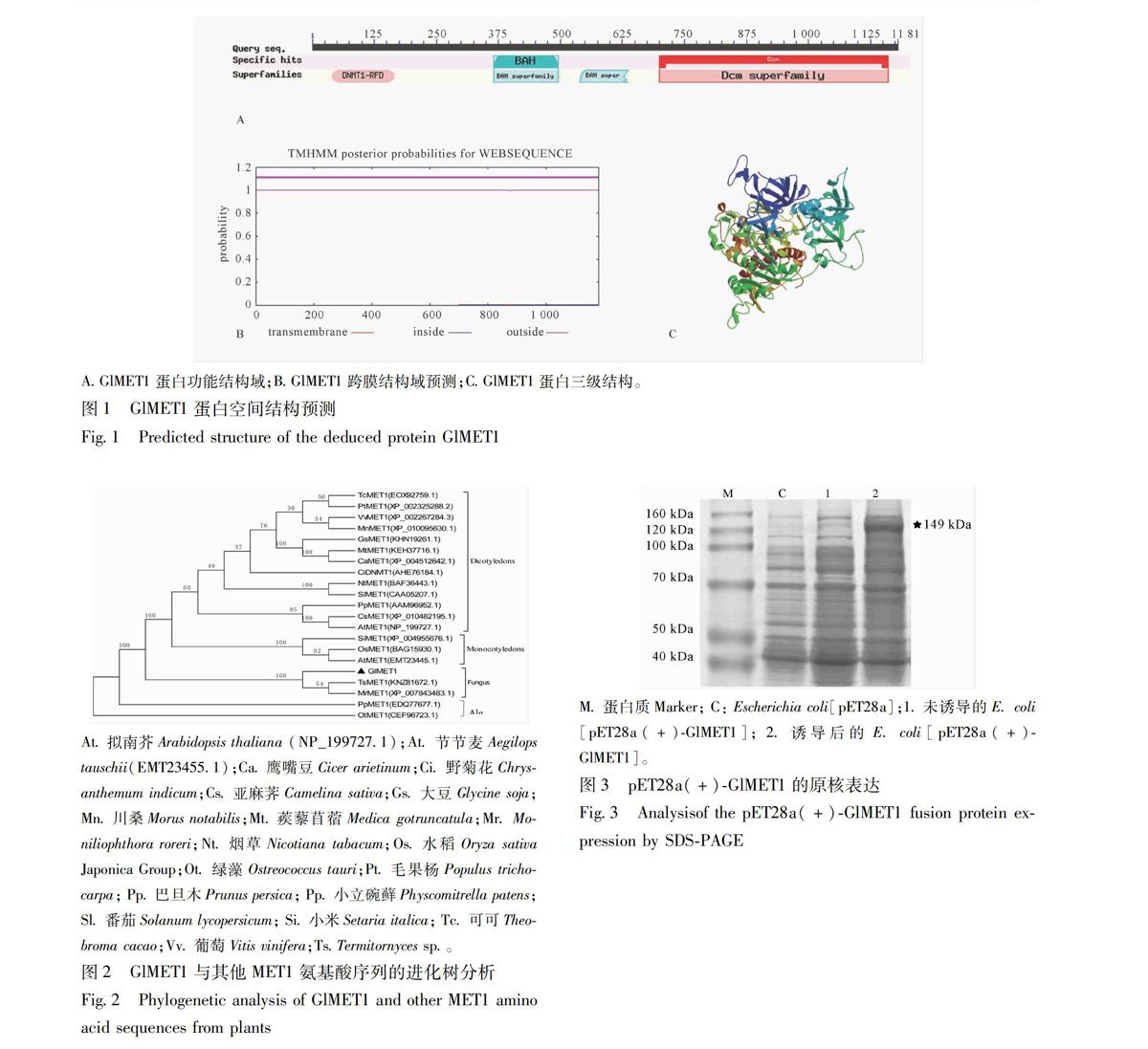
利用NCBI的 Conserved domains在线工具分析GlMET1蛋白的功能结构域,结果表明GlMET1具有4个功能域:39~166 aa的肽端与DNMT1RFD super family有较高的同源性,365~496 aa与39~637 aa均为保守的BAH结构域,700~1 160 aa与Dcm家族有较高的同源性。利用在线生物学工具TMHMM20 Server对进行蛋白跨膜结构域预测分析,结果显示无跨膜区。使用ExPASy在线服务器的SWISSMODEL Homology Modeling对GlMET1蛋白进行同源建模,得到了蛋白的三维空间模型,GlMET1蛋白模型4yoc1A得分为062,蛋白序列的相似性为2599%,见图1。
22GlMET1序列家族同源基因的系统进化分析
将GlMET1与GenBank中20种其他植物的同源蛋白进行比对, 在软件MEGA 606平台上采用相邻连接法构建进化树, 进行聚类关系分析,见图2。从进化树中,可以得知不同类植物在进化树中分别聚成不同的分支,双子叶植物的GlMET1聚为一个分支,进而与单子叶植物的MET1聚为一个大的分支,与GlMET1亲缘关系最近的为真菌植物Termitornyces sp和Moniliophthora roreri, 藻类植物的MET1与所有植物亲缘关系最远。
23pET28a(+)GlMET1原核诱导表达
将构建的pET28a(+)GlMET1质粒转化至表达菌BL21( DE3) 中,加入IPTG诱导表达,所得菌体经离心煮沸后进行SDSPAGE蛋白电泳分析,见图3。结果表明在149 kDa处出现目的蛋白条带,GlMET1基因的cDNA由3 545个碱基组成,编码133 kDa蛋白,原核表达载体pET28a上的HisTag标签大小为16 kDa,所以重组表达载体编码蛋白大约为149 kDa; 而阴性对照E coli[pET28a]诱导表达后在149 kDa处没有条带出现,表明含有GlMET1基因的重组质粒,在大肠杆菌BL21(DE3)中成功表达。
At拟南芥Arabidopsis thaliana (NP_1997271);At节节麦Aegilops tauschii(EMT234551);Ca鹰嘴豆Cicer arietinum;Ci野菊花Chrysanthemum indicum;Cs亚麻荠Camelina sativa;Gs大豆Glycine soja;Mn川桑Morus notabilis;Mt蒺藜苜蓿Medica gotruncatula;Mr Moniliophthora roreri;Nt烟草Nicotiana tabacum;Os水稻Oryza sativa Japonica Group;Ot 绿藻Ostreococcus tauri;Pt毛果杨Populus trichocarpa; Pp巴旦木Prunus persica; Pp小立碗藓Physcomitrella patens;Sl番茄Solanum lycopersicum; Si小米Setaria italica; Tc可可Theobroma cacao;Vv葡萄Vitis vinifera;TsTermitornyces sp。
24pET28a(+)GlMET1表达条件的优化
241诱导温度对pET28a(+)GlMET1蛋白表达的影响保持诱导时间12 h, IPTG浓度为04 mmol·L-1, A600为06不变,观察不同诱导温度16,20,25,30,37 ℃对pET28a(+)GlMET1蛋白表达的影响,见图4。温度为16,20 ℃时,pET28a(+)GlMET1有表达,且诱导温度为16 ℃时的表达量更高,但随着温度继续上升,pET28a(+)
M蛋白质Marker; C: Escherichia coli[pET28a];1未诱导的E coli[pET28a(+)GlMET1]; 2诱导后的E coli[pET28a(+)GlMET1]。
GlMET1在25,30,37 ℃均未产生表达,表明该蛋白适合低温诱导,最合适温度为16 ℃。
M.蛋白质Marker; C.Escherichia coli[pET28a];15E coli[pET28a(+)GlMET1],诱导温度分别为16,20,25,30,37 ℃。
242诱导时间对pET28a(+)GlMET1蛋白表达的影响保持诱导温度16 ℃,IPTG浓度为04 mmol·L-1,A600为06不变,观察不同诱导时间2,4,8,12,24 h对pET28a(+)GlMET1蛋白表达的影响,见图5。随着诱导时间增加,pET28a(+)GlMET1表达呈增加趋势,在诱导时间为12 h时其表达量最高,说明诱导时间有利于目的蛋白的表达。
M.蛋白Marker; C.Escherichia coli[pET28a];1~5E coli[pET28a(+)GlMET1],诱导时间分别为2,4,8,12,24 h。
243IPTG浓度对pET28a(+)GlMET1蛋白表达的影响保持诱导温度16 ℃,诱导时间12 h,A600为06不变,观察不同IPTG浓度0,01,02,04,06,08,10 mmol·L-1对pET28a(+)GlMET1蛋白表达的影响,见图6。随着IPTG浓度增加,pET28a(+)GlMET1表达没有明显变化,说明高浓度的IPTG不能增加目的蛋白的表达,低浓度的IPTG反而有利于目的蛋白的表达。
M蛋白Marker; C.Escherichia coli[pET28a];1~7E coli[pET28a(+)GlMET1],IPTG浓度分别为0,01,02,04,06,08,10 mmol·L-1。
图6pET28a(+)GlMET1在不同IPTG浓度处理下的表达
Fig6Expression of pET28a(+)GlMET1 protein at different IPTG concentration
244宿主菌吸光度A600对pET28a(+)GlMET1蛋白表达的影响保持诱导温度16 ℃,诱导时间12 h, IPTG浓度为04 mmol·L-1不变,选择诱导时宿主菌的吸光度(A600)分别为02,04,06,08和1时,观察pET28a(+)GlMET1蛋白的表达,见图7。随着宿主菌的吸光度(A600)增加,pET28a(+)GlMET1表达先上升后下降,在A600为08的时候目的蛋白的表达最高。
M蛋白Marker; C.Escherichia coli[pET28a];1~5 E coli[pET28a(+)GlMET1],宿主菌的吸光度(A600)分别为02,04,06,08,1。
图7pET28a(+)GlMET1在不同宿主菌密度下的表达
Fig7Expression of pET28a(+)GlMET1 protein in different culture densities before IPTG induction
25灵芝GlMET1的基因表达水平分析
为进一步了解灵芝甲基转移酶基因的表达模式,本研究通过Realtime PCR技术对安徽大别山区灵芝的3个栽培品种和1个野生品种进行了表达差异分析,见图8。结果显示GlMET1在不同品种灵芝中的表达水平有明显差异,且在不同生长时期也有差异。一方面,GlMET1在不同品种间的表达量依次为野生灵芝>霍芝1号>金芝1号>赤芝10号;另一方面,不同发育时期的GlMET1表达水平在4个品种中均表现出成熟期低于幼年期,说明GlMET1表达量随着灵芝的生长发育呈下降趋势。
3讨论
DNA甲基转移酶在植物和真菌的生长发育过程中具有重要的表观遗传调控作用,被认为是表观遗传中最关键酶之一。本研究首次通过灵芝转录组克隆得到灵芝DNA甲基转移酶GlMET1,并利用生物信息学的方法对其核酸及其推测的蛋白序列组成进行分析,对构建的pET28a(+)GlMET1重组质粒进行了原核表达蛋白优化分析,同时分析了不同生长时期及不同品种灵芝GlMET1的基因表达水平,
为深入研究DNA甲基转移酶蛋白的作用机制奠定了基础。
本研究将GlMET1基因与原核表达载体pET28a(+)构建了融合表达载体,并成功地在大肠杆菌BL21(DE3)中实现表达,对影响蛋白表达的四大因素诱导温度、诱导时间、IPTG浓度及诱导起始菌液浓度A600进行了优化分析,确定了pET28a(+)GlMET1蛋白最合适的表达条件为:诱导温度为16 ℃条件下,重组菌生长25 h(A600为08)时,IPTG浓度为02 mmol·L-1,诱导时间为12 h时,蛋白的表达量最高。这为后续研究GlMET1的蛋白功能奠定了基础。
在植物中第1个克隆的编码DNA甲基转移酶的基因MET1是在模式植物拟南芥中发现的,多项研究表明MET1的功能主要是维持DNA序列上的CG甲基化。拟南芥中MET1家族有4个成员,即METl,MET2a,MET2b以及MET3,且不同基因转录水平有较大不同,METl基因只在分生组织中转录,而MET2a,MET2b这2个基因的转录能够发生在所有组织中[12,16]。研究表明MET1类基因的在植物的特定器官中表达易受环境胁迫的影响,玉米不同组织中DNA甲基转移酶基因(ZmMET1) 的转录水平在冷胁迫条件下差异很大, 其中该基因在中胚轴中转录水平比较高, 而在根中几乎不表达[13]。本研究对不同品种和不同生长时期的灵芝MET1表达水平进行检测,发现GlMET1在不同品种灵芝中的表达水平有明显差异,且在幼年时期的表达水平均高于成熟时期,这暗示在灵芝的生长发育过程中MET1基因可能发挥着调控作用,具体调控机制有待深入研究。
[致谢]在灵芝样品收集过程中,得到安徽霍山衡济堂公司、安徽霍山一品堂公司和安徽金寨乔康药业的帮助。
[参考文献]
[1]史玉杰, 李庆贺, 刘晓辉 DNA 甲基化与基因表达调控研究进展[J] 中国生物工程杂志, 2013(7):17.
[2]Chan S W, Henderson I R, Jacobsen S E Gardening the genome: DNA methylation in Arabidopsis thaliana [J] Nat Rev Genet, 2005,6(5):351
[3]Martienssen R A, Colot V DNA methylation and epigenetic inheritance in plants and filamentous fungi[J] Science, 2001, 293(5532): 1070
[4]Jeltsch A Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases[J] Chem Bio Chem, 2002, 3(4): 274
[5]Finnegan E J, Kovac K A Plant DNA methyltransferases[J] Plant Mol Biol, 2000, 43(2/3): 189
[6]Finnegan E J, Peacock W J, Dennis E S DNA methylation, a key regulator of plant development and other processes[J] Curr Opin Genet Dev, 2000, 10(2): 217
[7]Liu W, Wang H, Pang X, et al Characterization and antioxidant activity of two lowmolecularweight polysaccharides purified from the fruiting bodies of Ganoderma lucidum[J]. Int J Biol Macromol, 2010, 46(4): 451
[8]Liu Z, Xing J, Huang Y, et al Activation effect of Ganoderma lucidum polysaccharides liposomes on murine peritoneal macrophages[J] Int J Biol Macromol, 2016,82:973
[9]Cheng S, Sliva D Ganoderma lucidum for cancer treatment we are close but still Not there[J] Integr Cancer Ther, 2015, 14(3): 249
[10]Mu D S, Li C, Shi L, et al Bioinformatic identification of potential MicroRNAs and their targets in the lingzhi or reishi medicinal mushroom Ganoderma lucidum (Higher Basidiomycetes)[J]. Int J Med Mushrooms, 2015, 17(8):783
[11]Xu Z, Xu J, Ji A, et al Genomewide selection of superior reference genes for expression studies in Ganoderma lucidum[J] Gene, 2015, 574(2): 352
[12]Chan S W L, Henderson I R, Jacobsen S E Gardening the genome: DNA methylation in Arabidopsis thaliana[J] Nat Rev Genet, 2005, 6(5): 351
[13]Steward N, Kusano T, Sano H Expression of ZmMET1, a gene encoding a DNA methyltransferase from maize, is associated not only with DNA replication in actively proliferating cells, but also with altered DNA methylation status in coldstressed quiescent cells[J] Nucleic Acids Res, 2000, 28(17): 3250
[14]常琳琳, 张志宏, 张运涛, 等 草莓 MET1 基因启动子克隆及瞬时表达分析[J] 西北植物学报, 2011, 31(6): 1110
[15]毛锐涛 马铃薯甲基转移酶和去甲基化酶预测及表达研究[D]呼和浩特:内蒙古大学, 2014
[16]Kishimoto N, Sakai H, Jackson J, et al Site specificity of the Arabidopsis METI DNA methyltransferase demonstrated through hypermethylation of the superman locus[J] Plant Mol Biol, 2001, 46(2): 171
[责任编辑吕冬梅]

