SYBR Green Ⅰ实时荧光定量PCR检测PRRSV方法的建立
罗忠永,王 印,杨泽晓
(四川农业大学动物医学院 动物检疫实验室,四川成都 611130)
研究论文
SYBR Green Ⅰ实时荧光定量PCR检测PRRSV方法的建立
罗忠永,王印*,杨泽晓
(四川农业大学动物医学院 动物检疫实验室,四川成都 611130)
摘要:根据编码PRRSV M蛋白的基因序列设计用于SYBR Green Ⅰ实时荧光定量PCR扩增的引物,通过温度梯度法优化反应体系;以不同科属常见猪致病性RNA病毒疫苗验证该检测方法的特异性,参考国内PRRSV分子流行病学特征,选择基因型代表性的2株疫苗株验证该检测方法的稳定性;以10倍梯度稀释的质粒为标准品扩增并构建标准曲线,探究该检测方法的灵敏度,随机挑取10份保存的PRRSV阳性病料和10份阴性病料同时使用农业部推荐的PRRSV RT-PCR检疫标准方法和建立的方法进行检测,以确定2种方法检测的符合率。结果显示,使用建立的方法可以在54.4℃ 80 min内完成对PRRSV的检测,该检测方法具有特异性和稳定性;检测灵敏度为6.8×10 拷贝/μL;以Ct值为横坐标,扩增序列拷贝浓度的对数值为纵坐标,得到标准曲线,方程为y=-3.411 x+39.002,R2=0.999;对随机挑取的10份PRRSV阳性病料和10份阴性病料进行检测,结果2种方法检测结果的符合率100%。结果表明,成功建立了PRRSV SYBR Green Ⅰ实时荧光定量PCR快速检测方法,为临床提供了一种很好的检测手段。
关键词:猪繁殖与呼吸综合征病毒;M蛋白;SYBR Green Ⅰ实时荧光定量PCR
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)是由猪繁殖与呼吸综合征病毒(PRRSV)引起的一种以母猪繁殖障碍和仔猪呼吸道疾病为特征的传染病。该病于1987年首次在美国发生,随后迅速在世界大范围流行[1]。我国自1996年暴发PRRS以来,已遍及全国各地,成为危害养猪业最严重的疫病之一[2-4]。2006年夏秋季节,我国南方部分省市相继暴发“猪高热病”疫情,经诊断该病主要致病病原为高致病性猪蓝耳病病毒(Highly pathogenic porcine reproductive and respiratory syndrome,HP-PRRSV),该病半年内几乎传遍了大半个中国,造成大批猪只死亡,引起巨大的经济损失[5]。PRRSV是一种表面相对光滑且有囊膜的病毒,其直径大小约50 nm~65 nm,属于套式病毒目(Nidovirales)、动脉炎病毒科(Arteriviridae)、动脉病毒属(Arterivirus)[6]。根据PRRSV抗原性、遗传特性和致病性将其分为2个基因型,欧洲Ⅰ型和美洲Ⅱ型,有研究表明美洲Ⅱ型是由欧洲Ⅰ型隔离变异而来[7]。PRRSV为单股正链不分节段的RNA病毒,基因组大小约为15 kb,结构为5′-UTR-ORF1a-ORF1b-ORF2-ORF3-ORF4-ORF5-ORF6-ORF7-UTR-poly(A)-3′,共含有10个开放性阅读框(ORFs)[8]。HP-PRRSV基因型属于美洲型,基因组结构与经典的PRRSV有所不同,其Nsp2基因有两处不连续的缺失,一处缺失3 nt,另一处缺失87 nt;氨基酸水平缺失30 aa[9]。病毒蛋白的表达机制是位于基因组3′端的结构基因通过负链不连续转录机制进行亚基因的转录,产生6个亚基因组mRNA表达蛋白[10]。亚基因组结构mRNA的转录产物具有与基因组相同的5′-UTR、3′-UTR及poly(A)尾巴[11]。ORF1a和ORF1b两个开放性阅读框长度约占基因组全长的80%,分别编码多聚蛋白pp1a和pp1ab,分别被裂解为9种和4种非结构蛋白[12]。ORF2-7分别编码病毒结构蛋白GP2a、GP2b、GP3、GP4、GP5、M和N蛋白[13]。主要结构蛋白为GP5、M和N蛋白。M白是非糖基化的囊膜蛋白,分子质量约为18 ku~19 ku,是所有毒株中最保守的结构蛋白,M蛋白不仅能刺激机体产生中和抗体,也能刺激猪产生最强的T淋巴细胞免疫应答,在细胞免疫中起着重要作用[14]。
荧光定量PCR是20世纪90年代中期发展的一种新型核酸定量技术,基于荧光能量传递技术,能量由供体发色基团转移到受体发色基团,受体荧光染料发出的荧光信号强度与DNA产量成正比。检测PCR过程的荧光信号,将该动态曲线与标准曲线对比可以获得靶序列的起始浓度;或者对比不同靶序列的动态扩增曲线,获得不同靶序列起始状态的相对含量。本研究依据编码PRRSV M蛋白基因保守序列设计引物,建立了SYBR Green Ⅰ实时荧光定量PCR快速定量检测PRRSV核酸的方法,并对该方法的特异性、稳定性、灵敏度以及与农业部推荐的PRRSV标准检测方法的结果进行比较,以期为临床PRRSV的检测提供一种较好的手段。
1材料与方法
1.1材料
NANODROP 2000分光光度计,Thermo公司产品;CFX ConnectTMOptics Module荧光定量PCR检测系统及其耗材,Bio-Rad公司产品;PRRSV、HP-PRRSV疫苗,上海海利生物技术股份有限公司产品;CSFV、JEV、PEDV疫苗,中牧实业股份有限公司产品;SYBR Premix DimerEraserTM,宝生物工程(大连)有限公司产品。
1.2方法
1.2.1引物设计与合成从GenBank下载编码PRRSV美洲型和欧洲型参考毒株的M蛋白基因序列或从全基因组序列中找出M蛋白基因序列,利用 DNA Star软件分析编码M蛋白的基因序列中的保守片段,利用Primer Premier6和Oligo7设计1对引物(表1)用于扩增编码PRRSV M蛋白基因154 bp片段,引物由英潍捷基(上海)贸易有限公司合成,PAGE纯化。
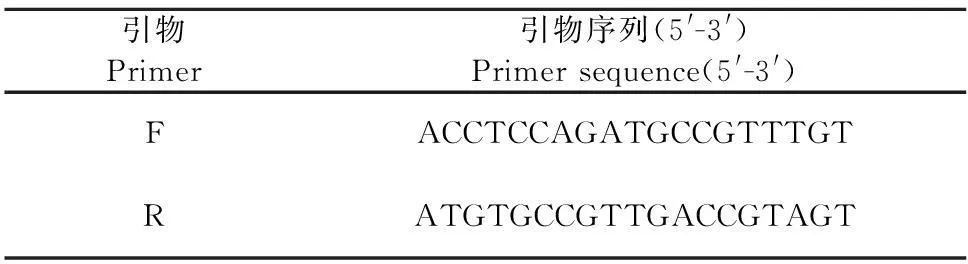
表1 PRRSV M基因片段扩增引物
1.2.2标准质粒的构建参照TaKaRa RNAiso Plus使用说明提取PRRSV CH-1R株疫苗RNA,使用TaKaRa PrimeScriptTMRT Reagent Kit进行病毒全基因组cDNA合成,以合成的cDNA为模板,引物F和R,使用2×PFU PCR MaserMix扩增目的片段,50 μL反应体系,退火温度54.4℃(由Primer Premier6和Oligo7软件分析给出)。取5 μL PCR产物经10 g/L agarose电泳确认后,剩余45 μL产物电泳分离后按通用型DNA纯化回收试剂盒说明书回收目的片段。使用Thermo NANODROP 2000分光光度计测量纯化后模板片段核酸浓度,参照加A反应液说明书对模板片段3′加A。将加A产物直接连接T-Vector pMDTM19(Simple),连接反应按DNA Ligation Kit Ver.2.1说明书操作,连接产物转化Dpα感受态细胞,蓝白斑筛选后挑取白色菌落过夜培养使用质粒小提取试剂盒提取质粒,用Thermo NANODROP 2000分光光度计测量质粒浓度,并将质粒送交英潍捷基(上海)贸易有限公司进行双向测序。
1.2.3检测体系的优化以1.2.2制备的质粒为模板,以Primer Premier6和Oligo7推荐的退火温度为中心利用棋盘法对退火温度进行优化。反应体系:DimerEraser(Perfect Real Time)2×10 μL,灭菌ddH2O 6 μL,引物F(浓度10 μmol/L) 1 μL,引物R(浓度10 μmol/L) 1 μL,10×梯度稀释(10-3、10-4、10-5、10-6)的标准质粒 2 μL。每个温度或梯度3个重复。反应条件:95℃ 3 min;95℃ 15 s,52.9℃~56.4℃ 20 s(共8个温度梯度,相邻梯度差别0.5℃),40个循环。
1.2.4特异性和稳定性检测参照TaKaRa RNAiso Plus使用说明分别提取PRRSV CH-1R株、HP-PRRSV NM1株、CSFV、JEV、PEDV等疫苗病毒核酸,合成病毒全基因组cDNA,检验检测所建立方法的特异性和稳定性,以灭菌ddH2O为模板的反应体系作为阴性对照。反应体系;DimerEraser(Perfect real time)2×10 μL,灭菌ddH2O 6 μL,引物F(浓度10 μmol/L) 1 μL,引物R(浓度10 μmol/L) 1 μL,模板2 μL。反应条件:95℃ 3 min;95℃ 10 s,54.4℃ 20 s,40个循环。反应结束后取5 μL产物经10 g/L agarose电泳,BIO-RAD Universal HoodⅡ凝胶成像系统观察。
1.2.5标准曲线的制作和灵敏度检测以1.2.2提取的质粒为标准品,计算标准品的拷贝浓度,以10倍梯度稀释标准品,每个梯度3个重复,检测建立方法的灵敏度,选取线性的拷贝浓度区间制作标准曲线,以灭菌ddH2O为模板的反应体系作为阴性对照。反应条件:95℃ 3 min;95℃ 15 s,54.4℃ 20 s,40个循环。反应体系:DimerEraser(Perfect real time)2×10 μL,灭菌ddH2O 6 μL,引物F(浓度10 μmol/L) 1 μL,引物R(浓度10 μmol/L) 1 μL,10倍梯度稀释(100~109)质粒 2 μL;每个梯度3个重复。
1.2.6检测结果的符合率随机挑取10份保存的PRRSV阳性病料和10份阴性病料同时使用农业部推荐的PRRSV RT-PCR检疫标准方法和建立的方法进行检测,确定2种方法检测的符合率。
2结果
2.1质粒测序结果
将质粒测序结果进行Blast比对,结果显示,扩增的M基因片段与GenBank上公布的PRRSVCH-1R相应序列同源性为100%,说明成功构建了正确的标准质粒(图1)。
2.2检测体系的优化
以Primer Premier6和Oligo7推荐的退火温度为中心进行退火温度的优化。结果显示在54.4℃一定温度梯度内目的片段均可以获得很好地特异性扩增(图2),同时也说明该检测体系特异性扩增的退火温度范围较宽,具有很好的适用性,综合考虑后选择54.4℃作为退火温度。
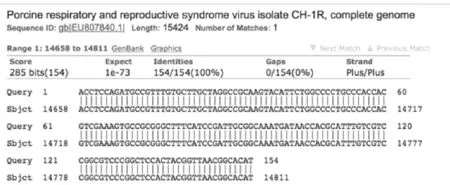
图1 序列同源性比对
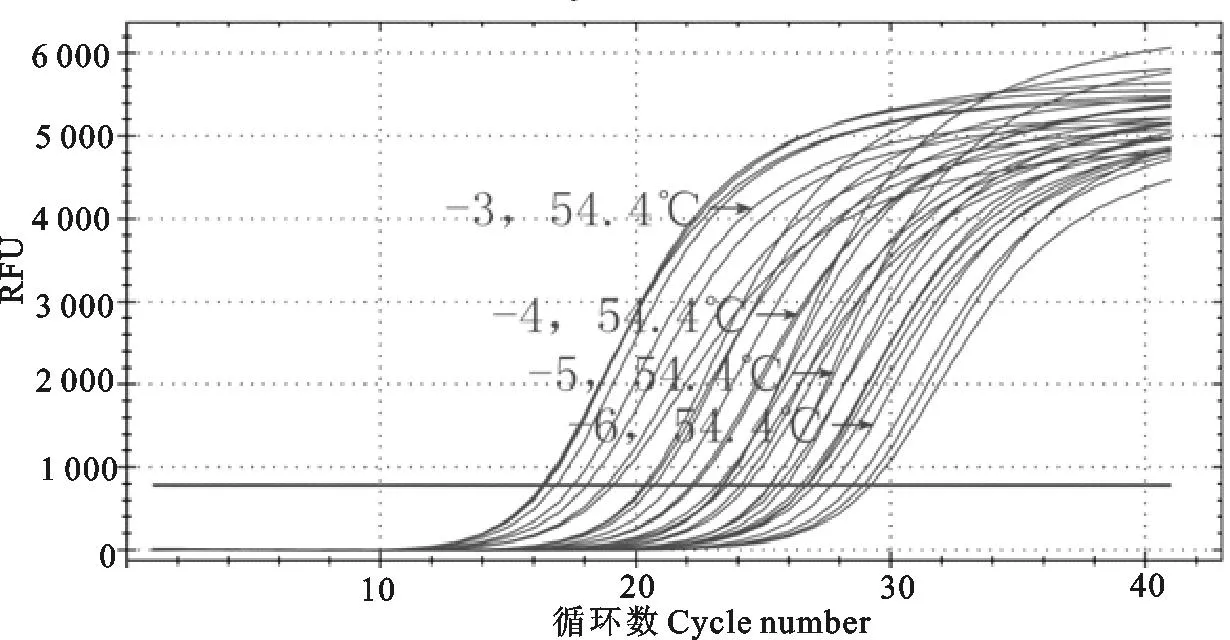
图2 退火温度优化的动态曲线
2.3特异性和稳定性测定结果
以CSFV、JEV、PEDV检验检测体系的特异性,以PRRSV CH-1R株和HP-PRRSV NM1株检验检测体系的稳定性。结果显示,该检测体系能很好地扩增PRRSV CH-1R株和HP-PRRSV NM1株154 bp片段(图3),而CSFV、JEV、PEDV和阴性对照均无扩增条带,动态曲线和熔解曲线特征符合预期,说明该检测体系具有很好的特异性和稳定性。
2.4标准曲线制作和灵敏度检测
以10倍梯度稀释标准品,每个梯度3个重复,检测建立的荧光定量PCR检测方法的灵敏度,选取线性的拷贝浓度区间制作标准曲线,以灭菌ddH2O为模板的反应体系作为阴性对照。结果显示,该检测方法的灵敏度为6.8×10 拷贝/μL。以Ct值为横坐标,扩增序列拷贝浓度对数值为纵坐标,得到标准曲线;方程为y=-3.411x+39.002,R2=0.999(图4和图5)。
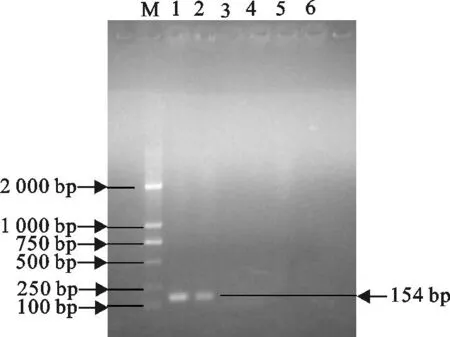
M.DNA 标准DL 2 000;1.PRRSV;2.HP-PRRSV;3.CSFV;4.JEV;5.PEDV;6.阴性对照
M.DNA Marker DL 2 000;1.PRRSV;2.HP-PRRSV;3.CSFV;4.JEV;5.PEDV;6.Negative control
图3引物有效性和特异性验证
Fig.3Verification of effectiveness and specificity of primers

0~9.100-9拷贝/μL;NC.阴性对照
0-9.100-9copies/μL;NC.Negative control
图4灵敏度检测扩增曲线
Fig.4Dynamic curve of sensitivity test

图5 制备的标准曲线
2.5与农业部推荐RT-PCR检疫标准的符合率
随机挑取10份保存的PRRSV阳性病料和阴性病料同时使用农业部推荐的PRRSV RT-PCR检疫标准方法和建立的方法进行检测,结果显示,10份PRRSV阳性病料2种检测方法的检测结果均为阳性,10份阴性病料2种检测方法的检测结果均为阴性;说明建立的方法的检测结果与农业部推荐的检测方法的结果符合率为100%。
3讨论
SYBR Green Ⅰ实时荧光定量PCR检测方法建立的难点在于选择合适的扩增靶序列,研究发现PRRSV各毒株核酸序列之间的变异很大,因此,为保证建立的检测方法具有稳定性和可应用性,必须在基因组内寻找可靠的保守序列。对PRRSV毒株全基因组序列分析发现Nsp2基因序列差异较大,基于Nsp2基因序列建立鉴别PRRSV高致病株和经典株SYBR Green-Ⅰ 实时荧光定量PCR方法需要设计简并引物,且较难获得稳定的反应体系[1];M蛋白基因则相对保守,是所有毒株中最保守的结构蛋白。本研究选择M基因的保守序列设计引物,没有选择Nsp2基因缺失区设计引物,因此无法区分PRRSV和HP-PRRSV。为了保证试验的严谨性,随机挑选PRRSV M蛋白基因30 nt左右的核酸片段进行Blast比对,未发现与其他生物体基因序列有同源片段,保证了M蛋白基因序列是PRRSV特有的。此外,笔者还通过核酸二级结构分析预测软件对M蛋白核酸序列进行二级结构分析,尽量避免在二级结构区设计扩增。同时,本研究中选择染料法而没有选择探针法,可以避免探针序列对反应体系的干扰,降低试验难度,保证检测体系的稳定性。本试验成功建立了SYBR Green Ⅰ实时荧光定量PCR检测PRRSV的方法,结果显示,在101拷贝/μL~109拷贝/μL有很好的线性关系,检测下限为10拷贝/μL。以CSFV、JEV、PEDV的cDNA为模板均无扩增条带,说明该对引物特异性良好;国内流行基因型疫苗株PRRSV CH-1R株和HP-PRRSV NM1株验证引物的稳定性;同时退火温度优化过程中发现该对检测体系特异性扩增的退火温度范围较宽,基于以上说明该检测体系具有较强的可应用性。随机挑取10份保存PRRSV阳性病料,经本检测方法检测结果均为阳性,10份阴性病料2种检测方法的检测结果均为阴性;与农业部推荐的RT-PCR检测方法检测结果的符合率为100%,说明建立的检测方法的检测结果的具有可信度。
我国自1996年暴发PRRS以来,已遍及全国各地,成为危害养猪业最严重的疫病之一。对PRRSV的快速准确检测是控制PRRS传播,保证食品卫生安全的重要措施。本研究选择PRRSV M蛋白基因保守序列设计引物,通过SYBR Green Ⅰ实时荧光定量PCR快速定量检测PRRSV核酸。该检测系统具有很高的特异性和稳定性;与农业部推荐检疫标准符合率为100%,为猪繁殖与呼吸综合征病毒提供了一个快速简便的检测方法。
参考文献:
[1]柴政.PRRSV高致病株和经典株SYBR Green-Ⅰ 实时荧光PCR鉴别方法的建立[D].陕西杨凌:西北农林科技大学,2010.
[2]郭宝清,陈章水,刘文兴,等.从疑似PRRS流产胎儿分离PRRSV的研究[J].中国畜禽传染病,1996(2):1-4.
[3]郭宝清,陈章水,刘文兴,等.应用免疫荧光法从国内生殖障碍综合征的血清中检测阳性抗体的研究[J].中国兽医科学,1996,26(3):3-5.
[4]蔡锦顺.PRRSV GP5与M蛋白重组犬2型腺病毒的构建及其免疫特性研究[D].吉林长春:吉林大学,2010.
[5]冷雪.高致病性猪繁殖与呼吸综合征病毒分离鉴定、遗传变异及致病性分析[D].吉林长春:中国农业科学院,2012.
[6]李志杰.高致病性猪蓝耳病病毒感染差异膜蛋白筛选及功能分析[D].吉林长春:吉林大学,2010.
[7]刘永宏.高致病性猪繁殖与呼吸综合征病理学研究及其病毒的序列分析[D].内蒙古呼和浩特:内蒙古农业大学,2010.
[8]薛青红.猪繁殖与呼吸综合征病毒分离鉴定、遗传变异分析及其体内拯救系统的初步研究[D].陕西杨凌:西北农林科技大学,2009.
[9]冷雪,李真光,王凤雪,等.高致病性猪繁殖与呼吸综合征病毒NM1株全基因组测序及致病性分析[J].中国兽医学报,2012(12):1752-1762.
[10]武小椿.PRRSV Shaanxi-1株分离鉴定、全基因测序及遗传进化分析[D].陕西杨凌:西北农林科技大学,2010.
[11]马苏.猪繁殖与呼吸综合征病毒GP5、M蛋白和N蛋白单克隆抗体的研制及生物学特性分析[D].江苏南京:南京农业大学,2006.
[12]周井祥.猪繁殖与呼吸综合征病毒GP5、M蛋白基因核酸疫苗和重组犬2型腺病毒的构建及其免疫特性研究[D].吉林长春:吉林大学,2007.
[13]王永志.猪繁殖与呼吸综合征病毒M、N基因克隆及N基因表达应用研究[D].四川雅安:四川农业大学,2006.
[14]刘明莉.PRRSV GP2和M蛋白免疫活性及其在感染细胞内分布的初步研究[D].河南郑州:河南农业大学,2009.
Establishment of SYBR Green Ⅰ Real-time Fluorescence Quantitative PCR Method for Detection of PRRSV
LUO Zhong-yong,WANG Yin,YANG Ze-xiao
(AnimalQuarantineLaboratory,CollegeofVeterinaryMedicine,SichuanAgricultureUniversity,Chengdu,Sichuan,611130,China)
Abstract:Primers were designed for SYBR Green Ⅰ real-time fluorescence quantitative PCR according to the PRRSV M protein.Different temperature gradient results were checked to optimize the reaction system.The specificity and stability of the detection method were verified through different genera of common swine pathogenic RNA virus vaccine and 2 representative genetype vaccine strains which were selected on the basis of the domestic molecular epidemiological characteristics of PRRSV.A standard curve was constructed and the sensitivity of this detection method was explored with the plasmid which was diluted in 10 times of gradient.10 conserved PRRSV positive samples and 10 conserved PRRSV negative samples were selected randomly to research the coincidence rate of the constructed detection and quarantine standards recommended by the Ministry of agriculture.The results showed that detection could be completed within 80 min when the temperature was set at 54.4℃,using the two-step detection system.The detection method has good specificity and stability.The detection sensitivity could as low as 6.8×102copies/L.The standard curve was the equation (y=-3.411x+39.002, R2 = 0.999) when the Ct value was selected as the abscissa and the amplified sequences of numerical was selected as ordinate.The detection result was in line with the quarantine standards recommended by the Ministry of Agriculture with the randomly selected porcine samples.The test results showed that the SYBR Green Ⅰ real-time fluorescence quantitative PCR method was established successfully which could provide a good means for clinical detection.
Key words:PRRSV;M protein; Green Ⅰ SYBR real-time fluorescence quantitative PCR
文章编号:1007-5038(2016)04-0001-05
中图分类号:S852.659.6
文献标识码:A
作者简介:罗忠永(1992-),男,河南潢川人,硕士研究生,主要从事分子生物学研究。*通讯作者
基金项目:“十二五”国家科技支持计划项目(2013BAD12B04)
收稿日期:2015-10-12

