D1/D2、MLST及MALDI-TOF MS在新生隐球菌分类中的应用**?
刘涛华,王颜颜,吕 倩,牟丽丽,陈 燕,康颖倩*
(贵州医科大学微生物学教研室,贵州贵阳 550004)
D1/D2、MLST及MALDI-TOF MS在新生隐球菌分类中的应用**?
刘涛华**,王颜颜,吕倩,牟丽丽,陈燕,康颖倩***
(贵州医科大学微生物学教研室,贵州贵阳550004)
[摘要]目的:比较单一D1/D2区域测定、多位点测序分型技术(MLST)和基质辅助激光解吸-飞行时间质谱技术(MALDI-TOF-MS)在新生隐球菌(Cryptococcus podzolicus)及与之邻近的其他种属的分类研究的稳定性和可靠性。方法:挑选以Cryptococcus podzolicus为主及与之相邻的其他种和属的菌株共20株及1株outgroup模式菌株,运用酵母菌基因组提取试剂盒提取其DNA,PCR扩增5个管家基因(ITS,D1/D2,rpb1,rpb2,tef1)并测序,以Bayesian法(MrBayes3.2.1)、最大似然法、邻接法(MEGA 6.0软件)分别构建5个管家基因及D1/D2基因序列的系统发育树;应用MALDI-TOF-MS采集蛋白指纹图谱,在BrukerDaltonics数据库(BDAL)和CBS真菌保藏中心数据库信息的基础上,对20株菌进行聚类分型,构建聚类分型树状图。结果: 3种方法所得树状图在种间的层面上,均出现了a、b、c 3个分支;在种内的层面上,特别是a这一分支,均为Cryptococcus podzolicus,单一D1/D2区域所构建的系统发育树分化程度不大,各菌株间无明显的分类多样性,而MLST及MALDI-TOF MS所得到的树状图在种内均出现了明显的分类多样性及亲缘关系的差异性。结论: MLST及MALDI-TOF MS均可作为隐球菌属中菌株分类鉴别中有效的分子生物学方法,能可靠地揭示隐球菌属中各菌种的种间及种内的遗传进化关系,MALDI-TOF-MS比MLST则更为快捷准确。
[关键词]多位点测序分型;基质辅助激光解吸飞行时间质谱;新隐球菌;分类法;发育生物学;进化
[筑科合同(2011103) 16号];贵州省国际科技合作计划项目[黔科合外G字(2014) 7006]
**贵州医科大学2013级硕士研究生
***通信作者E-mail: joycekangtokyo@ qq.com
网络出版时间: 2016-02-23网络出版地址: http: / /www.cnki.net/kcms/detail/52.5012.R.20160223.2055.054.html
隐球菌属(Cryptococcus)属于担子菌门银耳纲银耳目银耳科,隐球菌属下约有37个种,通常栖息在土壤中,属中的新生隐球菌(Cryptococcus neoformans)可使爱滋病患者罹患严重的脑膜炎或脑膜脑炎。对于Cryptococcus的分类研究,传统的方法主要是根据菌体的外部形态特征和生理生化特征,敏感性和特异性差,准确度不高。本课题前期研究发现,Cryptococcus podzolicus在进化树附近出现了一些其他属的菌,如Bullera和Trimorphomyc。Bandoni和Boekhout等[1-2]发现Trimorphomyc与Cryptococcus podzolicus除了生殖结构不同外,形态上也不一样,而Bullera没有这些差异,很难通过传统形态学的分类方法对其进行分类。Keisha Findley等[3]通过MLST技术对致病性格特隐球菌(Cryptococcus gattii)[4]和Cryptococcus neoformans之间的关系以及其在系统发育树中的具体位置展开了研究。基质辅助激光解吸-飞行时间质谱(matrix-assisted laser desorption/ ionization time of flight mass spectrometry,MALDI-TOF-MS)是近几年发展起来的一种新型软电离生物质谱,它能完成菌体多种成分的分析,包括蛋白质、脂类、脂多糖和脂寡糖、DNA、多肽及其他能被离子化的分子。MALDI-TOF-MS具有快速、准确、灵敏、分辨率高、高质量检测范围等优点,成为临床诊断、食品生产、环境监测以及军事领域研究的一种有力手段[5-6]。本研究通过单一D1/D2区域测定、多位点测序分型技术(MLST)和基质辅助激光解吸-飞行时间质谱技术(MALDITOF-MS)在Cryptococcus podzolicus及与之邻近的其他种属分类的比较研究,旨在探讨适合Cryptococcus分类的分子分类方法。
1 材料与方法
1.1实验菌株与试剂
挑选以Cryptococcus podzolicus为主及与之相邻的其他种和属的菌株共20株,包括Cryptococcus podzolicus、Cryptococcus flavus、Cryptococcus paraflavus、Bullera miyagiana、Bullera sakaeratica及Trimorphomyces papilionaceus,由荷兰真菌多样性研究中心、日本千叶大学真菌研究所惠赠。在进化树上所加的outgroup选择了Cryptococcus neoformans,CBS 132这一模式菌株,其数据来自于GenBank(见表1)。PDA和YPD培养基均购自日本Wako Pure Chemical Industries Ltd公司,基因组提取试剂盒购自天根生化科技公司,PCR相关试剂均购自上海生工生物工程有限公司。

表1 21株实验菌株种名Tab.1 List of 21 strains in the experiment
1.2多位点测序分型
1.2.1菌株培养及DNA的提取所有菌株均用YPD液体培养基复苏,接种在PDA培养基上,温度为25℃,培养24 h,挑取菌落;参照酵母菌基因组提取试剂盒操作说明书提取基因组DNA模板。
1.2.2 PCR扩增及凝胶电泳分析扩增的5个基因(ITS,D1/D2,rpb1,rpb2及tef1),PCR反应引物见表2,PCR扩增条件见表3。将扩增产物在1.5%的琼脂糖凝胶中电泳分离,100 V稳压20 min,在紫外透射仪下观察。PCR产物送往上海生工生物工程有限公司进行测序。

表2 MLST测序位点及对应引物Tab.2 Primers of MLST loci
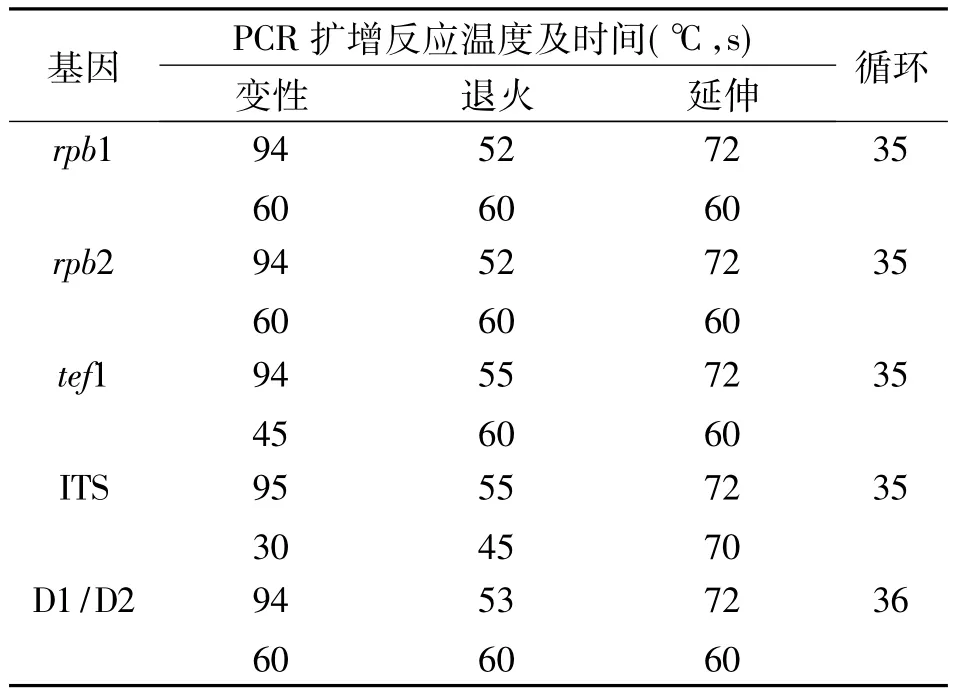
表3 PCR扩增反应条件Tab.3 PCR amplification reaction conditions
1.2.3 DNA序列系统发育分析对所获得的序列,以Bayesian法(MrBayes3.2.1)、最大似然法、邻接法(MEGA 6.0软件)分别构建ITS和5个管家基因(ITS,D1/D2,rpb1,rpb2,tef1)的系统发育树进行比较分析。
1.3 MALDI-TOF-MS检测
1.3.1样品处理取菌株新鲜培养物,在Eppendorf管中加入纯净水300 μL,挑取适量(5~10 mg)菌落混匀,再加入无水乙醇900 μL,混匀后以12 000 r/min离心2 min,弃去上清液,加入70%甲酸50 μL混匀,再加入乙腈50 μL混匀,以12 000 r/min离心2 min,吸取上清液,涂布于96孔样品板上,自然晾干1 h后,用基质溶液α-氰基-4-羟基肉桂酸(α-cyano-4-hydroxycinnamic acid,HCCA)覆盖菌苔,每孔1 μL。晾干后进行质谱分析。
1.3.2数据采集将上样的样品板小心置于板孔中,有样品面朝上,盖上盖子,抽真空。打开仪器控制软件FlexControl 3.3.108.0,调好校准仪器参数,采集样品的质谱图,保存数据,通过MALDI-TOF MS Biotyper RTC 3.0软件进行分析。
1.3.3 MALDI-TOF-MS结果判断所采集的质谱图与BrukerDaltonics数据库(BDAL)和CBS真菌保藏中心数据库中标准图进行比对,鉴定分值≥2.0表示可鉴定到种水平,2.300~3.000表示菌种鉴定的可信度较高,1.700~1.999表示可鉴定到属,0.000~1.699表示不可信的鉴定。
1.3.4聚类分析对21株菌的蛋白质质量图谱进行聚类分析,采用相似分值构建系统树。
2 结果
2.1分子分类
2.1.1单一D1/D2区域系统发育将所获得的序列,以Bayesian法(MrBayes3.2.1)构建单一的D1/D2基因的系统发育树(见图1),以最大似然法、邻接法(MEGA 6.0软件)分别进行比较,图中所示数值分别表示Bayesian方法的后验概率值,邻接法的引导百分率和最大似然法的引导百分率。从图1的系统发育进化树可见,在种间的层面上,主要出现了a、b、c 3个分支,而在种内的层面上,特别是a这一分支,均为Cryptococcus podzolicus,其分化程度不大,各菌株无明显的分类多样性,其亲缘关系无明显差异。
2.1.2多位点序列系统发育将所获得的序列,以Bayesian法(MrBayes3.2.1)构建4个管家基因(ITS,D1/D2,rpb1,rpb2)的系统发育树(见图2),以最大似然法、邻接法(MEGA 6.0软件)分别进行比较,图中所示数值分别表示Bayesian方法的后验概率值,邻接法的引导百分率和最大似然法的引导百分率。从图2的系统发育树可见通过MLST所构建的系统发育进化树在种间的层面上,均出现a、b、c 3个分支,而种内的层面上,其区分程度更高,特别是a这一分支,均为Cryptococcus podzolicus,各菌株之间的亲缘关系较大,出现了种内的分类多样性。
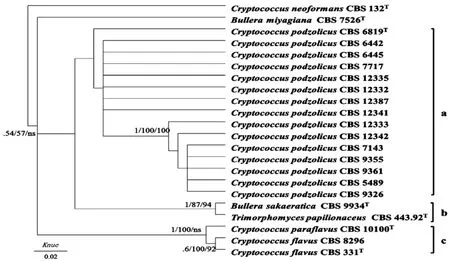
图1 基于D1/D2区域构建的系统发育树Fig.1 The phylogenetic relationships among species of Cryptococcus inferred from sequences of D1/D2

图2 基于多位点序列(ITS,LSU,rpb1,rpb2)构建的系统发育树Fig.2 The phylogenetic relationships among species of Cryptococcus inferred from a five-gene data set including sequences of ITS,LSU,rpb1,rpb2
2.2 MALDI-TOF-MS
对所有菌株的蛋白质质量图谱进行聚类分析,采用相似分值构建系统树,得到MSP聚类分析树状树。在种间的比较层面,出现a、b、c 3个分支,a这一分支里面都是Cryptococcus podzolicus,从图3中可清晰的看到Cryptococcus podzolicus中各菌株之间的进化关系,出现了明显的分类多样性,各菌株之间的亲缘关系差异较大。

图3 MSP聚类分析树状树Fig.3 Clustering classification tree of 21 srains of Cryptococcus by MALDI-TOF-MS
3 讨论
在绝大多数的真核生物中,表现出了极为广泛的序列多态性,即使亲缘关系非常近,也能显示不同的进化特征,但有的真菌由于进化顺序、变异等原因,在间隔区及核糖体大亚基序列上差异性较小[13]。前期的隐球菌的分类鉴定研究发现,ITS及D1/D2序列虽然在物种属内、近缘属间乃至科内系统进化关系研究中有一定的价值,但不适合属内种及种群的分类。本研究主要选取了Cryptococcus podzolicus和与之邻近的一些其他种和属的菌株,比较分析单一D1/D2区域、MALDI-TOF-MS及MLST 3种技术的稳定性和准确性。
在MLST研究方面,本文中所使用的菌株大部分都可得到其目的片段,但还是存在4株菌株的tef1基因片段测序失败,可能是该基因序列本身存在问题,比较难进行PCR扩增或者是测序反应,Schoch等[14]在研究过程中也发现了这一问题,因此本研究构建4个管家基因(ITS,D1/D2,rpb1,rpb2)的系统发育树和单一D1/D2区域的系统发育树进行对比研究。从图1和图2的系统发育树可见均出现a、b、c 3个分支,各菌株的大体位置相似,但两者又存在这一些差异。相似的地方是Cryptococcus podzolicus都在同a分支上,b这一分支也相同,不同之处在于MLST所构建的系统发育树各菌株间的区分程度更高,从树中更加清晰的了解Cryptococcus podzolicus种内菌株之间的进化关系,另外一个差异则是Bullera miyagiana的位置,从MLST所得的系统发育树来看,其应该更靠近b这一分支,由此可见间隔区及核糖体大亚基在序列上差异性较小,不适合属内种及种群的分类,而MLST在隐球菌的种间及种内分类研究则更加稳定和可靠。
在MALDI-TOF-MS研究方面,根据所得到的MSP聚类分析树状图与单一D1/D2区域构建的系统发育树比较分析,二者均出现了a、b、c 3个分支,相似的地方是Cryptococcus podzolicus都在同一分支上,b这一分支也相同,不同之处在于MALDITOF-MS所得到的MSP聚类分析树各菌株间的区分程度更高,从树中更加清晰的了解Cryptococcus podzolicus种内菌株之间的进化关系,还有则是Bullera miyagiawa的位置,其应该更靠近b这一分支。由此可见MALDI-TOF-MS相比与传统的D1/D2区域分析,在隐球菌的种间及种内分类研究则更加稳定和可靠。
MALDI-TOF-MS结合MLST和D1/D2区域分析可见,图3中的Ⅰ和Ⅱ簇菌有2株Cryptococcus podzolicus菌成为独立的1个分支,而在图2中,均出现了同样的分支,综合目前荷兰真菌多样性研究中心已测得的生理生化数据,发现第Ⅰ簇的菌株在C23Glycerol和C28D-Glucitol的碳源利用实验中,其结果与其他Cryptococcus podzolicus菌株的结果存在差别,而第Ⅱ簇的菌株在C21Inulin、C28Ribitol和C38DL-Lactate的碳源利用试验及N1Nitrate、N6Creatine和N9Imidazole的氮源利用试验与其他Cryptococcus podzolicus菌株的结果有所不同,这充分说明MALDI-TOF技术及MLST技术在种内具有较强的区分能力。第Ⅲ簇的菌株包括了Cryptococcus paraflavus和Cryptococcus flavus两个种的分支,在生理生化实验中也可发现在C9L-Rhamnose、C11Maltose、C13Me a-D-Glucoside、C15Salicin、C25Ribitol、C29D-Mannitol、C30Galactitol和C31myo-Inositol的碳源利用试验及N2Nitrite、N3Ethylamine和N5Cadaverine的氮源利用试验结果存在着明显的差异,这说明MALDI-TOF-MS技术及MLST技术在种间也具有较强的区分能力。
综上,MLST及MALDI-TOF MS均可作为隐球菌属分类鉴别中有效的辅助鉴别的分子生物学方法,相比于传统的生理生化试验及间隔区和核糖体大亚基序列比较,二者均可准确地揭示隐球菌种间及种内各菌种的遗传进化关系,相比于MLST技术较长的实验周期及某些基因扩增和测序结果的不确定性,MALDI-TOF-MS技术则更为快捷,成功率更高。
4 参考文献
[1]Liu XZ,Wang QM,Theelen B,et al.Phylogeny of tremellomycetous yeasts and related dimorphic and filamentous basidiomycetes reconstructed from multiple gene sequence analyses[J].Studies in Mycology,2015(1) : 1-26.
[2]Black KE,Baden LR.Fungal infections of the CNS: treatment strategies for the immunocompromised patient[J].CNS Drugs,2007(4) : 293-318.
[3]Kurtzman,Cletus,Jack W.Fell,and Teun Boekhout, eds.The yeasts: a taxonomic study[M].Elsevier,2011 (1).
[4]康颖倩,朱键,罗振华,等.IGS基因及RAPD标记分析在加特隐球酵母基因分类中的应用[J].菌物学报,2009(5) : 731-736.
[5]Wieser A,Schneider L,Jung J,et al.MALDI-TOF MS in microbiological diagnostics-identification of microorganisms and beyond (mini review)[J].Applied microbiology and biotechnology,2012(3) : 965-974.
[6]陈秀金,尹红红,匡华,等.沙门氏菌MALDI-TOF-MS蛋白质指纹图谱分析方法的研究[J].食品与生物技术学报,2012(11) : 1189-1198.
[7]Stiller JW,Hall BD.The origin of red algae: implications for plastid evolution[J].Proceedings of the National A-cademy of Sciences,1997(9) : 4520-4525.
[8]Matheny PB,Liu YJ,Ammirati JF,et al.Using RPB1 sequences to improve phylogenetic inference among mushrooms (Inocybe,Agaricales)[J].American Journal of Botany,2002(4) : 688-698.
[9]Liu YJ,Whelen S,Hall BD.Phylogenetic relationships among ascomycetes: evidence from an RNA polymerse II subunit[J].Molecular Biology and Evolution,1999 (12) : 1799-1808.
[10]Rehner SA,Buckley E.A Beauveria phylogeny inferred from nuclear ITS and EF1-α sequences: evidence for cryptic diversification and links to Cordyceps teleomorphs [J].Mycologia,2005(1) : 84-98.
[11]Kasai K,Nakamura Y,White R.Amplification of a variable number of tandem repeats (VNTR) locus (pMCT118) by the polymerase chain reaction (PCR) and its application to forensic science[J].Journal of forensic sciences,1990(5) : 1196-1200.
[12]O’Donnell DE,Webb KA.Exertional breathlessness in patients with chronic airflow limitation[J].Am Rev Respir Dis,1993(148) : 1351-1357.
[13]Gao L,Feng C,Li B,et al.Dection of Tilletia controversa using immunofluorescent monoclonal antibodies[J].Journal of applied microbiology,2015(2) : 497-505.
[14]Schoch CL,Seifert KA,Huhndorf S,et al.Nuclear ribosomal internal transcribed spacer (ITS) region as a universal DNA barcode marker for Fungi[J].Proceedings of the National Academy of Sciences,2012(16) : 6241-6246.
(2015-08-10收稿,2015-11-23修回)
中文编辑:刘平;英文编辑:刘华
·基础研究·
Application Research of Phylogenetic Analysis of Cryptococcus podzolicus with D1/D2,MLST and MALDI-TOF MS
LIU Taohua,WANG Yanyan,LU Qian,MOU Lili,CHEN Yan,KANG Yingqian
(Department of Microbiology,Guizhou Medical University,Guiyang 550004,Guizhou,China)
[Abstract]Objective: To compare and analyze the stability and reliability of multilocus sequence typing (MLST) and matrix-assisted laser desorption/ionization time of flight mass spectrometry (MALDI-TOF-MS) methods in taxonomic study of Cryptococcus,especially for Cryptococcus podzolicus.Methods: Twenty strains including Cryptococcus podzolicus and its adjacent strains were selected for the evaluation in this study; DNA was extracted using yeast genome extraction kit and 5 genes(ITS,D1/D2,rpb1,rpb2,tef1) were amplified and sequenced.The phylogeny tree was constructed by Bayesian,maximum parsimony and neighbor-joining analyses.The protein fingerprint acquisition parameters were collected by MALDI-TOF-MS method.Clustering classification was conducted for the twenty strains and the phylogenetic tree was constructed based on BDAL and CBS fungi preservation center database.Results: There existed three clusters(a,b and c) in inter species level that can be observed on the phylogenetic trees made by three different molecular methods; In intraspecific level,all Crypto-book=140,ebook=21coccus podzolicus strains were classified into Cluster a,in which there was no apparent taxonomic diversity for the strains in the phylogenetic tree constructed by D1/D2,while the evolution distances among Cryptococcus podzolicus strains were easily identified by both MLST and MALDI-TOF-MS methods.Conclusion: MALDI-TOF-MS methods can be more effective for identification and classification of Cryptococcus species.Moreover,MALDI-TOF-MS is more efficient and accurate than MLST.
[Key words]multilocus sequence typing; matrix-assisted laser desorption/ionization time of flight mass spectrometry; Cryptococcus podzolicus; classification; developmental biology; evolution
*[基金项目]国家自然科学基金(31060006,31260029) ;贵州省社会发展科技攻关项目[黔科合SY字(2011) 3017号];贵阳市科技局社会发展与民生计划
[中图分类号]R379.5
[文献标识码]A
[文章编号]1000-2707(2016) 02-0139-06

