基于位置权重矩阵的核小体识别及功能分析
岁品品,邢旭东,王 宏,崔 颖
( 哈尔滨医科大学生物信息科学与技术学院, 哈尔滨 150081)
基于位置权重矩阵的核小体识别及功能分析
岁品品,邢旭东,王宏,崔颖*
( 哈尔滨医科大学生物信息科学与技术学院, 哈尔滨 150081)
摘要:为研究高通量的人类CD4+T细胞的核小体定位模式,使用迭代算法对核小体定位模式进行分类,并利用位置权重矩阵方法分别构建稳定核小体定位序列、动态核小体定位序列和连接区序列模型,通过十倍交叉验证评估模型性能,并与Segal方法与弯曲度方法进行比较,发现位置权重矩阵方法在敏感性、精度和准确性方面都具有一定优越性。同时采用滑窗法在全基因组选取候选序列进行核小体识别,挖掘核小体定位相关基因,并进行基因生物学进程功能富集分析,发现稳定与动态核小体、真实与潜在核小体对应的基因所参与调控的生物学过程各有不同,但也有一些生物学过程为不同类别核小体所共有,例如对细胞内大分子的调控功能。
关键词:核小体定位;位置权重矩阵;基因功能富集分析
核小体是真核生物染色质的基本组成单位。真核细胞内大约75%~90%的DNA与组蛋白相互缠绕。核小体在DNA序列上的确切位置对DNA序列参与的生物学功能有重要的影响。核小体定位在基因转录调控、DNA复制与修复、可变剪接等基本生命过程中都扮演着重要的角色[1-2],然而,在全基因组上核小体的精确位置却不是一成不变的,即在DNA序列上核小体定位呈现动态性,其定位过程、在转录过程中的调控功能非常复杂[3-4]。随着测序技术的快速发展,ChIP-chip与ChIP-seq等高通量技术已经绘制出核小体定位图谱,为研究核小体定位及其功能奠定了基础。同时,这些高分辨率的核小体定位图谱给采用生物信息方法预测活体内核小体定位提供了丰富的数据样本,已经开发出来多种预测算法识别核小体定位。
本文利用位置权重矩阵构建核小体定位模型,在全基因组上识别核小体定位,挖掘核小体定位基因,通过基因富集分析挖掘到核小体参与调控的生物功能,这有利于加强人们对核小体在全基因组的定位模式的全面认识,能够增加对核小体生物功能的了解,对核小体定位机制的研究以及核小体与基因调控的关系有一定的指导作用。
1材料和方法
1.1数据来源与处理
人类全基因组数据来源于UCSC[5],并计算全基因组中四种碱基的背景频率。人类核小体定位数据来自于Dustin E. Schones和 Kairong Cui所做的工作[6],人类全基因组基因定位数据从ensemble数据库中[7]。将每条染色体的每一个基因信息分类统计,找出基因的起止位置。根据Segal等人的工作,下载到Segal模型所用的酵母核小体数据[8]。
1.2方法
为了减少DNA序列本身的碱基偏好性对模型的影响,本文把位置频率矩阵转换为位置权重矩阵(Position Weight Matrix)。通过引入全基因组背景频率bi(i∈{A,G,C,T})来消除DNA序列本身碱基组成的偏好性根据公式(1)构建位置权重矩阵模型元素:
(1)
(2)
(3)
其中,qi,j是碱基i在核小体序列第j(j=(1,2,3...147))个位置出现的频率。元素值Si,j表示碱基i(A,C,G,T)在核小体序列第j位置上的权重值,根据核小体序列集合,获得位置权重矩阵模型S,为4×147的矩阵,根据公式(3)计算候选序列与模型的相似性,计算其相似性得分,得分越高说明相似性越强。本文利用上述方法分别对稳定核小体、动态核小体、连接区序列构建位置权重矩阵模型,并分别计算每条候选序列与三个模型的相似性得分,相似性得分最高者判断为相应模型对应的集合。
2结果
2.1核小体定位模式
利用迭代匹配算法,即将休眠状态下的核小体起止位置与激活状态下的核小体起止位置进行匹配,获得4种核小体定位模式。(1)如果核小体定位在激活状态下相对于休眠状态未发生任何位置的改变,则该核小体定位定义为完全稳定模式(Completely Stable Mode,CSM);(2)如果核小体定位在激活状态下相对于休眠状态向左或向右移动小于147 bp,则定义为滑动模式(Shift Mode,SM);(3)如果核小体定位在激活状态下相对于休眠状态向左或向右移动超过147 bp,则定义为完全动态核小体定位(Completely Dynamic Mode, CDM);(4)如果核小体定位在激活状态下相对于休眠状态下无核小体定位,则定义为核小体缺失模式(Delete Mode,DEM)(见图1)。
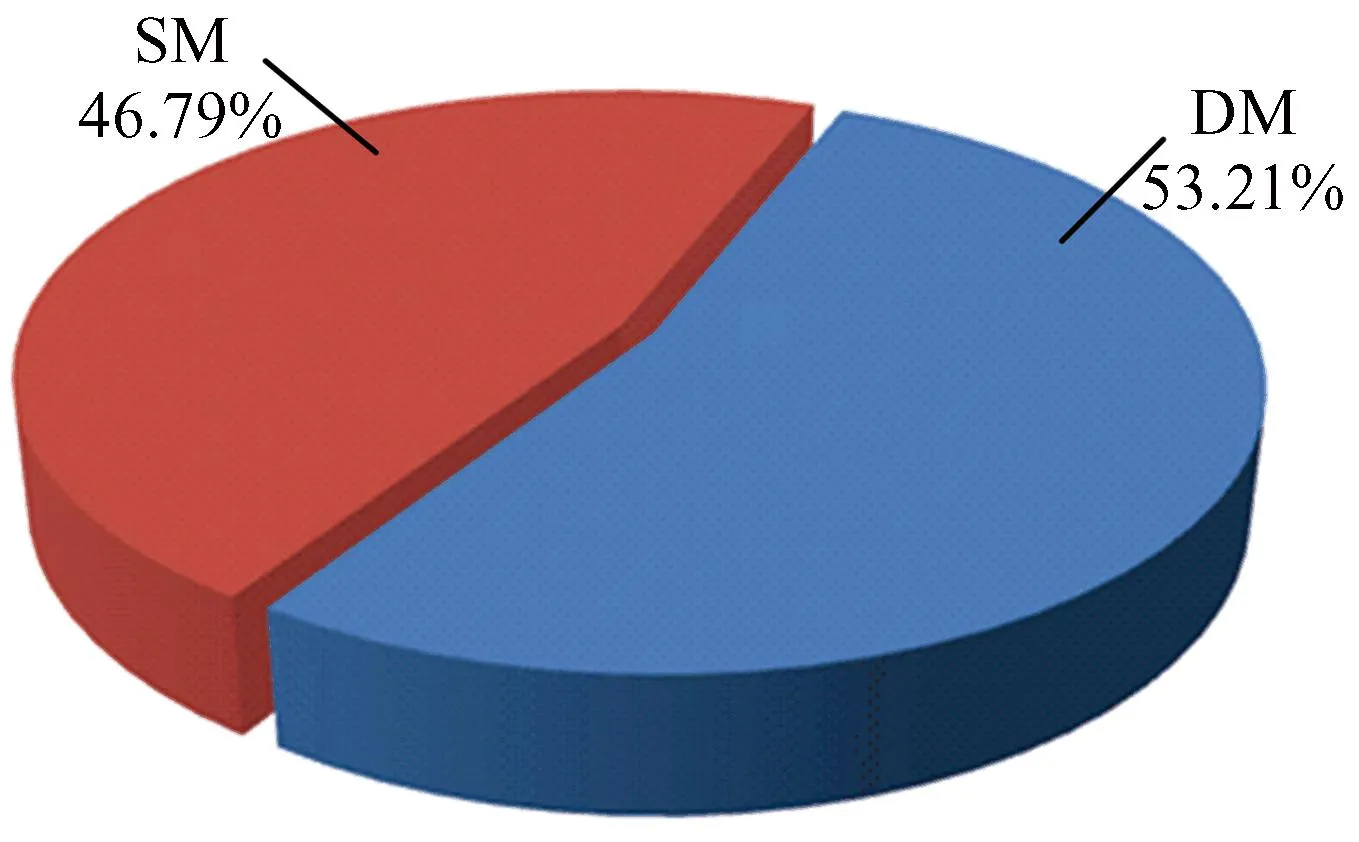
图1 核小体定位稳定模式和动态模式Fig.1 The nucleosome position of stable and dynamic pattern
本文分析核小体定位4种模式的DNA序列,发现CSM和SM存在很大相似性,其模式在DNA序列上的位置变化相对较小,因此将CSM和SM归为稳定模式(Stable Model, SM),而CDM和DM模式中核小定位呈现非常大的动态性,两者可能是同时相互协调发挥调控作用,因此将CDM和DEM归为动态模式(Dynamic Model, DM)。获得稳定模式核小体约53.21%,动态模式核小体定位约46.79%,核小体定位的多种模型可能和具体的生物过程有关。
2.2模型建立与模型比较
分别构建稳定核小体位置权重矩阵(Stable Nucleosome Position Weight Matrix, SNPM)、动态核小体位置权重矩阵(Dynamic Nucleosome Position Weight Matrix, DNPM)和连接序列位置权重矩阵(Linker Sequence Position Weight Matrix, LSM),并使用Wilcoxon-test检验三个模型间的差异是否具有显著性,对三个模型中4种碱基在1到147位置的权重差异性如表1和图2所示,结果表明两两模型间4种碱基的差异具有显著性,即三个模型之间存在显著差异,此差异为利用三模型识别核小体提供依据。
那天碰巧星期五,临下班时欧阳锋接到钱多多的一个电话。钱多多在电话中邀欧阳锋去鸿运酒楼吃海鲜。欧阳锋显得有些犹豫。电话那头的钱多没等欧阳锋回绝,说,欧阳锋,你不会连这点面子也不给吧?——你在单位门口等着,我马上让司机过去接你!

表1 模型间的差异性检验结果
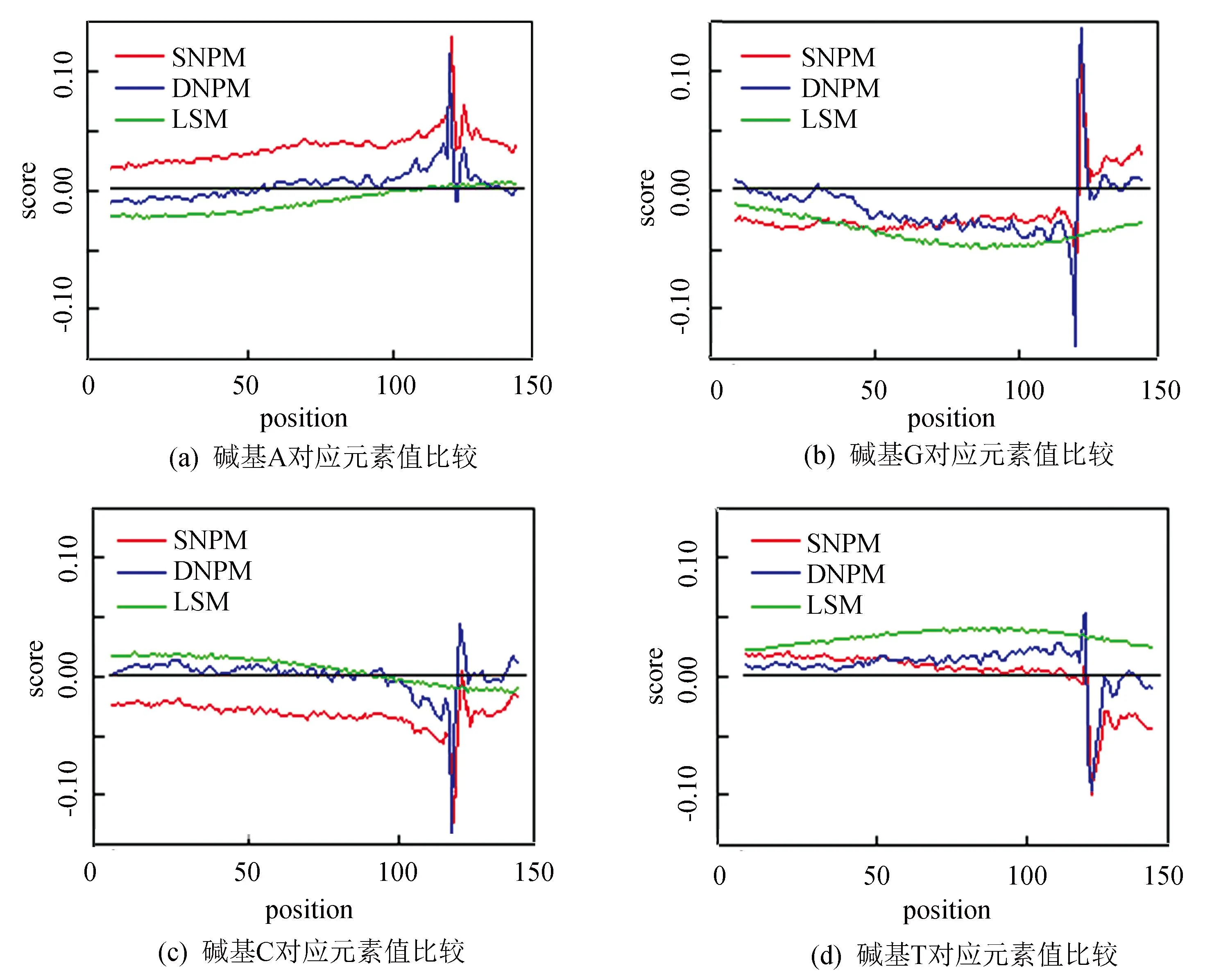
图2 模型间4种碱基对应位置元素值对比Fig.2 The compare of 4 types of bases according to related position in different models
注:彩图见电子版(http://swxxx.alljournals.cn/index.aspx)(2016年第1期)。
2.3模型性能评估
本文采用十倍交叉验证方法对模型的性能进行了评估,其性能评估指标为敏感性、特异性、精度和准确性如下列公式所示。
敏感性:Sensitivity=TP/(TP+FN)
(4)
特异性:Specificity=TN/(TN+FP)
(5)
精度:Precision=TP/(TP+FP)
(6)
准确性Accuracy=(TP+TN)/(TP+FN+FP+TN)
(7)
其中TP为真阳性数目、FP为假阳性数目、TN为真阴性数目和FN为假阴性数目十倍交叉验证,并与Segal[9]和弯曲度模型比较结果如表2所示。通过文献查找,Segal模型预测的敏感性为68.04%和(阳性)准确性42.32%。对Segal模型所用到的核小体数据进行处理,共得到60 073条酿酒酵母核小体序列和10 030条酿酒酵母连接区序列,利用位置权重矩阵方法进行模型评估,结果敏感性约为63.8%,特异度约为61.2%,精度约为90.8%,准确性约为63.5%,综合四项评估指标,位置权重矩阵模型要优于Segal模型,与弯曲度谱方法比较。弯曲度谱方法的敏感性为69.85%和(阳性)准确率为59.51%,本文方法敏感性为71.96%和准确性为75.40%均优于弯曲度谱方法[10]。因此可以将位置权重矩阵方法应用到人类核小体识别当中。
在全基因组上采用滑窗法,以单碱基为步长,147 bp为窗口宽度来选取候选序列,并去掉含有“N”的候选序列,24条染色体上的总候选序列条数为28亿多条,个别染色体候选序列集在硬件存储大小达到30 G以上,这对于硬件设备是一个严峻的考验。将候选序列集分别投入到模型中,根据打分公式(3)分别计算候选序列与SNPM、DNPM及LSM三个模型的相似度得分,并将候选序列归类到相似度得分最高的模型分类中。由于候选序列是采用滑动窗口法以单碱基为步长进行提取的,这种方法使候选序列中存在非常大的数据冗余,这使模型的识别结果也存在一定的冗余,为消除这种冗余对模型识别结果的影响,本方对经模型识别后的结果进行了去冗余。去冗余方法:将每个结果中的核小体候选序列与相邻的核小体候选序列的重叠(超过73 bp)情况合并为核小体区域,否则不合并,将此结果若核小体识别区域完全覆盖实验核小体定位为正确识别结果即稳定核小体定位(Stability Nucleosome Positioning,SNP)和动态核小体定位(Dynamic Nucleosome Positioning,DNP),否则认为识别结果为可能存在的核小体定位即潜在的核小体定位,包括潜在稳定核小体定位(Potential Stability Nucleosome Positioning,PSNP)和潜在动态核小体定位(Potential Dynamic Nucleosome Positioning,PDNP)。
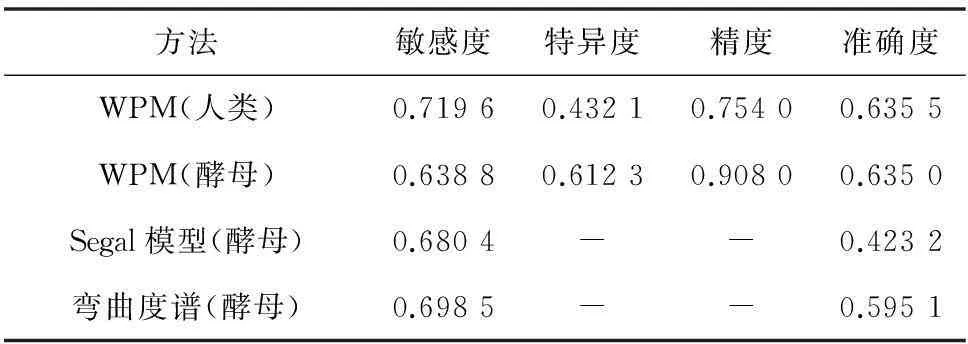
表2 模型性能比较
全基因组稳定核小体定位识别结果达到64%以上,全基因组动态核小体定位识别结果约为60%,模型预测的潜在稳定的核小体为35%以上,潜在的动态核小体定位结果为40%以上,此结果与模型评估的准确性基本一致,反应了模型不但有较好的发现真实核小体的能力,还可以有效地识别全基因组上潜在的核小体。
2.5挖掘核小体相关基因
为了分析核小体定位的功能,分别挖掘到核小体相关基因如图3所示。四类核小体相关的基因集合间存在很大交叠,但各集合也有相当一部分单独相关的基因存在。在真实核小体定位的相关基因集合中,大部分基因与真实核小体的两种定位(真实稳定核小体定位与真实动态核小体定位)都相关。
同样,在潜在核小体相关基因集合中,大部分基因与潜在核小体的两种定位(潜在稳定核小体定位于潜在动态核小体定位)都相关,说明大部分基因可能同时受到真实核小体不同定位或者是潜在核小体不同定位的调控作用。相比较而言,稳定核小体和潜在核小体相关基因之间的交集较小(真实稳定核小体定位与潜在稳定核小体定位之间,真实动态核小体定位与潜在动态核小体定位之间),说明真实核小体和潜在核小体同时调控同一个基因的机率相对较小。

图3 核小体定位相关基因Fig 3 The genes related to nucleosome positioning
注:SNP:真实稳定核小体;DNP:真实动态核小体;
PSNP:潜在稳定核小体;PDNP:潜在动态核小体。
Notes:SNP:Stability Nucleosome Positioning;DNP:Dynamic Nucleosome Positioning;PSNP:Potential Stability Nucleosome Positioning;PDNP:Potential Dynamic Nucleosome Positioning.
2.6功能富集分析
将四类核小体定位相关基因ID分别投入到DAVID9中进行Gene Ontology的Biological Process富集分析。为了使功能富集分析更加详尽减少冗余,选择Gene Ontology中的GOTERM_BP_4 ,显著性阈值P=0.001。并对显著性P值最小的前10个结果进行展示分析:
(1)如图4(a)所示,真实稳定核小体相关基因富集到的前10个生物学过程中涉及到细胞进程的调控(Positive regulation of cellular process、negative regulation of cellular process)、细胞内大分子调控(Biopolymer modification、cellular protein metabolic process、protein metabolic process、cellular macromolecule catabolic process)、细胞信号与通讯(Regulation of cell communication、regulation of signal transduction、intracellular signaling cascade)以及系统发育(Nervous system development)。
(2)如图4(b)真实动态核小体相关基因富集到的前10个生物学过程中,涉及到细胞进程的调控(Positive regulation of cellular process、negative regulation of cellular process)、细胞内大分子调控(Biopolymer modification、cellular protein metabolic process、positive regulation of macromolecule metabolic process、protein metabolic process positive regulation of macromolecule biosynthetic process、cellular macromolecule catabolic process)、细胞信号与通讯(Regulation of cell communication、regulation of signal transduction)说明真实核小体中,稳定核小体与动态核小体在调控功能上基本相似,除了在各生物学功能的显著性存在一定的差异外,真实稳定核小体还参与神经系统的发育。
(3)如图4(c)所示,潜在稳定核小体相关基因富集到的生物学过程满足显著性阈值的功能有9个。涉及到分化(Keratinocyte differentiation、epidermal cell differentiation、epithelial cell differentiation)、发育(Epidermis development、ectoderm development)、细胞内大分子调控(Cellular macromolecule biosynthetic process、regulation of macromolecule biosynthetic process)、转录事件(Transcription)、RNA代谢(Regulation of RNA metabolic process)。
(4)如图4(d)所示,潜在动态核小体相关基因富集到的生物学过程满足显著性阈值的功能也只有9个。分别参与的功能为分化(Epidermal cell differentiation、keratinocyte differentiation、epithelial cell differentiation)、发育(Epidermis development、ectoderm development、organ development、tissue development)、细胞内大分子调控(Cellular macromolecule biosynthetic process、protein-lipid complex assembly)。说明真实动态核小体和潜在动态核小体除在细胞大分子调控功能上类似、其他功能有很大差异。四种核小体都参与的功能为细胞内大分子的调控功能。

图4 基因富集分析结果Fig.4 The result of functional enrichment analysis
3结果与讨论
本文通过建立位置权重矩阵模型来识别核小体定位。研究结果显示,位置权重矩阵模型具有较高的敏感性和准确性,但假阳性率仍然比较高,原因可能是候选序列中每一条真正核小体前后都有与真正核小体相近的打分,但较高的假阳性也为挖掘试验中没有发现的核小体奠定了基础。另外,通过对挖掘得到的核小体相关基因进行功能富集分析,发现稳定与动态核小体、真实与潜在核小体对应的基因所参与调控的生物学过程各有不同,但也有一些生物学过程为不同类别核小体所共有,例如对细胞内大分子的调控功能。利用位置权重矩阵模型对在全基因组内选取的候选序列进行识别,除了发现实验中的已经发现的真实和稳定核小体之外,还挖掘到了一些具有核小体可能性的序列。对不同类别核小体相关的基因进行功能富集分析,发现真实与潜在、稳定与动态核小体区域相关的基因所参与调控的生物学过程这对核小体定位机制以及核小体与基因调控的关系的研究有一定的指导意义。我们推测一方面细胞通过全基因组范围内核小体定位模式的一致性来维持细胞的正常功能,另一方面细胞通过内部各类核小体定位模式的差异来发挥核小体的调控作用。不同的生长阶段、生理条件下细胞内基因的表达水平可能存在不同,其受很多因素的调控[12]。核小体通过具体的动态位置变化来隐蔽或暴露DNA上的蛋白结合位点,这些蛋白结合位点往往与转录因子等和基因表达紧密相关的蛋白质相结合来调控基因表达。虽然至今仍不能确定核小体定位的动态变化是引起基因表达水平变化的决定因素,但是至少两者之间存在着紧密的联系,值得进一步探索。
参考文献
[1]陈伟. 核小体定位对RNA剪接的影响及组蛋白变体的识别[D]. 呼和浩特:内蒙古大学, 2010.
CHEN Wei.The effect of nucleosome positioning on RNA splicing and the regcognition of histone rariants[D].Hohhot:Inner Mongolia University,2010.
[2] 蔡禄, 赵秀娟. 核小体定位研究进展[J]. 生物物理学报, 2009,25(6): 385-395.
CAI Lu, ZHAO Xiujuan. Advances in nucleosome positioning [J].Acta Biophysica Sinica,2009, 25(6): 385-395.
[3]SCHONES D E, CUI K, CUDDAPAH S, et al. Dynamic regulation of nucleosome positioning in the human genome[J]. Cell, 2008, 132(5): 887-898.
[4]JIANG C, PUGH B F.Nucleosome positioning and gene regulation:advances through genomics[J].Nature Reviews Genetics, 2009, 10(3): 161-172.
[5]KENT W J, SUGNET C W, FUREY T S, et al. The human genome browser at UCSC[J]. Genome Research, 2002, 12(6): 996-1006.
[6]ZHANG Y, SHIN H, SONG J S, et al. Identifying positioned nucleosomes with epigenetic marks in human from ChIP-Seq[J]. BMC Genomics, 2008, 9(1): 537.
[7]HUBBARD T J P,AKEN B L,BEAL K, et al. Ensembl 2007[J]. Nucleic Acids Research, 2007, 35(Suppl 1): D610-D617.
[8]SEGAL E, FONDUFE-MITTENDORF Y, CHEN L, et al. A genomic code for nucleosome positioning[J]. Nature, 2006, 442(7104): 772-778.
[9]GLYNN D J R , BRAD T S , DOUGLAS A H, et al. DAVID: Database for annotation,visualization,and integrated discovery[J]. Genome Biology, 2003, 4:R60(9): 54-56.
[10]SEGAL E, FONDUFE-MITTENDORF Y, CHEN L, et al. A genomic code for nucleosome positioning[J]. Nature, 2006, 442(7104): 772-778.
[11]张德金, 刘宏德, 袁志栋, 等. 基于Web技术的核小体在线预测平台实现[J]. 微计算机信息, 2010, 26(36): 185-187.
ZHANG Dejin,LIU Hongde,YUAN Zhidong,et al.Construction of an on-line platform of predicting nucleosomes based on web techniques[J].Microcomputer Information, 2010, 26(36): 185-187.
[12] TEIF V B,VAINSHTEIN Y,CAUDRON-HERGER M, et al. Genome-wide nucleosome positioning during embryonic stem cell development[J].Nature Structural & Molecular Biology, 2012, 19(11):1185-1192.
Nucleosome positioning identification and functional analysis on the position weight matrix
SUI Pinpin,XING Xudong,WANG Hong,CUI Ying*
(CollegeofBioinformaticsScienceandTechnology,HarbinMedicalUniversity,Harbin150081,China)
Abstract:This study was based on high throughout nucleosome positioning data of CD4+T cell in human genome to investigate the model of nucleosome positioning and category the nucleosomes.We constructed three the models by using position weight matrix, including stable nucleosome model, dynamic nucleosome model and linker sequences model respectively. Ten-fold cross validation was used to evaluate the performance of the three models, and the assessment results were compared with Segal model and curvature profile method.It was found that the position weight matrix method was superior to the other two methods in terms of sensitivity, precision and accuracy. At the same time the sliding window method is adopted to select candidate sequences in the genome to identify the nucleosomes. Furthermore we mined the related genes of nucleosome positioning and completed enrichment analysis of gene functions and found that different nucleosome positioning modes involved in both a certain similarity and difference in regulation function in biological processes.Whereas there are some biological processes are co-regulated by different nucleosome positioning modes,such as regulation of macromolecule.
Keywords:Nucleosome; Position weight matrix; Enrichment analysis of gene function
中图分类号:Q523
文献标志码:A
文章编号:1672-5565(2016)01-001-06
doi:10.3969/j.issn.1672-5565.2016.01.01
作者简介:岁品品,男,本科生,研究方向:生物信息学;E-mail:1447806377@qq.com.*通信作者:崔颖 ,女,讲师,研究方向:生物信息学;E-mail:cuiying204@163.com.
基金项目:黑龙江省卫生厅科研课题资助(2013129)。
收稿日期:2015-11-08;修回日期:2015-12-11.

