凡纳滨对虾TOR蛋白下游信号传导因子eIF4E2和eIF4E1A基因克隆与功能分析
辛 芳, 王 雷 刘 梅 王宝杰 蒋克勇 孙国琼
(1. 中国科学院海洋研究所 实验海洋生物学重点实验室, 青岛海洋科学与技术国家实验室, 海洋生物学与生物技术功能实验室, 山东 青岛 266071; 2. 中国科学院大学, 北京 100049)
凡纳滨对虾TOR蛋白下游信号传导因子eIF4E2和eIF4E1A基因克隆与功能分析
辛 芳1,2, 王 雷1, 刘 梅1, 王宝杰1, 蒋克勇1, 孙国琼1
(1. 中国科学院海洋研究所 实验海洋生物学重点实验室, 青岛海洋科学与技术国家实验室, 海洋生物学与生物技术功能实验室, 山东 青岛 266071; 2. 中国科学院大学, 北京 100049)
为研究mTOR信号通路在凡纳滨对虾(Litopenaeus vannamei)氨基酸代谢中的调控作用, 本研究利用RACE技术首次克隆获得了凡纳滨对虾肌肉组织中eif4e2和eif4e1a基因的全长cDNA序列。eif4e2基因cDNA序列全长1 069 bp, 开放阅读框699 bp, 编码232个氨基酸; eif4e1a基因cDNA序列全长3579bp, 开放阅读框627bp, 编码208个氨基酸。氨基酸序列比对和进化分析均证实eIF4E2和eIF4E1A为目前已知的eIF4E家族蛋白的同源蛋白。利用实时荧光定量PCR的方法研究了eif4e2和eif4e1a基因在凡纳滨对虾不同组织中的表达差异以及注射雷帕霉素或氨基酸后肌肉中eif4e2和eif4e1a基因表达量的变化情况, 探讨了eif4e2和eif4e1a基因在细胞生长中的调控作用。结果表明, 凡纳滨对虾eif4e2和eif4e1a基因在眼柄、肝胰脏、肠道、胃、鳃丝和肌肉等组织中均有表达; 其中eif4e2基因在肌肉中的表达量显著高于其他组织(P<0.05), eif4e1a基因在肠道和肌肉中都有较高表达量, 且在肠道中的表达量显著高于其在肌肉中的表达量(P<0.05); 注射雷帕霉素后2 h内肌肉中eif4e2和eif4e1a基因表达量都出现显著下降(P<0.05); 肌肉中eif4e1a基因的表达量在单独注射亮氨酸或精氨酸4 h内均未出现变化, 但在同时注射亮氨酸和精氨酸后表达量显著增加(P<0.05); 肌肉中eif4e2基因在注射亮氨酸、精氨酸及亮氨酸加精氨酸后表达量都明显提高(P<0.05), 且同时注射亮氨酸和精氨酸后, 基因表达变量明显比单独注射亮氨酸或精氨酸后变化量大。本研究从动物分子营养学角度出发, 克隆了凡纳滨对虾mTOR信号通路中eif4e2和eif4e1a两个基因, 并证明其能够通过mTOR信号通路接收不同氨基酸信号来调控细胞生长, 对于深入了解对虾的生长和代谢调控机制、饲料配方的优化以及建立合理的养殖管理模式都具有重要的意义。
eIF4E; 凡纳滨对虾(Litopenaeus vannamei); mTOR; 氨基酸; 生长
动物细胞生长调控是一个受多因素影响的复杂过程, 它不仅受到动物生长不同时期的限制, 营养条件等细胞内外环境条件也都是重要的影响因素。只有在营养元素充足且外界理化条件合适的情况下,动物细胞才能进行快速的营养代谢, 并获得最快生长[1]。在机体细胞中, mTOR(mechanistic target of rapamycin)信号通路接收来自于营养素、生长因子或者环境胁迫等信号, 通过调控细胞的合成代谢和分解代谢, 实现对细胞生长和生理活动的精确调控,其中最典型的就是对蛋白质翻译的调控[2-3]。另外, mTOR信号通路也调控脂类物质合成以补充新增殖细胞的细胞膜形成[4]; 当细胞中能量水平较低时, 通过激活H1F1α的转录和翻译促进糖酵解产生能量[5];还参与抑制细胞的自我吞噬和溶酶体形成[6]。mTOR信号通路接收营养素信号, 并对下游效应因子做出调控主要是由细胞环境中氨基酸浓度决定的[7], 并且有研究表明: 亮氨酸和精氨酸对mTOR信号通路的调节作用尤其显著[8-9]。
eIF4E(Eukaryotic translation initiation factor 4E)是mTOR信号通路下游的信号传导因子, 能够通过eIF4E-BP(eIF4E binding protein)接收来自TOR蛋白的信号, 而起到调节细胞内蛋白质合成的重要作用[10]。eIF4E作为蛋白质翻译起始复合物eIF4F的关键部分,是调节细胞内蛋白质合成的关键因子[11]。当蛋白质翻译起始时, eIF4E首先结合到mRNA的5′帽结构上,然后募集eIF4G和eIF4A, 形成eIF4F复合物, 再将核糖体小亚基招募到mRNA上开始肽链合成[12]。Altmann等[13]克隆获得了小鼠(Mus musculus)eif4e基因的cDNA序列, 并发现当用小鼠eif4e基因代替酵母细胞中eif4e基因时, 酵母仍然有正常的细胞活性,并能够进行生长和繁殖, 说明eIF4E与其他翻译因子之间的相互作用是高度保守的。2004年, Javier等[14]从斑马鱼(Danio rerio)中克隆了两个eif4e家族的基因, 并发现eif4e1a和eif4e1b两个基因在功能上有较大差异, eif4e1a广泛表达于斑马鱼的各个组织, eif4e1b只在早期胚胎的性腺和肌肉中表达。
凡纳滨对虾是世界对虾养殖的主要经济品种,目前针对其营养调控以及细胞生长的分子营养学研究鲜有报道。开展水产养殖动物分子营养学研究可以通过对不同营养状态下动物个体基因表达谱的差异分析, 发现更多用于评价营养状态的分子标记物,有助于确定水产动物的营养需要量[15]。
在斑马鱼和小鼠等模式生物eif4e基因研究的基础上, 本研究克隆获得了凡纳滨对虾eif4e2和eif4e1a基因(根据其序列与已知序列的同源性命名)的cDNA序列, 分别对其进行了比对和分析; 用实时荧光定量PCR的方法检测了eif4e2和eif4e1a基因的组织特异性表达以及雷帕霉素(Rapamycin)、亮氨酸(Leu)和精氨酸(Arg)对eif4e2和eif4e1a基因在肌肉组织中表达水平的影响。研究证实了eif4e2和eif4e1a基因存在于凡纳滨对虾的mTOR信号通路中, 且能够接收氨基酸信号调控蛋白质合成, 进而促进细胞生长。为研究对虾等甲壳动物的营养调控机理提供了重要的理论基础, 也为水产养殖实际生产中对虾饲料配方调配和水产养殖动物生长机制研究提供了参考。
1 材料与方法
1.1 实验动物
实验用凡纳滨对虾体长9.0 cm±1.0 cm, 养殖和实验均于青岛市胶南瑞兹海珍品养殖基地进行。实验过程中, 对虾养殖在约1 m3的白色方形水槽中,自然光照, 盐度为30, 持续充气, 室温。
1.2 基因克隆
1.2.1 引物设计与合成
根据课题组前期对凡纳滨对虾进行转录组测序分析的结果, 对数据库中注释为mTOR信号通路的基因序列, 利用primer Premier 5.0 设计5′RACE和3′RACE实验所需特异性引物以及实时荧光定量PCR引物, 见表1。引物均由南京金斯瑞生物科技有限公司合成。

表1 用于基因克隆和荧光定量的引物序列Tab. 1 Nucleotide primers used in gene cloning and real-time PCR
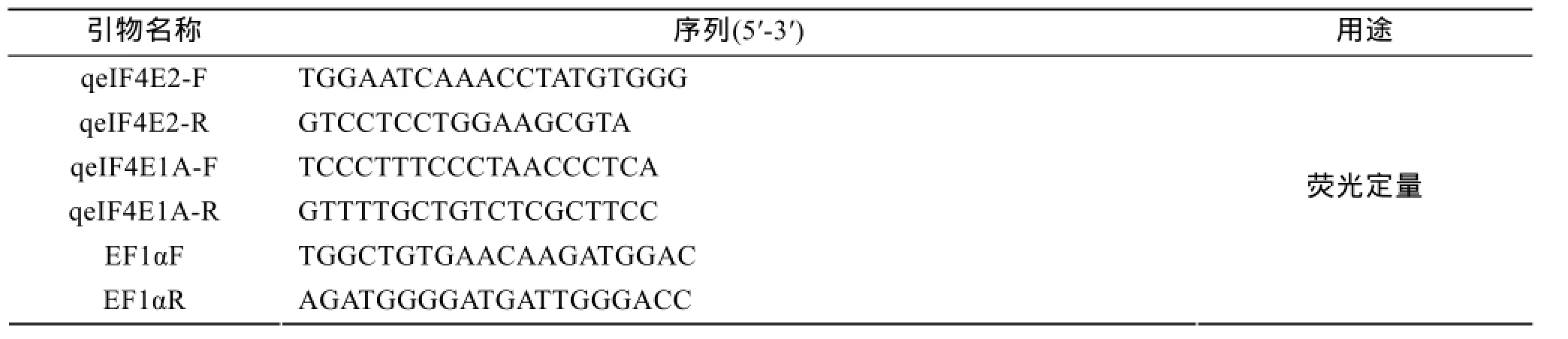
引物名称 序列(5′-3′) 用途qeIF4E2-F TGGAATCAAACCTATGTGGG qeIF4E2-R GTCCTCCTGGAAGCGTA qeIF4E1A-F TCCCTTTCCCTAACCCTCA qeIF4E1A-R GTTTTGCTGTCTCGCTTCC EF1αF TGGCTGTGAACAAGATGGAC EF1αR AGATGGGGATGATTGGGACC荧光定量
1.2.2 cDNA全长基因克隆
取凡纳滨对虾肌肉组织约50 mg, 用Trizol法抽提总RNA, 以oligo d (T)或3′CDS为引物, 用Takara M-MLV反转录合成cDNA, 储存于–20℃冰箱中。
3′RACE: 以3′CDS为引物反转录合成cDNA模板, UPM/特异性引物3F1和NUP/特异性引物3F2为引物进行巣式PCR, 扩增得到3′端序列。
5′RACE: 以oligo d(T)为引物反转录合成cDNA,经纯化后, 用Takara TdT酶在cDNA末端加poly(C),以此为模板, oligo d(G)和特异性引物5R1、5R2、5R3为引物进行巣式PCR, 扩增得到5′端序列。
取60 μL获得的PCR产物进行琼脂糖凝胶电泳,切下目的条带, 用3S柱式琼脂糖DNA小量快速纯化试剂盒回收。回收的产物连接到Trans-T1载体上,转入Trans DH5α大肠杆菌中进行序列测定, 序列测定由南京金斯瑞生物科技有限公司完成。
将测序获得的3′端序列、5′端序列以及转录组获得序列进行拼接, 得到基因的cDNA全长。
1.2.3 基因的生物信息学分析
利用NCBI网站查找同源基因序列和氨基酸序列;开放阅读框的寻找利用ORF Finder (Open Reading Frame Finder)在线软件; 序列翻译和蛋白质相似性分析利用DNAMAN和ClustalW等软件; 系统进化树构建利用MEGA5.1软件的最大似然法进行; 利用http: //smart.embl-heidelberg.de/网站提供的蛋白质序列分析工具查找功能域。
1.3 基因表达的组织分布
随机挑选4只凡纳滨对虾, 分别取眼柄、肝胰脏、肠道、胃、鳃丝和肌肉等新鲜组织约50 mg放于Trizol中, 采用Trizol法提取总RNA, 利用Takara Reverse Transcriptase M-MLV(RnaseH)反转录合成cDNA, 用于组织差异表达荧光定量。
1.4 雷帕霉素和氨基酸注射实验设计
进行雷帕霉素应答实验时, 将凡纳滨对虾随机分成两组养殖于室内水槽中。第一组注射500 mmol/L的雷帕霉素, 第二组注射75% DMSO(雷帕霉素的溶剂), 每尾虾自倒数第二腹节肌肉注射100 μL。注射开始记为0 h, 分别在0.5、1、2、3和4 h取样。雷帕霉素注射浓度和取样时间根据Oshiro等[16]mTOR信号通路相关研究及实验室原有研究[17]确定。每尾虾取第一腹节肌肉组织约50 mg, 提取总RNA, 每组每个时间点取4只虾作为平行。
进行氨基酸应答实验时, 将凡纳滨对虾随机分成4组。第一组注射0.1 mol/L亮氨酸, 第二组注射0.1 mol/L精氨酸, 第三组注射0.1 mol/L亮氨酸+精氨酸(1︰1), 第四组注射0.01 mol/L pH7.4 PBS溶液(氨基酸溶剂), 每尾虾自尾节处腹腔注射 100 μL。从0 时起, 分别在 0.5、1、2, 3和4 h取样。注射浓度和取样时间根据Hara等[18-19]mTOR信号通路相关研究及实验室原有研究确定。每尾虾取第一腹节肌肉组织约 50 mg, 提取组织总 RNA, 每组每个时间点取4只虾作为平行。
1.5 实时荧光定量PCR分析
采用SYBR Green I嵌合荧光法进行Real-time PCR, 20 μL反应体系为: 2×TransStart Top Green qPCR SuperMix 10 μL, 正反向引物(10 nmol/L)各0.4 μL, cDNA 1 μL, ddH2O 8.2 μL。反应条件: 95℃-30 s; 95℃-5 s, 60℃-30 s, 45个循环; 结束。以凡纳滨对虾EF1α为内参基因, 运用Line-Gene K软件得到各样品均一化处理的Ct值, 采用2–ΔΔCt法求各样品mRNA的相对表达量, 然后运用Excel和SPSS软件对数据进行统计分析, 并运用Origin Pro 8.0绘图。
2 实验结果
2.1 基因序列分析
将扩增获得的3′端序列、5′端序列以及转录组测序获得的部分序列拼接得到: eIF4E2的cDNA 全长序列, 共1 069 bp, 开放阅读框699 bp, 3′非翻译区74 bp,5′非翻译区296bp, 翻译得到232个氨基酸, 见图1; eIF4E1A的cDNA 全长序列, 共3 579 bp, 开放阅读框627 bp, 3′非翻译区42 bp, 5′非翻译区2 910 bp, 翻译得到208个氨基酸, 见图2。eife2基因和eif4e1a基因编码的蛋白质均含有一个典型的保守IF4E结构域, 见图3和图4。

图1 凡纳滨对虾eif4e2完整cDNA序列及推断氨基酸序列Fig. 1 Complete nucleotide and deduced amino acid sequence of eif4e2 from L. vannamei
2.2 蛋白质氨基酸序列分析利用MEGA5.1软件的最大似然法将相关已有研究报道的动物eIF4E氨基酸序列与本研究凡纳滨对虾的两个eIF4E家族氨基酸序列分别构建进化树,见图5和图6。eIF4E2氨基酸序列进化树显示, 凡纳滨对虾与日本对虾单独聚为一支, 亲缘关系最近,其次与其他昆虫纲节肢动物亲缘关系较近, 与哺乳动物亲缘关系较远。eIF4E1A氨基酸序列进化树显示,凡纳滨对虾与其他昆虫纲节肢动物聚为一个分支,同时凡纳滨对虾单独为一个小分支; 其他软体动物和脊椎动物聚于另一分支。
将凡纳滨对虾的eIF4E2推测氨基酸序列与日本对虾(Marsupenaeus japonicas)、湿木白蚁(Zootermopsis nevadensis)、毕氏粗角蚁(Cerapachys biroi)、小鼠、人类(Homo sapiens)、黄牛(Bos taurus)等的eIF4E2氨基酸序列比对, 相似性分别为99%、72%、65%、62%、62%和60%。将凡纳滨对虾的eIF4E1A推测氨基酸序列与湿木白蚁、尼日尔蚁(Lasius niger)、蚂蚁(Cerapachys biroi)、蜜蜂(Melipona quadrifasciata)、加洲海兔(Aplysia californica)、太平洋牡蛎(Crassostrea gigas)、非洲爪蟾(Xenopus laevis)、斑马鱼、胡瓜鱼(Osmerus mordax)等的eIF4E1A(或eIF4E1)氨基酸序列比对, 相似性分别为64%、61%、63%、61%、63%、65%、63%、61%和62%。将本研究获得的eIF4E2和eIF4E1A推测氨基酸序列进行比对, 其相似性为35%。利用Swiss-Modle预测的两个eIF4E蛋白质三维结构都含有8个反向平行的β折叠和3个长α螺旋。


图2 凡纳滨对虾eif4e1a完整cDNA 序列及推断氨基酸序列Fig. 2 Complete nucleotide and deduced amino acid sequence of eif4e1a from L. vannamei

图3 eIF4E2中IF4E超级家族结构域的位置Fig. 3 Position of domain IF4E superfamily in deduced protein eIF4E2

图4 eIF4E1A中IF4E超级家族结构域的位置Fig. 4 Position of domain IF4E superfamily in deduced protein eIF4E1A
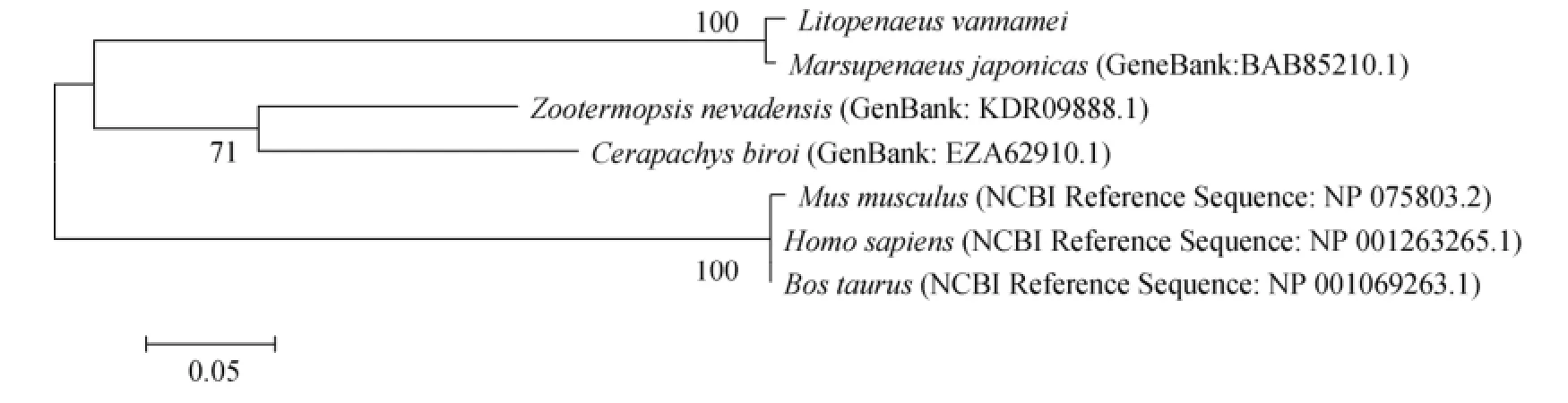
图5 基于eIF4E2氨基酸序列的系统进化树Fig. 5 Phylogenetic tree based on eIF4E2 amino acid sequence
2.3 eif4e2和eif4e1a基因的组织表达分析
利用实时荧光定量PCR分析了eif4e2和eif4e1a基因在不同组织中的表达情况。结果显示, eif4e2和eif4e1a基因在眼柄、肝胰腺、肠道、胃、鳃丝和肌肉等不同组织中均有表达(图7)。eif4e2基因在肌肉中的表达显著高于其他组织, 其次是肠道、胃和肝胰腺, 眼柄和鳃丝中表达量较低; eif4e1a基因在肠道和肌肉中的表达显著高于其他组织, 且在肠道中的表达高于肌肉, 在眼柄、肝胰腺、胃和鳃丝中的表达量没有显著性差异。
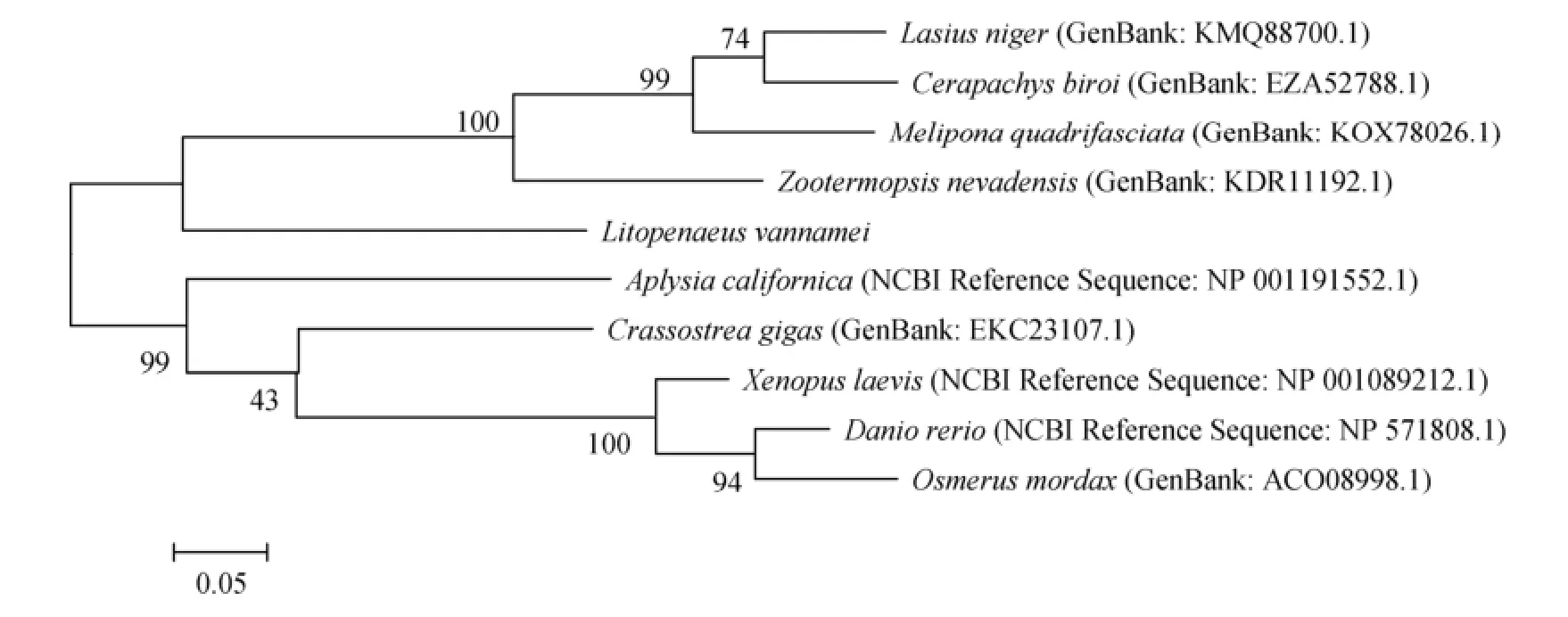
图6 基于eIF4E1A氨基酸序列的系统进化树Fig. 6 Phylogenetic tree based on eIF4E1A amino acid sequence
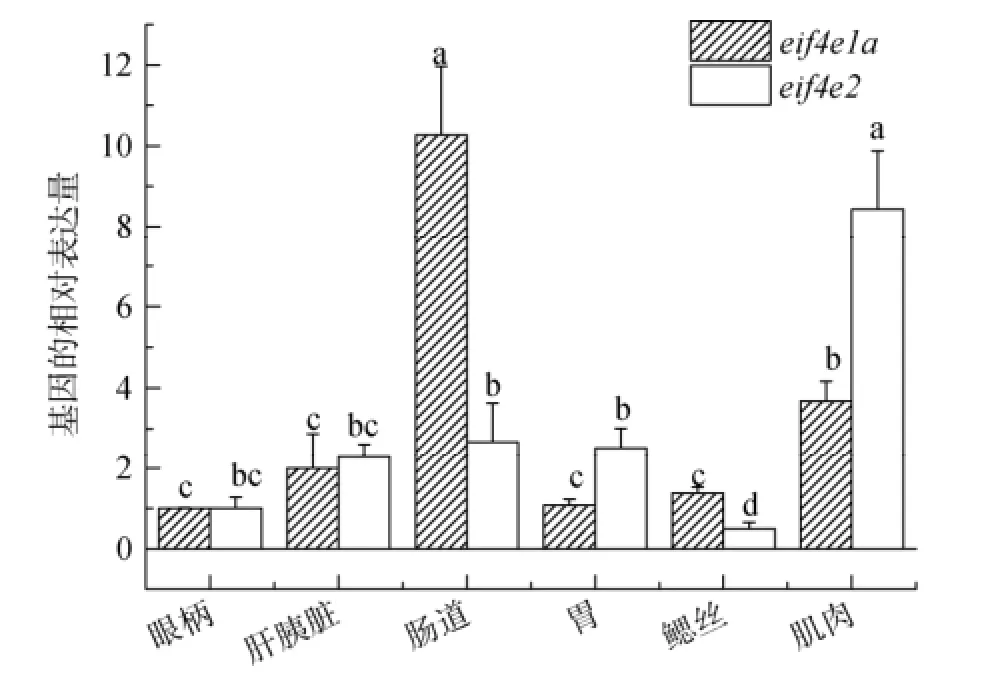
图7 凡纳滨对虾eif4e2和eif4e1a基因在不同组织中的相对表达量Fig. 7 Relative expression level of eif4e2 and eif4e1a in different tissues of L. vannamei

图8 eif4e2和eif4e1a基因应答雷帕霉素注射的相对表达量Fig. 8 Relative expression levels of eif4e2 and eif4e1a in muscle responding to rapamycin
2.4 基因应答雷帕霉素注射的表达分析
为了验证凡纳滨对虾eIF4E家族因子是否存在于mTOR信号通路中且受TOR蛋白的调控, 选择TOR蛋白抑制剂雷帕霉素进行肌肉注射。通过实时荧光定量PCR分析了注射雷帕霉素后, 凡纳滨对虾肌肉组织中eif4e2和eif4e1a两个eIF4E家族基因表达量的变化。以0时不做处理的对虾肌肉组织中基因表达量为对照, 计算和分析注射雷帕霉素和DMSO(对照组)0.5、1、2、3、和4 h后两个eif4e家族基因表达量的变化。结果如图8所示, 注射雷帕霉素后, 凡纳滨对虾肌肉中两个eif4e家族基因的表达量都受到显著影响, eif4e1a在注射雷帕霉素0.5 h后表达量出现显著下降(P<0.05), 直到第三小时才恢复注射前水平。eif4e2在注射雷帕霉素1 h后表达量出现显著下降(P<0.05), 直到第4小时才恢复注射前水平。
2.5 基因应答氨基酸注射的表达分析
为了进一步验证凡纳滨对虾eIF4E家族因子通过mTOR信号通路接收细胞外氨基酸信号来发挥作用, 选择mTOR信号通路较为敏感的亮氨酸和精氨酸进行腹腔注射。通过Reatime-PCR实时荧光定量的相对定量法分析了注射不同氨基酸后, 凡纳滨对虾肌肉中eif4e2和eif4e1a两个基因表达量的变化。以0时不做处理的对虾肌肉组织中基因表达量为对照, 计算和分析注射亮氨酸(Leu)、精氨酸(Arg)、亮氨酸加精氨酸(LeuArg)和PBS(对照组)0.5、1、2和4 h后两个eif4e基因表达量的变化。如图9所示, 注射亮氨酸后, eif4e1a的表达量在4 h内没有出现显著性变化, 而eif4e2的表达量在1 h后显著升高; 注射精氨酸后, eif4e1a的表达量在4 h内没有出现显著性变化,而eif4e2的表达量在第4小时出现显著升高(P<0.05);同时注射亮氨酸和精氨酸后, eif4e2和eif4e1a基因在0.5 h都出现极显著升高(P<0.01), 且升高值显著大于只注射一种氨基酸时eif4e表达量的升高值。
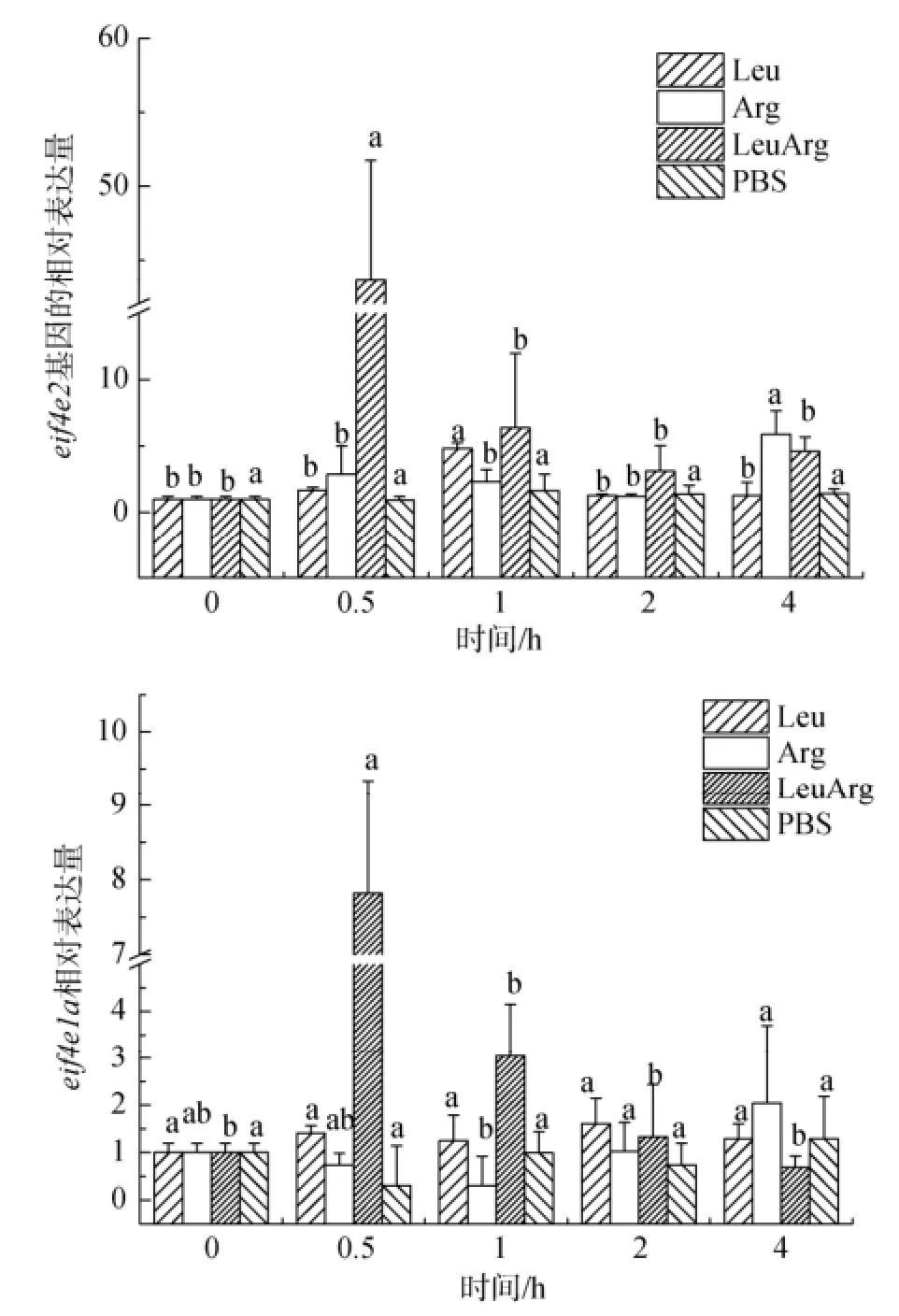
图9 eif4e2和eif4e1a基因应答氨基酸注射的相对表达量Fig. 9 Relative expression levels of eif4e2 and eif4e1a in muscle responding to amino acid
3 讨论
现有关于eIF4E家族基因的研究报道, 大多是进行cDNA序列克隆和进化分析, 对其功能的研究报道还较少。关于eif4e基因家族功能的研究主要有:线虫(Caenorhabditis elegans)eIF4E家族不同成员在不同细胞状态下与mRNA帽结构发生相互作用进而调控蛋白质翻译[20-21], 表明eIF4E家族成员的多样性分化不能简单地用功能冗余来解释; 在斑马鱼中, eIF4E1A和eIF4E1B蛋白都有与mRNA帽结构及eIF4G相结合的氨基酸残基, 但是只有eIF4E1A能与mRNA帽结构及eIF4G发生相互作用起始蛋白质合成,而eIF4E1B不能与之发生相互作用[14]。Diane等[22]研究发现, 在没有雷帕霉素存在的情况下, 哺乳动物细胞中eif4e的过量表达会促进细胞生长。迄今尚未见甲壳动物eif4e基因家族的研究报道, 本研究在甲壳动物中克隆了eif4e基因家族成员eif4e2和eif4e1a并对其功能进行了初步研究。
3.1 基因序列比较分析
通过基因克隆获得凡纳滨对虾eif4e2和eif4e1a基因cDNA全长序列, 将其氨基酸序列分别与其他动物的eIF4E氨基酸序列进行比对, eIFE2氨基酸序列与其他已报道动物eIF4E2的序列相似性在62%~ 99%, eIFE1A氨基酸序列与其他已报道动物eIF4E1 (或eIF4E1A)的序列相似性在61%~64%, 证明了eif4e2和eif4e1a基因分别与其他已报道动物eif4e2和eif4e1a基因是同源的。系统进化树分析把凡纳滨对虾eIF4E2与日本对虾eIF4E2单独聚为一支, eIF4E1A也单独聚为一支, 都与昆虫纲节肢动物聚在不同分支, 说明eif4e基因在节肢动物的甲壳纲和昆虫纲之间存在较大的进化变异。
根据NCBI保守结构域预测, 凡纳滨对虾eIFE2和eIFE1A蛋白质都含有IF4E结构域。Marcotrigiano等[23]利用X射线蛋白质结构分析发现, eIF4E蛋白质呈杯状, 包括1个弯曲、8个反向平行β片层和3个长α螺旋, 本研究利用Swiss-Modle预测的凡纳滨对虾的两个eIF4E蛋白质三维结构都与Marcotrigiano等的研究结果一致, 两个eIF4E蛋白质的IF4E结构域具有相似的α/β结构, 呈杯状, 包括8条反向平行的β链弯曲形成的β片层以及3条长α螺旋。两个eIF4E家族蛋白预测结构中, 色氨酸残基多位于杯状结构的底部, Marcotrigiano 等[24]研究认为eIF4E蛋白质的杯状结构底部含有较多色氨酸残基, 可以便于与 mRNA 5′端帽结构及eIF4G结合, 进而保证eIF4E参与蛋白质翻译起始复合物的形成, 起到启动mRNA翻译的关键作用。
3.2 基因的组织表达分析
研究发现, eif4e1基因普遍存在于真核生物中,而eif4e2基因仅存在于后生动物中, 两个基因在动物各个组织中都有广泛表达[25]。本研究基因差异表达分析结果显示, 凡纳滨对虾eif4e2和eif4e1a基因在所检测的眼柄、肝胰脏、肠道、胃、鳃丝和肌肉等组织中均有表达; eif4e2基因在肌肉中有较高表达;而eif4e1a基因在肠道和肌肉中都有较高表达, 且在肠道中的表达高于肌肉。斑马鱼eif4e1a基因在各个组织中广泛表达, 但是其在肌肉中的表达量高于肠道、心脏和鱼鳍等组织中的表达[14]。eif4e基因在肌肉或肠道中的较高表达与其调节细胞内蛋白质代谢的生物学功能相对应: 肌肉作为动物体中生长最快的组织, 肌蛋白合成量最多; 肠道作为主要的营养消化和吸收的场所, 消化和吸收相关的代谢酶合成量较高。
3.3 注射雷帕霉素后基因功能分析
雷帕霉素在真核细胞内的受体是1个12 kDa的小分子蛋白质, 称为FK506结合蛋白12(FK506-binding protein 12, FKBP12), 而FKBP12-RAPA复合物的靶向物质是TOR, FKBP12与RAPA结合可以抑制TOR的活性[26]。TOR蛋白活性被抑制后, 磷酸化4EBP的能力降低, 低磷酸化状态的4EBP能够与eIF4E的结合, 因此eIF4E不能够参与蛋白质翻译起始复合物的形成, 从而抑制蛋白质合成[27]。本研究设计了雷帕霉素注射实验, 验证凡纳滨对虾eIF4E因子是否存在于mTOR信号通路中且受TOR蛋白的调控。结果发现, 与对照组相比, 注射雷帕霉素后凡纳滨对虾肌肉中eif4e2和eif4e1a基因的表达量在2 h内都出现显著下降, 表明在凡纳滨对虾体内eif4e2和eif4e1a基因表达量与mTOR信号通路密切相关,在TOR蛋白活性受到抑制后, 其表达量下降, 从而导致细胞蛋白合成作用降低, 最终影响细胞的生长。
3.4 注射氨基酸后基因功能分析
Peponi等[28]通过体外细胞培养研究表明TOR蛋白能磷酸化4E-BP1, 从而使其失去与eIF4E结合的活性, 增加elf4e的表达, 促进与细胞生长相关蛋白的翻译。研究发现在小鼠肝脏细胞中, 亮氨酸和精氨酸通过激活mTOR信号通路促进蛋白质的合成, 并能抑制肝脏细胞自噬, 同时也可能抑制肠上皮细胞自噬介导的蛋白质降解[29]。本研究通过等浓度氨基酸注射实验, 分析了凡纳滨对虾mTOR信号通路在接收氨基酸信号后, 肌肉中eif4e2和eif4e1a基因的表达量变化情况, 结果显示, 与PBS对照组相比, 肌肉中eif4e1a基因仅在同时注射亮氨酸加精氨酸后表达量发生变化; 肌肉中eif4e2基因在注射亮氨酸、精氨酸及亮氨酸加精氨酸后表达量都发生明显变化,且同时注射亮氨酸加精氨酸后, 基因表达变量明显比单独注射亮氨酸或精氨酸后变化量大。据此推测,亮氨酸和精氨酸对凡纳滨对虾肌肉mTOR信号通路的激活作用可能是叠加的。有关于小鼠胚胎发育的研究发现, 亮氨酸和精氨酸对胚胎滋养层TOR蛋白复合物的激活作用是叠加的, 然而亮氨酸和精氨酸对囊胚发育的激活作用是相互独立的[30]。另外, 根据凡纳滨对虾eif4e2和eif4e1a基因应答氨基酸注射后在肌肉中的表达差异, 及其在不同组织中的差异表达, 推测它们可能调控不同类型蛋白质的合成, 或者不止有mTOR信号通路调控其活性。
4 总结
本研究从动物分子营养学角度出发, 克隆了凡纳滨对虾mTOR信号通路中eif4e2和eif4e1a两个基因, 并分析了他们的氨基酸序列结构特征和进化地位; 同时研究了营养素对凡纳滨对虾基因表达的调控作用, 为营养素在动物体内的生理功能提供了更深入的认识。
[1] 仁和, 占秀安. 水产动物氨基酸营养研究进展[J]. 饲料研究, 2006, 2: 41-43. Ren He, Zhan Xiuan. Research progress on amino acid nutrition of aquatic animals[J]. Feed Research, 2006, 2: 41-43.
[2] Kim J, Guan K L. Amino acid signaling in TOR activation[J]. Annu Rev Biochem, 2011, 80: 1001-1032.
[3] Ma X J M, Blenis J. Molecular mechanisms of mTOR-mediated translational control[J]. Nature Reviews Molecular Cell Biology, 2009, 10(5): 307-318.
[4] Laplante M, Sabatini D M. An emerging role of mTOR in lipid biosynthesis[J]. Current Biology, 2009, 19(22): R1046-R1052.
[5] Duvel K, Yecies J L, Menon S, et al. Activation of a metabolic gene regulatory network downstream of mTOR complex 1[J]. Molecular Cell, 2010, 39(2): 171-183.
[6] Koren I, Reem E, Kimchi A. DAP1, a novel substrate of mTOR, negatively regulates autophagy[J]. Current Biology, 2010, 20(12): 1093-1098.
[7] Proud C G. Regulation of mammalian translation factors by nutrients[J]. Biochem, 2002, 269: 5338-5349.
[8] Kimball S R. Regulation of translation initiation by amino acids in eukaryotic cells[J]. Prog Mol Subcell Biol, 2001, 26: 155-184.
[9] Hara K. Amino acid sufficiency and mTOR regulate p70 S6 kinase and eIF4E-BP1 through a common effector mechanism[J]. J Biol Chem, 1998, 273: 14484-14494.
[10] Laplante M, Sabatini D M. mTOR signaling in growth control and disease[J]. Cell, 2012, 149(2): 274-293.
[11] 夏良平, 曾宗渊. eIF4E作用及调节机制[J]. 国外医学分子生物学分册, 2001, 23(5): 280-282. Xia Liangping, Zeng Zongyuan. The function and regulation mechanism of eIF4E[J]. Foreign Medical Sciences Series of Molecular Biology, 2001, 23(5): 280-282.
[12] Sonenberg N, Hershey J W B, Mathews M B. Translational control of gene expression[M]. Cold Spring Harbor Laboratory Press, 2000: 39.
[13] Altmann M, Müller P P, Pelletier J, et al. A mammalian translation initiation factor can substitute for its yeast homologue in vivo[J]. Journal of Biological Chemistry, 1989, 264(21): 12145-12147.
[14] Javier R, Bhaveh J, Scott C F, et al. Two zebrafish eif4e family members are differentially expressed and functionally divergent[J]. The Journal of Biological Chemistry, 2004, 279(11): 10532-10541.
[15] 李茜, 李建国. 动物分子营养学的研究进展[J]. 黑龙江畜牧兽医, 2015, 1: 50-52. Li Qian, Li Jianguo. Research progress in molecular nutriology of animal[J]. Heilonjiang Animal Science and Veterinary Medicine, 2015, 1: 50-52.
[16] Oshiro N, Yoshino K, Hidayat S, et al. Dissociation of raptor from mTOR is a mechanism of rapamycin induced inhibition of mTOR function[J]. Genes to Cells 2004, 9(4): 359-366.
[17] Sun S, Wang B, Jiang K, et al. Target of rapamycin (TOR) in Fenneropenaeus chinensis: cDNA cloning, characterization, tissue expression and response to amino acids[J]. Aquaculture Nutrition, 2015, 21: 1-9.
[18] Hara K, Yonezawa K, Weng Q P, et al. Amino acid sufficiency and mTOR regulate p70 S6 kinase and eIF-4E BP1 through a common effector mechanism[J]. Journal of Biological Chemistry, 1998, 273(23): 14484-14494.
[19] Kimball S R, Shantz L M, Horetsky R L, et al. Leucine regulates translation of specific mRNAs in L6 myoblasts through mTOR-mediated changes in availability of eIF4E and phosphorylation of ribosomal protein S6[J]. Journal of Biological Chemistry, 1999, 274(17): 11647-11652.
[20] Keiper B D, Lamphear B J, Deshpande A M, et al. Functional characterization of five eIF4E isoforms in Caenorhabditis elegans[J]. Journal of Biological Chemistry, 2000, 275(14): 10590-10596.
[21] Miyoshi H, Dwyer D S, Keiper B D, et al. Discrimination between mono– and trimethylated cap structures by two isoforms of Caenorhabditis elegans eIF4E[J]. EMBO Journal, 2002, 21(17): 4680-4690.
[22] Fingar D C, Salama S, Tsou C, et al. Mammalian cell size is controlled by mTOR and its downstream targets S6K1 and 4EBP1/eIF4E[J]. Genes and Development, 2002, 16(12): 1472-1487.
[23] Marcotrigiano J, Gingras A C, Sonenberg N, et al. Cocrystal structure of the messenger RNA 5′cap- binding protein (eIF4E) bound to 7-methyl-GDP[J]. Cell, 1997, 89(6): 951-961.
[24] Marcotrigiano J, Gingras A C, et al. Cap-dependent translation initiation in eukaryotes is regulated by a molecular mimic of eIF4G[J]. Molecular Cell, 1999, 3: 707-716.
[25] Bhavesh J, Amy C, Rosemary J. Characterization of mammalian eIF4E-family members[J]. European Journal of Biochemistry, 2004, 271: 2189-2203.
[26] Harding M W, Galat A, Uehlingd E, et al. A receptor for the immunosuppressant FK506 is a cis-trans peptidylprolyl isomerase[J]. Nature, 1989, 341(6244): 758-760.
[27] Ma X M, Blenis J. Molecular mechanisms of mTOR-mediated translational control[J]. Nature Reviews Molecular Cell Biology, 2009, 10(5): 307-318.
[28] Peponi E, Drakos E, Reyes G, et al. Activation of mammalian target of rapamycin signaling promotes cell cycle progression and protects cells from apoptosis in mantle cell lymphoma[J]. The American Journal of Pathology, 2006, 169(6): 2171-2180.
[29] Blommaart E F, Luiken J J, Blommaart P J, et al. Phosphorylation of ribosomal protein S6 is inhibitory for autophagy in isolated rat hepatocytes[J]. J Biol Chem, 1995, 270: 2320-2326.
[30] Gonzalez I M, Martin P M, Burdsal C, et al. Leucine and arginine regulate trophoblast motility through mTOR-dependent and independent pathways in the preimplantation mouse embryo[J]. Developmental biology, 2012, 361(2): 286-300.
Received:Dec. 18, 2015
Cloning and functional analysis of TOR downstream signaling factors eIF4E2 and eIF4E1A of Litopenaeus vannamei
XIN Fang1,2, WANG Lei1, LIU Mei1, WANG Bao-jie1, JIANG Ke-yong1, SUN Guo-qiong1
(1. Institute of Oceanology, Chinese Academy of Sciences, Key Laboratory of Experimental Marine Biology, Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China; 2. University of Chinese Academy of Sciences, Beijing 100049, China)
eIF4E; Litopenaeus vannamei; mTOR; amino acid; growth
The aim of this study was to explore the regulation function of mTOR signaling in the amino acid metabolism of Litopenaeus vannamei. We cloned the full length cDNA of eif4e2 and eif4e1a genes from the muscle of L. vannamei for the first time. The cDNA of eif4e2 was 1069 bp and contained a 699 bp open reading frame that encoded 208 amino acids. The cDNA of eif4e1a was 3579 bp and contained a 627 bp open reading frame that encoded 232 amino acids. We individually compared the deduced eIF4E2 and eIF4E1A amino acid sequences of L. vannamei with other known sequences. The results confirmed their homology. We used a real-time polymerase chain reaction (PCR) to detect the tissue-specific expressions of eif4e2 and eif4e1a in L. vannamei and the responses of eif4e2 and eif4e1a in muscle to the injection of rapamcyin or amino acid (leucine or arginine). We also investigated the functions of eif4e2 and eif4e1a in the cell growth of L. vannamei. The results showed that the eif4e2 and eif4e1a genes were expressed in all the detected tissues, including the eyestalk, hepatopancreas, stomach, intestine, gill, and muscle. The relative expression level of eif4e2 was significantly higher than that of other tissues (P < 0.05). The relative expression levels of eif4e1a in intestine and muscle were both higher than those in other tissues, and that of the intestine was higher than that of the muscle (P < 0.05). The relative expression levels of eif4e2 and eif4e1a in muscle decreased after the injection of rapamcyin (P < 0.05). The relative expression level of eif4e1a was unchanged four hours after the injection of unitary leucine or arginine, while it increased significantly after the simultaneous injections of leucine and arginine (P < 0.05). The relative expression level of eif4e2 increased significantly after the injection of unitary leucine, unitary arginine, or both leucine and arginine (P < 0.05), and the relative expression level of eif4e2 after the simultaneous injections of leucine and arginine was significantly higher than that after injection of unitary leucine or arginine. In conclusion, with respect to animal molecular nutriology, we cloned full-length cDNA of eif4e2 and eif4e1a genes from the muscle of L. vannamei, and the results indicate that eif4e2 and eif4e1a can accept the amino acid signal and regulate cell growth in L. vannamei muscle. This study represents a significant contribution to our understanding of the mechanisms of growth and metabolism regulation, the optimization of feed formulas, and the establishment of a reasonable management model for shrimp culture development.
S917.4
A
1000-3096(2016)12-0071-11
10.11759//hykx 20151218002
(本文编辑: 谭雪静)
2015-12-18;
2016-03-25
国家自然科学基金项目(41406151)
[Foundation: Program of National Natural Science Funds of China, No.41406151]
辛芳(1989-), 女, 山东日照人, 硕士研究生, 主要从事水产动物分子营养学研究, 电话: 0532-82898723, E-mail: rzxinfang@163.com;刘梅, 通信作者, 电话: 0532-82898722, E-mail: liumei@qdio.ac.cn
——雷帕霉素

