日照海蝉DNA条形码及其分子系统进化分析
刘丰铭, 李金泽, 梁启冰*, 蔺胜娟
(1.山东省日照第一中学,山东日照 276826; 2.山东省日照青岛路中学,山东日照 276826)
日照海蝉DNA条形码及其分子系统进化分析
刘丰铭1, 李金泽1, 梁启冰1*, 蔺胜娟2*
(1.山东省日照第一中学,山东日照 276826; 2.山东省日照青岛路中学,山东日照 276826)
摘要[目的]研究COI基因区段作为DNA条形码在识别日照海蝉地理群体的可行性和有效性。[方法]以日照海蝉为研究对象,用引物LCO1490和HCO2198扩增线粒体COI基因片段序列,产物双向测序,用Clustal W进行DNA序列比对,用MEGA 6.0软件采用邻接法(Neighbor-Joing, NJ)和最大简约法(maximum parsimony, MP)构建分子系统发育树。[结果]获得大小为658 bp的COI基因,A、T、C、G的平均含量分别为30.24%、36.63%、16.41%、16.72%,其中A+T平均含量(66.87%)明显高于G+C平均含量(33.13%),NJ和MP系统发育树均表明日照海蝉属于蝉蟹总科(Hippoidea)眉足蟹科(Blepharipoda),与形态学的分属结果一致。[结论]采用COI基因序列作为DNA条形码进行海蝉分类鉴定具有一定的可行性。
关键词日照海蝉;DNA条形码;COI基因;系统进化
DNA Barcoding and Molecular Evolution of Rizhao Haichan
LIU Feng-ming1, LI Jin-ze1, LIANG Qi-bing1*, LIN Sheng-juan2*(1.Rizhao No.1 Middle School of Shandong, Rizhao, Shandong 276826; 2.Rizhao Qingdao Road Middle School, Rizhao, Shandong 276826)
Abstract [Objective] The aim was to study the feasibility and effectiveness of usingCOIparticular gene segment as the DNA barcoding in identifying Rizhao Haichan geographical group.[Method] With Rizhao Haichan as research object, the mitochondrial cytochrome C oxidase subunit I (COI) gene fragment was amplified by primers LCO1490 and HCO2198, the product was sequenced and the DNA sequence alignment was carried out by ClustalX.The phylogenetic trees were constructed using Neighbor-Joining method and Maximum Parsimony (on MEGA6.0).[Result] TheCOIgene was found 658 bp in size, and among all the sites, the average proportion of base A, T, C and G were 30.24%, 36.63%, 16.41% and 16.72%, respectively.A+T base pair accounted for 66.87% of the mitochondrialCOIgene, which was significantly higher than that of G+C base pair(33.13%).The results of NJ and MP method indicated that the related species could gather in the same branch, and the Rizhao Haichan belongs to the family Blepharipoda in superfamily Hippoidea.[Conclusion] DNA barcoding based on mitochondrialCOIgene sequence is applicable in the classification and identification of Anomura.
Key words Rizhao Haichan; DNA barcoding;COIgene; Phylogeny
日照海蝉,学名为解放眉足蟹,又称海蝉蟹,是一种介于虾和蟹之间的海洋甲壳动物。海蝉营养丰富,富含钙磷钾等矿物质。近年来由于海蝉被作为海鲜特色菜端上餐桌,消费者对其需求量大大增加,从而导致其采挖量随之升高;加之沿海污染物的排放破坏了其生存环境,严重影响了海蝉的繁殖成活率,致使海蝉的生存数量一直处于下降趋势。因此,对日照海蝉地方特有物种进行抢救性收集、保护已刻不容缓。
DNA条形码技术是通过对一个标准目的基因的DNA序列进行分析从而进行物种鉴定的技术,对于发现新物种和隐存种、鉴定濒危物种和鉴别外来入侵种具有重要意义。Hebert等[1-2]于2003年首次提出DNA条形码的概念,对其物种分类和鉴定意义予以肯定,并建议利用COI的特定区段作为DNA条形码的基础。COI基因相对保守,有足够的变异,序列长度合适,被广泛应用于动物遗传变异、系统发育分析及物种鉴定中[3]。研究表明,以COI基因为标记的DNA条形码能够准确地对海洋生物进行物种鉴定,可以选用COI基因作为各海洋生物条形码数据库的标准条形码。笔者分析日照地方物种海蝉COI基因片段核苷酸序列,并探讨该基因片段作为海蝉鉴定DNA条形码的可行性,以期了解海蝉的系统发育关系,为地方物种资源保护和利用提供依据。
1材料与方法
1.1试验材料海蝉样本采自山东日照,取其部分肌肉组织,用75%乙醇固定,4 ℃保存备用。
1.2试验方法基因组提取采用王茜等[4]的方法。PCR体系:灭菌蒸馏水37.75 μL,10×ExTaqBuffer 5.00 μL,2.5 mmol/L dNTP Mixture 4.00 μL,10 μmol/L LCO1490 2.50 μL,10 μmol/L HCO2198 2.50 μL,DNA模板2.00 μL TaKaRa ExTaqHS (5 U/μL)0.25 μL。
PCR反应条件:95 ℃预变性5 min;95 ℃变性30 s,47 ℃退火30 s,72 ℃延伸45 s,35个循环;72 ℃延伸7 min,PCR产物4 ℃保存。COI基因片段扩增引物分别为LCO1490: 5′-GGT CAA CAA ATC ATA AAG ATA TTG G-3′和HCO2198: 5′-TAA ACT TCA GGG TGA CCA AAA AAT CA-3′[5];PCR产物送上海生工生物工程技术有限公司纯化并双向测序。
1.3序列分析所得正反向序列利用SeqMan软件(DNAStar 7.2.1软件包)进行拼接,如正反序列出现不配对时,则需要根据荧光图谱进行手工校正,以确保每一个位点上的碱基都准确无误。将拼接和校正后得到的序列通过内置于Bioedit 7.0.9软件的claustal W程序进行排序,并去掉正反向引物序列。
将日照海蝉COI基因序列通过BLAST工具逐一与GenBank和BOLD数据库中可用的条形码序列进行比对,基于最大匹配率对序列进行确认,以确保物种鉴定的准确性。
利用MEGA 6.0软件,采用邻位相连法(NJ)和最大简约性法(MP)构建系统发育树并重复抽样分析(Replication=1 000)。
2结果与分析
2.1COI序列扩增结果以LCOI490和HC02198为PCR引物,能够成功扩增日照海蝉的COI基因序列,片段长度为709 bp(图1)。
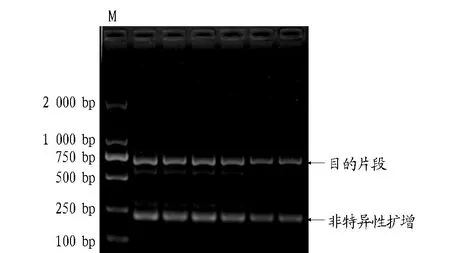
图1 COI基因片段PCR扩增结果Fig.1 PCR amplification results of COI gene fragment
2.2序列特征与分析除掉正反方向引物序列,LCOI490和HC02198扩增得到长度为658 bp的COI基因核苷酸片段(GenBank登录号KJ700466),且通过多重比较在序列中未发现插入和缺失位点。在COI基因中,A、T、C、G的平均含量分别为30.24%、36.63%、16.41%、16.72%,其中A+T平均含量(66.87%)明显高于G+C平均含量(33.13%),表现出碱基组成的偏倚性,这与无脊椎动物线粒体基因碱基组成特点相符合。

表1 异尾下目DNA条形码COI基因序列

图2 基于COI基因构建的邻位相连法(NJ)系统发育树Fig.2 The phylogenetic tree of neighbor-joining(NJ) method based on COI gene
2.3分子进化分析在NCBI中获得异尾下目各科属COI基因的序列(表1),基于COI序列,分别应用邻位相连法(NJ)和最大简约性法(MP)构建NJ和MP系统发育树(图2和图3),由图2、3可知,日照海蝉属于蝉蟹总科(Hippoidea)眉足蟹科(Blepharipoda),这说明分子系统学的结果与形态学的分属结果一致。
3结论与讨论
该研究表明,日照海蝉COI基因不存在碱基插入缺失现象,A+T含量偏高。A+T含量高的偏倚现象不仅存在于鱼类、贝类和头足类等水产动物中,在己报道的虾蟹类COI基因碱基组成中也普遍存在A+T含量偏倚现象,这也符合无脊椎动物线粒体DNA碱基组成的特点。
由于动物界线粒体(mt)DNA进化速率快于细胞核DNA,包括细胞色素B(COB)和细胞色素氧化酶第一亚基(COI)基因在内的mtDNA已被用于系统发育和系统地理学研究[6]。该研究采用COI的通用引物对日照海蝉进行扩增,可顺利获得相应序列,表明COI的通用引物在蝉蟹总科物种中具有普遍适用性。在该研究中,利用COI基因序列,通过条形码间隙和系统发育进化树都可以有效地进行鉴别,表明采用基于COI基因的DNA条形码技术进行分类鉴定是可行的。通过DNA条形码分子系统发育研究,明确了日照海蝉属于蝉蟹总科(Hippoidea)眉足蟹科(Blepharipoda),这与形态学分类鉴定结果一致[7]。
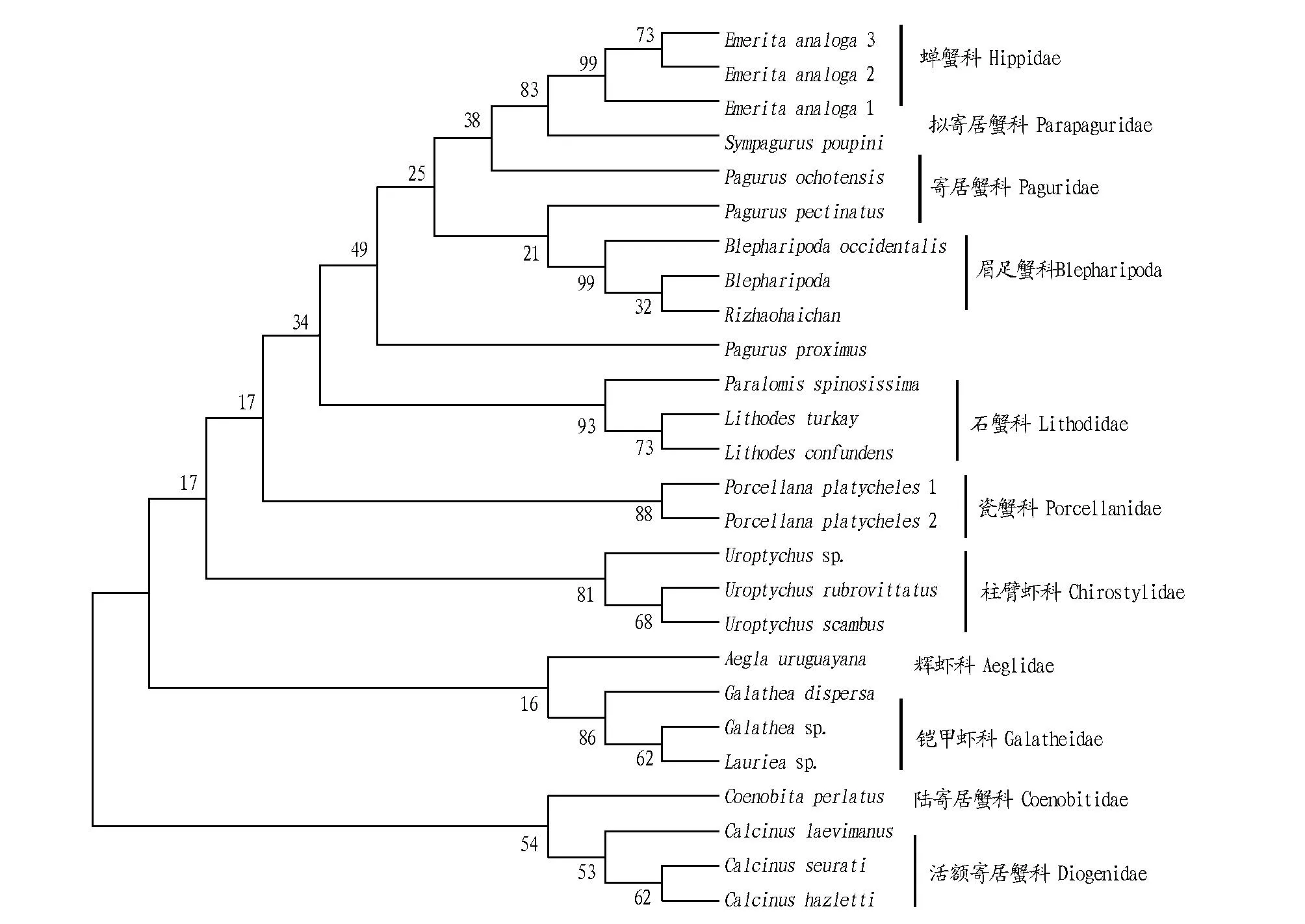
图3 基于COI基因构建的最大简约性法(MP)系统发育树Fig.3 The phylogenetic tree of maximum parsimony(MP) based on COI gene
DNA条形码技术获得了很多学者的认可并被广泛应用,可以弥补形态学物种鉴定的不足,快速对部分零散组织和形态特征不完整的样品进行鉴定,也不受生物生长发育时期的限制,是一种可以辅助形态学进行物种识别和鉴定的新兴技术,但DNA条形码技术不能完全取代传统的形态学鉴定方法。DNA条形码技术并不是基于DNA条形码序列而创建的一种新的分类系统,也不是部分学者认为的可以取代林奈的分类系统,而是一种快速识别物种和辅助传统分类法进行物种鉴定和发现新种的分类技术。
参考文献
[1] HEBERT P D,CYWINSKA A,BALL S L.Biological identifications through DNA barcodes[J].Proceedings of the royal society of london series B: Biological sciences, 2003, 270: 313-321.
[2] HEBERT P D,RATNASINGHAM S,WAARD J R.Barcoding animal life: Cytochromecoxidase subunitIdivergences among closely related species[J].Phil Trans R Soc Lond B, 2003, 270:96-99.
[3] 李敏, 席丽, 朱卫兵, 等.基于DNA条形码的中国普缘蝽属分类研究(半翅目:异翅亚目)[J].昆虫分类学报, 2010(1):36-42.
[4] 王茜, 金毅成.4种常见鲤科鱼类DNA条形码的研究[J].安徽农业科学, 2014(27):9281-9282.
[5] VRIJENHOEK R.DNA primers for amplification of mitochondrial cytochromecoxidase subunit I from diverse metazoan invertebrates[J].Mol Mar Biol Biotechnol, 1994,3: 294-299.
[6] AVISE J C,ELLIS D.Mitochondrial DNA and the evolutionary genetics of higher animals[J].Phil Trans R Soc London,1986, 312(1154):325-342.
[7] 王雪梅.解放眉足蟹(Blepharipoda liberate Shen)形态特征观察[J].渔业科学进展, 2015, 36(1):74-78.
收稿日期2016-01-08
作者简介刘丰铭(1998- ),男,山东日照人,高中生。*通讯作者,梁启冰,高级教师,硕士,从事信息与科技创新教育工作。*通讯作者,蔺胜娟,高级教师,从事数学与科技创新教育工作。
中图分类号S 188
文献标识码A
文章编号0517-6611(2016)03-133-03

