转lpaat和gpd1基因工程藻的快速筛选
王潮岗,李 逸,胡章立
深圳大学生命与海洋科学学院,深圳市海洋生物资源与生态环境重点实验室,广东深圳 518060

Received:2015-06-15;Accepted:2015-11-16
Foundation:National Natural Science Foundation of China (31470389, 31470431)
† Corresponding author:Professor Hu Zhangli. E-mail: huzl@szu.edu.cn
Citation:Wang Chaogang, Li Yi, and Hu Zhangli. Rapid screening of the transgenic algae haboringlpaatandgpd1[J]. Journal of Shenzhen University Science and Engineering, 2016, 33(1): 33-40.(in Chinese)

【生物工程 / Bioengineering】
转lpaat和gpd1基因工程藻的快速筛选
王潮岗,李逸,胡章立
深圳大学生命与海洋科学学院,深圳市海洋生物资源与生态环境重点实验室,广东深圳 518060
摘要:根据莱茵衣藻密码子偏好性对源于油菜的溶血磷脂酸酰基转移酶基因(lpaat)和酵母的甘油3-磷酸酰基转移酶基因(gpd1)进行优化,获得适合莱茵衣藻表达的c-lpaat和c-gpd1基因,将其连接到pH124质粒中,构建相应的衣藻表达载体.通过珠磨法进行遗传转化,经Zeomycin抗性筛选和聚合酶链式反应(polymerase chain reaction, PCR)验证,获得导入c-lpaat和c-gpd1的转基因藻.利用半定量反转录PCR(reverse transcription-PCR, RT-PCR)技术筛选高效表达c-lpaat和c-gpd1的转基因藻,目的基因的转录水平分别提高了56.38%和75.95%,其藻细胞的脂肪酸含量也得到相应增加,与基因表达情况一致.证明莱茵衣藻可以有效表达外源高等植物的基因,利用半定量RT-PCR可以快速筛选出潜在的高油脂含量转基因工程藻株.
关键词:基因工程;转基因藻;莱茵衣藻; lpaat基因; gpd1基因;三酰甘油合成
目前,生物柴油被认为是最理想的可再生能源之一,利用微藻制备生物柴油具有不占用耕地、易收易养和含油量高等特点,因而成为近年的研究热点.作为研究微藻脂肪酸代谢的最佳实验对象,莱茵衣藻具有遗传背景清晰和转化方法成熟等优势,可通过提高脂肪酸代谢路径中部分酶的活性来增加脂肪酸的含量[1-3].光合作用过程中得到的碳约有6.1%被用于草酰乙酸(oxalacetic acid,OAA)的合成,这一步由糖代谢路径中的限速酶——磷酸烯醇式丙酮酸羧化酶(phosphoenolpyruvate carboxylase,PEPC)催化;约24%的碳进入三羧酸循环(tricarboxylicacidcycle,TCA)路径;还有约24%的碳用于氨基酸合成;剩下的碳多用于脂肪酸合成.因此,可通过多个调节途径来调控脂肪酸的合成,包括过表达或抑制某些限速酶[4-6].
尽管利用微藻生产生物柴油具有多种优势,但进入商业化生产还面临许多问题,如脂肪酸含量未达到商业化生产水平、外源基因表达水平不高、需要构建高效表达载体等[7-9].微藻大规模培养技术及获得油脂含量达到商业开发水平的工程藻仍是整个生产环节的关键,采用更先进的微藻培养及采收装置、革新脂肪酸提取方法及创新相关工艺流程均可降低其生产成本.利用微藻生产生物柴油,未来关注的热点有:① Kennedy路径中各个酰基转移酶的改造;② 利用转录因子综合调控脂类代谢过程;③ 研究脂肪酸自动分泌的相关基因,以减少脂肪酸的提取成本[10].目前,溶血磷脂酸酰基转移酶(lysophosphatidic acid acyltransferase, LPAAT)和甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase, G3PDH)已被证明是三酰甘油合成途径中重要的合成酶,可以提高拟南芥等物种的油脂含量.本研究拟将经过密码子优化的油菜lpaat和酵母的gpd1基因导入莱茵衣藻cc-849的基因组中,通过调控莱茵衣藻细胞三酰甘油的合成来提高其油脂含量.
1实验方法
1.1材料和试剂
莱茵衣藻(Chlamydomonas reinhardtii cc-849)由美国杜克大学衣藻遗传研究中心提供,一直培养在Tris磷酸盐(Tris acetate phosphate,TAP)培养基中培养.莱茵衣藻表达载体pH124具有抗性筛选基因ble, 使宿主细胞获得腐草霉素和Zeomycin抗性,由深圳大学海洋生物资源与生态环境重点实验室保存. 其他材料包括:总基因组提取试剂盒(universal DNA extraction kit ver.3.0 Takara);RNA提取试剂盒(RNAiso plus for total RNA,Takara),反转试剂盒(SYBR®Premix ex taqTMII).
1.2载体构建
根据莱茵衣藻的密码子偏好性对油菜的lpaat基因(GenBank 登陆号:AY616009)和酿酒酵母的gpd1基因(GenBank 登陆号:Z24454)进行优化,分别得到适合莱茵衣藻表达目的片段c-lpaat和c-gpd1, 两端分别添加限制性内切酶位点Nhe Ⅰ和PmaC Ⅰ,送英韦创津生物公司合成.目的基因c-lpaat和c-gpd1分别经Nhe Ⅰ和PmaC Ⅰ酶切、电泳后获得,再连接到莱茵衣藻表达载体pH124的Nhe Ⅰ和PmaC Ⅰ位点中,分别获得pH124-c-lpaat和pH124-c-gpd1重组质粒,如图1所示.pH124载体中含有Hsp70A-RBCS2合成启动子、 RBCS2终止子和ble基因(使转化细胞具有腐草霉素抗性和博来霉素抗性).

图1 载体构建图Fig.1 (Color online)Construction of expression vector
1.3重组质粒的鉴定
采用试剂盒(QIAGEN®plasmid purification kit)提取质粒,分别采用聚合酶链式反应(polymerase chain reaction, PCR)和酶切进行质粒鉴定.
分别以载体两侧序列pH124-F和pH124-R为引物,通过PCR从质粒pH124-c-lpaat和pH124-c-gpd1中扩增出目的片段.PCR程序如下:94 ℃ 3 min;94 ℃ 1 min,58 ℃ 1min,72 ℃ 1 min,25个循环;72 ℃ 10 min.pH124-F:5′-TGACCTCCACTT TCAGCGACA-3′;pH124-R:5′-ACTTGAGAGCAGT ATCTTCCATCCA-3′.
通过Nhe Ⅰ和PmaC Ⅰ限制性内切酶消化重组质粒pH124-c-lpaat和pH124-c-gpd1,37 ℃温育1 h后,电泳进行分离,获得所插入的目的片段.
1.4莱茵衣藻的遗传转化
采用珠磨法对莱茵衣藻cc-849进行转化,转化方法参照文献[2].根据平板上长出的单克隆数与转化所用藻细胞数的比值,求得转化效率E为
E=A/(BV)
(1)
其中, A为单克隆数; B为转化所用藻细胞浓度; V为转化所用藻体积.
1.5转基因藻的PCR检测
采用总基因提取试剂盒提取总DNA,提取方法参照说明书.以提取的转基因藻基因组DNA为模板,c-lpaat-F/c-lpaat-R和c-gpd1-F/c-gpd1-R为引物进行PCR扩增,扩增程序为:95 ℃ 2 min;95 ℃ 30 s,60 ℃ 15 s,72 ℃ 15 s,25个循环;72 ℃ 5 min.引物序列见表1.

表1 引物列表
1.6转基因藻转录水平的分析
使用RNAiso plus for total RNA试剂盒(Takara)提取莱茵衣藻总RNA,方法参考说明书.提取获得一系列转化子的总RNA,经定量反转成cDNA后作为模板,以c-lpaat-F/c-lpaat-R和c-gpd1-F/c-gpd1-R为引物进行PCR扩增,获得目的基因片段.同时,以莱茵衣藻的β-actin基因为内参,对目的基因表达情况进行半定量分析,凝胶电泳所得结果用Photoshop软件进行灰暗度比对,根据结果绘制柱状图进行分析.
2实验结果
2.1莱茵衣藻表达载体的构建
将目的片段依据莱茵衣藻密码子优先性进行改造,两端分别连接保护碱基以及Nhe Ⅰ和PmaC Ⅰ酶切位点.在英韦创津生物公司合成长度为1 176碱基对(base pair, bp)的c-lpaat和c-gpd1基因(图2和图3),进行测序验证,结果表明合成的序列正确.通过双酶切分离目的片段,然后与衣藻转化载体pH124相连接(图1),转化入大肠杆菌中,涂平板,挑取单克隆,获得重组质粒pH124-c-lpaat和pH124-c-gpd1.PCR结果显示,所获得的重组质粒均含有目的基因,见图4,经过PCR筛选的重组质粒均获得测序确认.
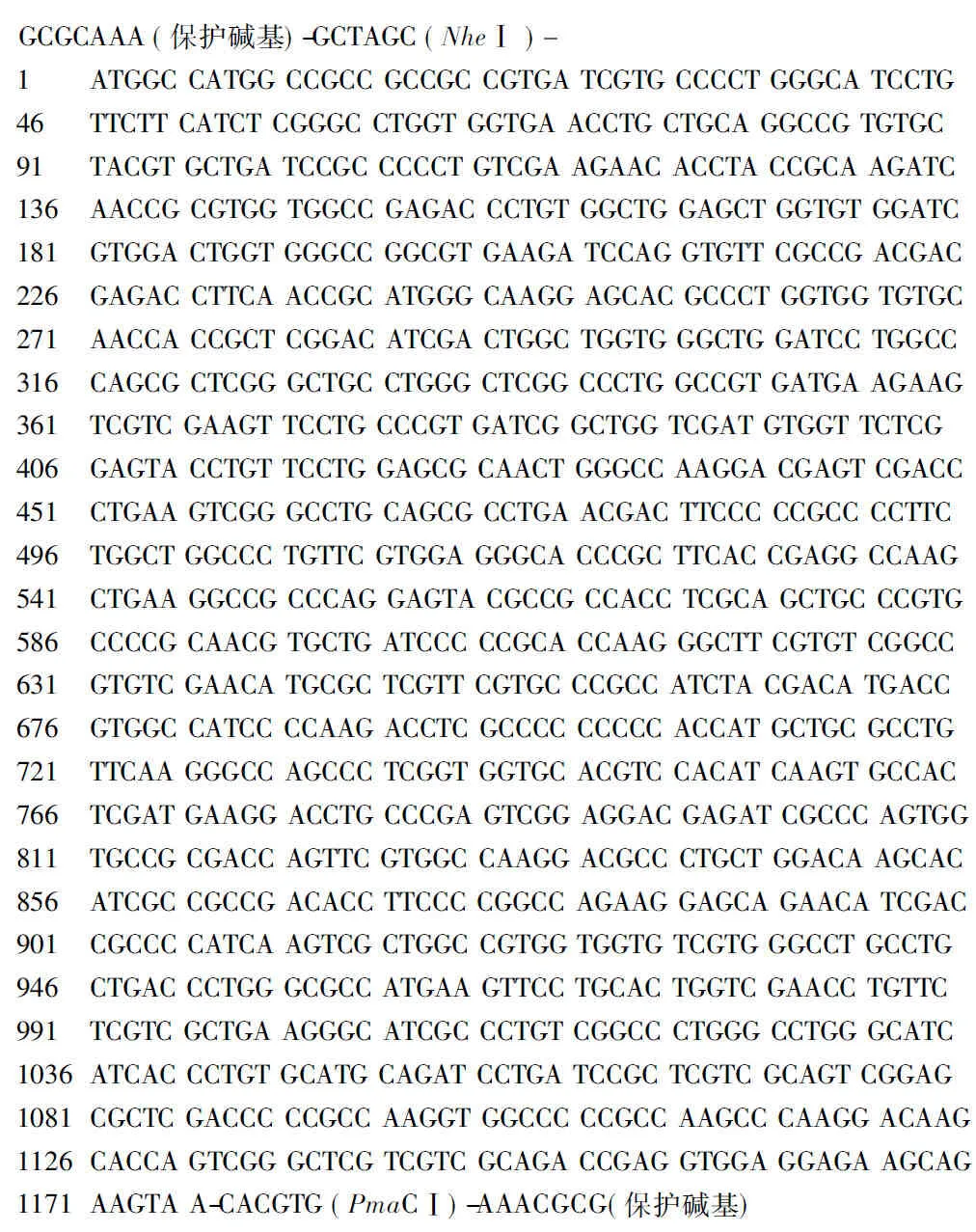
图2c-lpaat序列图
Fig.2Sequence ofc-lpaat
在设计目的片段时引入PmaC Ⅰ和Nhe Ⅰ限制性内切酶位点,重组质粒pH124-c-lpaat和pH124-c-gpd1经PmaCⅠ和NheⅠ限制性内切酶消化及电泳可获得1 176 bp的目的基因片段,琼脂糖凝胶电泳结果显示,已将c-lpaat和c-gpd1基因连接到载体pH124上,见图5.

图3c-gpd1序列图
Fig.3Sequence ofc-gpd1
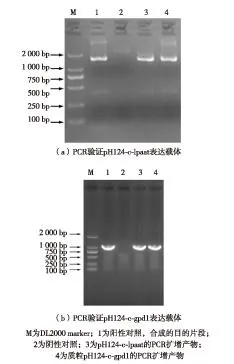
图4 重组质粒pH124-c-lpaat和pH24-c-gpd1的PCR验证Fig.4 PCR analysis of pH124-c-lpaat and pH124-c-gpd1

图5 重组质粒pH124-c-lpaat和pH124-c-gpd1的酶切验证Fig.5 Enzyme digestion analysis of pH124-c-lpaat and pH124-c-gpd1
2.2莱茵衣藻的遗传转化及筛选
采用珠磨法将质粒pH124-c-lpaat和pH124-c-gpd1分别转化到细胞壁缺陷的莱茵衣藻cc-849中,将转化后的藻细胞在含有10 μg/mL Zeomycin 的TAP( tris-acetate-phosphate)平板上进行筛选,获得绿色的单克隆藻株,如图4所示.多 次转化获得了大量的转化子,经PCR验证,获得成功转入c-lpaat基因和c-gpd1基因的转基因藻,其中,成功转入c-lpaat基因的转基因藻分别命名为Tranc-lpaat-J、K、L、M 、N、Q、1、2、3、4、5、14、15;成功转入c-gpd1基因的转基因藻分别命名为Tranc-gpd1-P、T、C、O、N、10、15、16;随后,对转化子的转录情况和油脂含量进行了分析.其中,pH124-c-lpaat载体的遗传转化效率E1=8.4×10-7;pH124-c-gpd1载体的遗传转化效率为E2=4.0×10-7.

图6 转基因衣藻的抗性平板筛选Fig.6 Screening of transformants by antibiotics resistance
转化子一直在含有10 μg/mL Zeomycin 的TAP平板上进行培养,提取基因组DNA,通过引物c-lpaat-F/c-lpaat-R以及c-gpd1-F/c-gpd1-R进行PCR检测,选取扩增出目的条带的转化子.PCR结果显示,可从获得的转化子中扩增出与目的基因大小一致的条带,如图7和图8,表明外源基因片段已经整合到莱茵衣藻cc-849的基因组中.多次转化获得了大量的转化子,经PCR验证成功转入c-lpaat基因的转化子有Tranc-lpaat-J、K、L、M 、N、Q、1、2、3、4、5、14、15,成功转入c-gpd1基因的转化子有Tranc-gpd1-P、T、C、O、N、10、15、16,随后对转化子的转录情况和油脂含量进行分析.

图7 c-lpaat转化子的PCR分析Fig.7 PCR analysis of the transformants contained c-lpaat

图8 c-gpd1转化子的PCR分析Fig.8 PCR analysis of transformants contained c-gpd1
2.3转基因藻中目的基因转录水平的分析
提取转基因藻的总RNA进行半定量反转录PCR(reverse transcription-PCR, RT-PCR)分析,琼脂糖凝胶电泳结果发现:各转化子均能扩增出与目的基因大小一致的条带,表明整合进莱茵衣藻基因组的c-lpaat和c-gpd1均能转录成mRNA,但是目的基因的表达量并不一致.为筛选出目的基因表达量最高的转化子,通过RT-PCR扩增出莱茵衣藻的β-actin基因作为内参,进行半定量RT-PCR的分析,如图9.
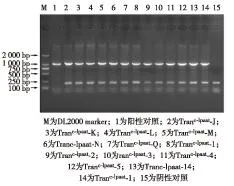
图9 转c-lpaat基因衣藻的RT-PCR分析Fig.9 RT-PCR analysis of the transgenic algae with c-lpaat
通过Photoshop软件对目的基因及β-actin基因的条带进行灰度比对,结果显示,Tranc-lpaat-15转化子的c-lpaat基因表达量最高,如图10.其中,转化子Tranc-lpaat-15的表达量比转化子Tranc-lpaat-Q增加了56.38%.

图10 Tranc-lpaat中c-lpaat基因相对表达量的分析Fig.10 Relative transcript levels of c-lpaat in Tranc-lpaat
利用气相色谱对转基因藻进行的油脂含量分析也发现:Tranc-lpaat-15的脂肪酸含量最高,达到120.06 μg/mg,与半定量RT-PCR分析的结果一致.
同理,通过半定量RT-PCR技术获得了Tranc-gpd1各转化子中c-gpd1的表达情况,结果显示,目的基因能在各转化子中得到有效转录,且表达量不一致,见图11.通过与内参基因β-acitn的比较来对目的基因的表达进行半定量分析,结果显示,Tranc-gpd1-T藻株中c-gpd1基因的表达量最高,其基因表达量比Tranc-gpd1-N转化子增加了75.95%,见图12.

图11 转c-gpd1基因衣藻的RT-PCR分析Fig.11 RT-PCR analysis of the transgenic algae with c-gpd1

图12 Tranc-gpd1中c-gpd1基因相对表达量的分析Fig.12 Relative transcript levels of c-gpd1 in transgenic algae
在后续的转基因藻油脂含量分析中发现:转基因藻Tranc-gpd1-T的脂肪酸含量最高,达到126.34 μg/mg,与半定量RT-PCR分析的结果一致.以上结果表明,通过密码子优化的c-lpaat和c-gpd1基因均能在莱茵衣藻中进行转录,目的基因在转基因藻中的表达量越大,其油脂含量越高.
3讨论
随着陆地资源的减少,水生资源的开发逐渐被人们关注,其中,微藻能源的开发是研究的重点.微藻是多样化的有机体,具有许多独特的代谢特性,如高光合传递速率、快速的生物量生产速率、适应多种环境和可以生产多种类的生物柴油等[11].基于微藻制备生物燃料的战略意义,许多国家都已投入了大量的人力和物力,目前已经取得了阶段性的成果.
三酰甘油(triacylglycerol,TAG)合成路径中有一系列的催化酶参与,对其功能的研究主要集中在拟南芥和油菜等高等植物和大肠杆菌、酵母中[12].酵母中的gpd1基因与油菜中的g3pdh基因的功能相同,研究发现,酵母的gpd1基因转入油菜种子中后,油菜种子中的含油量增加了40%[13].在哺乳动物中,G3PDH的酶活性与脂肪细胞呈正相关[14].由此推测,TAG路径中g3pdh基因起到关键作用.Lv等[15]发现,在胁迫条件下,莱茵衣藻脂肪酸含量升高的同时,lpaat基因的表达量增加;抑制lpaat基因后,中性脂含量降低了20%.因此, lpaat在TAG的路径中同样起到关键作用.将亲缘关系较近,且酶活性较高的酵母gpd1基因和油菜lpaat基因转入莱茵衣藻中,通过增加三酰甘油的合成效率,最终提高油脂在藻细胞中的含量.本研究证明了高等植物的外源基因在微藻中表达的可行性,且成功筛选出产油量较高的转化子.
选取两个外源基因片段,经过优化后与莱茵衣藻表达载体pH124连接,分别转入莱茵衣藻野生株中,成功获得了一系列转化子,且能在转录水平检测到目的基因的表达,说明c-lpaat与c-gpd1基因均在转基因衣藻中得到了有效转录.但是,不同藻株的表达量不一致,表明这两个基因在不同的转化株中表达效率不同,可能与外源基因在基因组中插入的位点有关,这与文献[16-17]报道的结果相似.通过多次的遗传转化,获得了近百株转化子,快速筛选出油脂含量高的转化子对本研究具有重要意义.本研究利用RT-PCR测定了这一系列转化子的基因表达量,比较之后发现Tranc-lpaat-15的基因表达量最高,较对照组提高了56.38%,其对应的脂肪酸含量提高幅度也是最大的;同样,Tranc-gpd1-T的基因表达量最高,较对照组提高了75.95%,其对应的脂肪酸含量提高幅度也是最大的.以上结果表明, c-lpaat与c-gpd1基因导入衣藻基因组后,较高的转录活性能获得更高的油脂含量, c-lpaat与c-gpd1基因促进了转化子中三酰甘油的合成,该研究通过RT-PCR手段,为快速筛选出目的基因转录活性高的转化子,并获得油脂含量得到提高的转基因藻提供了有效的技术手段,后续进行的关于基因表达量和脂肪酸含量变化关系的研究也获得了可喜的结果(将另撰文发表),显示了两者存在明显的相关性.
利用微藻作为原料生产生物柴油,关键是如何有效合成积累微藻中的脂肪酸.利用外源基因调控微藻产油和高等植物产油是一种非常普遍的方法,该方法比较成熟,目前已经成功在绿藻和硅藻等40多株藻中实施[18].随着微藻代谢途径的深入研究,利用基因工程调控相关代谢酶和转录调控因子的活性,成为了研究热点[19].
结语
利用新技术生产再生能源已成为当务之急,微藻产油因其众多优势而成为研究热点.利用基因工程改造转基因藻,可以有效调控其产油量.本研究选取两个外源基因,成功构建了表达载体,并将其转入莱茵衣藻cc-849中,获得了一系列转化株,采用半定量RT-PCR的技术对转化子进行快速的检测,可以快速筛选出潜在的高油脂含量转基因藻.

引文:王潮岗,李逸,胡章立. 转lpaat和gpd1基因工程藻的快速筛选[J]. 深圳大学学报理工版,2016,33(1):33-40.
参考文献/ References:
[1] Manikandan G,Kumar P S,Prakalathan R.Comparison of biodiesel production from macro and micro algae[J].International Journal of ChemTech Research,2014,6(9):4143-4147.
[2] Pflaster E L,Schwabe M J,Becker J,et al.A high-throughput fatty acid profiling screen reveals novel variations in fatty acid biosynthesis in Chlamydomonas reinhardtii and related algae[J].Eukaryotic Cell,2014,13(11):1431-1438.
[3] 王潮岗,黄惠珠,孙海珊,等.一种莱茵衣藻启动子功能检测系统的构建[J].深圳大学学报理工版,2012,29(6):534-540.
Wang Chaogang,Huang Huizhu,Sun Haishan,et al.Construction of Chlamydomonas reinhardtii system for analyzing the function of algal promoter[J]. Journal of Shenzhen University Science and Engineering,2012,29(6):534-540.(in Chinese)
[4] Joseph M,Di X.Metabolic and gene expression changes triggered by nitrogen deprivation in the photoautotrophically grown microalgae Chlamydomonas reinhardtii and Coccomyxa sp.C-169[J]. Phytochemistry,2012,75:50-59.
[5] Deng Xiaodong, Cai Jiajia, Li Yajun, et al.Expression and knockdown of the PEPC1 gene affect carbon flux in the biosynthesis of triacylglycerols by the green alga Chlamydomonas reinhardtii[J].Biotechnology Letters,2014,36(11):2199-2208.
[6] 田琪琳,施定基,贾晓会,等.“细菌型”pepc2基因反向表达载体构建及在莱茵衣藻中的表达[J].上海海洋大学学报,2013,22(5):665-671.
Tian Qilin,Shi Dingji,Jia Xiaohui,et al.The expression of recombinant vector of reverse “bacterial-type” pepc2 gene in Chlamydomonas reinhardtii[J].Journal of Shanghai Ocean University,2013,22(5):665-671.(in Chinese)
[7] Marchive C,Nikovics K,To A,et al.Transcriptional regulation of fatty acid production in higher plants: molecular bases and biotechnological outcomes[J].European Journal of Lipid Science and Technology,2014,116(10):1332-1343.
[8] Wu Shan,Xu Xiangrong, Sun Kaifeng, et al.Potential of microalgae as bioenergy and functional foods[J].International Journal of Environment and Bioenergy,2014,9(1):29-43.
[9] Fan Jianhua,Cui Yanbin,Zhou Yang,et al.The effect of nutrition pattern alteration on Chlorella pyrenoidosa growth, lipid biosynthesis-related gene transcription [J]. Bioresource Technology, 2014, 164: 214-220.
[10] Scranton M A, Ostrand J T, Fields F J, et al. Chlamydomonas as a model for biofuels and bio-products production[J].The Plant Journal,2015,82(3):523-531.
[11] Li-Beisson Y,Beisson F,Riekhof W.Metabolism of acyl-lipids in Chlamydomonas reinhardtii[J].The Plant Journal,2015,82(3):504-522.
[12] Davey M P, Horst I, Duong G H, et al. Triacylglyceride production and autophagous responses in Chlamydomonas reinhardtii depend on resource allocation and carbon source[J].Eukaryotic Cell,2014,13(3):392-400.
[13] Vigeolas H,Waldeck P,Zank T,et al.Increasing seed oil content in oil-seed rape (Brassica napus L.) by over-expression of a yeast glycerol-3-phosphate dehydrogenase under the control of a seed-specific promoter[J]. Plant Biotechnology Journal, 2007, 5(3):431-441.
[14] Yi Bao,Wang Jigui,Wang Shuang,et al.Overexpression of Banna mini-pig inbred line fatty acid binding protein 3 promotes adipogenesis in 3T3-L1 preadipocytes[J].Cell Biology International,2014,38(8):918-923.
[15] Lv(Lyu) Hexin,Qu Ge,Qi Xizhen,et al.Transcriptome analysis of Chlamydomonas reinhardtii during the process of lipid accumulation[J].Genomics,2013,101(4):229-237.
[16] Hu Zhangli,Fan Zhun,Zhao Zhonglin,et al. Stable expression of antibiotic-resistant gene ble from Streptoalloteichus hindustanus in the mitochondria of Chlamydomonas reinhardtii[J].PLOS One,2012, 7(4): e35542.
[17] 胡章立,李建成,程雪薇. 莱茵衣藻线粒体遗传系统及其外源基因表达[J]. 深圳大学学报理工版,2013,30(6) : 603-610.
Hu Zhangli,Li Jiancheng,Cheng Xuewei. The mitochondrial genetic system and exogenous gene expression of Chlamydomonas reinhardtii[J]. Journal of Shenzhen University Science and Engineering,2013,30(6) : 603-610.(in Chinese)
[18] Moody J W,McGinty C M,Quinn J C.Global evaluation of biofuel potential from microalgae[J].Proceedings of the National Academy of Sciences,2014,111(23):8691-8696.
[19] 刘飞飞,李秀波,方仙桃,等.三角褐指藻产油突变株的筛选[J].水生生物学报,2013,37(4):799-802.
Liu Feifei,Li Xiubo,Fang Xiantao,et al.Selection of oil-content mutants in Phaeodactylum tricornutum[J].Acta Hydrobiologica Sinca,2013,37(4):799-802.(in Chinese)
【中文责编:晨兮;英文责编:艾琳】
Rapid screening of the transgenic algae
haboringlpaatandgpd1
Wang Chaogang, Li Yi, and Hu Zhangli†
College of Life and Marine Science, Shenzhen Key Laboratory of Marine Bioresources and Ecology, Shenzhen University,
Shenzhen 518060, Guangdong Province, P.R.China
Abstract:Lpaat from Brassica napus and gpd1 from Saccharomyces cerevisiae were optimized according to the codon of Chlamydomonas reinhardtii to obtain c-lpaat and c-gpd1. The modified DNA fragments were amplified and inserted into plasmid pH124 to construct Chlamydomonas expression vectors. The expression vectors were then introduced into Chlamydomoas with the glass-bead-method and the transgenic algae containing c-lpaat and c-gpd1 was selected after screening with zeomycin resistance and polymerase chain reaction (PCR) analysis. Finally, the transgenic algae in which c-lpaat and c-gpd1 were expressed effectively was screened by semi-quantitative reverse transcription-PCR (RT-PCR). The transcription level of c-lpaat and c-gpd1 in transgenic algae was increased by 56.38% and 75.95%, respectively, resulting in correspondingly increased lipid content. In conclusion, the exogenous genes from the plants could be effectively expressed in Chlamydomonas reinhardtii and the transgenic algae with high lipid could be screened quickly by semi-quantitative RT-PCR. These results lay the foundation of quickly screening for transgenic algae with high lipid content.
Key words:genetic engineering; transgenic algae; Chlamydomonas reinhardtii; lpaat; gpd1; triacylglycerol synthesis
作者简介:王潮岗(1978—),男,深圳大学讲师、博士. 研究方向:微藻分子生物学.E-mail:charlesw@szu.edu.cn
基金项目:国家自然科学基金资助项目(31470389,31470431)
中图分类号:Q 786
文献标志码:A
doi:10.3724/SP.J.1249.2016.01033

