杜洛克猪HMGA1基因多态位点与生长、饲料利用性状的关联分析
蒲 蕾,刘 欣,岳静伟,张金山,王立刚,颜 华,侯欣华,高红梅,张 恬,张跃博,武群清,3,张龙超*,王立贤*
(1.中国农业科学院北京畜牧兽医研究所,北京100193;2.天津农学院动物科学与动物医学学院,天津300384;3.湖南农业大学动物科学技术学院,长沙410128)
杜洛克猪HMGA1基因多态位点与生长、饲料利用性状的关联分析
蒲 蕾1,2,刘 欣1,岳静伟1,张金山1,王立刚1,颜 华1,侯欣华1,高红梅1,张 恬1,张跃博1,武群清1,3,张龙超1*,王立贤1*
(1.中国农业科学院北京畜牧兽医研究所,北京100193;2.天津农学院动物科学与动物医学学院,天津300384;3.湖南农业大学动物科学技术学院,长沙410128)
摘 要:试验对HMGA1基因的两个SNPs位点进行分析,研究了HMGA1基因核苷酸多态性与生长、饲料利用性状的关联性。利用大白猪和民猪重测序结果比较发现了HMGA1基因的两个SNPs位点(g.-543T>C和g.1356C>T),利用Sequenom质谱测序平台对杜洛克猪g.-543T>C和g.1356C>T位点进行基因分型,并分析该位点多态性与生长、饲料利用性状的关联性。结果发现,杜洛克猪群体g.-543T>C位点,CT与CC基因型相比,90日龄体重升高了2.15kg(P<0.05),30kg体重日龄降低了3.21d(P<0.05),断奶重升高,剩余采食量降低,100kg体重日龄减少,30~100kg平均日增重、40~90kg平均日采食量、40~90kg平均日增重均降低。杜洛克猪群体g.1356C>T位点,CC与TT基因型相比,90日龄体重升高了0.37kg(P<0.05);平均日采食量降低0.13kg/d(P<0.05),100kg背膘厚降低0.43mm(P<0.05),剩余采食量降低了70.42g(P<0.05),30kg体重日龄降低了0.56d(P<0.05);40~90kg平均日采食量降低了0.17kg(P<0.05),断奶重升高,100kg体重日龄增大,30~100、40~90kg平均日增重均降低;CT与TT基因型结果与上结果类似。结果初步表明HMGA1基因两个SNPs位点的突变有利于杜洛克猪的生长、饲料利用效率的提高。
关键词:HMGA1基因;杜洛克猪;SNP;剩余采食量;饲料利用效率
饲料成本占养猪成本的60%以上,所以对饲料利用效率性状的改良有利于降低养猪成本[1]。剩余采食量(residual feed intake,RFI)性状是目前被关注的饲料利用效率指标之一[2]。与饲料转化率(feed conversion ratio,FCR)不同,剩余采食量是用来描述动物实际采食量与用于维持和增重所需的预测采食量的差异,是用来评价动物遗传背景决定的代谢差异[2]。
HMGA1属于高迁移率蛋白家族成员(high mobility group protein,HMG)。研究证实,HMGA1与癌症细胞的增殖分化有关,过表达HMGA1基因可以促进肿瘤细胞的转移[3-6];HMGA1基因在骨骼肌和脂肪组织中调控葡萄糖的转运及胰岛素信号通路[7-8];HMGA1超表达可导致转基因小鼠体重降低,脂质变薄[9]。另有研究报道称,HMGA1和黑皮质素受体4(melanocortin receptor 4,MC4R)基因的SNP互作与猪的生长、瘦肉量和脂肪含量均有一定关系[10-11]。机体三大物质(糖类、脂类和蛋白质)代谢的能力与猪的饲料利用效率有着密切关系[2]。而HMGA1基因在调节葡萄糖代谢和脂质代谢方面均有重要作用。因此,HMGA1可能与猪的饲料利用效率性状有关。
本试验对大白猪和民猪群体的重测序数据分析发现了HMGA1基因的两个单核苷酸多态性(single nucleotide polymorphism,SNP)位点,目前鲜有研究报道。采用质谱分型技术,对杜洛克猪群体进行HMGA1基因的两个SNPs位点的基因分型,并对该多态位点与生长、饲料利用性状进行关联分析,为饲料利用效率相关性状影响的功能研究及HMGA1的这两个位点作为标记辅助选择位点提供依据。
1 材料与方法
1.1主要试剂
琼脂糖(Agarose)购自TaKaRa公司;DEPC、蛋白酶K、DNA MarkerⅠ、DNA MarkerⅡ均购自天根生化科技(北京)有限公司;GoldView购自北京赛百盛基因技术有限公司;Tris饱和酚、三氯甲烷、无水乙醇(Ethanol)均购自北京化工厂。
1.2数据采集
所有的杜洛克猪进入有FIRE美国OSBORN自动喂料系统的圈舍中,测定获取日采食量及体重情况。所有的原始数据从FIRE软件收集至Excel表中,FCR性状为平均日采食量(average daily feed intake,ADFI)与平均日增重(average daily gain,ADG)的比值。RFI性状的计算参考美国爱荷华州立大学Onteru等[12]公式:
RFI=ADFI-[b1×(OnBW-30)+b2×(Off-BW-100)+b3×MetamidBW+b4×ADGA+b5 ×OffBFA)
式中,ADFI,测定期间的日采食量;Metamid-BW(meta middle body weight),猪的中间体重时需要的代谢能,MetamidBW=[(OnBW+OffBW)/2]0.75;ADGA,90~180d内的平均日增重;OffBFA(adjust off test back fat),测试结束时10~11肋背膘厚度调整到100kg体重时的值,即100kg体重背膘厚(100kg backfat,100kg BF);30kg体重日龄、100kg体重日龄和30~100kg平均日增重的校正公式来源于CCSI[13]。40~90kg平均日采食量和平均日增重均为检测真实值。
1.3 基因组DNA提取
采集所有杜洛克猪的耳样,将样品放入75%乙醇中,-20℃保存,以备后续基因组DNA的提取。基因组DNA采用酚-氯仿法提取,具体提取步骤见试剂盒说明。
1.4HMGA1基因的g.-543T>C和g.1356C>T基因型的判定
通过对大白猪和民猪的基因组DNA重测序所得结果进行比较分析,发现HMGA1基因的两个SNPs位点(g.-543T>C和g.1356T>C)。通过Ensemble软件(http://asia.ensembl.org/index.html)比对分析突变位置,确定SNP位点。
1.5Sequenom质谱分型
利用Sequenom质谱检测平台对试验样品进行HMGA1基因g.-543T>C和g.1356T>C位点的基因分型。根据GenBank中猪HMGA1基因RNA序列(登录号:NM_001185154)和DNA序列(登录号:NC_010449.4),利用AssayDesigner 3.1软件进行引物和探针设计,引物序列见表1。单管扩增引物稀释至100μmol/L,单管延伸引物稀释至500μmol/L。Sequenom MassArray系统基因分型步骤为:①PCR扩增反应;②SAP反应;③延伸反应;④树脂纯化;⑤上样,质谱反应;⑥Mass Array分析,并输出报告[14]。

表1 HMGA1基因SNPs位点质谱探针引物Table 1 Primers and probes for genotyping assay for SNPs ofHMGA1gene
1.6统计分析
对380头杜洛克猪HMGA1基因SNP位点与生长、饲料利用性状进行关联分析。使用SAS 9.2[15]数据处理软件中的广义混合线性模型(general linear mixed model,GLMM),以最小二乘法展开基因型与生长、饲料利用性状显著性检验,获得杜洛克猪群体中显著关联位点。使用的模型为:
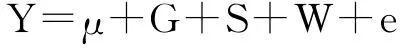
式中,Y,测得性状;μ,总体均数;G,基因型效应;S,性别固定效应;W,初测体重协变量;e,随机误差。
统计基因型,计算各群体中该位点的基因频率和基因型频率,并进行Hardy-Weinberg平衡检测。计算多态信息含量(PIC)、群体杂合度(He)、有效等位基因数(Ne),并做卡方(χ2)检验。
2 结果与分析
2.1生长、饲料利用性状表型分析
本试验中杜洛克猪群体各生长、饲料利用性状数据表型统计结果见表2。由表2可知,杜洛克猪群体猪的初生重为1.79kg;断奶重为7.78kg。90日龄体重为29.49kg;平均日采食量为1.74kg/d;平均日增重为0.66kg/d;100kg背膘厚为7.41mm;剩余采食量为-0.42g/d;饲料转化率为2.70%;30kg体重日龄为90.80d;100kg体重日龄为196.47d;300~100kg平均日增重为0.67kg/d;40~90kg平均日采食量为1.93kg/d;40~90kg平均日增重为0.78kg/d。
2.2HMGA1基因的多态性基因型频率分析
HMGA1基因5′-flank区域内发现了1个SNP位点(g.-543T>C);内含子区域发现1个SNP位点(g.1356C>T)。群体基因型频率和等位基因频率见表3。由表3可知,在g.-543T>C位点,C和T等位基因频率分别为90.79%和9.21%;在c.1356C>T位点,C和T等位基因频率分别为21.84%和78.16%。g.-543T>C和g.1356 C>T多态位点的多态信息含量(PIC)、杂合度(He)、有效等位基因数(Ne)及卡方(χ2)检验的分析结果中,杜洛克猪群体中该位点均达到Hardy-Weinberg平衡状态(P>0.05);均表现出低度到中度多态性(中度0.25<PIC<0.5;低度PIC<0.25)。根据等位基因频率和杂合度分析,发现两个位点均为低等杂合度SNP位点。
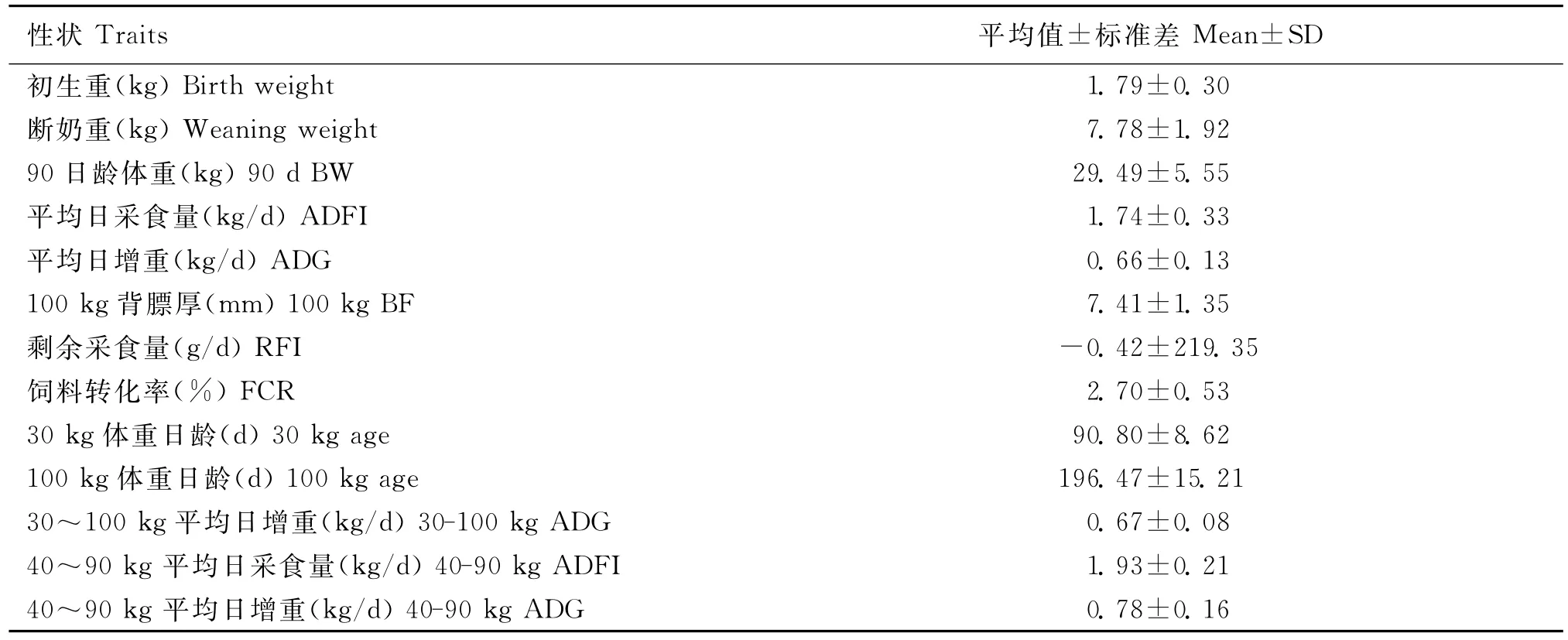
表2 饲料利用效率相关性状的描述性统计Table 2 Statistical description of data set for feed efficiency related traits
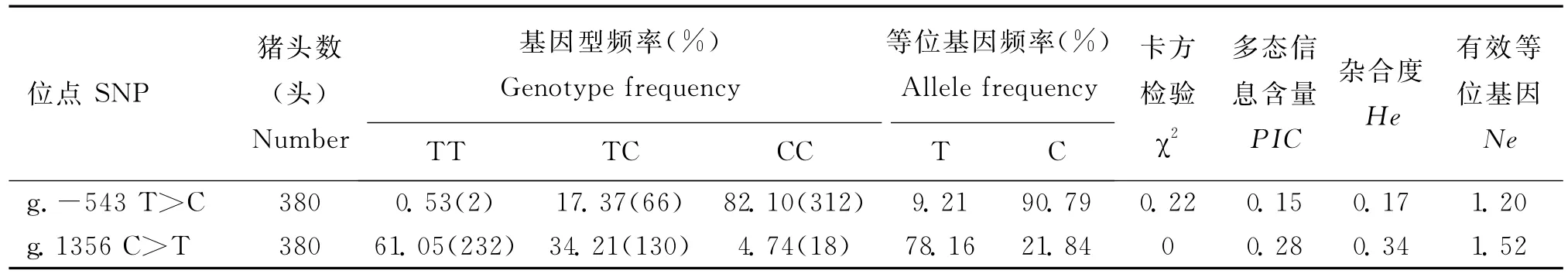
表3 杜洛克猪HMGA1基因的SNPs多态信息Table 3 The polymorphism information ofHMGA1gene SNPs in Durocs pigs
2.3SNPs与生长、饲料利用性状的关联分析
将HMGA1基因的两个SNPs位点基因型与杜洛克猪生长、饲料利用性状进行关联分析,结果见表4、5。由表4可知,杜洛克猪g.-543T>C位点,TT与CC基因型个体相比,90日龄体重降低了2.63kg(P<0.05),30kg体重日龄升高了4.11d(P<0.05),100kg体重日龄升高了16.21d(P<0.05),40~90kg平均日采食量降低了0.25kg(P<0.05),断奶重升高,剩余采食量、30~100kg平均日增重、40~90kg平均日增重均降低;CT与CC基因型相比,90日龄体重升高了2.15kg(P<0.05),30kg体重日龄降低了3.21d(P<0.05),断奶重升高,剩余采食量降低,100kg体重日龄减少,30~100kg平均日增重、40~90kg平均日采食量、40~90kg平均日增重均降低。
由表5可知,杜洛克猪g.1356C>T位点,CC与TT基因型相比,90日龄体重升高了0.37kg(P<0.05);平均日采食量降低0.13kg/d(P<0.05),100kg背膘厚降低0.43mm(P<0.05),剩余采食量降低70.42g(P<0.05),30kg体重日龄降低了0.56d(P<0.05);40~90kg平均日采食量降低了0.17kg(P<0.05),断奶重升高,100kg体重日龄增大,30~100、40~90kg平均日增重均降低;CT与TT基因型相比,90日龄体重升高了0.99kg(P<0.05);30kg体重日龄降低了1.54d(P<0.05),断奶重升高,100kg体重日龄增大,30~100、40~90kg平均日增重均降低。

表4 HMGA1基因g.-543T>C位点基因型与生长、饲料利用性状的关联分析Table 4 Association ofHMGA1gene g.-543T>C genotype with different feed efficiency related traits

表5 HMGA1基因g.1356C>T位点基因型与生长及饲料利用效率性状的关联分析Table 5 Association ofHMGA1gene g.1356C>T genotype with different feed efficiency related traits
3 讨 论
研究发现,HMGA1蛋白可以从细胞核转移至线粒体,与线粒体DNA的D-loop环结合,影响线粒体基因的转录和功能维持[16],且HMGA1的表达水平也影响了线粒体的功能和线粒体DNA的损伤修复效率[17]。机体能量消耗是通过线粒体的能量转化完成的,有研究称饲料利用效率高的动物体内线粒体的电子偶联比其他动物好,能更好地利用能量[18]。HMGA1是视黄醇结合蛋白4(retinol-binding protein 4,RBP4)基因的重要调节子,cAMPHMGA1-RBP4通路在葡萄糖的体内内稳态中发挥重要作用[11]。另外,HMGA1在胰岛β细胞的功能维持和胰岛素的生产方面发挥重要作用[19]。HMGA1基因在白色脂肪和棕色脂肪的形成中发挥重要作用,它可以抑制脂肪细胞分化,降低脂肪量[9]。HMGA1基因的多态性与肌肉组织的脂肪酸的成分比例显著关联[11]。猪的剩余采食量性状与猪的背部脂肪含量和瘦肉率具有很强的相关性[20]。所以HMGA1可能在调控饲料利用效率方面发挥一定作用。
通过对大白猪和民猪重测序结果进行比较分析,发现了HMGA1基因的两个SNPs位点:g.-543T>C和g.1356C>T,且HMGA1基因的这两个SNPs位点与猪生长、饲料利用性状的相关性鲜有报道。由于g.-543T>C位点的TT基因型个体只有两个,所以统计的代表性不强,故此部分结果并不做深入分析。本研究结果分析发现,杜洛克猪HMGA1基因g.-543T>C位点的CT与CC基因型相比90日龄体重更大,30kg体重日龄、100kg体重日龄和40~90kg平均日采食量更小,但该位点3种基因型与40~90kg平均日增重均无显著相关性。即对HMGA1基因g.-543T>C位点CT基因型进行选择可能对90日龄体重、30kg体重日龄、100kg体重日龄和40~90kg平均日采食量产生积极的影响,并不显著影响40~90kg平均日增重。杜洛克猪HMGA1基因g.1356C>T位点CT、CC基因型与TT基因型相比,90日龄体重更大,平均日采食量更小,30kg体重日龄更小,并不显著影响平均日增重的大小。由于g.1356 C>T位点CC基因型个体较CT和TT基因型个体表现出更薄的背膘厚、更低的剩余采食量和40~90kg平均日采食量,并不影响40~90kg平均日增重的大小。即这两个位点的突变可以使杜洛克猪在不影响平均日增重的同时降低同期间内的平均日采食量,可以使90日龄体重更重,30kg体重日龄和100kg体重日龄更小,尤其g.1356C>T位点还可以使杜洛克猪有更薄的背膘厚和更低的剩余采食量。
另有研究发现,HMGA1基因的其他SNP位点与猪的生长、饲料利用性状有关。研究报道,HMGA1基因的SNP位点与MC4R基因的SNP位点有一定关联互作关系,此互作关系与猪的生长、瘦肉量和脂肪含量有一定相关性[10]。HMGA1基因的c187-170C>T位点的多态性在低剩余采食量选择系和对照组在大白猪群体中突变频率差异极显著,但并没有发现此位点与饲料利用及其他肉质性状有相关性[21]。HMGA1基因的c.576C>T多态性位点与背膘厚呈现显著相关性,与平均日增重和瘦肉率并无显著相关性[10]。将HMGA1基因c.576C>T位点与MC4R基因(Asp298Asn)的突变位点进行综合关联分析,发现c.576C>T位点与大白猪的平均日增重显著相关[10]。本研究中,HMGA1基因g.1356C>T位点与背膘性状有一定相关性,但与上研究不同的是HMGA1基因g.-543T>C和g.1356C>T位点与杜洛克猪群体各个阶段的平均日增重均无显著相关性。
4 小 结
HMGA1基因g.-543T>C和g.1356C>T位点突变可以使杜洛克猪在不影响平均日增重的同时降低同期间内的平均日采食量,可以使90日龄体重更重,30kg体重日龄和100kg体重日龄更小。尤其g.1356C>T位点还可以使杜洛克猪有更薄的背膘厚和更低的剩余采食量。HMGA1基因的这两个SNP位点可作为分子标记,利用基因辅助选择的方法选择有利的基因型留种,提高育种改良效率。
参考文献:
[1] 英国养猪委员会.陶莉译.饲料成本对英国养猪生产行业的影响[J].国外畜牧学-猪与禽,2011,31(2):3-11.
[2] 蒲 蕾,岳慧洁,张龙超,等.猪饲料利用效率的研究进展[J].中国农业大学学报(自然科学版),2016,21(4):77-85.
[3] Huso T H,Resar L M.The high mobility group A1molecular switch:Turning on cancer-can we turn it off[J].ExpertOpinTherTargets,2014,18(5):541-553.
[4] Di Cello F,Hillion J,Kowalski J.Cyclooxygenase inhibitors block uterine tumorigenesis in HMGA1a transgenic mice and human xenografts[J].MolCancer Ther,2008,7(7):2090.
[5] Zhong J,Liu C,Chen Y J,et al.The association between S100A13and HMGA1in the modulation of thyroid cancer proliferation and invasion[J].JTransl Med,2016,23,14(1):80.
[6] Zhou W B,Zhong C N,Luo X P,et al.miR-625suppresses cell proliferation and migration by targeting HMGA1in breast cancer[J].BiochemBiophysRes Commun,2016,470(4):838-844.
[7] Chiefari E,Paonessa F,Iiritano S,et al.The cAMPHMGA1-RBP4system:A novel biochemical pathway for modulating glucose homeostasis[J].BMCBiol,2009,7:24.
[8] Di Marcantonio D,Galli D,Carubbi C,et al.PKCεas a novel promoter of skeletal muscle differentiation and regeneration[J].ExpCellRes,2015,339(1):10-19.
[9] Arce-Cerezo A,García M,Rodríguez-Nuevo A,et al.HMGA1overexpression in adipose tissue impairs adipogenesis and prevents diet-induced obesity and insulin resistance[J].SciRep,2015,5:14487.
[10] Kim K S,Lee J J,Shin H Y,et al.Association of melanocortin 4receptor(MC4R)and high mobility group AT-hook 1(HMGA1)polymorphisms with pig growth and fat deposition traits[J].AnimGenet,2006,37:419-421.
[11] Hong J,Kim D,Cho K,et al.Effects of genetic variants for the swineFABP3,HMGA1,MC4R,IGF2,andFABP4genes on fatty acid composition[J].Meat Sci,2015,110:46-51.
[12] Onteru S K,Gorbach D M,Young J M,et al.Whole genome association studies of residual feed intake and related traits in the pig[J].PLoSOne,2013,8(6):1-13.
[13] Canadian Center for Swine Improvement(CCSI).Canadian swine improvement program guidelines[Z],2010.
[14] 梁 晶,王红卫,程利安,等.北京黑猪PPARD基因G32E多态位点与脂肪沉积相关性状的关联分析[J].中国畜牧兽医,2015,42(7):1793-1799.
[15] Statistical Analysis Systems.SAS/STAT User’s Guide(V9.2).Statistical Analysis Systems Institute,Inc.2008.
[16] Dement G A,Maloney S C,Reeves R.Nuclear HMGA1 nonhistone chromatin proteins directly influence mitochondrial transcription,maintenance,and function[J].ExpCellRes,2007,313(1):77-87.
[17] Mao L,Wertzler K J,Maloney S C,et al.HMGA1 levels influence mitochondrial function and mitochondrial DNA repair efficiency[J].MolCellBiol,2009,29(20):5426-5440.
[18] Kolath W H,Kerley M S,Golden J W,et al.The relationship between mitochondrial function and residual feed intake in Angus steers[J].JournalofAnimal Science,2006,84(4):861-865.
[19] Arcidiacono B,Iiritano S,Chiefari E,et al.Cooperation between HMGA1,PDX-1,and MafA is essential for glucose-induced insulin transcription in pancreatic beta cells[J].FrontEndocrinol(Lausanne),2015,5:237.
[20] Richardson E C,Herd R M,Oddy V H,et al.Body composition and implications for heat production of Angus steer progeny of parents selected for and against residual feed intake[J].AustralianJournal ExplorationAgriculture,2001,41(7):1065-1072.
[21] Fan B,Lkhagvadorj S,Cai W,et al.Identification of genetic markers associated with residual feed intake and meat quality traits in the pig[J].MeatSci,2010,84(4):645-650.
(责任编辑 晋大鹏)
中图分类号:S813.3
文献标识码:A
文章编号:1671-7236(2016)12-3268-07
doi:10.16431/j.cnki.1671-7236.2016.12.028
收稿日期:2016-05-04
基金项目:中国农业科学院科技创新工程(ASTIP-IAS02);国家“十二五”科技支撑计划项目(2015BAD03B02-2);国家现代农业产业技术体系(CARS-36)
作者简介:蒲 蕾(1987-),女,陕西西安人,博士,研究方向:猪遗传育种,E-mail:pulei87@126.com
通信作者:*张龙超,男,河北定州人,副研究员,研究方向:猪分子遗传育种,E-mail:zhlchias@163.com王立贤,男,山西朔州人,研究员,研究方向:猪遗传育种,E-mail:iaswlx@263.net
Study onHMGA1Gene Polymorphism Site and Its Association with Feed Efficiency Related Traits in Duroc Pigs
PU Lei1,2,LIU Xin1,YUE Jing-wei1,ZHANG Jin-shan1,WANG Li-gang1,YAN Hua1,HOU Xin-hua1,GAO Hong-mei1,ZHANG Tian1,ZHANG Yue-bo1,WU Qun-qing1,3,ZHANG Long-chao1*,WANG Li-xian1*
(1.InstituteofAnimalSciences,ChineseAcademyofAgriculturalSciences,Beijing100193,China;2.CollegeofAnimalScienceandVeterinaryMedicine,TianjinAgriculturalUniversity,Tianjin300384,China;3.CollegeofAnimalScienceTechnology,HunanAgriculturalUniversity,Changsha410128,China)
Abstract:The objective of this study was to analyze two SNPs ofHMGA1gene,and investigate its association with feed efficiency related traits in Duroc populations.HMGA1gene SNPs(g.-543T>C and g.1356C>T)in Large White pig and Min pig were detected by re-sequencing technology,SNP genotyping of g.-543T>C and g.1356C>T sites in Duroc populations were performed using Sequenom mass spectrometry,and the association ofHMGA1gene SNPs with growth and feed efficiency related traits in Duroc populations were analyzed.The results in-dicated that the individuals of CT genotype in g.-543T>C increased 2.15kg on 90dbody weight(P<0.05),decreased 3.21dat 30kg weight age(P<0.05),weaning weight was increased and 100kg age were decreased,residual feed intake,30to 100kg average daily gain(ADG),40to 90kg average daily feed intake(ADFI)and 40to 90kg ADG were decreased,comparing with CC individuals.Moreover the individuals of CC genotype in g.1356C>T had significantly increased 90dbody weight(0.37kg,P<0.05),ADFI(less 0.13kg,P<0.05),back fat(BF,less 0.43mm,P<0.05),RFI(less 70.40g,P<0.05),30kg age(less 0.56d,P<0.05)and 40to 90kg ADFI(less 0.17kg,P<0.05)were significantly decreased;Weaning weight and 100kg age were increased;30to 100kg ADG,40to 90kg ADG were decreased than TT genotype individuals.And the CT genotype individual in g.1356C>T had the similar results with CC genotype individuals that compared with TT genotype individuals.All in all,the two SNPs ofHMGA1gene would be helpful in feed efficiency related traits in Duroc population.
Key words:HMGA1gene;Duroc pig;SNP;residual feed intake;feed utilization efficiency

