猪乙脑病毒BHK-21细胞传代株BSF.ZZ-1-p60和BSF.ZZ-3-p60的全基因组序列分析
滕 蔓,胡 博,冯丽丽,樊剑鸣,郅玉宝,李秀杰,邓瑞广,罗 俊*
(1.河南省农业科学院 动物免疫学重点实验室/河南省动物免疫学重点实验室/农业部动物免疫学重点实验室,河南 郑州 450002; 2.河南省农业科学院 农业经济与信息研究所,河南 郑州450002;3.郑州大学 公共卫生学院,河南 郑州450001)
猪乙脑病毒BHK-21细胞传代株BSF.ZZ-1-p60和BSF.ZZ-3-p60的全基因组序列分析
滕 蔓1,胡 博1,冯丽丽2,樊剑鸣3,郅玉宝1,李秀杰1,邓瑞广1,罗 俊1*
(1.河南省农业科学院 动物免疫学重点实验室/河南省动物免疫学重点实验室/农业部动物免疫学重点实验室,河南 郑州 450002; 2.河南省农业科学院 农业经济与信息研究所,河南 郑州450002;3.郑州大学 公共卫生学院,河南 郑州450001)
为研究猪源乙脑病毒分离株基因遗传稳定性,对猪源乙脑病毒分离株BSF.ZZ-1和BSF.ZZ-3在BHK-21细胞上连续传代60次后的病毒全基因组进行序列测定及分析。结果表明,BSF.ZZ-1-p60、BSF.ZZ-3-p60基因组全长均与各自的亲本毒株BSF.ZZ-1、BSF.ZZ-3一致,均为10 977 nt,在2个病毒基因组的10 701 nt位点处均插入1个碱基G。与亲本毒株相比,2个细胞传代毒株的病毒基因组经连续传代后趋于稳定,均有10个位点发生氨基酸突变,这些突变主要集中在E蛋白区域。核苷酸序列同源性比对分析发现,与其他JEV参考毒株相比,BSF.ZZ-1-p60和BSF.ZZ-3-p60与弱毒疫苗株SA14-14-2的核苷酸序列同源性较高,分别为99.6%和95.9%,其氨基酸序列与猪源野毒分离株SH0601和HW的氨基酸序列同源性较高,分别为98.2%、86.4%和98.3%、86.5%。
猪; 乙脑病毒; BHK-21细胞; 细胞传代; 病毒基因组; 序列分析
日本流行性乙型脑炎(Japanese encephalitis,JE)是由日本流行性乙型脑炎病毒(Japanese encephalitis virus,JEV)感染人、猪、马等多种动物引起的一种急性人畜共患传染病[1]。JE简称乙脑,该病主要发生于学龄前儿童及老龄人群,患者或感染动物的临床症状主要表现为神经性脑炎,痊愈后往往留下严重的后遗症。蚊虫是JEV的自然宿主和重要传播媒介,尤其是三带喙库蚊,病毒可随蚊虫冬眠而越冬存活。猪是JEV的重要贮存和扩增宿主,经由蚊虫叮咬后,JEV可在人群和猪群之间形成人—蚊—猪的循环传播[2]。不同日龄猪均可感染JEV,发病猪主要表现为妊娠母猪流产、木乃伊胎、新生仔猪脑炎或种公猪睾丸炎,导致猪群繁殖障碍及生产性能下降,给养猪业造成严重的经济损失,同时也增加了人群公共卫生安全的风险。
JEV是一种单股正链RNA病毒,在分类上属于黄病毒科(Flaviridae)、黄病毒属(Flavivirus),病毒基因组全长为11 kb,5′末端有一个I型帽子结构(m7GpppAmp),3′末端多为CU-OH,无poly A尾巴[3]。JEV共编码3个结构蛋白(C-PrM/M-E)和7个非结构蛋白(NS1-NS2a-NS2b-NS3-NS4a-NS4b-NS5),各基因区段间无重叠。此前,笔者所在课题组从河南省某规模化种猪场睾丸肿大的发病公猪精液中分离到2个JEV流行毒株BSF.ZZ-1和BSF.ZZ-3,基于prM和E基因的系统发生树分析显示,这2个毒株均属于基因Ⅲ型[4-5]。将这2个猪源分离株在BHK-21细胞上进行连续传代,对不同代次毒株E基因序列分析的结果表明,连续传代60次后,病毒E基因已基本稳定[6]。前期研究未对这2个猪源分离株的全基因遗传稳定性进行测定,为此,本研究对BSF.ZZ-1和BSF.ZZ-3的细胞传代毒株BSF.ZZ-1-p60和BSE.ZZ-3-p60的全基因组分别进行了序列测定,以期探讨细胞连续传代后整个病毒基因组的变化情况,为后续疫苗研究奠定基础。
1 材料和方法
1.1 细胞、毒株及试剂
BHK-21细胞系由本实验室保存;JEV分离株BSF.ZZ-1和BSF.ZZ-3毒株均为本实验室分离保存[4-5];新生牛血清(NCS)购自杭州四季青公司;TRIzol购自Invitrogen公司;M-MLV逆转录酶、pMD19-T 载体、DNA 回收试剂盒、ExTaq预混酶、RNA酶抑制剂、E.coliDH 5α 感受态细菌、dNTP均由TaKaRa公司提供。
1.2 病毒传代
常规方法复苏BHK-21细胞,按文献[7]的方法制备细胞单层,然后将BSF.ZZ-1和BSF.ZZ-3毒株在BHK-21细胞上连续培养,传代60次后收集子代病毒培养物,分别命名为BSF.ZZ-1-p60和BSF.ZZ-3-p60,-70 ℃冻存备用。
1.3 引物设计
根据GenBank已报道的JEV参考毒株全基因组序列,用Primer Premier 5.0软件设计8对保守的JEV全基因组重叠引物(表1),引物由生物工程(上海)股份有限公司合成。
表1 用于JEV病毒全基因组RT-PCR扩增的引物

引物编号引物名称类型引物序列(5'—3')引物长度/bpPCR产物长度/bp1JEVpC/M-F115'CTGTGTGAACTTCTTGGCTTAG22967JEVpC/M-R9783'AACTGTAAGCCGGAGCGACCAA222JEVpE-F9585'TTGGTCGCTCCGGCTTACAG201574JEVpE-R25323'GCCACTTCCACACCTCATCT203JEVpNS1-F25265'AAGTGGCATCTTCGTGCACA201149JEVpNS1-R36753'CCCAAGCATCAGCACAAGTA204JEVpNS2-F36615'GTGCTGATGCTTGGGGGCAT201366JEVpNS2-R50273'AGAATGGGTGAGCCGGATGT205JEVpNS3-F50055'GGAACATCCGGCTCACCCAT201495JEVpNS3-R65003'CATGCGACCGAGCACCTCTA206JEVpNS4-F64875'GTGCTCGGTCGCATGCCTGA201313JEVpNS4-R78003'GCGTGCTTCAGTGCGGTCCA207JEVpNS5/1-F77805'AGGTGGACCGCACTGAAGCA201650JEVpNS5/1-R94303'CATGACCTTGACCACTTTGT208JEVpNS5/2-F94005'ACAGGCACAAAGTGGTCAAG201578JEVpNS5/2-R109783'AGATCCTGTGTTCTTCCTCA20
1.4 RT-PCR
各取300 μL BSF.ZZ-1-p60和BSF.ZZ-3-p60病毒液,采用TRIzol法提取总RNA。用表1中的3′引物作为反转录引物,分别制备cDNA。 20 μL PCR体系包括:ExTaq预混酶10 μL、cDNA模板 1 μL、5′及3′引物各1 μL、 d3H2O 7 μL。PCR反应条件设定为:94 ℃预变性4 min;94 ℃变性30 s、55 ℃复性30 s、72 ℃延伸2 min,共30个循环;72 ℃延伸10 min。最后用1%琼脂糖凝胶电泳分析PCR产物。
1.5 基因克隆及测序
采用1%琼脂糖凝胶电泳分离PCR产物并回收目的片段,连接pMD19-T载体后转化至DH 5α感受态细胞,涂氨苄青霉素抗性的LB平板,37 ℃培养过夜。菌落PCR筛选阳性克隆,PCR反应体系和条件同1.4,最后将阳性菌送样至生物工程(上海)股份有限公司测序。
1.6 全基因组序列分析
用DNAstar MegAlign软件将测序获得的8个基因片段分别与部分JEV参考毒株(表2)全基因组序列进行比对,分析其序列同源性,然后用SeqMan软件将基因片段拼接成全基因组。最后分析BSF.ZZ-1-p60和BSF.ZZ-3-p60与6个参考毒株的全基因组核苷酸及氨基酸序列同源性,并结合15个强、弱参考毒株的基因组对关键氨基酸位点进行比对分析。
表2 JEV参考毒株的背景信息

编号毒株基因型登录号年份国家与地区来源1P3ⅢU470321949北京人2SA14ⅢU141631954西安人3Beijing-1ⅢL489611949北京人4HWⅢAY8499391988湖北猪5HWeⅢEF1075231988湖北猪6SA14-12-1-7ⅢAF4164571960s中国蚊虫7SH0601ⅢEF5438612006上海猪8JaGAr01ⅢAF0690761959日本蚊虫9P20778ⅢAF0802511958印度人10SA14-14-2ⅢAF3151191960s中国蚊虫11JEV-at222ⅢAB196924不详日本人12JEV-at31ⅢAB196923不详日本人13JaOH0566ⅢAY5088131966日本人14CH2195LAⅢAF2214991994台湾蚊虫15ML17ⅢAY5088121981日本疫苗16GP78ⅢAF0757231978印度人
2 结果与分析
2.1 JEV细胞传代毒株基因组的RT-PCR扩增及测序结果
将猪源JEV分离株BSF.ZZ-1和BSF.ZZ-3在BHK-21细胞上连续传代60次,分别提取病毒培养物总RNA,然后用JEV特异性引物(表1)对2个传代毒株的病毒全基因组进行RT-PCR,分别扩增JEV的C/M、E、NS1、NS2、NS3、NS4、NS5-1和NS5-2基因片段。电泳分析结果表明,所扩增的PCR产物条带均与预期大小相符(图1)。PCR产物经克隆测序,结果显示,2个毒株的8个基因组片段与SA14-14-2的核苷酸序列同源性均在98%以上。
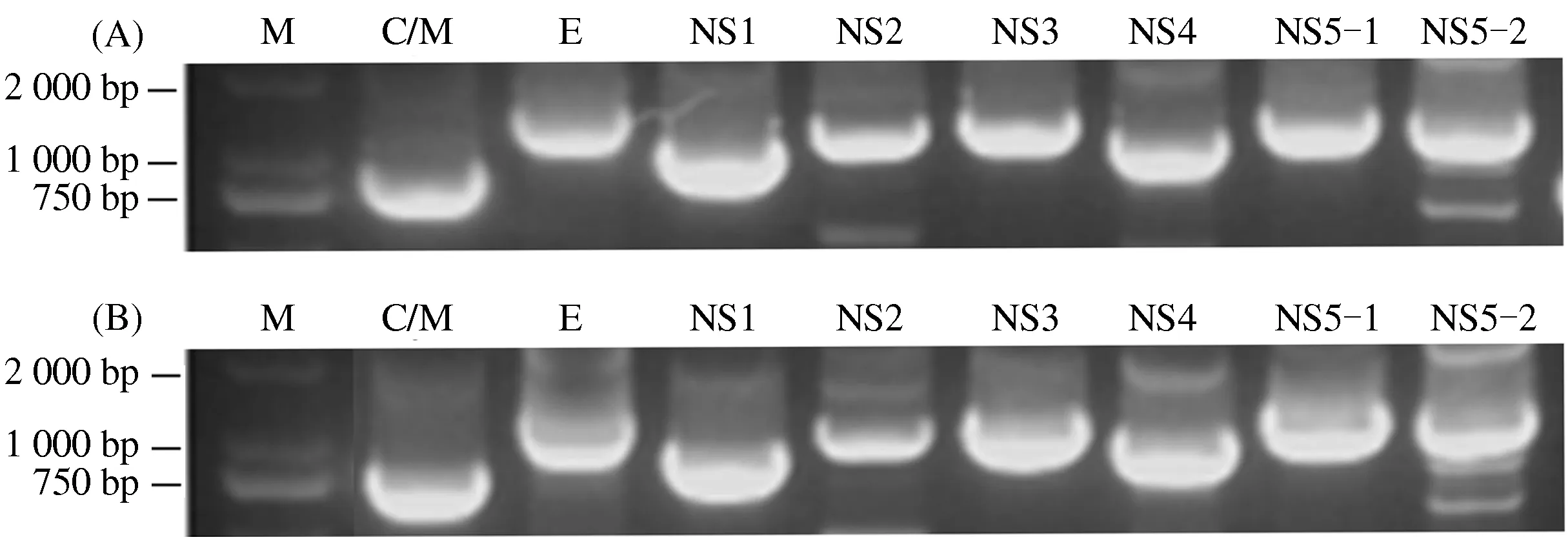
(A)为BSF.ZZ-1-p60; (B)为BSF.ZZ-3-p60
2.2 JEV细胞传代毒株全基因组的序列拼接及分析结果
对克隆获得的8个病毒基因片段序列分别进行全基因组拼接,分析结果显示,BSF.ZZ-1-p60和BSF.ZZ-3-p60的病毒基因组全长均为10 977 nt,在2个病毒基因组的10 701 nt位点处均有1个插入碱基G。BSF.ZZ-1-p60的A、G、T、C比例分别为27.67%、28.42%、21.01%、22.90%;BSF.ZZ-3-p60的A、G、T、C比例分别为27.75%、28.35%、20.99%、22.91%。2个毒株5′ 非编码区均含有96个核苷酸,均有1个97位至10 392位的单一开放阅读框,编码3 432个氨基酸残基。
2.3 JEV细胞传代毒株基因组与参考毒株的序列同源性比对分析结果
将BSF.ZZ-1-p60、BSF.ZZ-3-p60的病毒全基因组序列分别与表2中的强毒株SA14、弱毒疫苗株SA14-14-2、中间减毒株SA14-12-1-7以及3个猪源分离强毒株SH0601、HWe和HW进行同源性比对分析,结果表明,BSF.ZZ-1-p60和BSF.ZZ-3-p60的全基因组核苷酸序列同源性与SA14、SA14-14-2、SA14-12-1-7疫苗株较高,其中与SA14-14-2同源性最高,分别为99.6%和95.9%,但与SH0601、HWe和HW的同源性较低(表3)。6个参考毒株的全基因组氨基酸序列与BSF.ZZ-1-p60的同源性均较高,而与BSF.ZZ-3-p60的氨基酸序列同源性均较低。BSF.ZZ-1-p60和BSF.ZZ-3-p60与SH0601和HW的氨基酸序列同源性较高,分别为98.2%、86.4%和98.3%、86.5%。

表3 JEV细胞传代毒株与6个参考毒株的基因组序列同源性分析 %
2.4 JEV细胞传代毒株病毒全基因组关键氨基酸残基位点差异分析
将BSF.ZZ-1-p60、BSF.ZZ-3-p60与表2中的10个强毒株、5个弱毒株及其亲本毒株的全基因组氨基酸序列进行比对,分析关键氨基酸位点的差异(表4)。与SA14等强毒株和弱毒株相比,2个细胞传代毒株及其亲本毒株的氨基酸序列特征与疫苗毒株SA14-14-2和中间减毒株SA14-12-1-7更接近;与亲本毒株BSF.ZZ-1和BSF.ZZ-3相比,2个细胞传代毒株各自均发生了10个位点的氨基酸突变,主要集中在E蛋白区域;2个细胞传代毒株之间也有多个氨基酸位点差异,如E107、E138、E176、E279等。上述结果表明,BSF.ZZ-1-p60和BSF.ZZ-3-p60的病毒全基因组氨基酸序列与弱毒疫苗株SA14-14-2差异最小。
表4 BSF.ZZ-1-p60、BSF.ZZ-3-p60与15个参考毒株全基因组关键氨基酸位点差异分析
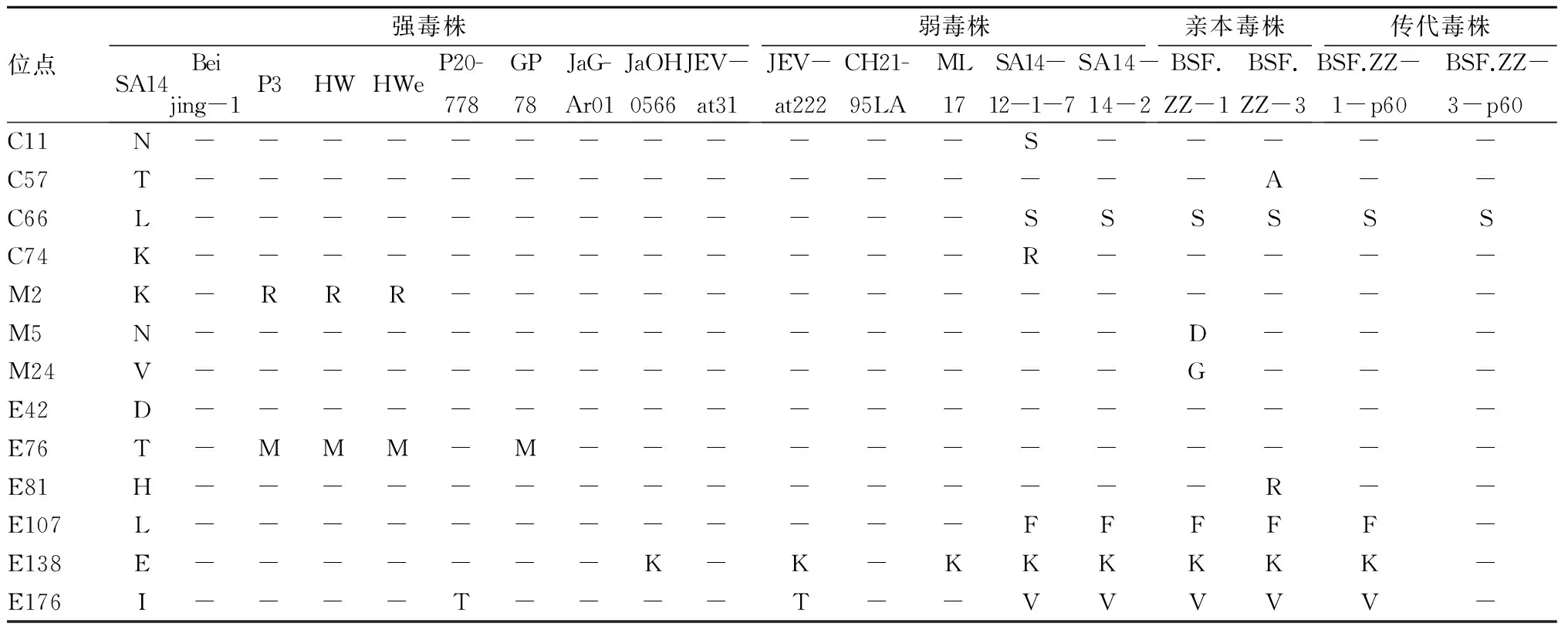
位点强毒株SA14Beijing-1P3HWHWeP20-778GP78JaG-Ar01JaOH0566JEV-at31弱毒株JEV-at222CH21-95LAML17SA14-12-1-7 SA14- 14-2亲本毒株BSF.ZZ-1BSF.ZZ-3传代毒株BSF.ZZ-1-p60 BSF.ZZ-3-p60C11N------------S-----C57T---------------A--C66L------------SSSSSSC74K------------R-----M2K-RRR--------------M5N--------------D---M24V--------------G---E42D------------------E76T-MMM-M------------E81H---------------R--E107L------------FFFFF-E138E-------K-K-KKKKKK-E176I----T----T--VVVVV-
续表4 BSF.ZZ-1-p60、BSF.ZZ-3-p60与15个参考毒株全基因组关键氨基酸位点差异分析

位点强毒株SA14Beijing-1P3HWHWeP20-778GP78JaG-Ar01JaOH0566JEV-at31弱毒株JEV-at222CH21-95LAML17SA14-12-1-7 SA14- 14-2亲本毒株BSF.ZZ-1BSF.ZZ-3传代毒株BSF.ZZ-1-p60 BSF.ZZ-3-p60E177T-------------AAA-AE244E------------GGGG--E264Q------------HHHHHHE271V------------------E279K-------------MMM-ME315A-------------VVVVVE439K------------RRRRRRE447G-------------DD---NS1-22V----I----------IHHNS1-235DG-GGGGGGGGGG------NS1-292SG-GGGGGGGGGGG-----NS1-339R-------------MMMMMNS1-351D-------------HHHHHNS1-354N------------KKKKKKNS1-392A-------------VVVVVNS2a-1A-----------S------NS2a-121HQ-QQQQQQQQQQ------NS2b-63E-------------DDDDDNS2b-65D-----E------GGGGGGNS3-31L-----F---------F--NS3-59M------------VVVVVVNS3-105A------------GGGGGGNS3-235A-------------VVVVVNS3-246S--------------T---NS3-474H------------------NS4a-64T---------------A--NS4a-230V--------------A---NS4a-255I------------VVVVVVNS5-231H---------------Y--NS5-268H-YYY--YYYY-Y-YYYGGNS5-557E------------------NS5-635Q------------------NS5-639Q-------------HHHHHNS5-671V-------------AAAAANS5-855L--------------F---
注:“-”表示该位点的氨基酸残基与SA14相应位点的氨基酸残基一致。
3 讨论
JE最早发现于日本,之后在东南亚也相继报道,调查显示,JE最初可能发生于印度尼西亚或马来西亚[8-9]。与其他病毒相比,JEV病毒基因组编码的大多数基因较为保守,更多研究主要集中于prM或E基因。此前研究表明,基因Ⅰ型JEV毒株的流行分布日渐广泛,有超过基因Ⅲ型的趋势[10-11]。在长期疫苗免疫压力及病毒自身进化过程中,JEV是否可能发生病毒变异或毒力增强,仍需要更加密切、广泛的流行病学调查及疫情监测。猪是JEV最重要的储存和扩增宿主[2],因此研发高效的猪用乙脑疫苗、做好猪群的免疫接种,对于有效控制人群乙脑的流行至关重要。在此前研究中[4-6],笔者所在课题组已从临床病例猪中分离了2个JEV流行毒株BSF.ZZ-1和BSF.ZZ-3,并将其在BHK-21细胞上进行了连续60次的传代培养,对传代毒株E基因的序列分析发现病毒传代已趋稳定。因此在本研究中,对BSF.ZZ-1-p60和BSF.ZZ-3-p60这2个细胞传代毒株的全基因组进行了序列测定和分析,结果表明,与亲本毒株相比,连续传代的子代病毒基因组有多个氨基酸残基位点的稳定突变,其中E蛋白突变较多。与强毒株SA14和弱毒株SA14-12-1-7、SA14-14-2[12-13]相比,BSF.ZZ-1-p60和BSF.ZZ-3-p60与疫苗株SA14-14-2的核苷酸序列同源性最高。此前研究表明,影响JEV毒力的除了E基因起关键作用外,还包括C基因和一些非结构基因。氨基酸残基位点E107、E138、E176、E177、E264、E279、E315、E439、NS2b-63、NS3-105和NS4b-106等被认为是JEV减毒过程中的关键突变位点,与JEV毒力直接相关[14]。本研究显示,BSF.ZZ-1-p60和BSF.ZZ-3-p60基因组中这些位点大多与疫苗株SA14-14-2一致,与SA14不一致。从病毒全基因组氨基酸序列同源性来看,BSF.ZZ-1-p60和BSF.ZZ-3-p60与猪源分离株的同源性更高,这为今后进一步筛选猪用乙脑疫苗候选毒株奠定了良好的基础。
[1] 殷震,刘景华.动物病毒学[M].2版.北京:科学出版社,1997:470-473.
[2] Konno J,Endo K,Agatsuma H,etal.Cyclic outbreaks of Japanese encephalitis among pigs and humans[J].American Journal of Epidemiology,1966,84(2):292-300.
[3] 项秉懿,陈苏民.乙型脑炎病毒的分子生物学研究进展[J].中国病毒学,1993,8(3):199-206.
[4] 王兴涛,罗俊,滕蔓,等.猪流行性乙型脑炎病毒种猪精液分离株的鉴定及进化分析[J].河南农业科学,2011,40(5):152-157.
[5] 禹乐乐,滕蔓,罗俊,等.猪源乙型脑炎病毒河南分离株的全基因组测序及进化分析[J].华北农学报,2012,27(5):184-190.
[6] 胡博,滕蔓,禹乐乐,等.猪乙脑病毒分离株BSF.ZZ-1和BSF.ZZ-3在BHK-21细胞上的传代培养及E基因序列稳定性分析[J].华北农学报,2013,28(6):71-76.
[7] Teng M,Luo J,Xing G X,etal.Main factors influencing the efficient propagation of virulent or attenuated strains of Japanese encephalitis virus in BHK-21 cells[J].Indian Journal of Virology,2012,23(3):344-348.
[8] Solomon T,Ni H,Beasley D W,etal.Origin and evolution of Japanese encephalitis virus in southeast Asia[J].Journal of Virology,2003,77(5):3091-3098.
[9] Mackenzie J S,Williams D T,Smith D W.Japanese encephalitis virus:The geographic distribution,incidence,and spread of a virus with a propensity to emerge in new areas[M]//Tabor Emerging viruses in human populations.Holard:Elsevier,2007:201-268.
[10] Chen S P.Molecular phylogenetic and evolutionary analysis of Japanese encephalitis virus in China[J].Epidemiology and Infection,2012,140(1):1637-1643.
[11] Teng M,Luo J,Fan J M,etal.Molecular characterization of Japanese encephalitis viruses circulating in pigs and mosquitoes on pig farms in the Chinese province of Henan[J].Virus Genes,2013,46(1):170-174.
[12] Xin Y Y,Ming Z G,Peng G Y,etal.Safety of a live-attenuated Japanese encephalitis virus vaccine (SA14-14-2) for children[J].The American Journal of Tropical Medicine and Hygiene,1988,39(2):214-217.
[13] 俞永新,武佩芬,熬坚,等.一株免疫性进一步提高的乙脑活疫苗减毒株的选育Ⅰ,SA14-14-2 弱毒株的某些生物学特性[J].中华微生物学和免疫学杂志,1981,1(2):77-83.
[14] 李静,俞永新,安琪,等.乙型脑炎病毒减毒活疫苗株 (SA14-14-2) 神经毒力减弱的分子基础[J].中国病毒病杂志[J].2011,1(1):51-57.
Sequence Analysis of Full-length Genomes of Two Japanese Encephalitis Virus Strains BSF.ZZ-1-p60 and BSF.ZZ-3-p60 Passaged on BHK-21 Cells
TENG Man1,HU Bo1,FENG Lili2,FAN Jianming3,ZHI Yubao1,LI Xiujie1,DENG Ruiguang1,LUO Jun1*
(1.Henan Provincial Key Laboratory of Animal Immunology/Key Laboratory of Animal Immunology of the Ministry of Agriculture,Henan Academy of Agricultural Sciences,Zhengzhou 450002,China; 2.Institute of Agricultural Economics and Information,Henan Academy of Agriculture Sciences,Zhengzhou 450002,China; 3.College of Public Health,Zhengzhou University,Zhengzhou 450001,China)
In this study,the full-length genomes of two Japanese encephalitis virus (JEV) strains,BSF.ZZ-1-p60 and BSF.ZZ-3-p60 that were isolated from pigs and passaged on BHK-21 Cells,were sequenced and compared with reference strains.The results showed that same to the parental viruses,both of the genomes of BSF.ZZ-1-p60 and BSF.ZZ-3-p60 were 10 977 nt long,with an insert base of G at the 10 701 nt site.Compared to the parental strains,the viral genomes of BSF.ZZ-1-p60 and BSF.ZZ-3-p60 were stabilized after passaged on BHK-21 cells for 60 times,with a total of 10 amino acid mutations which we mainly occurred in E proteins in both strains.Compared to the reference JEV strains,both BSF.ZZ-1-p60 and BSF.ZZ-3-p60 showed highest nucleotide sequence homologies (99.6% and 95.9%) to vaccine strain SA14-14-2, while for the amino acid sequences,both strains showed highest homologies (98.2%,86.4% and 98.3%,86.5% ) to that of the pig-originated strains SH0601 and HW,respectively.
pig; JEV; BHK-21 cell; cell passage; viral genome; sequence analysis
2015-11-20
国家公益性行业(农业)科研专项(201203082-5);河南省科技攻关计划项目(152102110127);河南省农业技术推广财政补助资金项目(YCN201519111);河南省农科院科研发展专项资金项目(20157817)
滕 蔓(1977-),女,河南周口人,助理研究员,博士,主要从事动物疫病快速检测技术研究及产品研发。 E-mail:tm135@aliyun.com
*通讯作者:罗 俊(1978-),男,河南罗山人,研究员,博士,主要从事动物病毒流行病学、病原学、致病机制及快速检测技术研究。E-mail:luojun593@aliyun.com
S855.3
A
1004-3268(2016)03-0135-06

