利用微卫星标记指导红鳍东方鲀亲本选配
刘永新,刘奕,周勤,张红涛
(1.中国水产科学研究院,北京 100141;2.中国水产科学研究院 珠江水产研究所,广东 广州 510380;3.中国水产科学研究院 北戴河中心实验站,河北 秦皇岛 066100)
利用微卫星标记指导红鳍东方鲀亲本选配
刘永新1,刘奕2,周勤3,张红涛3
(1.中国水产科学研究院,北京 100141;2.中国水产科学研究院 珠江水产研究所,广东 广州 510380;3.中国水产科学研究院 北戴河中心实验站,河北 秦皇岛 066100)
摘要:为制定红鳍东方鲀家系配组方案提供可行性指导,利用微卫星标记辅助红鳍东方鲀Takifugu rubripes家系的建立,选择34个微卫星标记对红鳍东方鲀两个群体进行遗传评估。结果表明:A群体的平均等位基因数(Na)和Nei基因多样性指数(He)分别为6.647 0和0.711 5,B群体的相应值分别为4.647 1和0.646 1,且两个群体之间的遗传差异达到显著性水平(P<0.05);群体间的遗传分化系数(GST)为0.050 7,基因流(Nm)为4.679 6, 表明红鳍东方鲀群体间存在低程度的遗传分化和一定程度的基因交流;分子方差分析(AMOVA)表明,遗传变异主要存在于群体内,所占比例为78.49%,而群体之间仅占21.51%;根据群体间Nei氏遗传距离构建UPGMA系统树,群体内个体之间的遗传距离为0.11~0.82,依据遗传距离的远近将全部个体分成多个分支,不同分支内含有数个个体;各项遗传参数表明,试验群体具备一定程度的遗传变异,不同个体之间拥有相对较远的亲缘关系,可以根据个体之间遗传距离制定育种计划,建立家系进行选育研究。研究表明,利用微卫星标记信息构建红鳍东方鲀家系的方法是切实可行的。
关键词:红鳍东方鲀;群体;微卫星;遗传距离
红鳍东方鲀Takifugurubripes隶属于硬骨鱼纲Osteichthyes、鲀形目Tetraodontiformes、鲀亚目Tetraodontoidei、鲀总科Tetraodntoidea、鲀科Tetraodontidae、东方鲀属Takifugu,俗称河豚,主要分布于中国的渤海、黄海和东海,以及日本沿海和朝鲜半岛[1]。红鳍东方鲀肉质细嫩、味道鲜美、高蛋白、低脂肪并含有丰富的维生素和微量元素,素有“鱼中之王”的美誉[2-5]。红鳍东方鲀的养殖始于20世纪80年代日本,其养殖方式以陆上水池人工养殖为主,但养殖产量无法满足日本民众的需求,因此,每年还需从中国进口大量的商品鱼。中国的红鳍东方鲀人工养殖始于1991年, 但由于中国自然资源量明显小于日本,而且红鳍东方鲀和假睛东方鲀Takifugupseudommus两个物种具有相近的繁殖季节和栖息海区,非常容易发生混交,从而为中国的红鳍东方鲀养殖生产和市场稳定带来了不利影响[6]。国内养殖的品种价格不高、种质不纯,出口受限制是养殖户面临的实际问题。中国水产科学研究院黄海水产研究所在1996年首次引进日本红鳍东方鲀种群,在国内确立了纯种引进技术,同时形成了全人工纯种育苗的大规模生产。迄今为止,国内有很多养殖公司仍然采用从日本引种的方式进行生产,在繁育了1~2代后养殖苗种明显出现生长速度缓慢、抗逆性降低的现象,为了解决这些问题只能再次从国外引种,因此,红鳍东方鲀养殖业受国外种质资源控制的影响非常严重。如何利用引进群体和国内本土资源开展良种选育研究,尽快培育出满足市场需求的优良品种并提供长期稳定的供应,是当前育种工作者亟待解决的根本问题。
国内对红鳍东方鲀遗传的研究虽有所开展,但多以形态性状[7-9]、群体结构和生化遗传变异[10-11]、分子标记开发[12-14]和种内杂交[15-16]等研究为主,未见有系统性的优良品种选育进程报道。到目前为止,没有红鳍东方鲀品种通过国家原良种审定委员会审定并予以推广养殖。选择育种技术是水产动物品种遗传改良过程中应用最为广泛的方法,杂交育种和分子标记育种等育种技术都要以选择育种为基础来开展。即使是现在,部分水产动物品种都已完成了基因组测序,随之而产生的全基因组育种技术也同样离不开选择育种。从已有研究基础来看,红鳍东方鲀主要经济性状的遗传改良采用常规的选择育种技术更为适合。开展选择育种首先需要构建具有丰富遗传变异的基础群体;其次,要借助分子标记技术分析群体的遗传结构,判断遗传变异度和群体内个体之间的遗传距离,制定相应的亲本配组方案,建立选育家系[17]。多种分子标记都能够进行群体遗传结构分析,随机扩增片段长度多态性DNA、扩增片段长度多态性和微卫星DNA技术先后在不同历史时期应用于水产动物遗传研究中。现阶段,微卫星DNA分子标记是进行群体分析的首选标记,众多水产动物品种如牙鲆Paralichthysolivaceus[18]、大菱鲆Scophthalmusmaximus[19]、黄颡鱼Pseudobagrusfulvidraco[20]、大黄鱼Pseudosciaenacrocea[21]、吉富罗非鱼Oreochromisniloticus[22]、赤点石斑鱼Epinephelusakaara[23]等都利用这种标记分析了目标群体的遗传基础。本研究中,以从日本进口的红鳍东方鲀受精卵发育至性成熟的亲本组成育种群体,利用34个微卫星DNA分子标记分析该群体的遗传结构,了解其遗传变异水平并进行UPGMA系统聚类,确定个体之间的遗传距离,旨在为大规模建立选育家系和开展分子标记辅助育种研究工作提供基础数据和理论指导。
1材料与方法
1.1材料
试验用红鳍东方鲀亲鱼共计225尾,其中199尾来自河北省唐山市天河水产养殖公司(记为A群体),26尾来自河北省昌黎县河鲀鱼良种场(记为B群体)。天河养殖有限公司从1995—1999年,每年由日本直接引进受精卵进行苗种孵化,并从事养殖,2000年时上述引进的苗种达到性成熟,将这些成熟的河鲀作为亲鱼;之后为防止近交衰退,每两到三年由日本引进一次受精卵更新种鱼,试验所采集的A群体为该公司养殖至今保有的亲鱼群体。河北省昌黎县河鲀鱼良种场亲鱼B群体也是由日本进口的受精卵经孵化、培育至性成熟的个体产生的后代,但其引种时间不同于天河养殖有限公司,为2005—2007年。所有亲鱼年龄为4~6龄,体质量为0.8~3.5 kg。采集亲鱼尾部鳍条用于提取基因组DNA。
1.2方法
1.2.1微卫星标记的筛选与PCR扩增选择由日本学者[24]和中国学者[10-12]开发的34个高多态性微卫星标记进行试验分析。标记名称、引物序列和退火温度列于表1。由上海生工生物工程服务有限公司负责引物合成。利用TIANGEN海洋动物基因组DNA提取试剂盒进行基因组DNA的提取。提取完成后,采用琼脂糖凝胶电泳检测DNA样品的质量,用核酸蛋白测定仪检测 DNA 浓度(A260/280值在1.8~2.0 之间较好);之后,稀释 DNA 至 50 ng/μL,于冰箱(-20 ℃)中保存备用。PCR反应体系(共25 μL):10×Buffer 2.5 μL,Mg2+(25 mmol/L)1 μL,dNTPs(各2 mmol/L)1 μL,上、下游引物(10 μmol/L)各1 μL,模板DNA (50 ng/μL)1 μL,TaqDNA聚合酶1 U,用ddH2O补足至25 μL。PCR反应程序:94 ℃下预变性3 min;94 ℃下变性30 s,退火30 s,72 ℃下延伸30 s,共进行30个循环;最后在72 ℃下延伸10 min。在AB9700型PCR仪上进行PCR扩增。
1.2.2PCR扩增产物的检测PCR扩增产物经8%非变性聚丙烯酰胺凝胶电泳检测,上样缓冲液由0.25%的溴酚蓝和40%的蔗糖水溶液组成。电泳后用1%硝酸银染色10 min,显色液显色10 min。凝胶在HP scanjet G4010扫描仪上成像,用Gel-Pro Analyzer 4.5凝胶分析软件对电泳谱带进行数据采集和分析。
1.3数据处理
每对微卫星引物检测一个位点,每条多态性带为一个等位基因。利用Popgene 3.2软件[25]统计每个微卫星标记的等位基因数(Number of alleles,Na)、Nei基因多样性指数(Nei’s gene diversity index,He)和Nei遗传距离(Genetic Distance,GD)[26]。采用Wilcoxon 配对检验法进行A群体和B群体等位基因数和Nei 基因多样性指数的显著性检验。根据Nei遗传距离进行非加权类平均法(UPGMA)系统聚类。基于每一个位点的等位基因差异,采用Arlequin 3.0软件[27]中的分子方差分析程序(AMOVA)计算F统计量(F-statistics,Fst),分析红鳍东方鲀群体的遗传分化,采用Excoffier 等[28]的非参数置换方法(3000次)进行Fst的显著性检验。
2结果与分析
2.1红鳍东方鲀群体的遗传参数
利用34个微卫星标记进行红鳍东方鲀群体的遗传分析,统计每个位点的等位基因数和Nei基因

表1 34个微卫星标记的名称、引物序列和退火温度
多样性指数。从表2可见,不同的微卫星位点具有较大的差异性。A群体的等位基因数为3~13,B群体的为0~12;A群体的Nei基因多样性指数为0.456 6~0.875 0,B群体的为0~0.840 2。从表3 可知,两个群体的等位基因数和Nei 基因多样性指数的平均值均较高, 表明这两个群体资源均具有较丰富的多样性。Wilcoxon 配对测验表明,A群体平均等位基因数(6.647 0)和Nei基因多样性指数(0.711 5)均高于B群体(4.647 1和0.646 1),且具有显著性差异(P<0.05)。由此表明,A群体具有更加丰富的遗传多样性。
2.2红鳍东方鲀群体的遗传分化
根据 Nei基因多样性指数计算两个群体总的遗传分化系数(GST)为0.050 7,由GST计算种群间的基因流值(Nm)为4.679 6,说明这两个红鳍东方鲀群体之间存在着一定程度的基因交流。分子方差分析(AMOVA)结果表明,遗传变异主要存在于群体内部,所占比例为78.49%,而群体之间的遗传变异仅占21.51%(表4)。
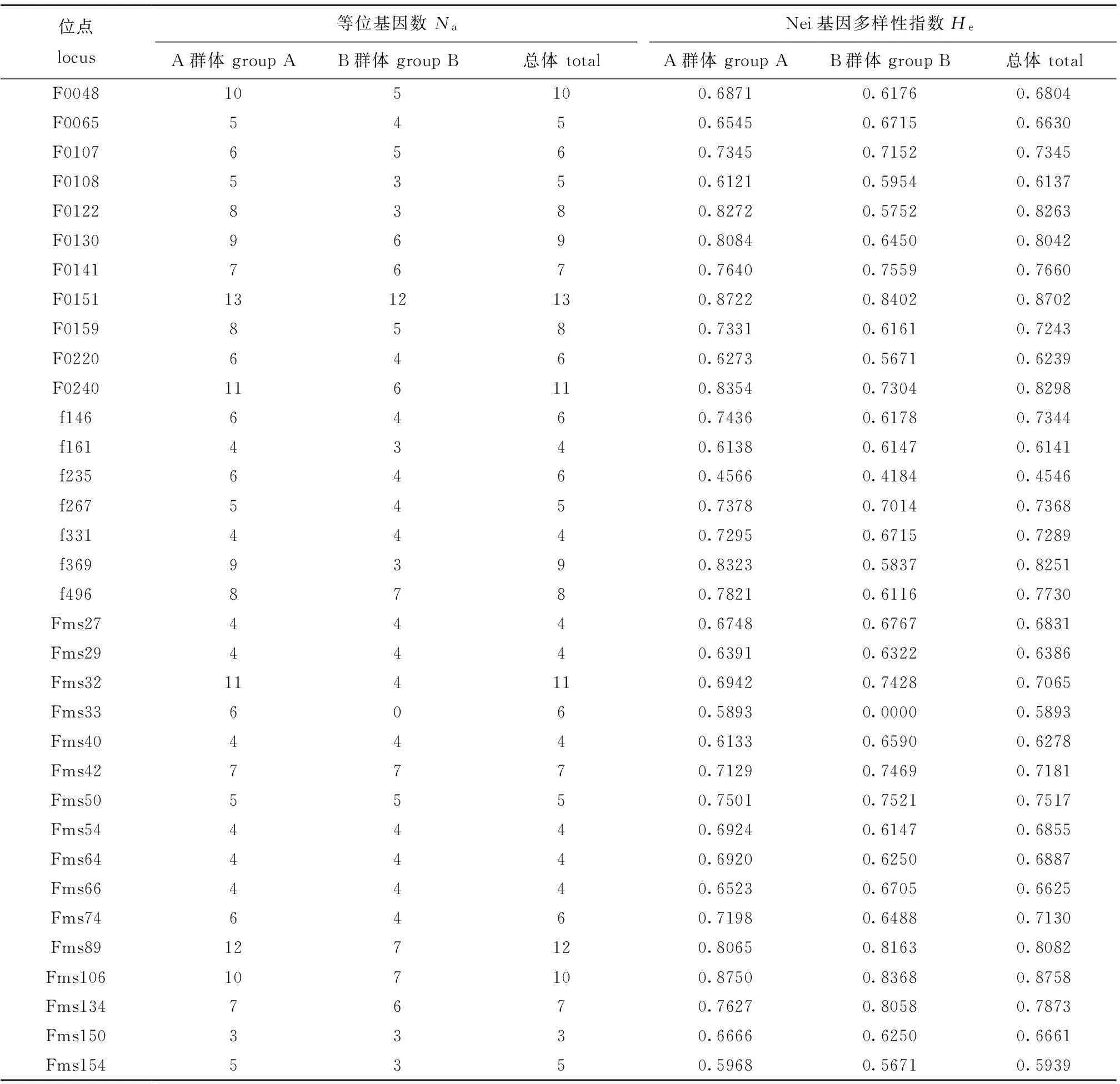
表2 红鳍东方鲀两个群体在34个微卫星位点的等位基因数和Nei基因多样性指数
表3红鳍东方鲀两个群体在34个微卫星位点的平均等位基因数和Nei基因多样性指数
Tab.3Mean number of alleles and average Nei’s gene diversity index at 34 microsatellite loci in two populations of redfin puffer

群体group样本数numberofsample等位基因数Na平均值±标准差mean±S.D.P值PvalueNei基因多样性指数He平均值±标准差mean±S.D.P值PvalueAB199266.6470±2.6501a4.6471±1.9677b0.00050.7115±0.0914a0.6461±0.1445b0.0240
注:同列中标有不同小写字母者表示组间有显著性差异(P<0.05)
Note:The means with different letters within the same column are significant differences at the 0.05 probability level
2.3聚类分析

表4 红鳍东方鲀群体的分子方差分析
两个群体共计225个个体的Nei遗传距离为0.11~0.82,表明这些个体之间既有遗传差异较大的个体,也存在遗传差异较小的个体。基于Nei遗传距离进行UPGMA系统聚类(图1),在Nei遗传距离为0.82处可以明显地划分为两类:A分支和B分支,A分支包含221个个体,而B分支仅包含来自B群体的4个个体(B23~B26)。在遗传距离为0.66处同样明显地分成两类,较大的分支为C分支,包括192个个体,较小的分支为D分支,包括29个个体。C分支在遗传距离为0.65处分为E、F两个分支,分别包含95和97个个体,其中E分支在遗传距离为0.59处又分为I和J两个分支。而F分支在遗传距离为0.61处分为G和H两个分支,其中G分支在遗传距离为0.57处分为K和L两个分支,而H分支在遗传距离为0.59处分为M和N两个分支。C分支在遗传距离小于0.60处细化成的6个分支I、J、K、L、M和N所包含的个体数依次为48、47、26、21、48和2个,其中I、J和K分支基本全部由来自A群体的个体组成,而大多数来自B群体的个体集合在M分支内。在遗传距离为0.11~0.60时,I、J、K、L、M和N分支内的个体按照遗传距离的远近程度聚集在不同的小分支内。
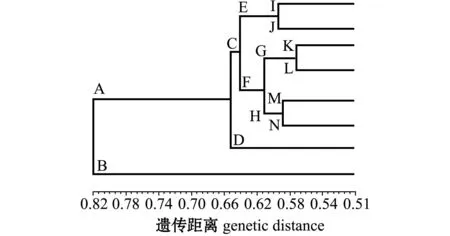
图1 亲鱼群体的亲缘关系聚类图Fig.1 Cluster dengrodogram of parental population
3讨论
3.1群体遗传学参数
传统的水产动物选择育种在进行家系构建时往往仅根据个体表型性状是否优良确定其是否作为繁殖用亲本。伴随着分子生物学技术日新月异的发展,不同种类的分子标记已经引入到家系选育过程中,通过判断基础群体的遗传变异和群体内个体之间的遗传距离,可为亲本个体的选择和配组方案的制定提供参考依据。利用这种方法组建家系的鱼类品种有建鲤Cyprinuscarpio[29]、团头鲂Megalobramaamblycephala[30]、虹鳟Oncorhynchusmykiss[31]等。遗传多样性是种群进化和遗传改良的前提和基础[32],物种的遗传多样性越高,其获得的遗传进展就越大。本研究中,所选群体的平均等位基因数和Nei基因多样性指数分别为4.647 1和0.646 1以上,与已有报道的红鳍东方鲀日本群体和秦皇岛群体的参数基本相同[10],而显著高于公司养殖群体和实验室养殖群体[11]。需要指出的是,虽然本试验中选择了几百个个体进行分析,但这些个体全部来自引进的受精卵,不敢确定一定存在遗传漂变,同时也无法确认是否能够代表日本原来的自然群体。为此,本研究中还选择了万玉美等[10]进行红鳍东方鲀两个自然群体遗传学研究时所用的微卫星标记,因为他们选择和评估这些标记,依据的是一个能代表自然群体的理想群体。除了上述两个遗传指标外,笔者也计算了Hardy-Weinberg遗传偏离指数,发现在当前群体中存在平衡偏离问题,而依据标记计算遗传多样性,描述群体结构的意义并不大,因为如果放归这些个体,这一结构马上会解体而不存在。对指导配组工作而言,试验的主要目的不是描述多样性和群体结构,而是获取个体之间遗传差异的信息,为实际育种服务。以本研究中所选的遗传参数指标等位基因数为例,每个位点的等位基因数都不相同,这其中就存在一个取样饱和度的问题。一个位点有十几个等位基因,尽管有几百个个体,仍会有很多等位基因只有一个,频率极低,这时群体内个体数量就不是30个,而需要300个甚至更多,因此,不是等位基因数愈多越好,只是在寻找个体差异的时候,等位基因数是越多越好。这里所强调的不是等位基因数量对群体研究的要求,而是等位基因数量对寻找个体差异的重要意义。
3.2群体内个体选配
分子方差分析表明,目标群体的大部分遗传变异存在于群体内部,少量的遗传差异存在于群体之间。由于AMOVA分析不必作Hardy-Weinberg假设,避免了数学假设带来的系统误差,其结合UPGMA系统聚类结果,可以更好地辅助微卫星标记进行个体遗传差异评估,作为建立家系的依据。鱼类的亲缘关系远近程度常用遗传距离来衡量,根据遗传距离对亲鱼进行聚类分析,能够为亲本配组和杂种优势预测提供参考依据[32]。本研究中的UPGMA系统聚类结果中,A分支和B分支在遗传距离为0.82处分开,这表明两个分支内的个体具有较远的亲缘关系,原则上可以作为配组对象。但是,B分支内个体数量过少,仅包括4个个体。这在实际育种工作中就会面临两个问题,其一,在繁殖季节亲鱼一般都需要进行营养强化,从而保证其能够孕育健康、优质的精子和卵子。亲鱼强化过程需要较为精湛的培育技术,在这一过程中雄性亲鱼容易产生活力不错的精子,但对雌性亲鱼往往不能保证全部待产个体都能够产生好的卵子,卵子质量较差或者生殖腺膨大却不能产出卵子的情况时有发生,从而导致预先设计的交配方案不能实施。因此,对于仅包含几个个体的B分支,一旦个体性腺发育不理想则无法进行配组。其二,即使B分支内个体性腺正常发育,但其数量过少,很容易成为一个单性别小群体,在配组时,只能做正交或反交的单向设计,且组合数也较少,产生性状优良组合的几率也将降低。此外,AMOVA分析结果表明,主要的遗传变异来自于群体内部而非群体之间,因此,应尽量考虑设计A群体或B群体内的个体配组。鉴于上述几点考虑,分支D、I、J、K、L、M和N之间的遗传距离(0.57~0.65)相对较远,这些分支内的个体可设计交配方案。这7个分支内还存在细化的小分支,这些小分支的遗传距离范围为0.11~0.57。有文献报道,家系生长在一定范围内随着亲本遗传距离的增大而逐渐增加,但是随着遗传距离的进一步增大,生长有下降的趋势。这表明配组亲本的遗传距离与生长之间存在一个阈值点,一旦超过这个阈值点,即使遗传距离再增加其生长优势反而会降低,这一论断在鲤中已经取得试验验证[33],除水产物种外,对部分作物的研究也得出了相似的结论[34-37]。参考这些研究结果,在本试验群体中对亲本遗传距离为0.11~0.60范围内的分支也可设计交配,一方面可以获得更多的组合,有助于在大规模组合中优中选优,另一方面可以验证红鳍东方鲀这一物种是否也存在类似的“阈值”理论。一旦发现阈值点,在今后的育种工作中便可集中于这一区域进行配组设计,从而加速优良组合的产生,用于新品种或新品系的筛选。本试验中对红鳍东方鲀群体遗传的分析结果,可以直接用于指导亲鱼配组实践,开展大规模家系选育,进行生长、抗病、品质、性别等多元化性状的遗传改良研究。育种材料是进行选育的根本所在,本研究中仅收集了日本群体的后代,如果想要获得持续、稳步的遗传进展还应收集中国不同海域的自然资源或继续引进韩国、朝鲜等外来资源,不断增加基础群体的遗传变异,才能为尽快培育出满足养殖需求的新品种奠定坚实基础。
参考文献:
[1]孟庆闻,苏锦祥,缪学祖.鱼类分类学[M].北京:中国农业出版社,1995:970.
[2]卢敏德,葛志亮.对我国4种东方鲀营养价值的评价[J].中国水产科学,1999,6(4):90-94.
[3]Aparicio S,Chapman J,Stupka E,et al.Whole-genome shotgun assembly and analysis of the genome ofFugurubripes[J].Science,2002,297:1301-1310.
[4]刘秀云,康大海,孙效文,等.红鳍东方鲀F1代混养群体的基因型鉴定及QTL分析[J].水产科学,2008,27(12):641-644.
[5]孙绪文,王群,颜显辉,等.土霉素在红鳍东方鲀体内的残留及休药期研究[J].渔业科学进展,2009,30(6):75-80.
[6]陆丽君.红鳍东方鲀国内主要养殖区养殖群体遗传多样性研究及与性别相关微卫星标记筛选[D].上海:上海海洋大学,2012.
[7]王新安,马爱军,庄志猛,等.红鳍东方鲀(Takifugurubripes)形态性状对体重的影响效果[J].海洋与湖沼,2013,44(1):135-140.
[8]王新安,马爱军,庄志猛,等.红鳍东方鲀体型性状选育指标的综合判定[J].渔业科学进展,2012,33(6):10-15.
[9]姜志强,何旭颖,王冰,等.不同家系红鳍东方鲀形态差异研究[J].现代农业科技,2012,17(1):255-257.
[10]万玉美,王蕾,谭照君,等.红鳍东方鲀两个群体的遗传结构及与经济性状的相关性分析[J].淡水渔业,2011,41(5):9-16.
[11]陈艳翠,高天翔,陈四清,等.红鳍东方鲀养殖群体生化遗传变异初步研究[J].海洋水产研究,2007,28(4):14-20.
[12]郝君,孙效文,孟雪松.红鳍东方鲀微卫星DNA多态性初步分析[J].上海水产大学学报,2006,15(1):21-24.
[13]崔建洲,申雪艳,杨官品,等.红鳍东方鲀基因组微卫星特征分析[J].中国海洋大学学报,2006,36(2):249-254.
[14]郝君,孙效文,孟雪松.红鳍东方鲀BAC数据库和ESTs数据库中微卫星的筛选与应用[J].大连水产学院学报,2007,22(2):97-101.
[15]万玉美,赵海涛,张福崇.红鳍东方鲀、菊黄东方鲀及其杂交F1幼鱼耗氧率与窒息点研究[J].水产科学,2013,32(1):21-25.
[16]范文涛,刘海金,赵文江,等.菊黄东方鲀♀× 红鳍东方鲀♂杂交后代早期形态特征及生长速度的比较[J].水产学报,2011,35(7):1065-1071.
[17]谷晶晶,朱迎军,孟雪松,等.微卫星标记对红鳍东方鲀繁殖的指导应用及遗传分析[J].水产科学,2010,29(9):527-531.
[18]马晓冰,王桂兴,刘海金,等.秦皇岛海域野生牙鲆群体遗传多样性分析[J].中国水产科学,2012,19(6):963-969.
[19]侯仕营,马爱军,王新安,等.大菱鲆4个引进地理群体遗传多样性的微卫星分析[J].渔业科学进展,2011,32(1):16-23.
[20]王婷婷,宋学宏,许爱国,等.应用微卫星标记分析4个黄颡鱼群体的遗传多样性[J].江苏农业科学,2012,40(4):41-45.
[21]陈淑吟,徐士霞,张志勇,等.大黄鱼野生群体与养殖群体遗传多样性研究[J].海洋科学,2011,35(12):82-87.
[22]刘海情,郭昱嵩,王中铎,等.利用微卫星分析吉富罗非鱼群体的遗传多样性[J].南京农业学报,2012,43(1):94-98.
[23]陈省平,丁少雄,陈嘉慧,等.赤点石斑鱼群体遗传结构的微卫星分析[J].中山大学学报,2012,51(3):83-89.
[24]古川聪史.红鳍东方鲀与高生长性状有关的遗传学及分子生物学的研究[D].东京:东京大学,2009.
[25]Raymond M,Rousset F.GENEPOP (version 1.2):population genetics software for exact tests and ecumenicism[J].Journal of Heredity,1995,86(3):248.
[26]Nei M.Analysis of gene diversity in subdivided populations[J].Proceedings of the National Academy of Sciences of the United States of America,1973,70(12):3321-3323.
[27]Excoffier L,Laval L G,Schneider S.Arlequin ver 3.0:an integrated software package for population genetics data analysis[J/OL].Evolutionary Bioinformatics Online,2005(1):47-50.
[28]Excoffier L,Smouse P,Quattro J.Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondrial DNA restriction data[J].Genetics,1992,131(2):479-491.
[29]李建林,唐永凯,李红霞,等.6个建鲤家系的遗传结构及不同亲缘关系个体间的遗传差异分析[J].大连海洋大学学报,2013,28(2):166-170.
[30]曾聪,张耀,曹小娟,等.团头鲂3个地理种群杂交效果的配合力和微卫星标记预测[J].水产学报,2012,36(6):809-814.
[31]王炳谦,谷伟,高会江,等.利用配合力和微卫星标记预测虹鳟品系间的杂交优势[J].中国水产科学,2009,16(2):206-213.
[32]陈灵芝.中国的生物多样性现状及其保护对策[M].北京:科学出版社,1993:93-113.
[33]鲁翠云,曹顶臣,孙效文,等.微卫星分子标记辅助镜鲤家系构建[J].中国水产科学,2008,15(6):893-901.
[34]李明爽,洪脱,龚永生,等.杂种优势预测研究进展[J].中国农学通报,2008,24(1):117-122.
[35]陈平华,张辉,陈如凯.利用RAPD 技术进行甘蔗育种亲本辅助选择[J].分子植物育种,2004,2(5):675-681.
[36]高世斌,荣廷昭,李晚忱,等.19 个玉米自交系的数量性状和AFLP遗传差异比较研究[J].华北农学报,2004,19(2):24-27.
[37]李伯群,余国东,石有明,等.两系杂交小麦杂种优势、配合力及遗传距离分析[J].麦类作物学报,2003,23(3):17-21.
Application of microsatellite markers in parental apolegamy
of redfin pufferTakifugurubripes
LIU Yong-xin1, LIU Yi2, ZHOU Qin3, ZHANG Hong-tao3
(1.Chinese Academy of Fishery Sciences, Beijing 100141, China; 2.Pearl River Fisheries Research Institute, Chinese Academy of Fishery Sciences, Guangzhou 510380, China; 3.Beidaihe Central Experiment Station, Chinese Academy of Fishery Sciences, Qinhuangdao 066100, China)
Abstract:A family mating plan of redfin puffer Takifugu rubripes was established by parental apolegamy assisted by microsatellite markers and by analysis of genetic variations and distances of parental populations, and genetic evaluation was carried out in two populations of the redfin puffer by a set of 34 microsatellite markers. The results showed that there were significantly higher allele number (6.647 0) and Nei’s gene diversity index (0.711 5) in population A than those (allele number 4.647 1 and Nei’s gene diversity index 0.646 1) in population B, significant genetic differences between the two populations (P<0.05). The genetic differentiation coefficient GSTof 0.050 7 and Nmof 4.679 6 indicated that there were inferior differentiation and some degree of gene exchange among the redfin puffer populations. AMOVA revealed that there was 78.49% variation within the population while there was 21.51% variation between the two populations. The UPGMA tree established by genetic distance indicated that the genetic distance (GD) among individuals within the population was varied from 0.11 to 0.82. All individuals were divided into different branches on basis of genetic distance magnitude. Each genetic parameter demonstrated that there were genetic variations to some extent in experimental population and distant relatives among individuals. The breeding schedule can be made according to the genetic distances among individuals, and then families can be produced for selective breeding. The findings indicate that the method is feasible for establishment of families in redfin puffer with microsatellite marker information.
Key words:Takifugu rubripes; population; microsatellite; genetic distance
通信作者:周勤(1963—),高级工程师。E-mail:laozhou529@hotmail.com
作者简介:刘永新(1979—),博士,副研究员。E-mail:liuyx@cafs.ac.cn
基金项目:国家“十二五”科技支撑计划项目(2012BAD26B01);中国水产科学研究院基本科研业务费项目(2013A0505)
收稿日期:2014-06-27
中图分类号:S917
文献标志码:A
文章编号:2095-1388(2015)02-0113-07
DOI:10.3969/J.ISSN.2095-1388.2015.02.001

