肺鳞癌差异性长链非编码RNA表达谱分析及初步验证
潘钦石,陈坚,许刚,丁鸿燕,黄卡特,王瑜敏
(温州医科大学附属第一医院,浙江 温州 325015,1.医学检验中心;2.病理科)
·论 著·
肺鳞癌差异性长链非编码RNA表达谱分析及初步验证
潘钦石1,陈坚1,许刚1,丁鸿燕1,黄卡特2,王瑜敏1
(温州医科大学附属第一医院,浙江 温州 325015,1.医学检验中心;2.病理科)
目的:探讨及分析肺鳞癌差异性长链非编码RNA(lncRNA)的表达谱并进行初步验证。方法:采用Arraystar高通量lncRNAs基因芯片研究20例肺鳞癌混合组织及对应的癌旁组织,获得lncRNAs差异性表达谱。同时采用实时荧光定量PCR方法对40例肺鳞癌相关的lncRNA分子进行验证。结果:本实验所用的基因芯片包含30 586个lncRNAs和26 109个mRNAs探针;与癌旁组织比较肺鳞癌混合组织出现了1 741条lncRNAs(≥2或≤0.5倍差异)和1 716条mRNAs(≥2或≤0.5倍差异)差异性表达;信号通路分析显示了P53信号通路、细胞周期等发生明显变化。我们初步证实了10个lncRNAs分子在肺鳞癌组织中的表达与基因芯片一致,其中KRT16P2、BC016831是表达差异最为明显的lncRNAs分子。结论:肺鳞癌存在明显的lncRNAs差异性表达,这些lncRNAs及某些信号通路可能在肺鳞癌发生和发展中发挥重要作用。
肺肿瘤;癌,非小细胞肺;长链非编码RNA;基因芯片
根据《2012中国肿瘤登记年报》,2009年中国肺癌的发病率和病死率分列男性肿瘤首位,女性肿瘤第二位和第一位[1]。尽管手术切除术、化学治疗和放射治疗技术在不断改进,但肺癌仍极易复发和致命[2]。非小细胞肺癌(non-small cell lung cancer,NSCLC)在肺癌中最为多见,约占肺癌总数的80%。肺鳞癌是属于NSCLC中的一种常见的类型。目前对于NSCLC治疗的整体效果仍很不理想,5年生存率只有10%~15%[3-4]。
长链非编码RNA(long non-coding RNA,lncRNA)为转录本长度大于200核苷酸的RNA分子,不编码蛋白,存在于核内或胞浆内[5]。其表达异常能够引起一系列疾病包括肿瘤的发生发展[6]。研究表明lncRNAs参与了肺癌的发生发展过程[7],为研究肺癌的生物学行为和发病机制提供了重要契机。目前国内外学者对lncRNAs在非小细胞肺癌中的研究尚处于起步阶段,仅筛选及鉴定部分与肺癌相关的lncRNAs[8-11]。但与肺癌相关lncRNAs有待于进一步深入挖掘、研究及阐明其作用机制。
在本项研究中,我们比较了肺鳞癌混合组织及对应的癌旁组织的lncRNAs表达,获得了差异性lncRNAs表达谱并进行深入分析。
1 材料和方法
1.1 病例选择及标本收集 收集本院从2013年1月至2013年12月肺鳞癌组织60例及对应的癌旁组织标本,其中男32例,女28例,年龄34~81岁。所有病例均经过病理学检查确诊。标本选用本院心胸外科手术切除并经病理确诊且临床资料完整的肺鳞癌组织标本,同时取癌旁组织(距病变组织5 cm以上取材,均经病理证实为正常肺组织);置-196 ℃液氮中保存。其中20例肺鳞癌组织及对应的癌旁组织用于lncRNAs基因芯片检测,另外40例肺鳞癌组织及对应的癌旁组织用于SYBR Green I实时荧光定量反转录PCR(reverse transcription-PCR,RTPCR)验证。所有标本留取均通过患者或患者家属知情同意及本院医学伦理委员会批准。
1.2 组织RNA的提取 Trizol试剂盒一步法分离提取总RNA按照Trizol试剂盒(Invitrogen Life Technologies公司,美国)说明书步骤进行。将2 μ L RNA溶解于98 μ L DEPC水中,用紫外分光光度计测定其在波长260 nm和280 nm处吸光度值(OD260和OD280),OD260/OD280比值在1.6~2.0之间,表示RNA样品纯净。以下式计算样品中的RNA浓度:RNA(μ g/mL)=OD260值×40 μ g/mL×稀释倍数。以1.2%琼脂糖凝胶电泳检查RNA的完整性,剩余RNA置于-70 ℃冰箱内保存。
1.3 lncRNAs基因芯片检测及分析 将20例的肺鳞癌组织RNA进行混合为肺鳞癌组,对应的癌旁组织RNA进行混合为癌旁组;lncRNAs检测采用微阵列分析,采用安捷伦阵列平台,即人类lncRNAs阵列V3.0(8×60K,Arraystar),包含30 586 lncRNAs和26 109 mRNAs的探针;这些lncRNAs是来自最备受尊崇的公共转录组数据库(RefSeq,UCSC knowngenes,Gencode等)及著名出版物。由安捷伦扫描仪G2505C.Agilent特征提取软件(11.0.1.1版)扫描来分析获得的芯片阵列图像。分位数标准化和后续的数据处理采用GeneSpring GX V12.0软件包(安捷伦科技)。该基因芯片检测由中国上海康成生物科技公司完成。
1.4 功能化组的分析 GO分析是根据基因本体论(www.geneontology.org),包括三个结构化网络定义的术语,用来描述基因产物属性,P≤0.05表示有意义。根据最新KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库,我们分析差异表达mRNAs的信号通路,P≤0.05表示有意义。
1.5 候选lncRNAs基因的RT-PCR验证 综合差异倍数、基因位点比对、邻近编码基因信息分析等多重筛选条件,初步确定了10个感兴趣lncRNAs候选基因,采用SYBR Green I RT-PCR方法检测40例肺鳞癌及对应癌旁组织,以验证这些lncRNAs分子是否与芯片结果一致。具体操作为将步骤1.2的RNA进一步反转录成cDNA,采用SYBR Green I RT-PCR方法扩增目的片段并实时检测产物的荧光强度,内参使用人甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)基因;根据2-△△CT公式来计算出这些lncRNAs分子在肺鳞癌组织中的相对表达量。
1.6 统计学处理方法 应用SPSS 17.0统计学软件对实验数据进行处理。两两比较采用Mann-Whitney U检验。P<0.05为差异有统计学意义。
2 结果
2.1 lncRNAs芯片结果 通过高通量lncRNAs芯片对肺鳞癌组及癌旁组标本进行检测,差异倍数≥2.0者被认为存在差异性表达。结果显示共检测出差异性表达lncRNAs 1 741条,其中上调lncRNAs 858条,下调lncRNAs 883条;我们对三种lncRNAs亚类进一步进行分析,结果如下所示(见图1-3,表1-2)。

图1 经标准化处理后肺鳞癌组与癌旁组芯片的基础荧光信号强度一致

图2 肺鳞癌组与癌旁组差异性lncRNAs表达谱差异呈正相关

图3 肺鳞癌组及癌旁组lncRNAs分层聚类
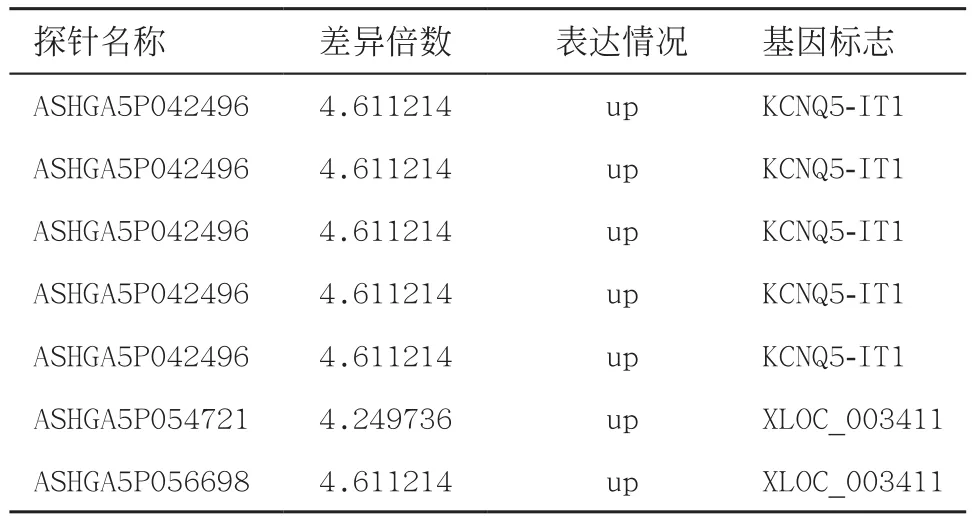
表1 肺鳞癌组差异性表达上调的部分lncRNAs
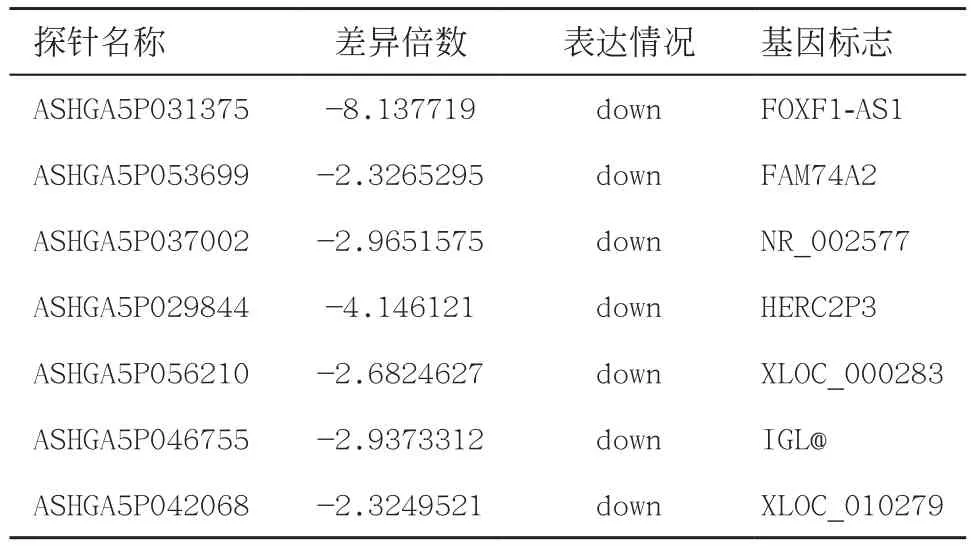
表2 肺鳞癌组差异性表达下调的部分lncRNAs
2.1.1 lncRNAs差异性表达谱研究:结果显示与癌旁组比较,肺鳞癌组差异性表达lncRNAs有343条,其中上调的lncRNAs有167条,下调的lncRNAs有176条;同时也获得lncRNAs所对应可能调控的临近编码基因的数据。部分lncRNAs及邻近调控编码基因数据如下所示(见表3)。

表3 肺鳞癌组差异性表达的部分lncRNAs及邻近调控编码基因
2.1.2 增强样lncRNAs差异性表达谱研究:结果显示与癌旁组比较,肺鳞癌组共有48条增强样lncRNAs差异性表达,其中10条增强样lncRNAs出现上调,另外38条增强样lncRNAs出现下调。
2.1.3 盒基因簇(hox gene cluste,HOX)集群分析:包含了针对lncRNAs和mRNAs的4个HOX基因座的所有探针表达数据,该数据表包含了HOXC11、HOXB-AS3、HOXA2、HOXB-AS3等83个HOX集群。
2.2 差异性表达mRNAs结果 同样采用该高通量芯片对肺鳞癌组及癌旁组标本进行检测,差异倍数≥2.0者被认为存在差异表达。结果显示共检测到差异性表达mRNAs 1 716条,其中上调mRNAs 982条,下调mRNAs 734条。
2.3 GO分析结果 下调基因涉及Biological process的有228个基因,涉及Cellular component的基因55个,涉及Molecular function的基因72个。上调基因涉及Biological process的有573个基因,涉及Cellular component的基因99个,涉及Molecular function的基因119个。
2.4 通路分析结果 上调通路有34个,涉及到P53信号通路、细胞周期等(见图4)。而下调通路有35个,涉及到链球菌感染、细胞黏附分子等(见图5)。
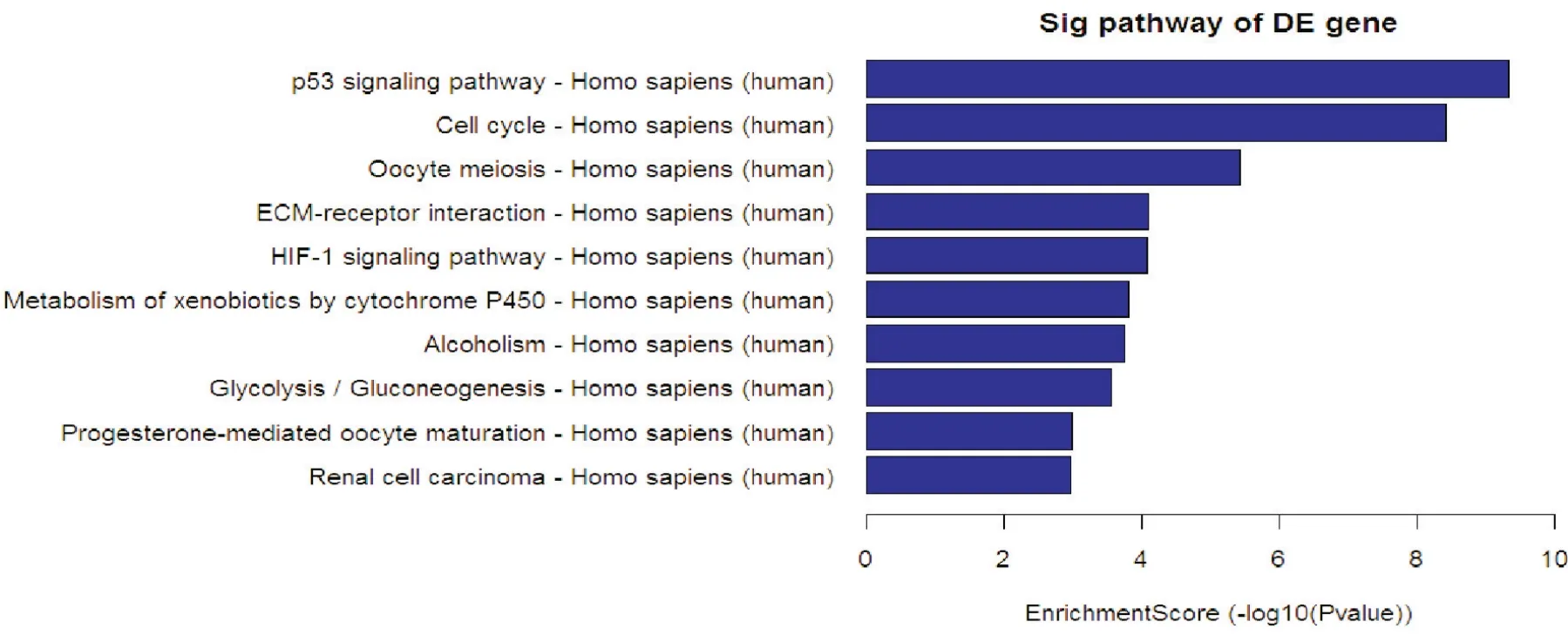
图4 肺鳞癌组差异性表达上调的mRNAs通路分析

图5 肺鳞癌组差异性表达下调的mRNAs通路分析
2.5 SYBR Green I RT-PCR验证结果 初步筛选出10个lncRNAs分子,包括RP11-385J1.2、KRT16P2、CTD-2194D22.3、AC025627.9、AC006050.3、BC016831、Z82214.3、XIST、uc.468、FOXF1-AS1,我们采用SYBR Green I RT-PCR检测发现这些lncRNAs分子在基因芯片和组织表达水平基本一致(见图6)。其中KRT16P2、BC016831为差异性表达最明显的lncRNAs分子;KRT16P2在肺鳞癌组织的表达水平明显高于癌旁组织(Mann-Whitney U=148.12,P= 0.017),而BC016831在肺鳞癌组织的表达水平明显低于癌旁组织(Mann-Whitney U=151.31,P=0.009),两者差异均有统计学意义(见图7)。

图6 10个肺鳞癌相关lnRNAs分子RT-PCR的验证结果
3 讨论
LncRNAs在许多生物过程中发挥重要作用,包括X染色体失活、基因印记、干细胞维护等,但它们的作用机制并不相同[12-13]。很多证据已经确认lncRNAs是在癌症的基因表达中最重要的控制因素[14]。研究表明,lncRNAs在肿瘤的发生和发展中发挥非常重要的作用[15]。lncRNAs差异地表达于正常细胞和肿瘤细胞,因为lncRNAs是非常重要的一类基因表达调控因子,这将不可避免地导致基因表达和肿瘤发生的异常表达水平。

图7 KRT16P2、BC016831在40例肺鳞癌组织及癌旁组织的表达水平比较
本研究发现,与癌旁组织比较,肺鳞癌组织出现了858条lncRNAs上调和883条lncRNAs下调,显示了肺鳞癌存在一系列lncRNAs表达异常,这表明了lncRNAs在肺鳞癌的发病机制中具有潜在作用。我们进一步对lncRNAs不同亚类进行分析,结果显示与癌旁组织比较,肺鳞癌组织出现167条上调的lncRNAs和176条下调的lncRNAs异常表达,10条上调增强样lncRNAs和38条下调增强样lncRNAs异常表达。此外我们的数据还包括针对lncRNAs和编码基因转录的HOX集群分析。
GO分析表明上调mRNAs的数量明显多于下调的mRNAs。通路分析显示有34个上调信号途径(包括P53信号通路、细胞周期等)和35个表达下调途径(包括链球菌感染、细胞黏附分子等),这些途径可能在肺鳞癌的发生和发展中发挥重要的作用。针对如此海量lncRNAs数据,我们结合多重条件初步筛选出10个感兴趣的lncRNAs分子,包括RP11-385J1.2、KRT16P2、CTD-2194D22.3、AC025627.9、AC006050.3、BC016831、Z82214.3、XIST、uc.468、FOXF1-AS1,我们进一步采用SYBR Green I RT-PCR检测发现这些lncRNAs分子在基因芯片和组织中表达水平基本一致。其中KRT16P2、BC016831在肺鳞癌组织表达差异最为明显,肺鳞癌组织KRT16P2表达水平明显高于癌旁组织,而BC016831在肺鳞癌组织表达水平明显低于癌旁组织。我们后续将对KRT16P2、BC016831在肺鳞癌中的分子生物学功能及分子机制进一步进行研究。
总之,我们的研究结果显示,与正常肺组织比较,肺鳞癌出现明显lncRNAs异常表达;另外一些信号传导途径可能参与肺鳞癌的发生和发展过程。这些可能为肺鳞癌的研究和治疗提供新的生物学标志物或新的作用靶点。
[1] 郝捷, 赵平, 陈万青. 2012中国肿瘤登记年报[M]. 北京: 军事医学科学出版社, 2012: 216.
[2] Gridelli C, Rossi A, Maione P. Treatment of non-smallcell lung cancer:state of the art and development of new biologic agents[J]. Oncogene, 2003, 22(42): 6629-6638.
[3] Martinet Y, Hirsch FR, Martinet N, et al. Clinical and biological basis of lung cancer prevention[M]. Basel: Birka¨user, 1998: 297-304.
[4] Custodio A, Méndez M, Provencio M. Targeted therapies for advanced non-small-cell lung cancer: current status and future implications[J]. Cancer Treat Rev, 2012, 38(1): 36-53.
[5] Ponting CP, Oliver PL, Reik W. Evolution and functions of long noncoding RNAs[J]. Cell, 2009, 136(4): 629-641.
[6] Wapinski O, Chang HY. Long noncoding RNAs and human disease[J]. Trends Cell Biol, 2011, 21(6): 354-361.
[7] Enfield KS, Pikor LA, Martinez VD, et al. Mechanistic roles of noncoding RNAs in lung cancer biology and their clinical implications[J]. Genet Res Int, 2012, 2012: 737416.
[8] Thai P, Statt S, Chen CH, et al. Characterization of a novel long noncoding RNA, SCAL1, induced by cigarette smoke and elevated in lung cancer cell lines[J]. Am J Respir Cell Mol Biol, 2013, 49(2): 204-211.
[9] Yang Y, Li H, Hou S, et al. The noncoding RNA expression profle and the effect of lncRNA AK126698 on cisplatin resistance in non-small-cell lung cancer cell[J]. PLoS One, 2013, 8(5): e65309.
[10] Han L, Kong R, Yin DD, et al. Low expression of long noncoding RNA GAS6-AS1 predicts a poor prognosis in patients with NSCLC[J]. Med Oncol, 2013, 30(4): 694.
[11] Gutschner T, Hämmerle M, Eissmann M, et al. The noncoding RNA MALAT1 is a critical regulator of the metastasis phenotype of lung cancer cells[J]. Cancer Res, 2013, 73(3): 1180-1189.
[12] Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs: insights into functions[J]. Nat Rev Genet, 2009, 10(3): 155-159.
[13] Wang KC, Chang HY. Molecular mechanisms of long noncoding RNAs[J]. Mol Cell, 2011, 43(6): 904-914.
[14] Khachane AN, Harrison PM. Mining mammalian transcript data for functional long non-coding RNAs[J]. PLoS one, 2010, 5(4): e10316.
[15] 李琪儿, 叶国良, 郭俊明. lncRNA: 肿瘤分子诊断中的一颗新星[J]. 中国生物化学与分子生物学报, 2014, 30(3): 233-240.
(本文编辑:吴健敏)
LncRNAs expression profiles of lung squamous cell carcinoma revealed by microarray and valida-tion
PAN Qinshi, CHEN Jian, XU Gang, DING Hongyan, HUANG Kate, WANG Yumin.
1.Center for Clinical Laboratory, the First Affiliated Hospital of Wenzhou Medical University, Wenzhou, 325015; 2.Department of Pathology, the First Affiliated Hospital of Wenzhou Medical University, Wenzhou, 325015
Objective: To analyze lncRNA expression profles in lung squamous cell carcinoma and prelimiary validate. Methods: LncRNAs and mRNAs expression profles in lung squamous cell carcinoma samples compared with NT samples were studied by high-throughput microarray. The interesting candidate lncRNAs were verifed by real-time quantitative PCR. Results: Using abundant and varied probes, 30 586 lncRNAs and 26 109 coding transcripts were able to assess in our microarray. From the data we found there were thousands of lncRNAs and mRNAs that differentially expressed ($2 fold-change) and 1 741 lncRNAs and 1 716 mRNAs differentially expressed in lung squamous cell carcinoma samples compared with NT. P53, cell cycle, streptococcal infection, cell adhesion molecules signaling pathways may be involved in the pathogenesis of the lung squamous cell carcinoma. We also found, via quantitative PCR, that 10 lncRNAs were aberrantly expressed in lung squamous cell carcinoma compared with matched histologically normal lung tissues. Among these, KRT16P2 and BC016831 were the most aberrantly expressed lncRNAs, as estimated by quantitative PCR in 40 pairs of lung squamous cell carcinoma and NT samples. Conclusion: The results reveal lncRNAs expression patterns in lung squamous cell carcinoma by microarray. These lncRNAs and some pathways may play a key role in tumor development and occurrence.
lung neoplasms; carcinoma, non-small-cell lung; lncRNAs; microarray
R446.8
ADOI: 10.3969/j.issn.2095-9400.2015.03.001
2014-10-14
国家自然科学基金资助项目(81401736);温州市科技局科研基金资助项目(Y20110041,Y20130170,Y20140024,Y20140032)。
潘钦石(1977-),男,浙江苍南人,主管技师。
王瑜敏,副主任技师,Email:wym0577@163.com。

