外显子组测序对1例青壮年猝死的分子遗传学分析
王纯,王辉,许心舒,许传超,赖小平,陈锐,林汉光,邱升元
(1.成都体育学院,四川成都610041;2.许昌市公安局刑事科学技术研究所,河南许昌 461002;3.广州市刑事科学技术研究所,广东广州 510030;4.广东医学院,广东东莞 523808)
外显子组测序对1例青壮年猝死的分子遗传学分析
王纯1,王辉2,许心舒3,许传超4,赖小平4,陈锐4,林汉光4,邱升元4
(1.成都体育学院,四川成都610041;2.许昌市公安局刑事科学技术研究所,河南许昌 461002;3.广州市刑事科学技术研究所,广东广州 510030;4.广东医学院,广东东莞 523808)
目的以1例青壮年不明原因猝死综合征(sudden unexplained death syndrome,SUDS)案例为研究对象,采用全外显子组测序技术,在全外显子组水平寻找与SUDS相关的致病基因突变。方法对1例常规尸体解剖及病理学检验未发现明显致死性病理改变的SUDS病例样本,利用Ion Torrent PGMTM系统进行全外显子组测序。测序数据以hg19为参照序列,并通过PhyloP、PolyPhen2、SIFT等软件进行突变功能分析。最后设置三重条件过滤筛选有意义的单核苷酸变异:选取错义突变-等位基因频率<1%-蛋白质功能预测。结果共发现4个罕见的可疑致病性单核苷酸变异。结合尸体解剖及病理学检验的结果,确定1个“高危害性”突变MYOM2(8_2054058_G/A)。PolyPhen2、SIFT的预测均为“有害”。结论利用二代测序技术进行全外显子组水平的基因突变检测和分析,可以为SUDS病例的死因分析提供新的方法和思路。MYOM2基因新可能是SUDS的致病基因,但其具体机制仍需进一步研究。
法医遗传学;法医病理学;突变;不明原因猝死综合征;全外显子组测序
心源性猝死(sudden cardiac death,SCD)是目前最常见的死亡原因之一。据不完全统计,我国的SCD发病率约为41.8/100000[1],以此推算每年发生在我国的SCD病例约50万。大多数SCD病例在随后的解剖及病理检验中可发现冠心病、心肌炎等明确的病理改变。但有部分病例,在解剖及病理学检查中,均不能发现明显的致死性形态学改变,即解剖阴性的不明原因猝死(conventional autopsy-negative sudden unexplained death),这类病例多见于青壮年群体和儿童(0~35岁),因此也称作青壮年不明原因猝死综合征
(sudden unexplained death syndrome,SUDS)[2]。
近年来,研究者发现SUDS的发病特点与一些特殊类型的心脏疾病——离子通道病/心肌病(channelopathies/cardiomyopathies)[3-4]的特点相似,如患者通常表现“健康”,不知道自己的病情,心源性猝死常为首发症状。离子通道病/心肌病是多种疾病的总称,包括长QT综合征(long QT syndrome,LQTS)、Brugada综合征(Brugada syndrome,Brugada S)、儿茶酚胺敏感性多形性室速(catecholaminergic polymorphic ventricular tachycardia,CPVT)等心肌离子通道病[5-7],以及肥厚型心肌病(hypertrophic cardiomyopathy,HCM)、扩张型心肌病(dilated cardiomyopathy,DCM)、致心律失常型右心室心肌病(arrthythmogenic right ventricular cardiomyopathy,ARVC)等心肌病[8-10]。随着分子遗传学研究的深入,研究者发现这类疾病都具有一定的遗传背景,分子遗传学检测和分析成为对此类疾病进行明确诊断的必要手段。
与临床诊断明确的各种离子通道病/心肌病不同,法医面临的SUDS常缺乏生前的临床检查资料,而常规的解剖和病理学检验亦难以发现明确的病理改变。因此,在无法从病理学层面明确死者的心肌病类型时,进行分子遗传学检测时也无法明确待检测的目标基因范围。由于与SUDS相关的致病基因多达近百个[3,11],仅与HCM相关的致病基因就有数十个,涉及的突变类型多达上千种[12]。如果利用传统分子生物学技术对这些基因进行突变筛查,无疑是个成本极高且费时费力的工作。因此,SUDS的遗传学检验至今未能普及。
外显子组测序技术[13]是以二代测序技术为平台的高通量基因组分析技术,利用外显子序列特异性捕获技术将基因组全部外显子区域DNA捕捉并富集后进行的高通量测序分析,一轮检测即可得到一份样本外显子组序列信息,有效数据可以涵盖目标区域95%以上的序列。随着二代测序技术的成本逐渐降低[14-15],外显子组测序有望成为最有效的SNP及基因突变的检测手段。本研究利用外显子组二代测序技术对1例SUDS进行遗传学检测,以期发现其可疑致病突变基因,为死因不明、解剖阴性案例的法医病理学诊断探索新途径。
1 材料与方法
1.1 研究对象
猝死病例1例,男性,19岁。运动后突然倒地死亡,尸体解剖未发现可以解释其死亡原因的典型形态学改变,并通过问其亲属已排除家族遗传病史。
1.2 解剖及病理学检验
死者经系统的法医学尸体解剖,并取心脏、大脑、肝、肾、肺等主要器官行组织病理学检验。取心血进行常见毒物和药物检验。
1.3 外显子组测序
1.3.1 基因组DNA提取
取尸体骨骼肌组织50mg,利用细胞/组织基因组DNA提取试剂盒(DP1901,北京百泰克公司),提取基因组DNA。
1.3.2 构建样本DNA的外显子文库
按照Ion Torrent PGMTM的TargetSeqTMExome Enrichment操作手册程序,构建样本DNA的外显子文库,主要包括以下步骤:
(1)利用AMPure XP核酸纯化试剂盒及Ion ShearTMPlus Enzyme MixⅡ对提取的基因组DNA进行纯化和片段化(180~220bp);
(2)利用Agencourt AMPure XP磁珠纯化系统对片段化的DNA进行纯化;
(3)对纯化的片段化DNA进行质检(Agilent Bioanalyzer),质检合格后的DNA片段进行Adapters连接和缺口修复及纯化;
(4)利用Ion TargetSeqTMExome试剂盒对纯化的DNA片段库进行PCR扩增,扩增产物经磁珠纯化;
(5)在Agilent 2100 Bioanalyzer平台上,利用Agilent High Sensitivity DNA试剂盒对扩增的DNA库产物进行质控;
(6)外显子靶序列片段的富集:利用Ion TargetSeq Exome Probe Pool、TargetSeq Hybridization Solution A及Hybridization Enhancer B等对通过质控的DNA文库进行外显子靶序列片段的杂交捕获,利用Ion TargetSeq Amplification试剂盒对捕获富集到的外显子文库片段进行PCR扩增,产物进行磁珠纯化;
(7)用Ion PITMTemplate OT2 200试剂盒(No. 4482286)准备引物,检测exome-enriched library的片段长度分布,进行高通量测序前的模板准备(定量、稀释);
(8)用Ion Torrent PGMTM系统,以稀释好的外显子文库为模板,进行高通量平行测序。
以上试剂盒均购自美国Life Technology公司。
1.4 生物信息学分析
将样本外显子测序数据与参照序列(hg19)比对,进行SNP检测和注释、InDel检测与注释等生物信息学分析。再将单核苷酸多态性(SNP)或单核苷酸变异(single nucleotide variants,SNV)等位点与千人基因组计划、dbSNP等标准数据库信息进行比对,确定各SNP或SNV的基因频率。筛选出等位基因频率<1%的SNV,进一步确定发生在猝死相关基因[3,12]范围内
的SNV。
运用在线、生物大分子功能预测软件PolyPhen2和SIFT,以及基因序列保守性分析软件PhyloP和Grantham对外显子组测序结果中检测到的基因突变进行编码产物蛋白质的功能预测分析。
利用sanger法对上述步骤筛选出的基因突变进行测序验证。
2 结果
2.1 法医病理学检验
全身主要器官未发现明显致死性病理改变。心脏质量272g,外膜光滑、表面血管淤血;左右冠状动脉开口及主干无狭窄,主动脉根部内膜光滑;各瓣膜及各瓣口周径未见明显异常;左心室壁厚1.4 cm,右心室壁厚0.4cm,室间隔厚1.2cm。镜下:心外膜疏松,冠脉管壁未见明显异常,大血管空虚,小血管淤血;心肌间隙弥漫性增宽、疏松,心肌纤维弥漫性波浪样不规则扭曲、粗细不均,散在不规则心肌断裂,偶见灶性出血,间质血管淤血,未见明显炎症细胞浸润。常见毒(药)物检验均为阴性。
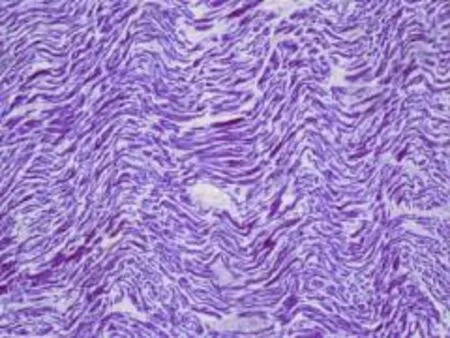
图1 心肌弥漫波浪样变,肌纤维断裂HE×40
2.2 外显子组测序
样本的基因组DNA经过外显子组测序及随后的生物信息学分析,在其基因组内各个外显子及其侧翼部分区域内共检出24230个SNP位点,其中,新发现的SNV位点有590个。
鉴于SUDS病例的发病率极低,且很多为散发病例,因此,本研究将SNV作为可疑致病突变的筛查对象,测序结果见表1。经过错义突变-等位基因频率<1%-蛋白质功能预测三重筛选后,得到4个可疑致病突变位点信息(表2)。

表1 样本外显子测序结果与数据库外显子及临近侧翼区域参考序列的比对
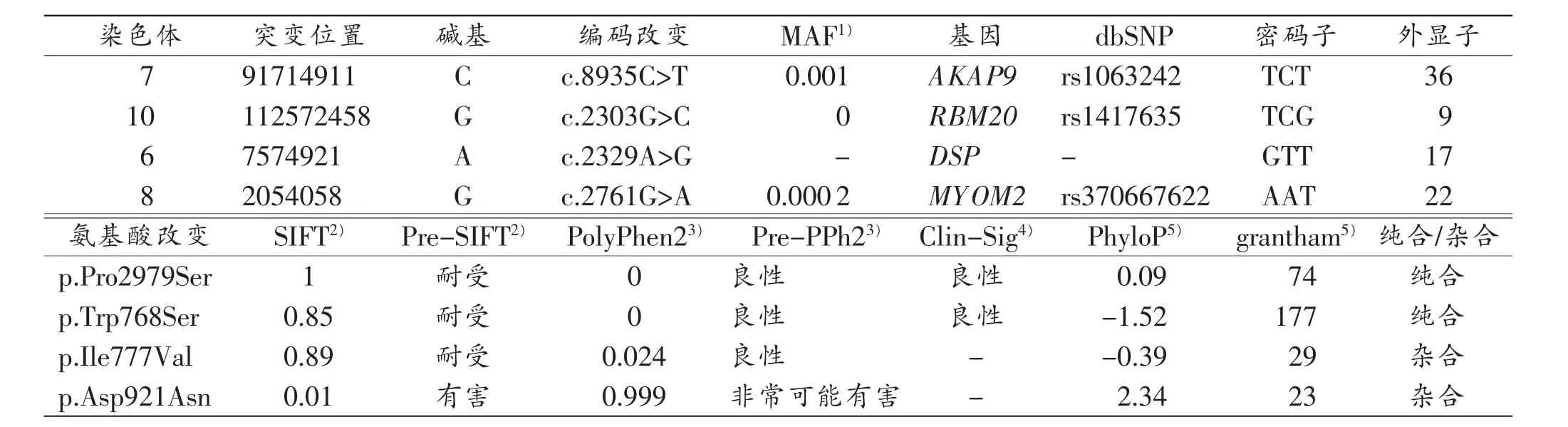
表2 检测样本在SUDS相关基因外显子区域发现的部分SNV(MAF<1%)
2.3 sanger法测序验证
对三重筛选后得到的4个SNV位点中两个新发现的SNV位点进行sanger法测序验证:
(1)8号染色体2054058位点(MYOM2基因)的G2761A的杂合突变;
(2)6号染色体7574921位点(DSP基因)的A2329G杂合突变。

图2 Sanger法对外显子组的验证结果
3 讨论
本例SUDS死者解剖及病理学检验未见各主要器官的致死性疾病和损伤,排除心肌炎、冠心病、先天性心脏病等基础性疾病。心肌弥漫性不规则扭曲、断裂及灶性出血符合急性心功能紊乱改变,脑、心、肺、肝、肾等全身器官高度淤血,符合急性心力衰竭致循环功能衰竭继发改变。死者在疲劳状态下突然倒地,迅速死亡,符合心源性猝死的发病特征。因此,在外显子组测序后的遗传学检验分析,主要的突变分析集中在与心血管系统结构与功能相关的基因范围内[3,12]。
由于本研究检测的样本只有1例(家属因心理压力未能配合提供DNA样本),在缺乏生前临床资料以及了解到无家族遗传背景的情况下,本研究设置了筛选SUDS相关的致病性SNV的条件:(1)选取处于91个基因[3,12]外显子区域的错义突变;(2)由于SUDS发病率低[2],可疑的致病突变应为罕见突变,因此选取等位基因频率<1%的SNV;(3)对第二步筛选到的突变进行dbSNP数据库检索及文献检索,选取新发现的突变和已经报道为致病性的突变;对于新发现的突变,则需要通过软件对其进行蛋白质功能影响预测,如SIFT及PolyPhen2等。
本研究样本中,外显子区域共检出两万多个SNV,在91个与心肌结构和功能相关的基因范围内,共筛选出错义SNV 23个,主要分布于AKAP9、BAG3、CASQ2、DSP、JAG1、MYH7B、MYPN、MYOM1、MYOM2、RBM20、TTN、TXNRD2共12个基因的外显子范围内。经dbSNP数据库信息筛选,排除已经被证实为良性的突变,剩余12个SNV。依据MAF<1%的标准进行筛选,最终得到4个罕见SNV。其中,2个位点(分别位于AKAP9基因和MYH7B基因)为已在dbSNP数据库注释的SNP位点,2个为新发突变(分别位于DSP基因和MYOM2基因)。
第一个新发突变位于心肌细胞M线结构的一种蛋白——M-protein(Myomesin 2)的编码基因MYOM2中。MYOM2基因编码的M-protein(Myomesin 2),相对分子质量165000,由1465个氨基酸组成。是心肌细胞M带的重要组成成分。通过与肌球蛋白、肌联蛋白、轻酶解肌球蛋白等的结合发挥生理功能。本研究发现的MYOM2基因的22号外显子的G2899A突变(rs370667622),使相应的密码子由GAT突变为AAT,最终可以使合成的多肽链上发生Asp921Asn突变。这个突变位于M-protein多肽链高度保守的Ig-like C2-type 3区域。经过SIFT、PolyPhen2的分析预测,该突变的结果为有害。根据以上预测结果,推测这个酸性氨基酸(Asp)被替换成中性(极性)氨基酸(Asn)的变异,将会影响M-protein与Myomesin、Titin等分子的亲和力,这对心肌细胞肌小节结构的稳定性将产生显著影响。在Rozanski等[16]的研究中发现,心肌细胞的M-protein的表达受甲状腺素T3的调控。T3对心肌细胞长期毒性效应的分子机制是T3可下调心肌细胞M-protein的表达,进而严重影响心肌细胞的收缩速度。由此可见,M-protein的正常表达对于心肌细胞正常功能的维持具有关键作用。本研究发现的存在于SUDS病例的M-protein变异,有可能是该病例的死亡原因之一。该突变与心肌细胞功能障碍之间的相关性还有待进一步通过实验来证实。
第二个新发突变是位于DSP基因的A2329G。DSP基因编码桥粒斑蛋白,桥粒斑蛋白是心肌细胞间连接结构-桥粒的主要成分,负责将中间丝锚定在桥粒斑,其N末端负责连接桥粒斑,C末端连接中间丝。DSP基因突变已经被证实与心肌病ARVC相关[17-18]。本研究发现的DSP基因17号外显子的错义突变A2329G,将导致多肽链中的Ile777Val变异。经过SIFT、PolyPhen2等软件的预测分析,结果为良性(benign)。这提示该突变对DSP的转录产物桥粒斑蛋白的功能影响不大。本研究中的SUDS死者的右心室心肌也并未发现如右心室心肌层被纤维脂肪组织取代,心肌间质脂肪浸润等明显的ARVC特征性病理改变,与软件预测相符。
第三个低频率SNV是AKAP9基因的C8935T(rs1063242,p.Pro2979Ser)。AKAP9基因产物Yotiao蛋白属于A激酶锚定蛋白家族的一员。在交感神经系统对心肌细胞动作电位时程进行调控的过程中,
AKAP9(Yotiao蛋白)可通过与KCNQ1钾离子通道蛋白的α亚单位形成复合体而影响钾离子电流Iks。KCNQ1基因突变可导致LQTS[19],Chen等[20]发现AKAP9分子的S1570L突变是一个LQTS家系的发病原因,该突变位于Yotiao蛋白与KCNQ1钾离子通道蛋白结合的区域,进而影响Iks,导致LQTS的临床体征。AKAP9基因的突变体与LQTS1相关,本研究所发现的Pro2979Ser突变位于AKAP9分子C端,不在与钾离子通道的结合区。PhyloP分析突变的位置并非序列保守区域,各种生物学分析结果均为“良性”。因此可初步判断,AKAP9(Yotiao)蛋白的Pro2979Ser突变对KCNQ1钾离子通道的Iks不产生明显影响,因此本SUDS病例死于LQTS的可能性不大。
第四个低频率SNV是RBM20。该基因编码产物通过与RNA结合调节转录产物的剪接修饰,是具有多种功能的基因通过选择性剪接表达出功能多样的蛋白质的重要调节蛋白。Refaat等[21]报道了RBM20参与心肌细胞肌节蛋白Titin的剪接调控,RBM20突变导致的剪接调控异常与DCM与相关。Brauch等[22]首先报道了在这个基因外显子9区域内,在一段高度保守的富含精氨酸/丝氨酸的结构域中发生的几个突变(R636S、R636H和S637G)与家族性扩张型心肌病相关联。Wells等[23]利用WES技术发现了RBM20基因的c.1907 G>A突变与DCM相关。在本例SUDS样本中发现的RBM20基因c.2303G>C(p.Trp768Ser)突变虽然也位于9号外显子,但突变位置(768位氨基酸)据Brauch报道的突变热点区域相距100多个氨基酸,位于保守性较低的区域(PhyloP和Grantham评分均显示此位点保守性较低)。该突变后果为色氨酸(Trp)突变为丝氨酸(Ser),SIFT及PolyPhen2等预测软件显示该突变的危害性非常低。因此我们认为这个RBM20基因的c.2303G>C(p.Trp768Ser)不是本SUDS的主要致病突变。
此外,与Roncarati等[24]的研究提示相同,SUDS相关的各种心肌病有可能不是单基因病,其症状体征更可能是多个“有害”的多态性/突变基因协同作用的结果。这提示,全面检测心肌细胞电生理功能及收缩舒张机械功能相关的各个基因,才能更加完整地解释此类疾病的分子机制。
综上所述,在本例SUDS样本中,通过“等位基因频率<1%的错义突变-心肌细胞表达基因”的三步筛选,得到一个新发现的MYOM2基因的Asp921Asn突变,可能是其猝死的主要分子基础。对于该突变对心肌细胞功能的影响,尚需进一步实验来验证。对于法医学实践中遇到的缺乏详细生前临床资料及家族遗传病信息的猝死病例,外显子组测序技术在鉴定致病突变及发现新突变领域具有不可替代的优势,是猝死案例遗传学检测的有力工具。
[1]Hua W,Zhang LF,Wu YF,et al.Incidence of sudden cardiac death in China:analysis of 4 regional populations[J].J Am Coll Cardiol,2009,54(12):1110-1118.
[2]Lim Z,Gibbs K,Potts JE,et al.A review of sudden unexpected death in the young in British Columbia[J]. Can J Cardiol,2010,26(1):22-26.
[3]Wilde AA,Behr ER.Genetic testing for inherited cardiac disease[J].Nat Rev Cardiol,2013,10(10):571-583.
[4]Ackerman MJ,Marcou CA,Tester DJ.Personalized medicine:genetic diagnosis for inherited cardiomyopathies/channelopathies[J].Rev Esp Cardiol(Engl Ed),2013,66(4):298-307.
[5]Goldenberg I,Moss AJ.Long QT syndrome[J].J Am Coll Cardiol,2008,51(24):2291-2300.
[6]Kaufman ES.Mechanisms and clinical management of inherited channelopathies:long QT syndrome,Brugada syndrome,catecholaminergic polymorphic ventricular tachycardia,and short QT syndrome[J].Heart Rhythm,2009,6(S8):S51-S55.
[7]Yl覿nen K,Poutanen T,Hiippala A,et al.Catecholaminergic polymorphic ventricular tachycardia[J]. Eur J Pediatr,2010,169(5):535-542.
[8]Mohiddin SA,Knight C.Interventional treatments for hypertrophic cardiomyopathy[J].Cardiovasc Ther,2012,30(3):e107-e114.
[9]McNally EM,Golbus JR,Puckelwartz MJ.Genetic mutations and mechanisms in dilated cardiomyopathy[J].J Clin Invest,2013,123(1):19-26.
[10]Marcus FI,Edson S,Towbin JA.Genetics of arrhythmogenic right ventricular cardiomyopathy:a practical guide for physicians[J].J Am Coll Cardiol,2013,61(19):1945-1948.
[11]Loporcaro CG,Tester DJ,Maleszewski JJ,et al. Confirmation of cause and manner of death via a comprehensive cardiac autopsy including whole exome next-generation sequencing[J].Arch Pathol Lab Med,2014,138(8):1083-1089.
[12]Efthimiadis GK,Pagourelias ED,Gossios T,et al. Hypertrophic cardiomyopathy in 2013:Current speculations and future perspectives[J].World J Cardiol,2014,6(2):26-37.
[13]Bick D,Dimmock D.Whole exome and whole genome sequencing[J].Curr Opin Pediatr,2011,23(6):594-600.
[14]Johansen Taber KA,Dickinson BD,Wilson M.The promise and challenges of next-generation genome sequencing for clinical care[J].JAMA Intern Med,2014,174(2):275-280.[15]Rabbani B,Tekin M,Mahdieh N.The promise of whole-exome sequencing in medical genetics[J].J Hum Genet,2014,59(1):5-15.
[16]Rozanski A,Takano AP,Kato PN,et al.M-protein is down-regulated in cardiac hypertrophy driven by thyroid hormone in rats[J].Mol Endocrinol,2013,27(12):2055-2065.
[17]Bauce B,Basso C,Rampazzo A,et al.Clinical profile of four families with arrhythmogenicright ventricular cardiomyopathy caused by dominant desmoplakin mutations[J].Eur Heart J,2005,26(16):1666-1675.
[18]den Haan AD,Tan BY,Zikusoka MN,et al.Comprehensivedesmosomemutationanalysisinnorth americans with arrhythmogenic right ventricular dysplasia/cardiomyopathy[J].Circ Cardiovasc Genet,2009,2(5):428-435.
[19]Herbert E,Trusz-Gluza M,Moric E,et al.KCNQ1 gene mutations and the respective genotype-phenotype correlations in the long QT syndrome[J].Med Sci Monit,2002,8(10):RA240-RA248.
[20]Chen L,Marquardt ML,Tester DJ,et al.Mutation of an A-kinase-anchoring protein causes long-QT syndrome[J].Proc Natl Acad Sci U S A,2007,104(52):20990-20995.
[21]Refaat MM,Lubitz SA,Makino S,et al.Genetic variation in the alternative splicing regulator RBM20 is associated with dilated cardiomyopathy[J].Heart Rhythm,2012,9(3):390-396.
[22]Brauch KM,Karst ML,Herron KJ,et al.Mutations in ribonucleic acid binding protein gene cause familial dilated cardiomyopathy[J].J Am Coll Cardiol,2009,54(10):930-941.
[23]Wells QS,Becker JR,Su YR,et al.Whole exome sequencing identifies a causal RBM20 mutation in a large pedigree with familial dilated cardiomyopathy[J]. Circ Cardiovasc Genet,2013,6(4):317-326.
[24]Roncarati R,Viviani Anselmi C,Krawitz P,et al. Doubly heterozygous LMNA and TTN mutations revealed by exome sequencing in a severe form of dilated cardiomyopathy[J].Eur J Hum Genet,2013,21(10):1105-1111.
(本文编辑:刘宁国)
Molecular Genetic Analysis of One Sudden Unexplained Death in the Young by Whole Exome Sequencing
WANG Chun1,WANG Hui2,XU Xin-shu3,XU Chuan-chao4,LAI Xiao-ping4,CHEN Rui4,LIN Hanguang4,QIU Sheng-yuan4
(1.Chengdu Sport University,Chengdu 610041,China;2.Institute of Criminal Science and Technology, Xuchang Public Security Bureau,Xuchang 461002,China;3.Guangzhou Institute of Criminal Science and Technology,Guangzhou 510030,China;4.Guangdong Medical College,Dongguan 523808,China)
Objective To find the mutation of disease-causing genes of sudden unexplained death syndrome(SUDS)in the young by whole exome sequencing in one case.Methods One SUDS case was found no obvious fatal pathological changes after conventional autopsy and pathological examination.The whole exome sequencing was performed with the Ion Torrent PGMTMSystem with hg19 as
equence for sequencing data.The functions of mutations were analyzed by PhyloP,PolyPhen2 and SIFT. A three-step bioinformatics filtering procedure was carried out to identify possible significative single nucleotide variation(SNV),which was missense mutation with allele frequency<1%of myocardial cell. Results Four rare suspicious pathogenic SNV were identified.Combined with the analysis of conventional autopsy and pathological examination,the mutation MYOM2(8_2054058_G/A)was assessed as highrisk deleterious mutation by PolyPhen2 and SIFT,respectively.Conclusion Based on the second generation sequencing technology,analysis of whole exome sequencing can be a new method for the death cause investigation of SUDS.The gene MYOM2 is a new candidate SUDS pathogenic gene for mechanism research.
forensic genetics;forensic pathology;mutation;sudden unexplained death syndrome;whole exome sequencing
DF795.2
A
10.3969/j.issn.1004-5619.2015.06.006
1004-5619(2015)06-0436-05
2011年四川省科技基础条件平台项目;广东省科技计划项目(2011B080701097)
王纯(1961—),女,教授,主要从事运动性猝死和运动医学研究;E-mail:993279039@qq.com
许传超,男,博士,讲师,主要从事分子遗传学研究;E-mail:xcchao5855@sina.com

