利用外转录间隔区对中国人参和西洋参进行分子鉴定
邓菁,张甜甜,王迪,金海珠,王洪涛
(烟台大学生命科学学院,山东烟台 264005)
利用外转录间隔区对中国人参和西洋参进行分子鉴定
邓菁,张甜甜,王迪,金海珠,王洪涛*
(烟台大学生命科学学院,山东烟台 264005)
人参和西洋参是我国两种重要的中药材。两者虽然形态相似,但药性差异较大。为了建立一种简单高效的中国人参和西洋参的分子鉴别方法,作者利用从核DNA的外转录间隔(ETS)发掘的人参和西洋参单核苷酸多态性位点(SNP),分别设计了人参和西洋参的等位基因特异性引物,并利用多重PCR对人参和西洋参进行了鉴定。结果表明,作者建立的多重PCR体系可以有效地对人参、西洋参及两者的混合品进行鉴定。
人参;西洋参;外转录间隔区;单核苷酸多态性
人参为多年生五加科草本植物(Panax ginseng)的根,是我国最重要的中药材之一。2012年9月4日,卫生部正式批准人工种植的人参为新资源食品,极大地推动了人参产业的发展。西洋参为五加科植物(Panax quinquefolius)的根,近年来在我国大量种植。两者虽然形态相似,但药性差异较大。人参性温,补气偏于助阳;而西洋参性凉,补气偏于养阴[1]。因此人参和西洋参的适用人群有异,混淆使用可能会适得其反。虽然目前市场上人参和西洋参价格差别不大,但由于西洋参总皂苷含量普遍高于人参[2],因此用西洋参冒充人参作为产品原料的情况较为多见。因此,为了保证人参和西洋参在临床上的正确使用,亟需建立一种对人参和西洋参进行快速有效地鉴别方法。
近年来,针对人参和西洋参的鉴别,国内外学者开展了大量的研究工作。根据人参和西洋参分别所含有的特征皂苷Rf和24(R)—拟人参皂苷F11,高效液相色谱和质谱被用于建立人参和西洋参的指纹图谱[3]。但两者的特征性成分含量均较低,而且滞留时间比较接近,并且皂苷的含量会受到外界环境及加工处理等因素的影响,很难用于人参和西洋参产品尤其是混合品的有效鉴定。随着分子生物学的快速发展,DNA分子标记技术也被广泛用于人参和西洋参的鉴定。比如RAPD[4]、AFLP[5]、RFLP[6]、SCAR[7]、SSR[8]、SNP[9]等。但RAPD和ISSR的稳定性及重现性受实验条件的影响较大;RFLP和AFLP需要对DNA进行限制性内切酶消化,且对DNA的质量要求较高。SSR虽然稳定性高,但是由于两者之间的DNA片段长度差异太小,需要用聚丙烯酰胺凝胶电泳和银染进行检测,操作较为繁琐。目前对人参和西洋参的分子鉴定研究主要集中在内转录间隔区(ITS)和5.8S rDNA,但中国人参种质资源非常丰富,部分农家品种并不能利用该区间与西洋参区分开来。ETS区位于26S rDNA和18S rDNA之间的基因间隔区(IGS)上[10],有报道表明ETS序列的进化速率比ITS序列要快[11]。本研究对人参核DNA的外转录间隔区(ETS)进行了分析,并利用从中发掘的SNP分子标记开发了中国人参和西洋参的简单高效的鉴别方法。
1 材料与方法
1.1 材料
选用了5种中国人参的农家品种:大马牙、二马牙、边条、长脖、黄果。西洋参则选用市售和山东文登种植的西洋参。样品由烟台大学药学院李桂生教授进行性状鉴定,来源和产地见表1。
1.2 基因组DNA的提取
以人参和西洋参的根为原料,利用液氮在研钵粉碎成粉末后,利用植物基因组DNA提取试剂盒(FOREGENE),严格按照说明书进行提取,产物用1%的琼脂糖凝胶电泳检测。
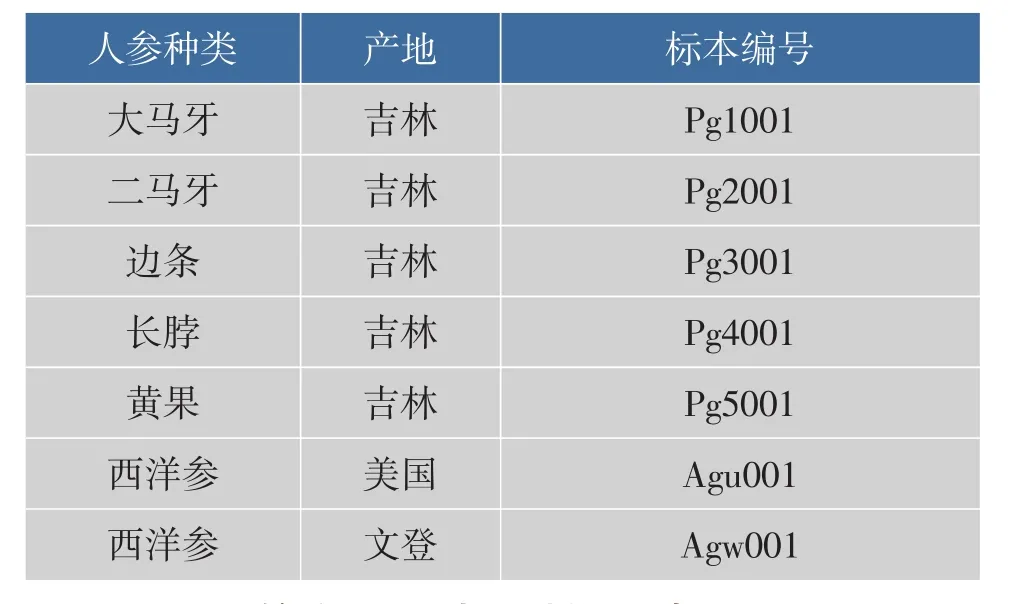
表1 采用的样品Table 1Samples used in this study
1.3 ETS区的序列比对及引物设计
根据Genbank中人参和西洋参的ETS序列(HQ650811和HQ650812),利用Clustal Omega在线软件进行序列比对。利用引物设计软件Primer Premier5设计人参的特异性引物PgF和西洋参的特异性引物AgF及反向引物ETSR。人参和西洋参特异性引物的3’末端人为引入错配碱基以保证引物的特异性。
1.4 人参和西洋参特异性引物退火温度的确定
用人参和西洋参的特异性引物PgF和AgF分别对人参和西洋参进行扩增,PCR(Eppendorf Mastercycler)反应体系为20µL,其中包括10 ng的模板DNA,0.5µmol/L ETSR,0.5µmol/L PgF或AgF,10µL 2X Premix DNA polymerase(Genotech)。PCR的反应条件为94℃预变性4 min,循环过程为94℃30 s,退火温度分别选择60、62和64℃,时间为30 s,72℃30 s,共35个循环,72℃延伸5 min,4℃保温结束反应。取3µL反应产物在含有溴化乙锭的琼脂糖凝胶上电泳并在凝胶成像系统(上海精科实业,WFH-201B)下检测,根据反应条带确定PgF和AgF用于多重PCR鉴定人参和西洋参的退火温度。
1.5 人参和西洋参的PCR鉴定
PCR反应体系为20µL,其中包括10 ng的模板DNA,0.5µmol/L ETSR,0.5µmol/L PgF,0.2µmol/L AgF,10µL 2X Premix DNA polymerase(Genotech)。PCR的反应条件为94℃预变性4 min,循环过程为94℃30 s,64℃30 s,72℃30 s,共35个循环,72℃延伸5 min,4℃保温结束反应。取3µL反应产物在含有溴化乙锭的琼脂糖凝胶上电泳并在凝胶成像系统下检测。
2 结果
2.1 人参和西洋参特异引物的设计
根据人参和西洋参ETS区的比对结果,发现在第55和第169的碱基位置上,人参都是T而西洋参都为A。根据这两个突变位点,分别设计西洋参和人参的正向特异性引物AgF和PgF。为了保证引物的特异性,将AgF的倒数第二位碱基由A改为T(GTGTTGGCATAGTGTACGTTA),PgF的倒数第三位碱基由C变为A(AGAGCAGTAAGCCTTGGAAA AT),使得特异性引物对非目标DNA有两个碱基的错配。同时设计引物ETSR(AGACAAGCATATGAC TACTGGCAGG)作为两个特异性引物的反向引物进行扩增。比对结果及引物的相对位置如图1所示。
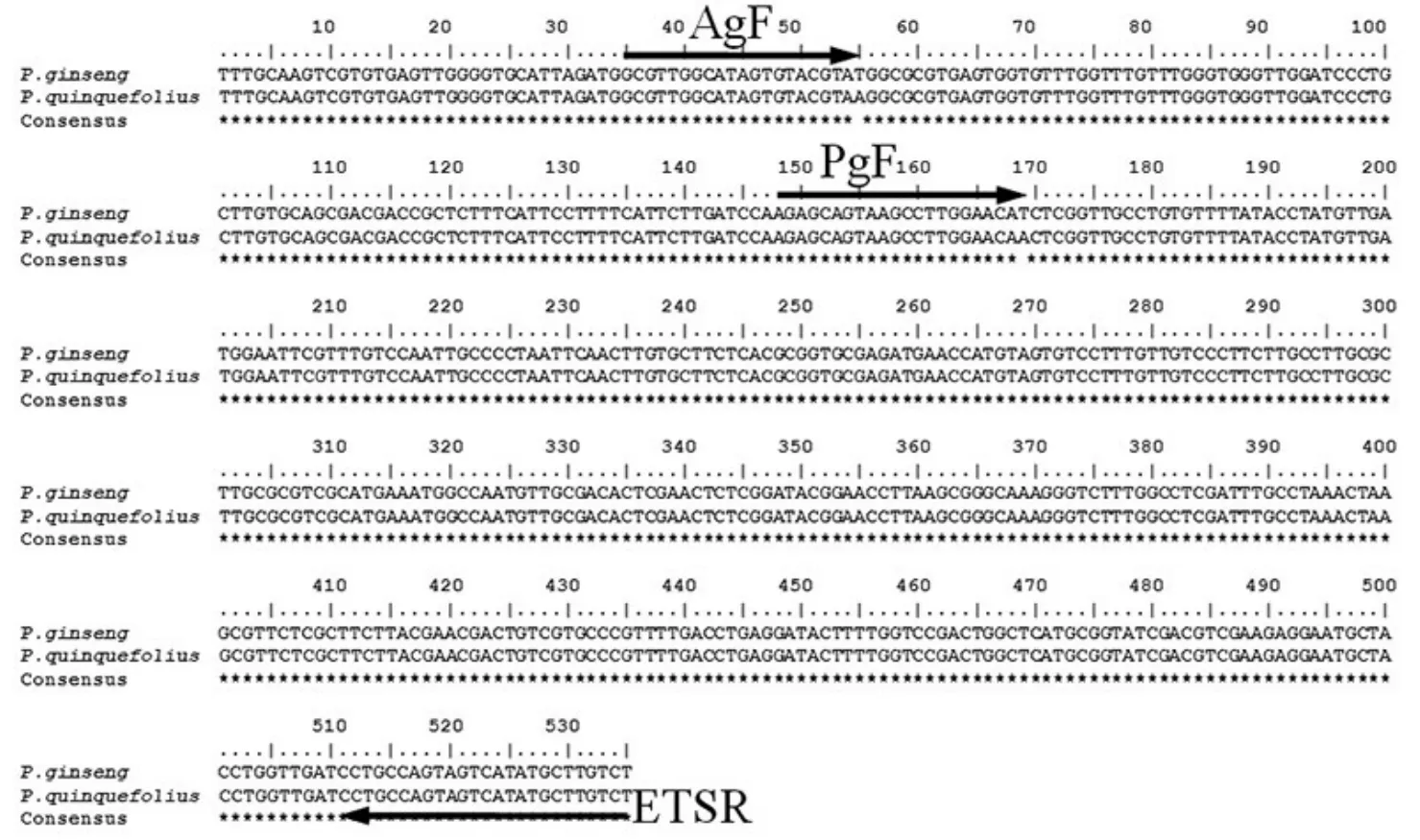
图1 中国人参与西洋参的比对结果Figure 1Comparison of ETS sequences of Panax ginseng and Panax quinquefolius.
2.2 人参和西洋参特异性引物退火温度的确定
用人参和西洋参的特异性引物分别对人参和西洋参进行扩增,如图2所示,对于人参的特异性引物PgF,3个不同的退火温度对人参的扩增效率几乎没有影响,而西洋参在64℃时无扩增产物出现。同样,对于西洋参的特异性引物AgF,3个不同的温度条件对西洋参的扩增效率几乎没有影响,而人参在64℃时无扩增条带。
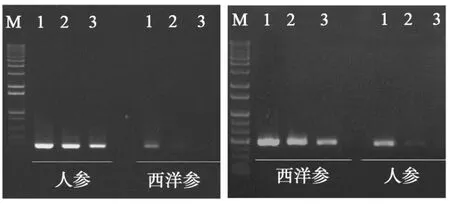
图2 人参与西洋参特异引物的退火温度检测。Figure 2Determination of annealing temperatures of specific primers.
因此,用于鉴定人参和西洋参的退火温度确定为64℃。
2.3 人参和西洋参的PCR鉴定
以中国人参的5个农家品种和西洋参基因组DNA为模板,利用人参和西洋参的两个正向特异性引物PgF和AgF及反向引物ETSR做多重PCR,引物的相对位置如图3所示,反应条件如1.5所述。
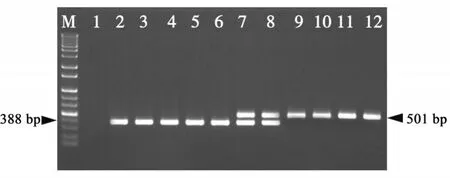
图3 中国人参和西洋参鉴定的凝胶电泳图谱。Figure 3Agarose gel image of authentication of Panax ginseng and Panax quinquefolius.
从凝胶成像结果可以看出,中国人参的5个农家品种全部扩增出388 bp的特异性条带,两个西洋参样品扩增出了501 bp的特异性条带,对于人参和西洋参的混合品则两种特异性条带都有扩增。在1.5所述的PCR条件下,对本实验进行了多次重复检测,鉴定结果的重现性及可靠性均非常理想。因此,利用本研究的多重PCR体系可对人参、西洋参及两者的混合品均能成功鉴定。
3 讨论
与其他分子标记相比,SNP分子标记分布广泛,遗传稳定,易于基因分型,非常适用于种间的快速鉴定。虽然有研究利用5.8S rDNA的SNP分子标记对人参和西洋参进行了鉴定,但中国人参种质资源非常丰富,有大量的农家品种作者发现并不能利用该区间与西洋参区分开来,因此,利用5.8S rDNA来区分人参和西洋参有局限性。
作者利用ETS区上人参和西洋参的SNP位点,分别设计了人参和西洋参的特异性引物并利用多重PCR对人参和西洋参进行了鉴定。为了保证引物的特异性,人为地在引物的3’末端引入了错配碱基。在适宜的退火温度下,错配碱基的引入大大降低了非特异性扩增的效率而对目标DNA的扩增效率几乎没有影响。作者建立的多重PCR体系可以在同一个反应中同时对人参、西洋参及两者的混合品进行鉴定,并且对模板DNA纯度的要求不高,结果的检测也只需要用琼脂糖凝胶电泳即可完成。因此,本方法对现在的人参和西洋参进行区别的化学分析法是一个有益的补充,并且可以作为中药材及其混伪品进行区分和鉴定的常规方法进行应用。
[1]詹鑫婕,田程,张媛,等.基于ITS2条形码SNPs的人参和西洋参PCR-SSCP分子鉴别研究[J].中国中药杂志,2012,(24):3748-3751.
Zhan Xinjie,Tian Cheng,Zhang Yuan,et al.PCR-SSCP molecular identification of Panax ginseng and P.quinquefolius based on ITS2 bar codinng SNPs[J].China journal of Chinese materia medica,2012,37(24):3748-3751.(in Chinese)
[2]Wang C-Z,Aung H H,Ni M,et al.Red American ginseng:ginsenoside constituents and antiproliferative activities of heat-processed Panax quinquefoliusroots[J].Planta medica,2007,73(7):669.
[3]Wang X,Sakuma T,Asafu-Adjaye E,et al.Determination of ginsenosides in plant extracts fromPanax ginsengandPanax quinquefoliusL.by LC/MS/MS[J].Analytical chemistry,1999,71(8):1579-1584.
[4]Mihalov J J,Marderosian A D,Pierce J C.DNA identification of commercial ginseng samples[J].Journal of agricultural and food chemistry,2000,48(8):3744-3752.
[5]Ha W,Shaw P,Liu J,et al.Authentication ofPanax ginsengandPanax quinquefoliususing amplified fragment length polymorphism(AFLP)and directed amplification of minisatellite region DNA(DAMD)[J].Journal of agricultural and food chemistry,2002,50(7):1871-1875.
[6]Um J-Y,Chung H-S,Kim M-S,et al.Molecular authentication of Panax ginseng species by RAPD analysis and PCR-RFLP[J]. Biological and Pharmaceutical Bulletin,2001,24(8):872-875.
[7]Wang J,Ha W-Y,Ngan F-N,et al.Application of sequence characterized amplified region(SCAR)analysis to authenticate Panax species and their adulterants[J].Planta medica,2001,67(08):781-783.
[8]陈子易,吕旭楠,程舟,等.微卫星标记在人参和西洋参鉴别中的应用[J].复旦学报:自然科学版,2011,(2):185-191.
CHEN Ziyi,LV Xunan,CHEN Zhou,et al.Application of microsatellite markers to differentiate Panax ginseng CA meyer from Panax quinquefoliusL.[J].Journal of Fudan University(Natural Science),2011,(2):185-191.(in Chinese)
[9]宋沁馨,冯芳,张心悦,等.SNP测定结合芯片电泳法快速鉴别人参和西洋参[J].药物分析杂志,2009,(1):1-5.
SONG Qinxin,FENG Fang,ZHANG Xinyue,et al.Rapid detection the SNP of Panax ginseng and P.quinquefolius by microchip electrophoresis[J].Chinese Journal of Pharmaceutical Analysis,2009,(1):1-5.(in Chinese)
[10]王川易,郭宝林.植物核基因组核糖体基因间隔区序列的结构特点及其在系统发育研究中的应用[J].武汉植物学研究,2008,(4):417-423.
WANG ChuanYi,GUO Baolin.The Characteristics of the frequently used nuclear robosome gene spacers and their utilizations in phylogenetic study of plants[J].Journal of Wuhan Botanical Research,2008,(4):417-423.(in Chinese)
[11]Markos S,Baldwin B G.Higher-level relationships and major lineages of Lessingia(Compositae,Astereae)based on nuclear rDNA internal and external transcribed spacer(ITS and ETS)sequences[J].Systematic Botany,2001,26(1):168-183.
Molecular Authentication of Panax ginseng and Panax quinquefolius by their Ribosomal DNA External Rranscribed Spacer Region
DENG Jing,ZHANG Tiantian,WANG Di,JIN Haizhu,WANG Hongtao*
(College of Life Science,Yantai University,Yantai 264005,China)
Panax ginseng and Panax quinquefolius are two important traditional Chinese medicine with similar morphology but different medicinal efficacy.This study aimed to develop a simple and effective analysis of molecular authentication of Panax ginseng and Panax quinquefolius.Two allelespecific primers of Panax ginseng and Panax quinquefolius were designed from their Single Nucleotide Polymorphism(SNP)created by ribosomal DNA(rDNA)external transcribed space(ETS). Multiplex polymerase chain reaction(Multiplex PCR)was conducted for identification of Panax ginseng and Panax quinquefolius.The results confirmed the effective authentication of Panax ginseng and Panax quinquefolius as well as their mixture by the established PCR method.
Panax ginseng;,Panax quinquefolius,ETS,SNP
R282.5
A
1673—1689(2015)04—0420—04
2014-08-23
山东省自然科学基金项目(2013ZRCQ021)
*通讯作者:王洪涛(1982-),男,山东聊城人,理学博士,讲师,主要从事中药材DNA分子标记研究。E-mail:wht1211@gmail.com

