西瓜枯萎病菌的快速检测与鉴定
曹月霞 凌键 谢丙炎 杨宇红
(中国农业科学院蔬菜花卉研究所,北京 100081)
西瓜枯萎病菌的快速检测与鉴定
曹月霞 凌键 谢丙炎 杨宇红
(中国农业科学院蔬菜花卉研究所,北京 100081)
通过西瓜枯萎病菌与其他专化型枯萎病菌及瓜类几种重要病原菌的比较基因组分析,获得了西瓜枯萎病菌的基因组特异序列。在此基础上,设计出特异引物,筛选可扩增出西瓜枯萎病菌特异性DNA条带的引物。将特异性引物和尖孢镰刀菌专化型的通用引物W106R/W106S结合,建立双重PCR检测体系。该双重PCR检测体系可以在一次PCR反应中快速、准确的检测出西瓜枯萎病菌,为通过分子方法快速鉴定西瓜枯萎病菌提供技术支持。
西瓜枯萎病菌;双重PCR;尖孢镰刀菌
西瓜枯萎病俗称蔓割病或萎蔫病,是由尖孢镰刀菌西瓜专化型(Fusarium oxysporum f. sp. niveum)引起的一种世界性瓜类土传病害[1-3]。它的发生极为普遍,对西瓜产量和品质构成了严重的威胁,是西瓜生产上的重要病害。目前,对这类维管束病害的有效防治方法主要是依靠抗病品种选育,但可供选育的抗病品种抗源单一,效率不高。赵丽明等[4]从西瓜根际土壤中筛选出对西瓜枯萎病菌防治效果好、无污染土壤中生存良好的放线菌菌株XF9、XF18和XF22,能较好的防治西瓜枯萎病。3种有益微生物为生物农药的研发奠定了基础,也为西瓜枯萎病上拮抗菌的拮抗机制奠定了理论基础。国际上公认的西瓜枯萎病菌有3个生理小种,即生理小种0号、1号和2号,其中2号生理小种的侵染性最强[5-7]。除以上3个生理小种外,还有其他的小种分化,生理小种3于2010年鉴定分离[8],但在中国尚未见报道,需进一步证实。传统上对西瓜枯萎病菌的鉴定主要是采用形态特征的比较法,即对不同地区的西瓜枯萎病菌分离物进行形态特征(如菌落颜色、菌落密度、大小孢子的大小与形状及菌落直径等)的比较。但利用传统的鉴定方法来鉴别病原菌准确性不高,已不能满足生产的要求。近年来,随着分子生物学技术的发展,包括AFLP、RFLP、RAPD及SCAR在内的分子检测技术在病原菌鉴定[9]、分类[10]及群体遗传多样性[11]研究中得到植物病理学家的高度重视。基于特异基因组设计的特异性引物已经广泛应用于病原菌的分子检测,前人基于特异性引物成功建立了快速检测双重PCR技术已用于检测水稻白叶枯病菌和细菌性条斑病菌[12]、小麦条纹花叶病毒和小麦花叶病毒[13]等植物病原菌的分子鉴定体系。目前在尖孢镰刀菌的分子检测方面,Zhang等[14]利用甘蓝枯萎病菌特异性引物Focs-1/Focs-2和尖孢镰刀菌通用引物W106R/W106F建立了检测甘蓝枯萎病菌的多重PCR检测体系。该技术既可应用于甘蓝枯萎病发病区病情的检测,也可从发病植株中检测出甘蓝枯萎病菌。而对于西瓜枯萎病菌的分子检测,Zhang等[15]把西瓜枯萎病菌和西瓜蔓枯病菌的2对特异性引物Fn-1/Fn-2 和Mn-1/Mn-2置于同一反应中,建立了检测西瓜枯萎病菌和蔓枯病菌的双重 PCR 体系。但双重PCR检测西瓜枯萎病菌和其他病原菌的方法在国内外都未见报道。
本研究拟通过设计西瓜枯萎病菌的特异引物,与尖孢镰刀菌的通用引物W106R/W106S结合,建立双重PCR检测体系,对西瓜枯萎病菌、尖孢镰刀菌其他专化型菌株和其他外围菌株进行专化性检测,以期为该病的预测预报并及时采取有效的防控措施提供依据。
1 材料与方法
1.1 材料
本研究供试菌株包括西瓜枯萎病菌的3个小种共7个菌株、尖孢镰刀菌其他专化型的10个菌株和其他5个外围菌株。其种名、来源及数量,见表1。

表1 供试西瓜枯萎病菌菌株的来源
1.2 方法
1.2.1 供试菌株培养 西瓜枯萎病菌在PDA培养基上(28℃)培养72 h后,从菌落的边缘挑取菌丝转至PDB液体培养基中(150 r/min,28℃)振荡培养3 d过滤收集菌丝,-80℃保存备用。
1.2.2 DNA的提取 用改良的CTAB法[16]提取病原菌的DNA。
1.2.3 引物的设计 根据西瓜枯萎病菌的基因组序列,比对近缘种以及其他不同致病菌的序列同源性,通过基因组BLAST的方法找到西瓜枯萎病菌的特异片段。用引物设计软件Primer 5.0设计出10对引物,详况,见表2。

表2 待筛选的生物序列
1.2.4 引物的PCR扩增 引物检测西瓜枯萎病菌的PCR反应总体系是25 μL:2×Taq Master Mix(Dye Plus)12.5 μL,10 μmol/L的引物各1 μL,DNA模板2 μL,ddH2O 8.5 μL。PCR的反应程序是:94℃预变性4 min;94℃变性40 s;60℃退火30 s;72℃延伸100 s,共30个循环;最后72℃延伸10 min。扩增结束后,取6 μL PCR产物经1%(50×TAE)琼脂糖凝胶电泳后,在凝胶成像仪上观察并拍照。
1.2.5 引物的特异性检测 用合成的引物对供试菌株的基因组DNA进行扩增,检测其特异性,PCR反应体系、反应程序同上。
1.2.6 双重PCR体系的建立 利用设计的检测西瓜枯萎病菌的特异性引物与尖孢镰刀菌其他专化型菌株的通用引物W106R/W106S[17]结合,建立双重PCR体系。通用引物W106R/W106S的序列为W106R(5'-GCAGTCGTACGTCATCGACC-3')和W106S(5'-CCATGGCAGATGGCGAGTCA-3'),该引物使尖孢镰刀菌其他专化型菌株扩增出一条729 bp的特异性条带[17]。
双重PCR反应总体系为25 μL:2×Taq Master Mix(Dye Plus)12.5 μL,西瓜枯萎病菌的特异引物(10 μmol/L)各1 μL,通用引物(10 μmol/L)各1 μL,DNA模板2 μL,ddH2O 6.5 μL。PCR反应程序:94℃预变性4 min;94℃变性40 s;60℃退火30 s;72℃延伸100 s,共30个循环;最后72℃延伸10 min。扩增结束后,取6 μL PCR产物经1%(50×TAE)琼脂糖凝胶电泳后,在凝胶成像仪上观察并拍照。
2 结果
2.1 引物的特异性筛选
利用设计的10对西瓜枯萎病菌特异性引物对供试的7个西瓜枯萎病菌菌株的DNA进行PCR扩增。结果(图1)显示,只有1、2、3、4和6号引物均可以产生比较单一的条带,而其他的引物则不能产生单一的条带,说明上述5对引物均具有较强特异性。
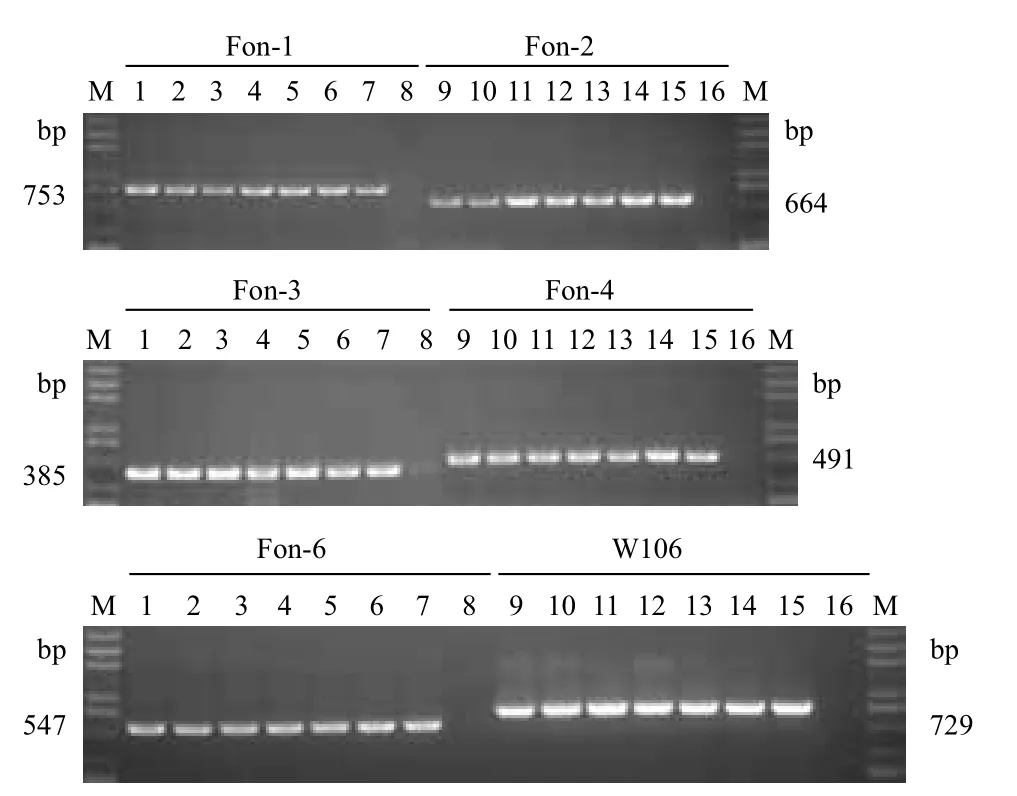
图1 利用编号为1至10的引物和通用引物W106R/ W106S扩增的特异性条带
2.2 特异性引物的检测
对筛选出的5对引物和供试的7个西瓜枯萎病菌菌株、10个尖孢镰刀菌其他专化型菌株和5个外围菌株的DNA进行PCR扩增,以ddH2O为对照。结果显示,1、2、3、4和6号引物可以使西瓜枯萎病菌产生相应的单一条带(图2,泳道1-7),而在尖孢镰刀菌其他专化型菌株和外围菌株中均不能产生条带(图2,泳道8-22)。通用引物可以使西瓜枯萎病菌和尖孢镰刀菌其他专化型菌株扩增出729 bp的条带(图2,泳道1-17),表明上述引物均具有特异性。

图2 利用编号为1、2、3、4、6的引物和通用引物W106R/W106S扩增的特异性条带
2.3 双重PCR检测
以5对特异性引物和通用引物(W106R/W106S)对7个西瓜枯萎病菌菌株以及15个对照菌株的基因组DNA进行双重PCR扩增,以ddH2O为对照。PCR产物琼脂糖凝胶电泳检测结果如图3所示,西瓜枯萎病菌可以产生两条不同的条带(图3,泳道1-7,1-17),尖孢镰刀菌其他专化型菌株只产生一条729 bp的条带(图3,泳道8-17),而外围菌株和无菌水不能扩增出条带(图3,泳道18-23)。研究结果显示,双重PCR检测结果与常规PCR检测结果一致,也进一步证明双重PCR检测技术的可靠性。
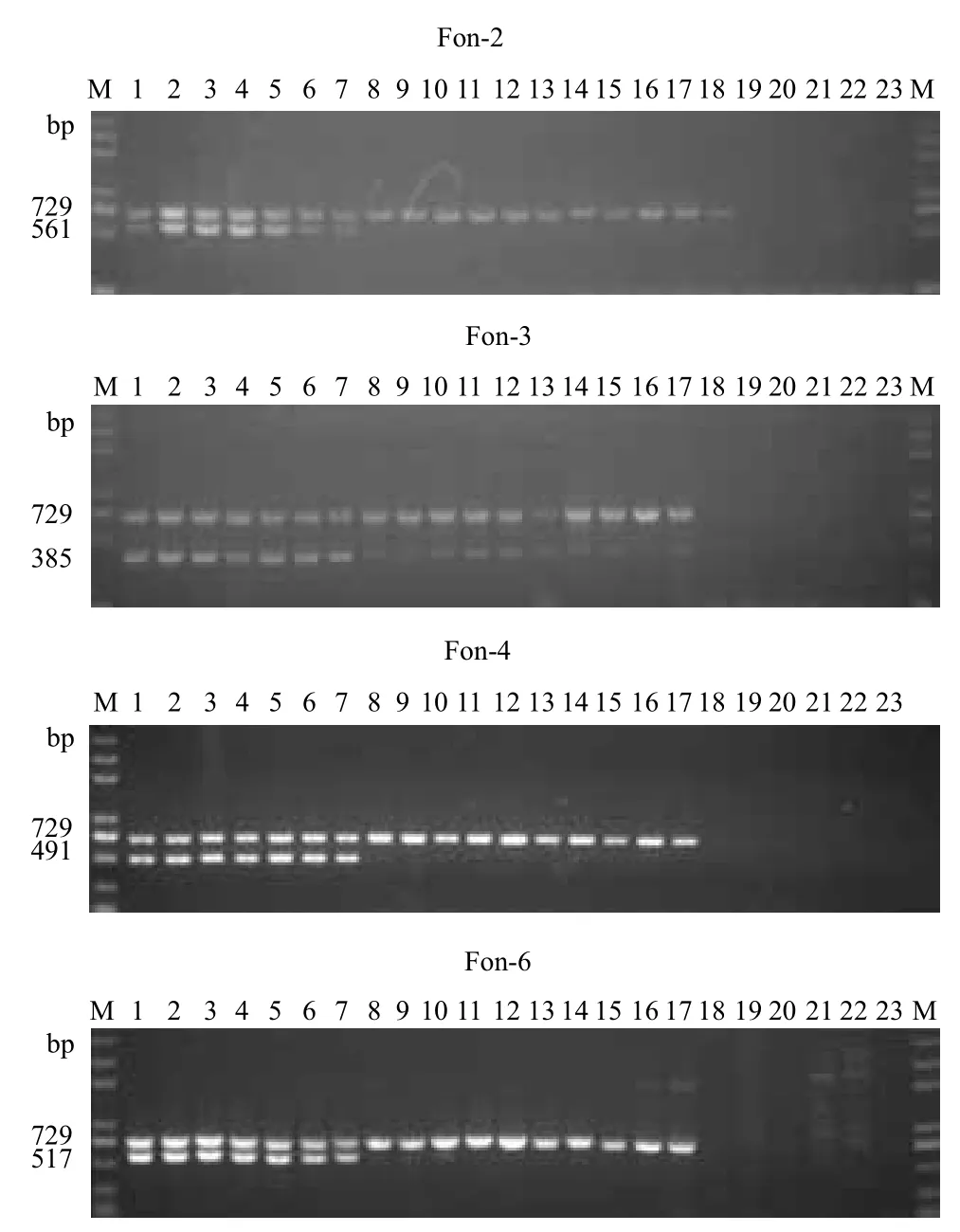
图3 利用编号为2、3、4、6的引物和通用引物W106R/ W106S扩增的特异性条带
3 讨论
双重PCR(Duplex PCR)是在一个PCR反应体系中加入两对特异性引物,同时扩增2个目的基因,能够在一个PCR反应中检测出两种病原菌,提高效率,而且还可以减少因实验操作所带来的假阳性等问题,能很好地用于植物病原菌间的检测。建立双重 PCR 体系的关键是引物设计[18],双重PCR反应中引物之间不可以发生相互作用,形成引物二聚体和非特异性的扩增。同时,二者扩增的片段长度要有一定的差距,电泳检测可以产生不同的条带。因此,双重PCR体系的建立很重要[19,20]。
检测不同植物病原菌的双重PCR反应在国内外均有报道,如陈庆河等[21]利用马铃薯晚疫病菌内转录间隔区序列设计了一对特异性引物INF1/INF2,与马铃薯青枯病菌特异性引物 759/760 组合建立双重 PCR 体系。Diallo 等[22]建立了检测胡萝卜软腐果胶杆菌马铃薯黑胫病亚种和菊亚种的双重 PCR体系,能够快速的对两种病原菌进行分子检测。Majumder 等[23]建立了检测洋葱黄矮病毒和葱潜隐病毒的双重 PCR 体系,该体系是在 RNA 水平上对洋葱黄矮病毒和葱潜隐病毒进行检测。
前人应用双重PCR方法只能检测出西瓜枯萎病菌和西瓜蔓枯病菌,而针对西瓜枯萎病菌与尖孢镰刀菌其他专化型的病原菌和其他病原菌的分子检测尚未报道。在本研究中,通过对西瓜枯萎病菌的全基因组测序,比对近缘种以及瓜类上几种重要病原菌的基因组序列,设计并筛选能扩增出西瓜枯萎病菌特异性DNA条带的引物。本研究设计的特异引物能够快速而准确的检测西瓜枯萎病菌,在西瓜枯萎病菌潜伏期和发病初期对其进行诊断、监测和病原菌鉴定,以便采取有效的防治方法,及时控制病害的传播和蔓延,减少经济损失,在实际生产上具有重要的指导作用。双重PCR具有效率高、成本低等优点,具有非常广阔的实际应用价值,同时本研究也为其他植物病害双重PCR检测方法的建立奠定了理论基础。
4 结论
本研究建立了一种双重PCR检测体系,通过设计和应用引物Fon-2R/ Fon-2F、Fon-3R/ Fon-3F、Fon-4R/ Fon-4F 和Fon-6R/ Fon-6F能检测出西瓜枯萎病菌。该检测体系具有高效、准确及经济实惠等优点,适用于西瓜田间土壤的带菌检测以及西瓜枯萎病菌的检验检疫和有效防治。
[1]Zheng Q, Bi Y, Yun XM, et al. Research progress on watermelon blight and its control[J]. Plant Protection, 2007, 27(2):11-12.
[2]Ding JC, Zhang QA, Fang L. Identification of resistance of watermelon varieties to Fusarium Wilt[J]. Chinese Vegetable,2005(6):27-29.
[3]Gu WH, Wang YH, Song RH. Preliminary study on Fusarium wilt pathogen races in Shanghai area[J]. Acta agriculture Shanghai,1994, 10(3):63--67.
[4]赵丽明, 丁延芹, 路晓萌, 等. 西瓜根际枯萎病拮抗放线菌的筛选及鉴定[J]. 生物技术通报, 2010(5):107-110.
[5]Martyn RD. Fusarium oxysporum f. sp. niveum Race 2:A highly aggressive new race to the United States[J]. Plant disease, 1987(3):71-73.
[6]Martyn RD, Netzer D. Resistance to races 0, 1, and 2 of Fusarium wilt of watermelon in Citrullus sp. PI296341-FR[J]. Hortscience,1991, 26:429-432.
[7]Wang HB, Wang M. Study of physiological races of Fusarium oxysporum f. sp. niveum[J]. Acta Agriculturae Boreali-Occidentalis Sinica, 1993, 2(4):67-70.
[8]Zhou XG, Everts KL, Bruton BD. Race 3, a new and highly virulent race of Fusarium oxysporum f. sp. niveum causing Fusarium wilt in watermelon[J]. Plant disease, 2010, 94(1):92-98.
[9]Nicholson P, Kezanoor HZ, 苏海. 用RAPD分析和DNA指纹鉴别玉米小斑病菌O.C.T三个小种 [J]. 植物病理学报, 1993, 23(2):114.
[10]Alalounakis DJ, Fragkiadakis GA. Genetic diversity of Fusarium oxysporum isolates from cucumber:differentiation by pathogenicity,vegetative compatibility and RAPD fingerprinting [J]. Ecology and Population Biology, 1999, 89(2):161-168.
[11] Tooley PW, O’Neill NR, Goley ED, et al. Assessment of diversity in Claviceps Africana and other Claviceps species by RAM and AFLP analyses [J]. Phytopathology, 2000, 90:1126-1130.
[12] 冯雯杰, 常清乐, 杨龙, 等. 水稻叶枯病和细菌性条斑病菌的分子标记筛选检测[J]. 植物病理学报, 2013, 43(6):581-589.
[13]Price JA, Smith J, Simmons A, et al. Multiplex real-time RT-PCR for detection of Wheat streak mosaic virus and Tritcum mosaic virus[J]. Journal of Virological Methods, 2010, 165(2):198-201.
[14]张吉祥, 凌键, 谢丙炎, 等.甘蓝枯萎病菌1号和2号生理小种的快速检测与鉴定[J].植物病理学报, 2014, 44(6):586-594.
[15]Zhang Z, Zhang J, Wang Y, et al. Molecular detection of Fusarium oxysporum f. sp. niveum and Mycosphaerella melonis in infected plant tissues and soil[J]. FEMS Microbiology Letters. 2005, 249(1):39-47.
[16]Liu D, Coloe S, Baird R, et al. Rapid mini-preparation of fungal DNA for PCR [J]. Journal of Clinical Microbiology, 2000, 38(1):471.
[17]LI MH, Yu HT, Wang HF, et al. Rapid Detection and Identification of Fusarium oxysporum f. sp. cubense Race 1 and Race 4(in Chinese)[J]. Scientia Agricultura Sinica, 2012, 45(19):3971-3979.
[18]Henegariu O, Heerema NA, Dlouhy SR, et al. Multiplex PCR:critical parameters and step-by-step protocol[J]. Biotechniques,1997, 23(3):504-511.
[19]Gilbert SA, Burton KM, Prins SE, et al. Typing of bovine viral diarrhea viruses directly from blood of persistently infected cattle by multiplex PCR[J]. Journal of Clinical Microbiology, 1999, 37(6):2020.
[20]Kent L, Mchugh TD, Billington O, et al. Demonstration of homology between IS6110 of Mycobacterium tuberculosis and DNAs of other Mycobacterium spp.[J]. Journal of Clinical Microbiology, 1995,33(9):2290.
[21]Cheng QH, Li BJ, Lan CZ, et al. Development and application of duplex PCR assay for detection of Phytophthora infestans and Ralstonia solanacearum[J]. Acta Phytopathologica Sinica, 2009,39(6):578-583.
[22]Diallo S, Latour X, Groboillot A, et al. Simultaneous and selective detection of two major soft rot pathogens of potato:Pectobacterium atrosepticum(Erwinia carotovora subsp. atrosepticum)and Dickeya spp.(Erwinia chrysanthemi)[J]. European Journal of Plant Pathology, 2009, 125(2):349-354.
[23]Majumder S, Baranwal VK, Joshi S. Simultaneous detection of Onion yellow dwarf virus and Shallot latent virus in infected leaves and cloves of Garlic by duplex RT-PCR[J]. Journal of Plant Pathology, 2008, 90(2):371-374.
(责任编辑 狄艳红)
Rapid Detection and Identification of Fusarium oxysporum f. sp. niveum
Cao Yuexia Ling Jian Xie Bingyan Yang Yuhong
(Institute of Vegetable and Flowers,Chinese Academy of Agricultural Sciences,Beijing 100081)
Based on the genome comparison analysis between Fusarium oxysporum f. sp. niveum(Fon)and other closely-related pathogens, the specific sequences of Fon were obtained. Then we developed specific primers that could amplify Fon-specific DNA bands. Combined the Fon-specific primers with the universal primer of Fusarium oxysporum (W106R/W106S), we established a duplex PCR method that might rapidly and accurately detect Fon by 1 PCR reaction, which is beneficial to technically support the rapid identification of Fon by method of molecular detection..
Fusarium oxysporum f. sp. niveum;duplex PCR;Fusarium oxysporum
10.13560/j.cnki.biotech.bull.1985.2015.07.009
2014-11-05
国家自然科学基金项目(31272003),国家科技支撑计划“果蔬重大病虫害防控技术研究与集成示范(2012BAD19B06),公益性行业(农业)科研专项(200903049),中国农业科学院科技创新工程(CAAS-ASTIP-IVFCAAS)
曹月霞,女,硕士研究生,研究方向:分子植物病理学;E-mail:caoyuexia815@163.com
凌键,男,副研究员,研究方向:植物病理学;E-mail:lingjian2005@126.com

