水稻miR169o及其靶基因OsNF-YAs对缺水胁迫的早期表达模式
陈禹彤 陈华民 余超 Amy Thein 田芳 何晨阳
(中国农业科学院植物保护研究所 植物病虫害生物学国家重点实验室,北京 100193)
水稻miR169o及其靶基因OsNF-YAs对缺水胁迫的早期表达模式
陈禹彤 陈华民 余超 Amy Thein 田芳 何晨阳
(中国农业科学院植物保护研究所植物病虫害生物学国家重点实验室,北京100193)
MicroRNAs(miRNAs)是一类小的非编码RNA,在植物逆境胁迫应答中发挥重要的调控作用。miR169可受干旱胁迫诱导表达,而过表达miR169则可以增强植物对干旱的耐受性。然而,miR169及其靶基因NF-YAs在水稻干旱胁迫条件下的表达动态至今尚不清楚。对水稻进行不同时间缺水处理,用qRT-PCR法定量测定了水稻根、茎、叶组织中miR169o及其靶基因表达的动态变化。结果表明,随着缺水处理时间的增加,水稻不同组织中miR169o表达量总体上有升高趋势;而靶基因(NF-YA1、NFYA2 和NF-YA3)表达模式基本与miR169o的表达模式相反,但并不全部对应。推测在水稻干旱胁迫早期反应中,miR169o可能主要调控了部分特定靶基因的表达。此外,miR169o在水稻根、茎、叶组织中的表达和丰度存在着明显的差异,具有组织特异性。
miR169o;OsNF-YAs;水稻;干旱胁迫;表达模式
MicroRNAs(miRNAs)是一类内源基因编码的单链小分子非编码RNA,长度为18-25个核苷酸,其主要通过两种机制调控其靶基因的表达:剪切靶基因mRNA或者抑制靶基因翻译[1-3]。miRNAs是植物应对生物和非生物逆境中的重要调控因子[4,5]。miR169是受多种胁迫(如干旱、氮饥饿、高盐、冷害等)条件调控的一个保守miRNA家族[6-8]。核因子Y(nuclear factor Y,NF-Y)是普遍存在的一类转录因子,由NF-YA、NF-YB和NF-YC三个亚基组成。NF-YA家族含有miR169靶标位点,miR169可以抑制NF-YA基因转录[9,10]。氮饥饿显著下调拟南芥中miR169的表达水平,上调靶基因NF-YA的表达,从而调控植物中氮素传感器NRT1.1的表达,因此调节了植物应对氮饥饿的反应[7]。高盐胁迫能够诱导拟南芥miR169的表达,其靶基因NF-YA作为CAAT结合因子,进一步参与基因表达调控和信号转导,从而抵抗高盐胁迫[11]。miR169还参与调控植物的花期,过量表达miR169d,抑制了靶基因AtNFYA2,从而减少FLC(Flowering Locus C,FLC)的表达,导致FLC的靶基因FT(FLOWERING LOCUS T)和LFY(LEAFY)表达量增加,从而促进拟南芥开花[12]。干旱胁迫下调拟南芥miR169a和miR169c的表达,共表达实验表明miR169a能更高效地抑制atNFYA5的mRNA水平。nfya5突变体和过表达ath-miR169a的转基因植株加快了叶片的失水速率,对干旱胁迫也更加敏感[9]。共表达实验和5' RACE实验都证明miR169可以剪切菜豆GmNFYA3,将GmNFYA3在拟南芥中过表达,则转基因植株叶片失水速率降低,抗旱性明显增强,而对高盐、ABA则更为敏感[10]。此外,miR169还参与了植物根发育的调控,通过miRNAs类似物和剪切位点突变的NF-YA基因的相关分析表明,miR169在根的生长和分支上发挥了特殊的调控作用。miR169defg通过抑制NF-YA2和NF-YA10的转录和蛋白翻译影响了拟南芥主根的生长和侧根的起始[13]。干旱胁迫诱导了sly-miR169在番茄体内的积累水平,相应地下调了siNF-YA1/2/3的转录水平,过量表达sly-miR169c的转基因植株降低了气孔的开放、呼吸速率和叶片的失水率,从而增强了对干旱的耐受性[14]。这些结果表明,尽管miR169是一个非常保守的家族,但miR169在不同物种中的功能并不完全一致,参与干旱调节的主效靶基因也可能并不完全相同。
干旱胁迫是水稻生长发育的重要环境影响因子之一,能够导致水稻减产,甚至死亡。目前,水稻干旱胁迫反应研究已经深入到分子水平,涉及抗旱基因克隆、定位及功能分析等[15]。水稻miR169家族中对植物调控作用主要包括miR169g和miR169n/o两部分,其中miR169n和miR169o位于同一个miRNA基因簇中,二者相距3 707个碱基[16]。然而,在干旱胁迫条件下,水稻miR169o及其靶基因早期表达变化及其组织特异性尚不清楚。
为了明确缺水或干旱胁迫条件下,水稻miR169及其靶基因的表达调控模式,本研究利用qRTPCR法检测了胁迫初期水稻根、茎、叶中miR169o以及靶基因OsNF-YA1(Os03g48970)、OsNF-YA2(Os12g42400)和OsNF-YA3(Os02g53620)的表达动态变化,以期明确缺水或干旱胁迫初期,miR169o及其靶基因的动态表达调控模式和组织特异性情况。
1 材料与方法
1.1 材料
水稻品种日本晴(Oryza sativa L. cv. Nipponbare)由本实验室保存;Trizol Regent购自Invitrogen公司;TaqMan® MicroRNA Reverse Transcription Kit 购自ABI公司;RQ1 RNase-Free DNase、GoScriptTMReverse Transcription System、GoTaq® qPCR Master Mix购自Promega公司;其他试剂均为国产分析纯;实验引物由北京华大基因科技有限公司合成。
水 稻 培 养 Hogland营 养 液[17]:10 mmol/L KH2PO4,2 mmol/L MgSO4,1 mmol/L CaCl2,0.1 mmol/L Fe-EDTA,50 μmol/L H3BO4,12 μmol/L MnSO4,1 μmol/L ZnCl2,1 μmol/L CuSO4,0.2 μmol/L Na2MoO4,pH5.5-6.0。
1.2 方法
1.2.1 水稻幼苗培养及其缺水胁迫处理 将水稻种子置于培养皿清水中,在37℃下催芽2 d。待幼芽长至1 cm长时,将其种植于泡沫板上,将泡沫板置于清水中,覆盖蛭石。4 d后将泡沫板移至含3 mmol/L KNO3的Hogland培养液中培养(28℃、16 h光照、8 h黑暗)。选取生长3周的水稻幼苗,将其从营养液中取出,用干燥吸水纸迅速吸除根部水分,进行脱水干旱胁迫处理0、0.5、1、2和4 h,然后采集根、茎和叶部样品,经液氮速冻后,置于-80℃保存备用。
1.2.2 水稻总RNA提取及cDNA合成 经缺水处理0、0.5、1、2和4 h的水稻根、茎和叶组织样品按照稍作改进的Trizol方法[18]进行水稻总RNA提取。经DNase处理后,用琼脂糖凝胶电泳检测RNA完整性及基因组DNA是否完全去除,用Nano Drop 2000分光光度计检测RNA浓度。miR169o反转录茎环引物为5'-GTCGTATCCAGTGCAGGGTCCGAGGTATTC GCACTGGATACGACTAGGCA-3',按照ABI TaqMan® MicroRNA Reverse Transcription试剂盒方法进行反转录;靶基因OsNF-YAs反转录引物为Oligo dT,按照GoScriptTMReverse Transcription System试剂盒方法进行反转录。
1.2.3 实时荧光定量PCR分析 利用Primer premier 5.0软件,设计miR169o、靶基因OsNF-YAs和内参基因U6的特异性引物(表1)。以合成的cDNA为模板,按照GoTaq® qPCR Master Mix试剂盒方法,用ABI 7500实时定量PCR仪进行qRT-PCR检测。对照样品为未经胁迫处理的水稻样品。miR169o及其靶基因相对表达量按照2-△△Ct法计算[19],其中△△Ct =(Ct样品-CtU6)TimeX -(Ct对照样品-CtU6)Time0,Time X表示任意时间点,Ct为荧光阈值。

表1 本研究用引物
2 结果
2.1 水稻根部miR169o及OsNF-YAs的表达
以U6为内参基因、未处理样品为对照,进行缺水处理水稻根部样品qRT-PCR检测。实验中miR169o、OsNFYAs和U6基因熔解曲线均为单一峰。分别计算各处理样品中基因的相对表达量。结果(图1)表明,水稻根部miR169o表达量总体上呈升高趋势,在缺水处理0.5 h时出现极显著差异,在缺水处理2 h时达到最高峰,之后逐步下降,但仍明显高于对照。OsNF-YA2的表达动态变化与miR169o的完全相反,符合miRNA与靶基因的对应关系;OsNFYA1的表达动态与miR169o相似;OsNF-YA3在缺水处理1-4 h之间的表达动态与miR169o相反。
2.2 水稻茎部miR169o及OsNF-YAs的表达
经缺水处理的水稻茎部miR169o表达动态与根部相似(图2),但总体表达变化幅度不超过5倍。缺水处理1 h表达量迅速升高,2 h时出现一个2倍的峰值,随后下降,但4 h时但仍明显高于对照。3个OsNF-YAs的表达动态变化趋势基本一致,均与miR169o相反,符合miRNA与靶基因的对应关系。
2.3 水稻叶部miR169o及其OsNF-YAs的表达
经缺水处理的水稻叶部miR169o的表达量整体呈上升趋势(图3)。缺水处理0-0.5 h迅速上升,在0.5 h时出现显著差异,0.5-1 h其表达基本维持此水平,随后迅速下降,在2 h时降至最底点,随后又上升至4倍峰值;OsNF-YA2和OsNF-YA3的表达在初始0.5 h显著下降,与miR169o呈相反趋势,但0.5-4 h的表达趋势同miR169o一致;OsNF-YA1的表达趋势与miR169o一致,同其在根部表达情况类似,不符合miRNA和靶基因的对应关系。
3 讨论
miRNA在植物生长发育和应对逆境胁迫反应中发挥着重要的调控作用。近年来,针对miR169家族及靶基因NF-YAs的研究涉及到拟南芥[20]、水稻[21]、番茄[14]、大豆[10]、山杨[22]等植物,主要包括早花[12]、干旱[14]、高盐[16]、低氮[7]等性状的调控方面。水稻中miR169家族包含17个成员,代表9个仅存在微小差别的不同的成熟序列。在高盐胁迫中,仅miR169g和miR169n/o受到诱导,其余miR169家族成员对盐胁迫无响应[16]。PEG 6000(polyethylene glycol 6000)模拟干旱处理后,芯片分析表明水稻miR169家族中几个成员都能被诱导表达,然而Northern blot仅能验证miR169fg的诱导表达[23],这可能是由于不同技术方法的灵敏度和特异性所决定的。通常来说,盐胁迫和干旱胁迫之间存在着密切的联系,尽管已经报道miR169o在高盐胁迫下诱导表达,但对于干旱胁迫下水稻miR169o的动力学表达变化尚无报道。本研究对缺水干旱胁迫初期水稻miR169o及其靶基因的动态变化趋势进行了监测。对于胁迫条件设置,本实验室未采用PEG6000处理法,而是对植物进行缺水处理来模拟干旱胁迫初期的情况,这种处理方式较PEG6000处理更为剧烈,在4 h时植株已经出现明显萎蔫症状。在干旱胁迫初期0-4 h内,尽管水稻miR169表达水平在根、茎、叶中都有不同程度升降,但其表达总体上呈上升趋势。

图1 经缺水处理的水稻根部m iR169o及OsNF-YAs表达
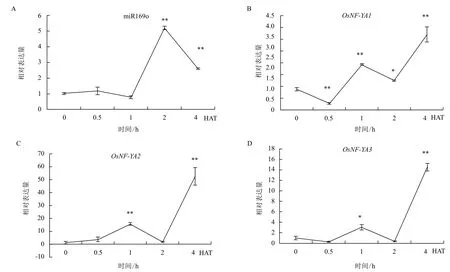
图2 经缺水处理的水稻茎部m iR169o及OsNF-YAs表达
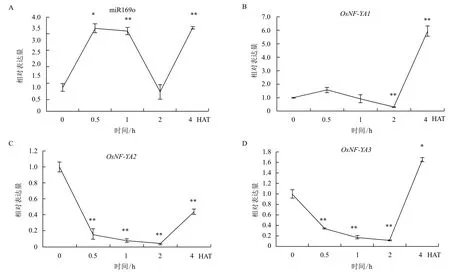
图3 经缺水处理的水稻叶部m iR169o及OsNF-YAs表达
本研究表明,干旱胁迫下miR169o在水稻不同组织中的表达具有明显的组织特异性。其在根、茎部的表达趋势基本一致,但根部变化幅度更大,可高达171倍。有报道在盐胁迫条件下玉米根部应答反应比叶部更快、更敏感[24]。本研究发现miR169o根部变化远高于茎部,可能是因为miR169o在干旱胁迫下根中的应答反应更为敏感。miR169o叶部表达趋势不同于根茎部,胁迫初始上调迅速,可能是干旱胁迫诱导了根部ABA积累、从根部运输到叶片,气孔开闭受抑制,从而减少水分流失[25-27]。因此,miR169o在叶部能迅速应答干旱胁迫,但其表达量变化范围远远小于根部。
本研究还发现,miR169与靶基因的表达变化趋势在茎部完全对应,但在根、叶部并不能完全对应。可能的原因是在干旱胁迫反应中,有其它miRNA、激素以及调控因子同时参与了调控过程[5]。有报道拟南芥NF-YAs家族分为3个亚组,不同亚组在根或地上部的积累量不同[7]。水稻OsNF-YAs家族也分为不同亚组,OsNF-YA1、OsNF-YA2和OsNFYA3处于不同亚组中,3个靶基因在根茎叶部表达趋势并不一致。其具体分布状况尚待研究。
4 结论
本研究定量分析了在干旱胁迫下水稻不同组织中miR169o及靶基因OsNF-YAs表达的动态变化,发现miR169o在干旱胁迫下表达量总体上升,其在根茎部动态变化趋势基本一致,根部在应对干旱胁迫应中更敏感,叶部反应更为迅速。靶基因OsNFYA1、OsNF-YA2、OsNF-YA3的表达在茎部与miR169o完全对应,而在根、叶部并不完全对应。
[1] Rogers K, Chen X. Biogenesis, turnover, and mode of action of plant microRNAs[J]. Plant Cell, 2013, 25(7):2383-2399.
[2] Bartel DP. MicroRNAs:genomics, biogenesis, mechanism, andfunction[J]. Cell, 2004, 116(2):281-297.
[3] Jones-Rhoades MW, Bartel DP, et al. MicroRNAs and their regulatory roles in plants[J]. Annu Rev Plant Biol, 2006, 57:19-53.
[4] Sunkar R, Chinnusamy V, Zhu J, et al. Small RNAs as big players in plant abiotic stress responses and nutrient deprivation[J]. Trends in Plant Science, 2007, 12(7):301-309.
[5] Khraiwesh B, Zhu JK, Zhu J. Role of miRNAs and siRNAs in biotic and abiotic stress responses of plants[J]. Biochim Biophys Acta,2012, 1819(2):137-48.
[6] Covarrubias AA, Reyes JL. Post-transcriptional gene regulation of salinity and drought responses by plant microRNAs[J]. Plant Cell Environ, 2010, 33(4):481-489.
[7] Zhao M, Ding H, Zhu JK, et al. Involvement of miR169 in the nitrogen-starvation responses in Arabidopsis[J]. New Phytol,2011, 190(4):906-915.
[8] Lundmark M, Korner CJ, Nielsen TH. Global analysis of microRNA in Arabidopsis in response to phosphate starvation as studied by locked nucleic acid-based microarrays[J]. Physiol Plant, 2010,140(1):57-68.
[9] Li WX, Oono Y, et al. The Arabidopsis NFYA5 transcription factor is regulated transcriptionally and posttranscriptionally to promote drought resistance[J]. Plant Cell, 2008, 20(8):2238-2251.
[10] Ni Z, Hu Z, Jiang Q, et al. GmNFYA3, a target gene of miR169, is a positive regulator of plant tolerance to drought stress[J]. Plant Mol Biol, 2013, 82(1-2):113-129.
[11] Liu HH, Tian X, Li YJ, et al. Microarray-based analysis of stressregulated microRNAs in Arabidopsis thaliana[J]. RNA, 2008,14(5):836-843.
[12] Xu MY, Zhang L, Li WW, et al. Stress-induced early flowering is mediated by miR169 in Arabidopsis thaliana[J]. J Exp Bot,2014, 65(1):89-101.
[13] Sorin C, Declerck M, Christ A, et al. A miR169 isoform regulates specific NF-YA targets and root architecture in Arabidopsis[J]. New Phytol, 2014, 202(4):1197-1211.
[14] Liu HJ, Chen LG, Zhu PP, et al. Effect of hyacinth mulching on rice(Oryza sativa L. )uptake and utilization of nitrogen[J]. Environmental Science, 2011, 32(5):1292-1298.
[15] 潘雅姣, 傅彬英, 王迪, 等. 水稻干旱胁迫诱导DNA甲基化时空变化特征分析[J]. 中国农业科学, 2009, 42:3009-3018.
[16] Zhao B, Ge L, Liang R, et al. Members of miR-169 family are induced by high salinity and transiently inhibit the NF-YA transcription factor[J]. BMC Mol Biol, 2009, 10:29.
[17] Hoagland DR, Arnon DI. The water-culture method for growing plants without soil[M]. 2nd ed. Circular. California Agricultural Experiment Station, 1950:347.
[18] Rio DC, Ares M, Hannon GJ, et al. Purification of RNA using TRIzol(TRI reagent)[J]. Cold Spring Harbor Protocols, 2010(6):pdb. prot5439.
[19] Livak KJ, Schmittgen TD. Analysis of relative gene expression data using Real-Time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4):402-408.
[20] Yang B, Ma HY, Wang XM, et al. Improvement of nitrogen accumulation and metabolism in rice(Oryza sativa L. )by the endophyte Phomopsis liquidambari[J]. Plant Physiol Biochem,2014, 82:172-182.
[21] Liu Q, Zhang YC, Wang CY, et al. Exp ression analysis of phytohormone-regulated microRNAs in rice, implying their regulation roles in plant hormone signaling[J]. FEBS Letters,2009, 583(4):723-728.
[22] Jia X, Wang WX, Ren L, et al. Differential and dynamic regulation of miR398 in response to ABA and salt stress in Populus tremula and Arabidopsis thaliana[J]. Plant Molecular Biology, 2009, 71(1-2):51-59.
[23] Zhao B, Liang R, Ge L, et al. Identification of drought-induced microRNAs in rice[J]. Biochem Biophys Res Commun, 2007,354(2):585-590.
[24] Jia W, Wang Y, et al. Salt-stress-induced ABA accumulation is more sensitively triggered in roots than in shoots[J]. Journal of Experimental Botany, 2002, 53(378):2201-2206.
[25] Daszkowska-Golec A, Szarejko I. The molecular basis of ABA-mediated plant response to drought[J]. Agricultwre and Biological Scieurs, 2013:103-133.
[26] Wilkinson S, Davies WJ. ABA-based chemical signalling:the co-ordination of responses to stress in plants[J]. Plant, Cell & Environment, 2002, 25(2):195-210.
[27] Chaves MM, Maroco JP, Pereira JS. Understanding plant responses to drought—from genes to the whole plant[J]. Functional Plant Biology, 2003, 30(3):239-264.
(责任编辑 李楠)
Dynam ic Expression of m iR169o and Its Target Genes OsNF-YAs in the Early Response to W ater Deficiency in Rice
Chen Yutong Chen Huamin Yu Chao Amy Thein Tian Fang He Chenyang
(State Key Laboratory for Biology of Plant Disease and Insect Pests,Institute of Plant Protection,Chinese Academy of Agricultural Science,Beijing100193)
MicroRNAs(miRNAs)is noncoding RNAs and play important roles in plant development and response to various environment stresses. Recent evidence have indicated that miR169 is upregulated response to drought stress, and overexpression of miR169 contributes to improve the tolerance to drought stress in plant. However, it is still unclear to the expression pattern of miR169 under drought stress and the regulatory mechanism response to drought stress in rice. In this study, the dynamic expression patterns of miR169o and its target gene NF-YAs(Nuclear Factor Y A subunit, NF-YA)in the root, stem and leaf of rice were analysed systematically by qRT-PCR after water deficiency treatment. Generally, miR169o showed up-regulated after water deficiency stress treatment compared to that before treatment, while target gene NF-YAs showed imperfect reverse expression pattern to miR169o. In addition, the expression pattern and abundance of miR169o in rice root, stem and leaf suggested that the expression of miR169o is tissue-specific.
miR169o;OsNF-YA s;rice;water deficiency;expression pattern
10.13560/j.cnki.biotech.bull.1985.2015.08.034
2015-03-09
国家转基因生物新品种培育重大专项(2014ZX08010-005,2014ZX08001-002),国家“863”计划(2012AA101504)
陈禹彤,女,硕士研究生,研究方向:分子植物病理学;E-mail:cythxmichelle@163.com
陈华民,副研究员,硕士生导师,研究方向:分子植物病理学;E-mail:hmchen@ippcaas.cn

