番茄HDACs家族基因在胁迫条件下的表达分析
李涛 苏慧慧 李植良 徐小万 王恒明 李颖 黎振兴

摘 要 组蛋白去乙酰化酶(Histone deacetylases,HDACs)家族基因在植物的生长发育、器官构建及逆境胁迫和激素信号应答中发挥重要作用。利用生物信息学方法对番茄的HDACs家族成员、分布及结构和功能等进行分析。结果表明,番茄HDACs家族包含15个成员,分为3个亚家族。遗传进化分析表明,番茄HDACs家族成员与拟南芥HDACs家族具有相似分类。利用实时荧光定量PCR对番茄HDACs家族基因的组织表达分析表明,HDACs具有组织特异性表达差异,SlHDT1、SlHDT2和SlHDT3在根中表达较高,而SlHDA1、SlHDA3、SlHDA5、SlHDA6在果实发育过程中表达较高;利用RT-PCR对番茄HDACs的胁迫响应分析表明,在盐、SA、ABA、高温和低温胁迫条件下,15个番茄HDACs成员的表达模式不同,其中部分基因的表达水平被显著地诱导增加或者降低,推测这些基因很可能参与了调控番茄逆境胁迫条件下的防御应答反应。结果将为进一步解析番茄HDACs家族基因的功能奠定基础。
关键词 番茄;组蛋白去乙酰化酶;表达分析;氧化胁迫;青枯病
中图分类号 S641.2 文献标识码 A
Abstract Histone acetylation and deacetylation play an important role in the regulation of eukaryotic gene expression, and implicated in plant development, organ formation, stress response, and hormonal signaling. In this study, our analysis revealed the presence of 15 genes encoding HDAC proteins in the tomato genome, the distribution on the chromosome, the structure and function of proteins were analyzed. HDAC proteins could be classified into 3 groups based on phylogenetic analysis. Ten of them belonged to RPD3/HDA1, and other members had similar classification with Arabidopsis thaliana, which was supported with the organization of predicted conserved putative motifs in HDAC proteins. The analysis of the developmental tomato expression profiling data with Q-PC, indicated that SlHDT1, SlHDT2 and SlHDT3 were highly expressed in roots, in addition, the transcripts of SlHDA1, SlHDA3, SlHDA5 and SlHDA6 were accumulated in fruits. Fifteen HDACs genes showed distinct expression patterns in response to stresses of salt, SA, ABA, high temperature and cold, the expressions of several genes were significantly up/down regulated, implying that these members might participate in regulating the defense response against abiotic stresses. The results would provide a very useful reference for the cloning and functional analysis of each member of HDAC gene family in Solanaceae crops.
Key words Tomato; Histone deacetylation; Gene expression; Oxidative stress; Bacterial wilt
doi 10.3969/j.issn.1000-2561.2015.11.012
植物在生长过程中会不断受到来自环境的生物和非生物胁迫,非生物胁迫如干旱、冷害、高盐等,生物胁迫如细菌、真菌、病毒等[1]。植物经胁迫诱导后发生一系列生理生化变化,如多种信号途径的激活,功能性调控蛋白的诱导表达等,来应变和抵抗这些不良环境对其生长发育的影响[2]。组蛋白修饰在基因表达调控方面扮演重要角色,组蛋白通过组蛋白乙酰转移酶(Histone acetyltransferase,HATs)促进基因表达,而组蛋白去乙酰化酶(Histone deacetylase,HDACs)抑制基因表达。组蛋白乙酰化酶(HATs)根据序列特征划分为5个不同的家族:GNAT、MYST、p300/CREB結合蛋白(CBP)辅激活物、TAF II250、HATs。组蛋白去乙酰化酶(HDACs)在植物中划分为RPD3/HDA1、SIR2和HD2这3个亚家族[3],其中前2个分别与酵母RPD3/HDA1、SIR2家族同源,而HD2家族是植物特有的家族[4]。有研究结果表明;组蛋白乙酰化和去乙酰化主要参与植物的生长发育,其中包括根发育[5]、花发育[6]、配子体发育[7]、器官生长过程中细胞的增殖等[8];也参与植物应对外界环境变化,如光信号[9]、盐胁迫[10]、冷害[11]、热胁迫[12]、脱落酸(abscisic acid,ABA)信号途径,以及一些其他的激素信号途径[13]。
前人对植物如拟南芥[3]、水稻[14]、玉米[15]、大麦[16]、葡萄[17]和番茄[18-21]组蛋白乙酰化基因家族进行生物信息学和基因功能研究;Pandey等[3]对植物组蛋白乙酰化家族基因进行了分析,并首次发现番茄HDT1101、HDT1102和HDT1103隶属于HD2家族。Aiese等[18]2013年通过全基因组系统分析了番茄中组蛋白甲基化和乙酰化家族成员进化关系,同时利用S. pennellii近等基因系(ILs)进行定位和果实发育过程中的表达分析,研究发现番茄中有32个组蛋白乙酰基转移酶(Histone acetyltransferase,HATs)和15个组蛋白去乙酰基酶(Histone deacetylase,HDACs),而且这些酶在番茄果实发育过程中具有不同的表达变化;卢晶霞[18-19]和Zhao等[20]2014年通过酵母双杂交分析发现,番茄HDAC可能参与番茄的果实发育。而番茄HDACs在氧化胁迫研究方面还未见报道[21]。鉴于HDACs家族的重要生理功能,本研究利用生物信息学方法对番茄HDACs家族开展氧化胁迫及接种青枯病条件下的表达进行分析,为番茄HDACs家族基因的功能分析提供基础信息,也为分子植物育种提供候选基因。
1 材料与方法
1.1 材料
番茄测序品种‘Heinz 1706种植于广东省农业科学院蔬菜研究所温室,在营养生长阶段取根、茎、叶,生殖生长阶段取花和成熟果实;选取‘Heinz 1706生长饱满的种子,播种于1/2 MS培养基置于光照培养箱培养,生长温度为28 ℃,光照3 000 lx,12 h的黑暗和光照,相对湿度75%,待种子萌发后移栽至1/2MS固体培养基,培养2周。选取生长一致幼苗各30株进行胁迫处理。盐处理:将幼苗置于200 mmol/L NaCl溶液中;ABA处理:将幼苗置于10 mol/L ABA溶液中;SA处理:将5 mmol/L SA喷洒植株叶面;42 ℃高温处理:将幼苗置于光照培养箱培养,温度为42 ℃,光照3 000 lx,相对湿度75%;4 ℃低温处理:将幼苗置于人工气候室,温度为4 ℃,光照3 000 lx,相对湿度75%;青枯菌接种参照苏慧慧等[22]方法,以上处理重复3次,样品分别于0、1、2、8 h取样,用液氮速冻后放入-80 ℃冰箱保存备用。
1.2 方法
1.2.1 番茄HDACs家族基因序列检索和鉴定 根据拟南芥中已经鉴定出来的HDACs基因及其编码的蛋白质序列[23],利用Hmmer3.1b1软件构建隐马氏模型序列,对从SOL[23](SGN, http://solgenomics.net, release v2.40)下载的番茄(Solannum lycopericum L.)蛋白数据进行检索和去冗余,得到候选蛋白序列。利用Pfam(http://www.sanger.ac.uk/Software/Pfam/search.shtml)[24]和SMART(http://smart.embl-heidelberg.de/)[25]對候选基因的氨基酸序列结构域进行鉴定,凡是含有HDACs基因保守结构域的蛋白即为番茄HDACs成员。在番茄基因组数据库SOL中查出了所有HDACs基因的开放阅读框长度、染色体位置、内含子个数等基本信息。利用在线工具Expasy(http://web.expasy.org/compute_pi/)进行等电点分析,利用PSORT(http://psort.ims.u-tokyo.ac.jp/form.html)[26]对番茄HDACs基因成员的亚细胞定位进行了分析。
1.2.2 番茄HDACs的分类和染色体定位分析 利用Clustal X2.1软件[27],对番茄和拟南芥HDACs成员进行多重序列比对,基于比对结果和拟南芥HDACs的分类依据,进行番茄HDACs的分类。
根据检索到的番茄HDACs基因组信息,利用番茄基因组数据(SGN,http://solgenomics.net,release v2.40),对番茄HDACs基因进行染色体定位分析。
1.2.3 番茄HDACs蛋白系统发生分析 从TAIR10(http://www.arabidopsis.org)数据库中下载拟南芥的HDACs蛋白全长序列,将候选番茄HDACs家族蛋白序列与拟南芥蛋白序列用Clustal X2.1软件进行多重匹配分析,基于比对结果,参照相关文件[28],利用MEGA5.05采用相邻连接法(Neighbor-Joining,NJ)构建系统发育树,并对构建的树进行自检(bootstrap),重复设定为1 000。
1.2.4 HDACs基因的表达分析 采用Trizol(Invitrogen)法提取总RNA,经DNaseI处理去除基因组DNA,3 g RNA经M-MLV(Rnase H-)反转录合成第1链cDNA,稀释后做模板。根据番茄数据库基因序列,利用Oligo 6.0设计引物(表1),由英潍捷基(上海)生物公司合成。HDACs组织表达利用荧光实时定量PCR反应试剂盒为SYBR Green Realtime PCR Master mix(Takara,大连),反应体系为SYBR Premix Ex TaqTM II(TliRNaseH Plus)(2×)10 μL,上下游引物(10 μmol/L)各0.8 μL,DNA模板2.0 μL,无菌双蒸馏水6.4 μL,反应总体积20.0 μL。反应条件:95 ℃预变性30 s;95 ℃变性5 s,60 ℃退火20 s,40个循环;融解曲线分析95 ℃ 0 s,65 ℃ 15 s,95 ℃ 0 s。采用2-△△Ct方法计算基因表达相对量,每个基因的表达反应重复3次,以Actin2作为内参基因。利用Roche Light Cycler 480软件根据PCR扩增曲线计算实时荧光定量PCR的扩增效率。
HDACs氧化胁迫采用半定量PCR反应。反应体系:10×PCR buffer 2 μL,dNTPs(2.5 mmol/L)1.6 μL,上下游引物(表2)(10 μmol/L)各1.0 μL,cDNA模板视浓度而定,最后添加ddH2O至总体积20 μL。反应条件:95 ℃预变性3 min;95 ℃变性30 s,Tm(不同基因不同退货温度)退火30 s,72 ℃延伸30 s,30个循环;2 ℃延伸10 min,16 ℃无限延伸。采用琼脂糖凝胶法进行检测,以Actin1作为内参基因。
2 结果与分析
2.1 番茄HDACs家族基因鉴定
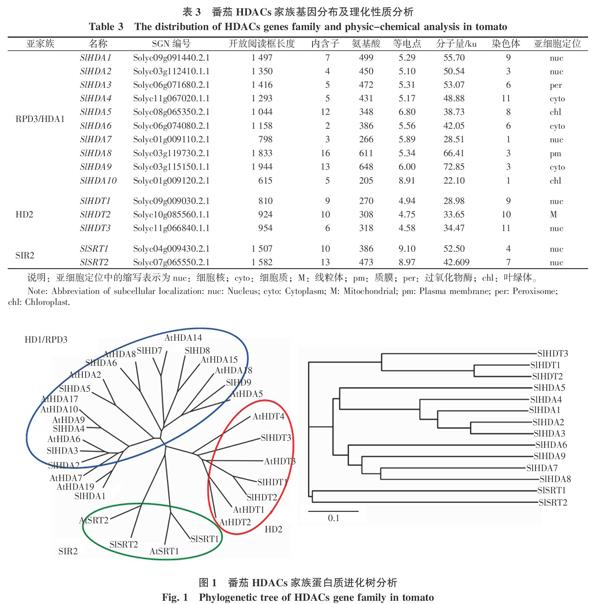
在番茄基因组数据库SGN v2.40对番茄HDACs家族基因进行鉴定,结合Pfam和SMART在线分析结构域,共得到15个含有HDACs结构域的HDACs蛋白序列(表3)。其中番茄HDACs氨基酸长度为205~648,分子量为22.10~72.84 ku。根据其发育进行树将其分为3个亚家族:RPD3/HDA1、SIR2、HD2,其中10个HDACs属于RPD3/HDA1亚家族成员,3个属于HD2亚家族成员,2个属于SIR2亞家族成员。亚细胞定位发现番茄中HDACs家族基因在细胞中多数成员定位在细胞核内,也有少部分定位在叶绿体、过氧化物酶和细胞质内。其中,SIR2亚家族的基因定位在细胞核内,这15个基因在染色体上分布不均匀(表3),在3号染色体上有3个基因分布,分别为SlHDA2、SlHDA8和 SlHDA9;SlHDA7和SlHDA10分布于2号染色体上;SlHDA3和SlHDA6分布在6号染色体上;SlHDT2和SlSRT1存在于10号染色体上;另外,SlSRT1在4号染色体上,SlSRT2在7号染色体上,SlHDA5在8号染色体上,SlHDT1在9号上,SlHDA5存在于12号染色体上。
由番茄和拟南芥HDACs家族系统进化分析结果表明:相同类型的HDACs在进化图中相离很近,如HD2亚家族中的SlHDT1、SlHDT2、AtHDT1和AtHDT2,同样,SIR亚家族的SlSTR1、SlSRT2、AtSRT1和AtSRT2相离较近(图1)。
2.2 番茄HDACs家族基因组织表达分析
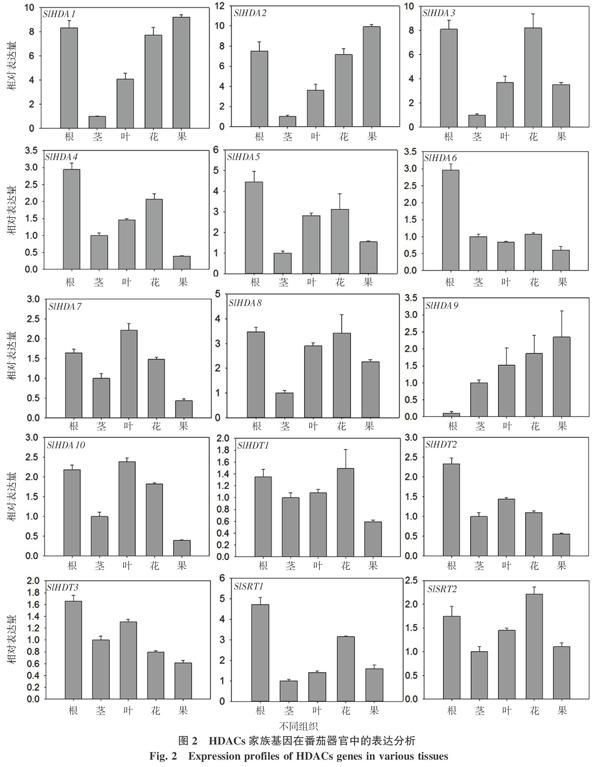
番茄HDACs基因组织表达分析见图2所示。结果表明:15个基因在各个组织中均有表达且具有组织表达特异性,其中,除了SlHDA9基因,其余HDACs家族基因在根组织中有较强的表达,尤其是SlHDA1、SlHDA3、SlHDA2、SlSRT1和SlHDA5表达非常强;而SlHDA1和SlHDA2在果实发育过程中表达最高,SlHDA5、SlHDA8和SlHDA9在果实发育过程中表达较高,而SlHDA4和SLHDA10在果实中表达较低;HD2亚家族的3个成员(SlHDT1、SlHDT2、SlHDT3)在根中的表达比其它组织高。SIR2亚家族的2个成员在根和花组织中表达较高。
2.3 番茄HDACs家族基因胁迫条件下的表达分析
番茄HDACs家族基因对胁迫的响应见图3所示。结果发现,在ABA处理条件下,HDACs家族基因具有不同的表达模式,其中,SlHDA2、SlHDA3、SlHDA4、SLHDA5、SlHDA7、SlHDA9和SlHDT1、SlHDT3持续上调表达;而SlHDA1、SlHDT2和SlSRT1表达下调。在盐处理条件下,SlHDA1、SlHDA3、SlHDA9、SlHDT3、SlSRT1上调表达;SlHDA4、 SlHDT1下调表达。在SA处理条件下,SlHDA3、SlHDA5、SlHDT2、SlHDT3表达上调;SlHDA8、SlHDT1和SlSRT1下调表达。高温处理和低温处理条件下,基因表达趋势相反,高温下调,低温上调。
为进一步研究番茄HDACs家族基因功能,用RT-PCR(图4)对番茄HDACs家族基因接种青枯菌后的表达差异进行分析。从图4中可知,番茄品种1 706接种青枯菌后在0、1、2 h短时间处理后,基因SlHDA3、SlHDA7、SlHDT2表达上调,而基因SlHDA8、SlHDT3、SlSRT1表达下调;番茄Henz1706接种青枯菌后在0、1、3、10 d,基因SlHDA5和SlHDT1上调表达,基因SlHDA3、SlHDA7和SlHDT2下调表达;需要指出的是基因SlHDA7、SlHDT2,在短时间处理内,前者表达上调,后者下调,但在长时间处理后,基因表达情况相反。
3 讨论与结论
组蛋白乙酰化和去乙酰化在植物生长发育和逆境胁迫响应方面发挥重要作用[30]。国内外学者在模式植物如拟南芥、玉米、水稻、大麦、葡萄和番茄等组蛋白乙酰化基因家族和基因功能方面开展了大量研究[3,31]。随着番茄基因组计划的完成[23],从全基因组层面鉴定和研究基因家族的分类、进化特征和功能预测是番茄功能基因研究的热点。本研究通过对番茄基因组组蛋白去乙酰化家族成员开展生物信息学分析,发现番茄基因组含有15个HDAC成员,分属于3个亚家族,且分布在8条染色体上;并对其蛋白进化关系、保守结构域、基序开展分析。在番茄HDACs家族基因研究方面,Aiese等[17]在番茄基因组中找到14个成员,其中RPD3/HDA1亚家族有9个成员,本研究与卢晶霞[18-19]和Zhao等[20]研究结果一致,发现番茄HDACs有15个成员且RPD3/HDA1有10个成员。
在组织表达研究方面,Aiese等[17]通过表达谱芯片分析了HDACs在果实发育过程中的表达模式,发现SlHDA9与拟南芥AtHDA5和AtHDA18表达模式相似,在根中表达较强,SlHDA5、SlHDA6和SlHDA7在果实1 cm到破熟期10 d的表达较高;SlHDA1和SlHDA3与拟南芥AtHDA6和AtHDA19氨基酸序列相似性较高在破熟10 d与成熟期表达较强,拟南芥AtHDA6和AtHDA19基因在花发育,配子发育和其它生物学过程中具有重要功能[31];SlSRT1主要在芽和1 cm果实大小表达较高,而SlSRT2在花和破熟10 d表达较高。SlHDT1和SlHDT2在果实1 cm和3 cm表达较强,而SlHDT3在果实3 cm和绿熟期表达较强。卢晶霞[18-19]与Zhao等[20]利用荧光定量PCR对15个HDACs家族基因开展组织表达分析发现:SlHDA1、SlHDA3和SlHDA4在花中表达较高而在果实发育过程中表达较低;SlHDA5在花和10 d的果实中表达较高而在果实发育和成熟期表达较低;SlHDA6在红熟期表达较高而在叶片、花和果实中表达较低;SlHDA7、SlHDA9和 SlHDA10在子叶、真叶和第五周叶子中均有高水平的表达;SlHDA8特异性的在授粉后10 d的果实中有较高水平的表达;SlHDT1和SlHDT3在根和下胚轴表达较高,SlSRT1和SlSRT2在真叶、花和花后10 d的果实表达较高。本研究结果表明,SlHDT1、SlHDT2和SlHDT3在根中表达较高,而SlHDA1、SlHDA3、SlHDA5、SlHDA6在果实发育过程中表达较高,SRI2的,2个成员在花中表达较高,以上结果与前人的研究结果基本一致,表明番茄HDACs家族成员在番茄不同生长发育阶段起着不同作用。
组蛋白去乙酰化酶在环境压力应答过程中扮演重要的角色[29]。Alinsug等[32]在利用拟南芥芯片数据分析结果表明:在盐胁迫处理条件下,AtHDA2、AtHDA6和AtHDA14表达上调,AtHDA5和AtHDA7表达下调;高温处理条件下,AtHDA5、AtHDA6、AtHDA7、AtHDA8和AtHDA14上调表达,AtHDA9显著下调表达;低温条件下,AtHDA18和AtHDA19显著上调表达,AtHDA2、AtHDA5、AtHDA6、AtHDA7、AtHDA8、AtHDA9和AtHDA14显著下调表达;在激素处理条件下,AtHDA7和AtHDA9受ABA和SA诱导下调表达,AtHDA5和AtHDA18受SA诱导显著上调;在生物胁迫研究方面,AtHDA6受线虫侵染显著表达上调,而在丁香假单胞菌则显著下调表达。后续有研究结果表明,拟南芥AtHDA6参与长期冷胁迫相关基因的调控且在抗冷胁迫累积响应中发挥重要功能,与野生型相比在冷胁迫效应累積的hda6突变体中,表现出抗冷胁迫效应增强现象[33];AtHDA7、AtSin3和AtHDA19三者组成转录抑制复合体,参与调控ABA和非生物胁迫响应[34]。本研究结果发现:SlHDA1、SlHDA3、SlHDA9和SlSRT1受盐胁迫上调表达,SlHDA4和SlHDT1下调表达;大多数番茄HDACs基因受高温诱导下调表达,低温上调表达;ABA处理条件下,SlHDA5、SlHDA7、SlHDT3上调表达,SlHDA1、SlHDA3、SlSRT1下调表达;SA处理条件下,SlHDA3、SlHDA5、SlHDT2、SlHDA3上调表达,SlHDA8、SlHDT1和SlSRT1下调表达;在青枯菌短时间(2 h)处理条件下,SlHDA3、SlHDA7和SlSRT1持续上调表达;SlHDA1、SlHDT1、SlHDT3和SlSRT1则是明显下调表达,而在青枯菌长时间(10 d)处理条件下,SlHDA5、SlHDT1、SlHDT3持续上调表达。综合结果表明,番茄组蛋白去乙酰化家族基因在生物和非生物胁迫条件下具有重要作用。
参考文献
[1] 李书粉, 孙富丛, 肖理慧, 等. 植物对非生物胁迫应答的转录因子及调控机制[J]. 西北植物学报, 2006, 26(6): 1 295-1 300.
[2] 王 迪, 傅彬英, 张立军. 植物表观遗传变化与环境压力研究进展[J]. 分子植物育种, 2008, 6(3): 569-573.
[3] Pandey R, Muller A, Napoli CA, et al. Analysis of histone acetyltransferase and histone deacetylase families of Arabidopsis thaliana suggests functional diversification of chromatin modification among multicellular eukaryotes[J]. Nucleic acids research, 2002, 30(23): 5 036-5 055.
[4] Lusser A, Brosch G, Loidl A, et al. Identification of maize histone deacetylase HD2 as an acidic nucleolar phosphoprotein[J]. Science, 1997, 277: 88-91.
[5] Kornet N, Scheres B. Members of the GCN5 histone acetyltransferase complex regulate PLETHORA-mediated root stem cell niche maintenance and transit amplifying cell proliferation in Arabidopsis[J]. The Plant cell, 2009, 21(4): 1 070-1 079.
[6] Han S K, Song J D, Noh Y S, et al. Role of plant CBP/p300-like genes in the regulation of flowering time[J]. The Plant journal, 2007, 49(1): 103-114.
[7] Latrasse D, Benhamed M, Henry Y, et al. The MYST histone acetyltransferases are essential for gametophyte development in Arabidopsis[J]. BMC plant biology, 2008, 8: 121.
[8] Nelissen H, Fleury D, Bruno L, et al. The elongata mutants identify a functional Elongator complex in plants with a role in cell proliferation during organ growth[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(21): 7 754-7 759.
[9] Alinsug M V, Chen F F, Luo M, et al. Subcellular localization of class II HDAs in Arabidopsis thaliana: nucleocytoplasmic shuttling of HDA15 is driven by light[J]. PloS one, 2012, 7(2): e30846.
[10] Luo M, Wang Y Y, Liu X, et al. HD2C interacts with HDA6 and is involved in ABA and salt stress response in Arabidopsis[J]. Journal of experimental botany, 2012, 63(8): 3 297-3 306.
[11] Kuang J F, Chen J Y, Luo M, et al. Histone deacetylase HD2 interacts with ERF1 and is involved in longan fruit senescence[J]. Journal of experimental botany, 2012, 63(1): 441-454.
[12] Bharti K, Von Koskull-Doring P, Bharti S, et al. Tomato heat stress transcription factor HsfB1 represents a novel type of general transcription coactivator with a histone-like motif interacting with the plant CREB binding protein ortholog HAC1[J]. The Plant cell 2004, 16(6): 1 521-1 535.
[13] Li W, Liu H, Cheng Z J, et al. DNA methylation and histone modifications regulate de novo shoot regeneration in Arabidopsis by modulating WUSCHEL expression and auxin signaling[J]. PLoS genetics, 2011, 7(8): e1002243.
[14] Liu X, Luo M, Zhang W, et al. Histone acetyltransferases in rice(Oryza sativa L.): phylogenetic analysis, subcellular localization and expression[J]. BMC plant biology, 2012, 12: 145.
[15] Kapazoglou A, Tondelli A, Papaefthimiou D, et al. Epigenetic chromatin modifiers in barley: IV. The study of barley Polycomb group(PcG)genes during seed development and in response to external ABA[J]. BMC plant biology, 2010, 10(1): 73.
[16] Aquea F, Timmermann T, Arce-Johnson P. Analysis of histone acetyltransferase and deacetylase families of Vitis vinifera[J]. Plant physiology and biochemistry: PPB/Societe francaise de physiologie vegetale, 2010, 48(2-3): 194-199.
[17] Aiese Cigliano R, Sanseverino W, Cremona G, et al. Genome-wide analysis of histone modifiers in tomato: gaining an insight into their developmental roles[J]. BMC Genomics, 2013, 14: 57.
[18] 盧晶霞. 番茄组蛋白去乙酰化酶基因家族分子特性与功能初步分析[D]. 北京: 中国科学院大学, 2014.
[19] 卢晶霞, 杨松光, 吴克强. 番茄组蛋白去乙酰化酶HD2家族生物信息学及表达模式分析[J]. 广东农业科学, 2014, 41(12): 138-143.
[20] Zhao L, Lu J, Zhang J, et al. Identification and characterization of histone deacetylases in tomato(Solanum lycopersicum)[J]. Front Plant Sci, 2014, 5: 760.
[21] 李 涛, 黎振兴, 李植良, 等. 番茄组蛋白去乙酰化家族基因的分离鉴定及其在抗青枯病中的功能研究[J]. 园艺学报, 2014, 41(S): 2 696.
[22] 苏慧慧, 李 涛, 黎振兴, 等. 广州地区番茄青枯病菌的系统发育分析及致病力评价[J]. 热带作物学报, 2014, 35(11): 2 284-2 288.
[23] The tomato genome consortium. The tomato genome sequence provides insights into fleshy fruit evolution[J]. Nature, 2012, 485: 635-641.
[24] Punta M, Coggill P C, Eberhardt R Y, et al. The Pfam protein families database[J]. Nucleic acids research, 2012, 40: 290-301.
[25] Letunic I, Doerks T, Bork P. SMART 7: recent updates to the protein domain annotation resource[J]. Nucleic acids research, 2012, 40(1): 302-305.
[26] Horton P, Park K J, Obayashi T, et al. WoLF PSORT: protein localization predictor[J]. Nucleic Acids Research, 2007, 35(2): 585-587.
[27] Thompson J D, Gibson T J, Plewniak F, et al. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic acids research, 1997, 25(24): 4 876-4 882.
[28] Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular biology and evolution, 2011, 28(10): 2 731-2 739.
[29] 潘麗娜. 表观遗传修饰调控非生物胁迫应答提高植物抗逆性[J]. 遗传, 2013, 35(6): 745-751.
[30] Liu X, Yang S, Zhao M, et al. Transcriptional repression by histone deacetylases in plants[J]. Mol Plant, 2014, 7(5): 764-772.
[31] Yuan L, Liu X, Luo M, et al. Involvement of histone modifications in plant abiotic stress responses[J]. J Integr Plant Biol, 2013, 55(10): 892-901.
[32] Alinsug M V, Yu C W, Wu K. Phylogenetic analysis, subcellular localization, and expression patterns of RPD3/HDA1 family histone deacetylases in plants[J]. BMC plant biology, 2009, 9(1): 37.
[33] To T K, Nakaminami K, Kim J M, et al. Arabidopsis HDA6 is required for freezing tolerance[J]. Biochemical and biophysical research communications, 2011, 406(3): 414-419.
[34] Chen L T, Wu K. Role of histone deacetylases HDA6 and HDA19 in ABA and abiotic stress response[J]. Plant signaling & behavior, 2010, 5(10): 1 318-1 320.

