基于发卡探针DNA/G—四链体模拟酶结构转化的高灵敏无标记荧光检测DNA
宋璐娜 张永花 +薄红艳 高强


摘 要 建立了基于目标DNA诱导的发卡探针DNA转化为G-四链体DNA过氧化物模拟酶的非标记荧光增强型DNA检测新体系。发卡探针DNA即分子信标探针由两部分构成,环状部分与目标DNA互补,茎部一端由一条富G序列构成。无目标DNA存在时,分子信标处于闭合状态,形成发卡结构; 富G序列由于部分处于杂交状态,无法形成G四链体。当目标DNA与发卡探针DNA杂交并打开发卡结构,富G序列解链并自发折叠成G-四链体结构。G-四链体结构与血红素结合形成DNA模拟酶,催化H2O2还原的同时将无荧光的底物10-乙酰基-3,7-二羟基吩恶嗪(ADHP)氧化成荧光产物。通过测量荧光信号,实现对目标DNA的定量检测。优化后的体系检测条件为pH 8.0, 10 mmol/L K+, 0.2 μmol/L Hemin, 50 μmol/L ADHP。在优化的条件下,目标DNA在0.005~1.0 nmol/L浓度范围内与体系荧光信号呈线性关系,检出限为3.0 pmol/L。本方法可以区分完全互补和单碱基错配的目标DNA。
关键词 荧光; DNA; 发卡探针; G-四链体模拟酶; 结构转化
1 引 言
近年来,DNA检测在临床诊断、基因变异识别、环境监测、食品安全以及生物学研究等领域的应用愈加广泛,显示出良好的应用前景,引起了研究者的广泛关注[1]。常见的DNA检测方法包括聚合酶链式反应 (PCR) 法[2]、质谱法[3]、光谱法[4]、色谱法[5]、电化学法[6]等。在光谱分析中,荧光法具有操作简单、检测快速、灵敏度高和选择好等优点,是DNA定量检测时应用最广泛的方法之一[7]。
分子信标(Molecular beacon,MB)是一种常用的特异性检测目标DNA或RNA的DNA探针[8]。常规的MB茎环结构的两端通常需要标记荧光基团和淬灭基团,在无目标物存在时闭合形成发卡式的茎环结构,淬灭基团通过荧光共振能量转移的方式淬灭发光基团发出的荧光。当目标DNA与MB的环状区特异杂交之后,闭合的茎环发卡结构被打开,发光基团因远离淬灭基团而发出荧光,根据荧光信号的强度来定量目标DNA。得益于发卡状茎环结构,基于MB的DNA检测方法通常具有较高的选择性和灵敏度,但标记过程较为复杂耗时。此外,目标DNA和MB荧光标记分子的1∶1的比例关系也严重制约了基于MB方法灵敏度的提高[9]。因此,开发高灵敏度的免标记MB逐渐引起研究者的关注[10]。
近年来,DNA过氧化物模拟酶(DNAzyme)因其表现出过氧化物酶的活性,且具有稳定性好、成本低、易合成、易修饰等优点而成为传感器信号放大研究的热点[11~13]。DNAzyme是富含G碱基的单链寡核苷酸在一定条件下折叠成G-四链体结构,再与血红素结合形成的复合物。利用DNAzyme的催化活性,可以实现多种物质的高灵敏度检测,如金属离子[14]、小分子[15]、蛋白质[16]和DNA[17]等。在大部分过氧化物模拟酶催化体系中,主要采用2,2-联氮基双( 3-乙基苯并噻唑啉-6-磺酸) 二铵盐( ABTS) 做为酶底物,通过比色法定量[18]。虽然比色分析法简单,但检测实际样品时干扰较多且灵敏度较荧光法低。因而开发基于DNAzyme催化活性的荧光检测体系逐渐引起了研究者的兴趣[19]。
本研究设计了免标记的发卡式荧光DNA分子探针,当探针与目标DNA特异性杂交后,发卡结构被打开,探针DNA的一部分会自发形成G-四链体,在血红素存在时表现出过氧化物酶的活性。以无荧光的10-乙酰基-3,7-二羟基吩恶嗪(ADHP)为模拟酶底物,利用其氧化产物具有荧光的性质实现了高灵敏的DNA检测。2 实验部分
2.1 仪器与试剂
UV-2400PC紫外可见分光光度计(日本岛津工公司); F-7000 荧光仪(日本日立公司); MIR系列PCR仪(东胜创新公司)。
ADHP、30% H2O2、三羟甲基氨基甲烷(Tris)、TritonX-100(Sigma-Aldrich公司); 血红素(Hemin)、二甲基亚砜(DMSO)、醋酸钾(Alfa Aesar公司); 实验用水均为超纯水(≥18.2 Ω·cm)。Tris-HAc(pH 8.0)缓冲液(10 mmol/L Tris,10 mmol/L HAc,0.05%(w/V)TritonX-100)。Hemin 用 DMSO 配成5 mol/L储备液,-20℃暗处保存。ADHP 用DMSO配成20 mmol/L储备液,冷冻保存。使用时用Tris-HAc (pH 8.0) 缓冲液稀释到所需浓度。
2.2 实验方法
将目标DNA样品溶液加入DNA探针溶液中,在室温下反应1 h,使探针发卡结构打开。加入适量Tris-HAc缓冲液( pH 8.0 )及血红素,在室温下反应40 min,以形成DNA过氧化物模拟酶体系。然后,将ADHP和H2O2溶液加入到溶液中,室温反应1 h。用荧光仪测定溶液荧光,激发波长为540 nm,发射波长为581 nm。
3 结果与讨论
3.1 检测原理
免标记荧光检测DNA的原理如图1所示。非标记的发卡型DNA探针的茎状部分一端是富含G碱基的序列,与分子信标另一端茎状部分互补。
无目标序列存在时,分子信标处于闭合状态形成发卡结构,富含G碱基的序列无法形成G-四链体。当加入目标DNA后,分子信标环状部分与目标DNA进行杂交导致发卡结构被打开,从而使富含G碱基的序列游离出来,在K+和Hemin作用下形成具有过氧化物酶活性的DNA模拟酶。当加入H2O2和底物ADHP,本身不发荧光的ADHP被氧化成具有荧光的物质,通过测量体系的荧光信号达到检测目标DNA的目的。这种免标记分子信标的优点在于不需要在DNA探针上标记荧光基团和猝灭基团,而且由于引入了过氧化物模拟酶的催化反应,放大了信号,提高了测定灵敏度。endprint
3.2 可行性验证
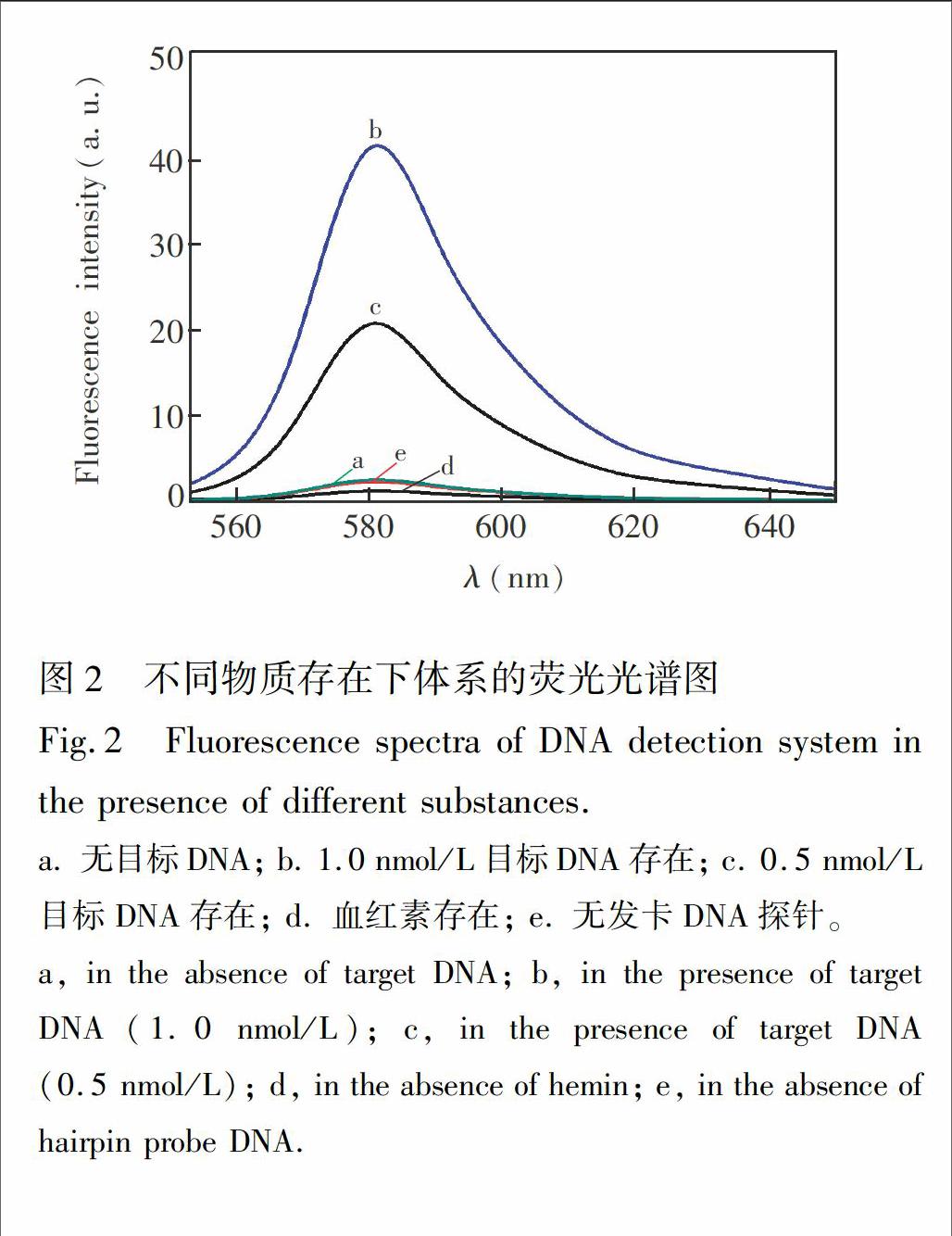
首先,对检测原理的可行性进行了验证。如图2所示,在没有目标DNA 时,体系具有微弱的荧光(图2曲线a)。由于血红素自身具有类过氧化物酶活性,但是催化活性极低,这一微弱荧光可能来自于血红素催化ADHP氧化为荧光产物。加入目标DNA 后,体系荧光信号明显增强,并且荧光强度随目标DNA的浓度的升高而增大(图2曲线b和c),荧光信号与目标DNA浓度具有相关性。为了证明荧光信号确实来自DNA模拟酶的催化作用,考察了溶液中没有血红素和没有DNA探针时体系的荧光,结果如图2曲线d和e所示。在没有血红素的情况下,体系并未表现出任何荧光,表明单独存在的G-四链体DNA本身并不会催化ADHP氧化。当没有DNA探针时,体系表现出类似于没有目标DNA时的微弱的荧光,表明目标DNA和血红素共存物也不能有效地催化无荧光的ADHP氧化为荧光产物。以上实验结果表明,只有在目标DNA存在下,与探针结合打开探针DNA的发卡结构,探针DNA的富G部分在K+存在下折叠成G-四链体DNA,然后与血红素结合形成复合物,从而表现出明显的过氧化物酶活性[19]。
3.3 实验条件的优化
为了提高目标DNA检测的灵敏度,对缓冲溶液pH值、K+浓度、血红素的浓度以及荧光底物的浓度进行了优化,结果如图3所示。从图3A可见,当缓冲溶液pH值从6.5增大到9.0,体系荧光强度随着pH值的增加先增加后减少,当pH=8.0时,荧光强度最大。这可能是由于溶液的pH值影响过氧化物模拟酶的活性。从图3B和3C可见,模拟酶的催化活性受溶液中K+、Hemin浓度的影响,这主要是由于K+影响G-四链体的折叠,而Hemin是模拟酶催化活性的核心[15],二者浓度都对模拟酶的活性有较大影响。当K+浓度为10 mmol/L,Hemin浓度为0.2 μmol/L时,体系荧光强度达到最大值。从图3D可见,随着荧光底物ADHP的浓度的增加,荧光强度不断增大,当ADHP浓度为50 μmol/L时,体系荧光强度达到稳定值。综上所述,优化的检测体系溶液组成为: pH 8.0, 10 mmol/L K+, 0.2 μmol/L Hemin, 50 μmol/L ADHP。
3.4 工作曲线和检出限
在优化的实验条件下,考察了体系的荧光强度和目标DNA浓度的关系。结果如图4所示,荧光强度随目标DNA浓度增加而增大。荧光强度和目标DNA浓度在5×10
12~1×10
9 mol/L范围内呈良好的线性关系(图4B),线性方程F=1.26+0.047c (pmol/L),相关系数r=0.954。依据信噪比为3(S/N=3),确定体系检出限为 3.0 pmol/L。本方法对DNA的检测限优于文献[20~24],证明本方法具有很高的灵敏度。
3.5 方法的选择性
发卡型DNA探针的最大特点在于其较高的单碱基变异区分能力。为了考察新型分子信标探针对完全互补和碱基错配的DNA序列的区分能力,将体系用于含单碱基错配、两碱基错配和非互补DNA序列的检测,结果如图5所示。相对于完全互补的目标DNA序列产生的荧光信号,单碱基错配DNA只产生了弱的荧光信号, 两碱基变异的DNA的荧光信号只有完全互补序列信号的十分之一, 非互补随机序列的目标DNA几乎没有引起体系荧光强度的变化。上述结果表明, 体系具有较高的单碱基区分能力,可以区分完全互补和碱基错配的DNA序列,具有较好的选择性。4 结 论
建立了高灵敏度、选择性荧光检测DNA的体系。设计了免标记的发卡型DNA分子探针,其特点在于当DNA探针发卡结构与目标DNA结合而打开后,探针序列的一部分会自发形成G-四链体,在血红素存在时表现出类过氧化物酶的活性。以无荧光的ADHP为模拟酶底物,利用其氧化产物具有荧光的性质实现了高灵敏度DNA检测。DNA模拟酶催化反应的引入,提高了检测的灵敏度,具有较高的灵敏度和较低的检出限(3.0 pmol/L)。本体系具有较好的选择性,可以区分完全互补和单碱基错配的DNA序列。
References
1 Taton T A, Mirkin C A, Letsinger R L. Science, 2000, 289(5485): 1757-1760
2 Zhang N, Appella D H. J. Am. Chem. Soc., 2007, 129(27): 8424-8425
3 Qiu F, Jiang D W, Ding Y B, Zhu J, Huang L Q L. Angew. Chem. Int. Ed., 2008, 47(27): 5009-5012
4 ZHENG Ai-Hua, ZHU Qing, XIANG Dong-Shan, HE Zhi-Ke. Chinese J. Anal. Chem., 2013, 41(3): 325-329
郑爱华, 朱 庆, 向东山, 何治柯. 分析化学, 2013, 41(3): 325-329
5 Zhang J J, Zhang L, Zhou K, Ye X, Liu C, Zhang L, Kang J, Cai C. Anal. Biochem., 2011, 413: 164-170
6 L Cui, KANG Tian-Fang, LU Li-Ping, XIONG Yue. Chinese J. Anal. Chem., 2012, 40(12): 1822-1826
吕 翠, 康天放, 鲁理平, 熊 岳. 分析化学, 2012, 40(12): 1822-1826
7 Wang F, Elbaz J, Willner I. J. Am. Chem. Soc., 2012, 134(12): 5504-5507endprint
8 Maxwell D J, Taylor J R, Nie S. J. Am. Chem. Soc., 2002, 124(32): 9606-9612
9 Zhang P, Beck T, Tan W H. Angew. Chem. Int. Ed., 2001, 40(2): 402-405
10 GUO Qiu-Ping, ZHAO Xia-Yu, XIE Qin, WANG Ke-Min, WAN Jun, YUAN Bao-Yin, TAN Yu-Yu. Chem. J. Chinese Universies, 2014, 35(8): 1646-1651
郭秋平, 赵下雨, 谢 琴, 王柯敏, 万 俊, 袁宝银, 谭誉宇. 高等学校化学学报, 2014, 35(8): 1646-1651
11 Zhang Y F, Li B X, Jin Y. Analyst, 2011, 136(16): 3268-3273
12 Wang F A, Freage L, Orbach R, Willner I. Anal. Chem., 2013, 85(17): 8196-8203
13 DUAN Na-Na, WANG Na, YANG Wei, KONG De-Ming. Chinese J. Anal. Chem., 2014, 42(10): 1414-1420
段娜娜, 王 娜, 杨 薇, 孔德明. 分析化学, 2014, 42(10): 1414-1420
14 Zhou X H, Kong D M, Shen H X. Anal. Chem., 2010, 82(3): 789-793
15 Yang C,Lates V, Prieto-Simón B, Marty J, Yang X R. Biosens. Bioelectron., 2012, 32: 208-212
16 Huang Y, Chen J, Zhao S L, Shi M, Chen Z F, Liang H. Anal. Chem., 2013, 85(9): 4423-4430
17 Deng M G, Zhang D, Zhou Y Y, Zhou X. J. Am. Chem. Soc., 2008, 130(19): 13095-13102
18 Shimron S, Wang F A, Orbach R, Willner I. Anal. Chem., 2012, 84(2): 1042-1048
19 Golub E, Freeman R, Niazov A, Willner I. Analyst, 2011, 136(21): 4397-4401
20 Ma J L, Yin B C, Le H N, Ye B C. ACS Appl. Mater. Interfaces, 2015, 7(23): 12856-12863
21 Hu Y L, Zhang L, Zhang Y, Wang B, Wang Y W, Fan Q L, Huang W, Wang L H. ACS Appl. Mater. Interfaces, 2015, 7(4): 2459-2466
22 Zhang L B, Zhu J B, Zhou Z X, Guo S J, Li J, Dong S J, Wang E K. Chem. Sci., 2013, 4(10): 4004-4010
23 Huang J H, Su X F, Li Z G. Anal. Chem., 2012, 84(14): 5939-5943
24 Xu L G, Zhu Y Y, Ma W, Kuang H, Liu L Q, Wang L B, Xu C L. J. Phys. Chem. C, 2011, 115(33): 16315-16321endprint

