CCl4诱导大鼠纤维化肝组织microRNA差异表达及其初步分析*
刘业方,张传涛,李白雪,郭尹玲△,王宝家,唐洪屈,王 政,张 凤,杨 宇
(1.成都中医药大学基础医学院,成都 610075;2.成都中医药大学附属医院感染科,成都 610072;3.重庆市垫江县中医院肿瘤科 408300)
MicroRNA是一种大小约21~23个碱基的单链小分子RNA,microRNA可调控细胞增殖、凋亡、脂代谢/脂肪酸代谢等相关通路,在肝纤维化的发生发展过程中发挥重要作用[1-2]。肝纤维化发生发展的中心环节是肝星状细胞(HSC)的激活与增殖;microRNA可协同细胞因子如血小板衍生生长因子(PDGF)、转化生长因子(TGF)、过氧化物酶体增殖激活受体(PPAR)等促进HSC活化增殖或凋亡,从而绘制成复杂的纤维化调控基因网络图[3-4],提示microRNA可能参与调控 HSC生物学行为从而影响肝纤维化的发生发展[5]。因此,本研究采用高通量测序分析技术研究四氯化碳(CCl4)诱导的大鼠纤维化肝脏中microRNA的差异表达及初步功能,旨在揭示肝纤维化发生的机制,为今后开展防治研究提供基础。
1 材料与方法
1.1 材料
1.1.1 动物 选用体质量120g左右的Wistar雄性大鼠20只,由成都中医药大学动物实验中心提供。
1.1.2 主要试剂 橄榄油(分析纯)和CCl4(分析纯)购自成都俱进生物技术有限公司;总miRNA提取试剂盒(Ca#t.AM1560Austin TX,US)、单端测序簇生成试剂盒(Illumina,GD-300-2001)、英杰公司 SYBR 金色Ⅱ核苷酸胶体染料(Invitrogen,719644)、6%尿素缓冲液聚丙烯酰胺凝胶(Invitrogen,11041164)、Qubit®dsDNA HS分析试剂盒(Invitrogen,Q32854)、安捷伦高灵敏度试剂盒(Aglient,5067-4626)等。
1.1.3 主要仪器 艾本德公司微型离心机(Eppendorf,5418)、美国ABI公司®GeneAmp®9700型PCR系统、英杰公司 Qubit®2.0荧光仪(Invitrogen,Q32866)、Agilent 2100生物分析仪等。
1.2 方法
1.2.1 分组及造模 20只大鼠随机分为对照组(n=10)及模型组(n=10)。采用CCl4复制大鼠肝纤维化动物模型,CCl4与橄榄油之比为2∶5。模型组大鼠首次皮下注射40%CCl4油剂0.5mL/100g,以后每次皮下注射40%CCl4油剂0.2 mL/100g,每周2次,根据体质量调整CCl4用量;共注射8周。
1.2.2 样品采集与处理 取1.5cm×1.5cm×0.2cm大小肝组织2块,1块脱蜡HE染色;其余肝组织液氮冻存,用于microRNA基因测序检测。
1.2.3 microRNA提取 取肝组织,抽提总 RNA,所得总RNA经使用Qubit®2.0仪光仪,应用QubitTMdsDNA HS试剂盒检测文库浓度。质控标准为:对于Agilent microRNA Chip &qPCR:RIN≥6.0and 28s/18s>0.7;其他:RIN≥7.0 and 28s/18s>0.7。
1.2.4 建库测序 使用small RNA Sample Pre Kit构建文库,选用single-read 1×50nt multiplex程序进行测序。并对测序结果进行质控。
1.2.5 质控情况 测序结果的质控标准为每个样品提供测序后原始reads(Rawreads)数不少于5M;每向碱基质量大于20(Q20)的比例不小于85%(注:Q20为bases of Q≥20/all bases of sequencing)。
1.2.6 测序数据分析 (1)对样本小RNA测序数据进行常规小RNA测序分析、样品间表达差异分析等。(2)应用DAVID Bioinformatics Resources6.7(http://david.abcc.ncifcrf.gov/)进行基因本(GO)、pathway分析、功能注释聚类分析获得差异基因相关功能的功能富集类。(3)差异性明显的GO、Pathway分析所包含的靶基因取交集,绘制基因网络图,得到核心调控作用的microRNA和被microRNA调控的关键靶基因[6]。网络图中基因用三角形来表示,microRNA用圆形来表示,相互作用用直线来表示。
1.3 统计学处理 采用SPSS17.0软件对数据进行统计学处理,对照组与模型组比较采用配对t检验,microRNA表达组间比较采用方差分析,以P<0.05为差异有统计学意义。
2 结 果
2.1 大鼠肝组织HE染色 对照组肝细胞,形态完整,未见炎症细胞浸润、变性坏死,未见纤维化增生(图1A);模型组肝脏可见点灶状炎细胞浸润,局灶或成片空泡变性或肝细胞点灶性坏死,大多数粗大纤维间隔形成包绕假小叶结构(图1B)。
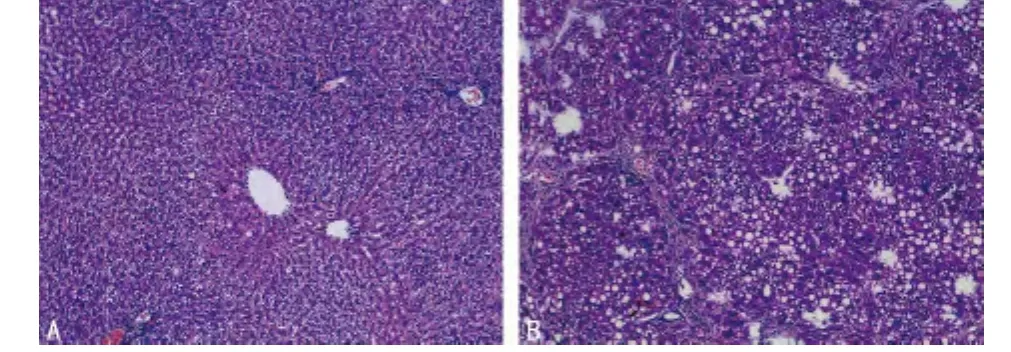
图1 HE染色结果(×200)
2.2 microRNA
2.2.1 microRNA差异谱 用火山图推断出差异microRNA的整体分布情况,从差异倍数(Fold change>1)和显著水平(P<0.05)两个方面进行评估,对差异microRNA进行筛选。共筛选到37个差异microRNA,其中模型组与对照组相比,上调29个(表1),下调8个(表2)。
2.2.2 microRNA靶基因的GO分析 模型组上调microRNA所对应的靶基因共参与84项显著性功能(表3),包括有机物质的应答、细胞凋亡的负向/正向调控、细胞增殖、细胞生长/发育/正向调控、细胞周期等;下调microRNA所对应的靶基因共参与55项显著性功能(表4),包括细胞体内化学、离子平衡、蛋白质的运输与定位、信号转导的负向/正向调控、细胞通讯、细胞增殖、负调控凋亡等。

表1 模型组较对照组表达上调的microRNA

表2 模型组较对照组表达下调的microRNA

表3 模型组较对照组表达上调的microRNA的靶基因GO分析

表4 模型组较对照组表达下调的microRNA的靶基因GO分析
2.2.3 microRNA靶基因参与的Pathway分析 模型组上调的microRNA靶基因参与的显著性信号通路71条(图2),包括PPAR信号通路、GnRH信号通路、癌症的相关通路、氨基酸的代谢、脂肪细胞因子的信号通路等;而下调靶基因参与的显著性信号通路44条(图3),包括钙信号通路、MAPK信号通路、VEGF信号通路、胰岛素信号通路、ABC转运受体等。
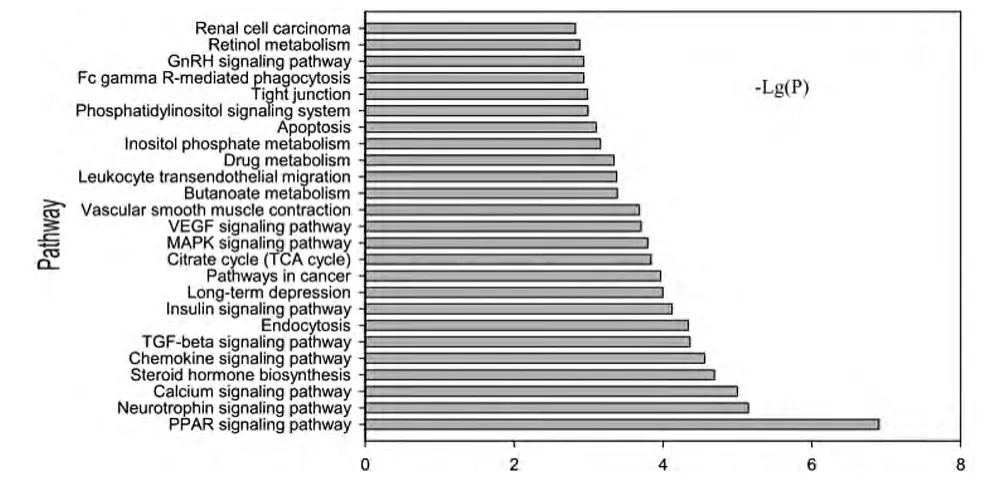
图2 模型组较对照组表达上调的microRNA的靶基因Pathway分析
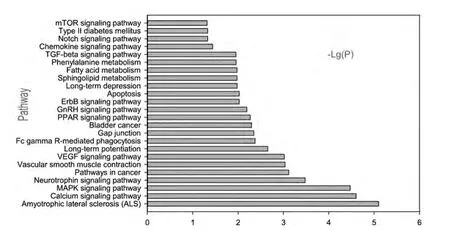
图3 模型组较对照组表达下调的microRNA的靶基因Pathway分析

图4 模型组较对照组表达上调的microRNA基因网络图
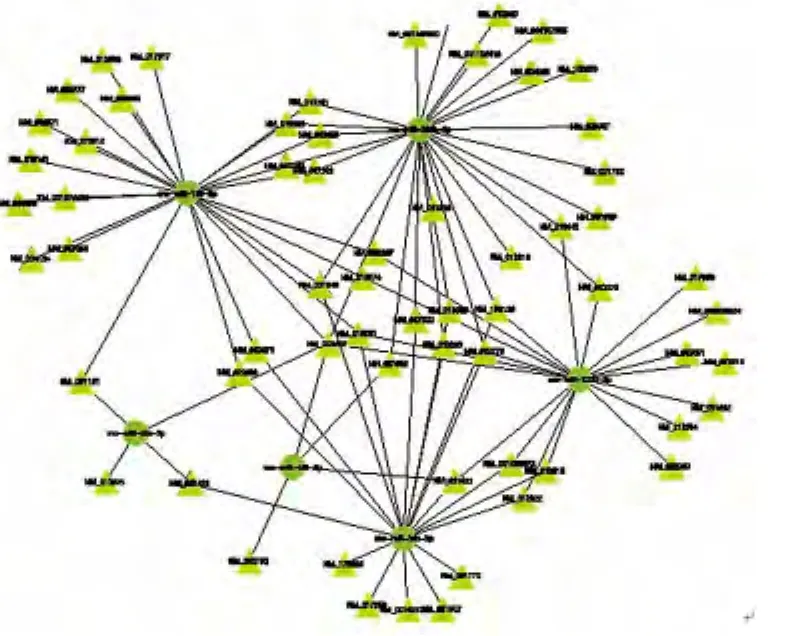
图5 模型组较对照组表达下调的microRNA基因网络图
2.2.4 差异microRNA与交集靶基因的调控网络构建 通过靶基因的功能显著性分析和信号通路的显著性分析,得到显著性GO和Pathway及其所属的靶基因,取显著性GO所属靶基因与显著性Pathway所属靶基因的交集,利用microRNA与靶基因之间的靶向调控关系,构建microRNA-基因网络图(图4、5)。发现上调的关键 microRNA主要包括 miR-184、miR-10b-5p、miR-199a-3p等,其调控的关键靶基因主要包括 NM_012671、NM_021843、NM_012775、NM_013095、NM_178866(gene bank accession number)等;下调的关键microRNA主要包括 miR-200b-3p、miR-199a-5p、miR-125b-5p等,其调控的关键靶基因主要包括及 NM_053679、NM_013095、NM_031055、NM_012775、NM_017322等。
3 讨 论
本实验通过比较正常大鼠与肝纤维化大鼠肝组织microRNA表达谱,模型组大鼠筛选出37个差异microRNA(差异倍数在2倍以上),上调29个,下调8个。其中miR-199a-3p、miR-199a-5p、miR-222-3p、miR-335等[7-10]目前已有研究报道与肝纤维化的形 成密切 相关。而let-7i-5p、miR-125b-5p、miR-155-5p等虽未查见与肝纤维化直接相关的报道,但有研究报道与其他脏器的纤维化相关,如let-7i-5p、miR-125b-5p与肺间质纤维化相关[11-12],miR-155-5p与原发性骨髓纤维化增生有密切联系[13]。本实验结果显示 miR-223-3p在对照组与模型组中的表达谱差异显著,提示该因子有可能与肝纤维化的形成相关。而目前的临床报道提示miR-223-3p可以明显作为丙肝肝癌早期和多种肝脏疾病导致的肝硬化的非侵入性生物标志物[14],而对其与肝纤维化形成的关系却未见报道。
本实验中发现的一些对照组与模型组表达谱有明显差异的microRNA,如 miR-130b-3p、miR-181b-5p、miR-204-5p、miR-193-3p、miR-152-5p、miR-375、miR-214-3p、miR-127-3p等,目前未见报道发现与纤维化有关,而在其他疾病领域有一定的研究和报道。如miR-130b-3p是在非小细胞肺癌中异常表达的小分子核糖核酸[15],miR-181b-5p可以作为星形细胞瘤肿瘤抑制基因[16],miR-375和 miR-214-3p可以早期预测口腔鳞状细胞癌[17]等。此外,本实验还发现了一些鲜有报道的microRNA,如 miR-203a-3p、miR-203b-3p、miR-582-3p、miR-411-5p、miR-92b-3p、miR-6329、miR-6216等,这些 microRNA 是否与肝纤维化形成有关尚不明确,值得进一步研究。
作者对筛选的差异microRNA靶基因功能进行GO分析发现,差异microRNA能够调控细胞增殖、凋亡、细胞周期等,还可能调控有机物质的应答、细胞体内化学、离子平衡、蛋白质的运输与定位等。Pathway分析结果提示,差异microRNA主要涉及增殖和凋亡相关信号通路、肝纤维化微循环相关通路两类,这两类信号通路与目前相关文献报道是一致的。其中增殖和凋亡相关信号通路,如细胞凋亡、Wnt信号通路、TGF-beta信号通路、Notch信号通路等已有大量研究报道证实其与肝纤维化形成密切相关[18-20];而本实验结果中提示肝纤维化微循环相关信号通路,如黏附连接、紧密连接、白细胞跨内皮迁移、磷脂酰肌醇信号系统等,虽有报道提示其在肝纤维化发病机制中起着重要作用,但相关研究报道比较少,值得进一步研究。此外,本次分析结果还发现了一些其他的信号通路,如趋化因子信号通路、T细胞信号通路、脂肪细胞因子的信号通路、胰岛素信号通路、ABC转运蛋白等;这些信号通路与肝纤维化发生的关系目前未见报道,而本次实验结果提示差异microRNA也有可能通过这些通路调控肝纤维化的发生,但需进一步实验研究。
利用图论的方法对网络中microRNA和基因在网络中的调控地位进行评价;发现在网络图中显著性最高的基因在信号通路中可能起着最显著的作用;如上调的microRNA调控的关键靶基因NM_021843受17个microRNA共同调控;NM_012671受16个microRNA共同调控;下调的microRNA调控的关键靶基因 NM_053679(DFFA)、NM_012775(TGF-β1)位于基因调控网络的中心,受5个microRNA共同调控;TGF-β1是细胞因子TGF-β超家族成员之一,因其能促进成纤维细胞的转化生长而得名;microRNA可能靶向DFFA、TGFβ1调控肝纤维化进程相关信号通路。
总之,肝纤维化发生十分复杂,本研究初步从microRNA角度发现一些可能与肝纤维化相关的新机制,不仅从microRNA调控角度进一步丰富了肝纤维化发病机制,还为今后开展肝纤维化防治研究提供了新方向,为肝纤维化的早期干预提供新的思路,具有重要现实意义和学术价值。
[1]Kim VN,Nam JW.Genomics of microRNA[J].Trends Genet,2006,22(3):165-173.
[2]Brennecke J,Cohen SM.Towards a complete description of the microRNA complement of animal genomes[J].Genome Biol,2003,4(9):228.
[3]Li G,Cai G,Li D,et al.MicroRNAs and liver disease:viral hepatitis,liver fibrosis and hepatocellular carcinoma[J].Postgrad Med J,2014,90(160):106-112.
[4]Noetel A,Kwiecinski M,Elfimova N,et al.microRNA are central players in anti-and profibrotic gene regulation during liver fibrosis[J].Front Physiol,2012,3(49):49.
[5]Guo CJ,Pan Q,Cheng T,et al.Changes in microRNAs associated with hepatic stellate cell activation status identify signaling pathways[J].FEBS J,2009,276(18):5163-5176.
[6]Joung JG,Hwang KB,Nam JW,et al.Discovery of microRNA-mRNA modules via population-based probabilistic learning[J].Bioinformatics,2007,23(9):1141-1147.
[7]Lee CG,Kim YW,Kim EH,et al.Farnesoid X receptor protects hepatocytes from injury by repressing miR-199a-3p,which increases levels of LKB1[J].Gastroenterology,2012,142(5):1206-1217,e7.
[8]Lino Cardenas CL,Henaoui IS,Courcot E,et al.miR-199a-5p is upregulated during fibrogenic response to tissue injury and mediates TGFbeta-induced lung fibroblast activation by targeting caveolin-1[J].PLoS Genet,2013,9(2):e1003291.
[9]Ogawa T,Enomoto M,Fujii H,et al.MicroRNA-221/222 upregulation indicates the activation of stellate cells and the progression of liver fibrosis[J].Gut,2012,61(11):1600-1609.
[10]Chen C,Wu CQ,Zhang ZQ,et al.Loss of expression of miR-335is implicated in hepatic stellate cell migration and activation[J].Exp Cell Res,2011,317(12):1714-1725.
[11]Huang Y,Dai Y,Zhang J,et al.Circulating microRNAs as potential biomarkers for smoking-related interstitial fibrosis[J].Biomarkers,2012,17(5):435-440.
[12]Giray BG,Emekdas G,Tezcan S,et al.Profiles of serum microRNAs;miR-125b-5p and miR223-3p serve as novel biomarkers for HBV-positive hepatocellular carcinoma[J].Mol Biol Rep,2014,41(7):4513-4519.
[13]Norfo R,Zini R,Pennucci V,et al.miRNA-mRNA integrative analysis in primary myelofibrosis CD34+cells:role of miR-155/JARID2axis in abnormal megakaryopoiesis[J].Blood,2014,124(13):e21-32.
[14]Oksuz Z,Serin MS,Kaplan E,et al.Serum microRNAs;miR-30c-5p,miR-223-3p,miR-302c-3p and miR-17-5p could be used as novel non-invasive biomarkers for HCV-positive cirrhosis and hepatocellular carcinoma[J].Mol Biol Rep,2015,42(3):713-720.
[15]Mitra R,Edmonds MD,Sun J,et al.Reproducible combinatorial regulatory networks elucidate novel oncogenic microRNAs in non-small cell lung cancer[J].RNA,2014,20(9):1356-1368.
[16]Zhi F,Wang Q,Deng D,et al.MiR-181b-5p downregulates NOVA1to suppress proliferation,migration and invasion and promote apoptosis in astrocytoma[J].PLoS One,2014,9(10):e109124.
[17]Yoon AJ,Wang S,Shen J,et al.Prognostic value of miR-375and miR-214-3p in early stage oral squamous cell carcinoma[J].Am J Transl Res,2014,6(5):580-592.
[18]Guichard C,Amaddeo G,Imbeaud S,et al.Integrated analysis of somatic mutations and focal copy-number changes identifies key genes and pathways in hepatocellular carcinoma[J].Nat Genet,2012,44(6):694-698.
[19]Zender L,Villanueva A,Tovar V,et al.Cancer gene discovery in hepatocellular carcinoma[J].J Hepatol,2010,52(6):921-929.
[20]Whittaker S,Marais R,Zhu AX.The role of signaling pathways in the development and treatment of hepatocellular carcinoma[J].Oncogene,2010,29(36):4989-5005.

