香蕉细菌性枯萎病菌超分支滚环扩增快速检测技术
王念武,陈劲松,沈建国,黄 振,翁瑞泉
(1.福清出入境检验检疫局,福建 福清350300;2.泉州出入境检验检疫局,福建 泉州362000;3.福建出入境检验检疫局,福建 福州350002;4.福州机场出入境检验检疫局,福建 福州350002)
香蕉细菌性枯萎病菌(Ralstonia solanacearum race 2,Rs2)是危害香蕉最严重的病原细菌,R. solanacearum 种以下有不同的race 型,根据寄主的不同而分为5 个races[1],香蕉细菌性枯萎病菌则属于其中的race 2。香蕉细菌性枯萎病菌主要侵害三倍体香蕉及赫蕉,可致使植株发生严重的枯萎,最终导致整株死亡[2,3]。香蕉细菌性枯萎病菌是我国禁止进境的检疫性有害生物,在我国未见分布。我国虽然对进境香蕉和香蕉种苗进行检疫,但对进境芭蕉属(Musa)和蝎尾蕉属(Heliconia)的景观类植物未进行该细菌的检测,而这些植物也可作为香蕉细菌性枯萎病菌寄主[4],对携带并传入该病菌存在一定的风险。
Nilsson et al[5]于1994年提出锁式探针检测技术。近年来,该技术以灵敏、特异、稳定等特点,广泛用于微生物鉴定、细胞原位检测、医学病原检测等领域。该技术可以在恒温下等温扩增[6],1 h 内达到109个拷贝数[7]。本研究根据香蕉细菌性枯萎病菌独有的插入序列ISRso19,设计特异性锁式探针,采用超分支滚环扩增(hyperbranched rolling circle amplification,HRCA)技术,对香蕉细菌性枯萎病菌的检测方法进行研究,以期为口岸把关和疫情监测提供新的检测方法。
1 材料与方法
1.1 供试菌株
香蕉细菌性枯萎病菌由中国检验检疫科学院赵文军研究员提供;番茄细菌性溃疡病菌(Clavibacter michiganensis subsp. Michiganensis)、杨桃细菌性斑点病菌(Pseudomonas syringae pv. Averrhoii)、番茄青枯病菌(R.solanacearum race 1)、枯草芽孢杆菌(Bacillus subtilis)、水稻细菌性条斑病菌(Xanthomonas oryzae pv.Oryzicola)、番茄细菌性叶斑病菌(P. syringae pv. Tomato)、柑橘溃疡病菌(X. citri)和西瓜果斑病菌(Acidovorax avenae subsp.Citrulli)由福建农林大学细菌实验室提供。
1.2 试剂与仪器
Taq DNA 连接酶(NEB 公司),核酸外切酶Ⅰ(NEB 公司),核酸外切酶Ⅲ(NEB 公司),Bst DNA 聚合酶(NEB 公司),2 ×PCR Master Mix(Tangen 公司),Sterilized ddH2O(NEB 公司),dNTPs(Promega 公司),100bp DNA Ladder(Tangen 公司),细菌基因组DNA 提取试剂盒(TIANGEN Bacterial DNA Kit)。PCR 仪(ABI 公司),凝胶成像系统,核酸蛋白仪(瑞典AMERSHAM,型号ULTROSPEC1100),电泳仪(瑞典AMERSHAM),离心机(Eppendorf 公司)。
1.3 锁式探针的设计
以Lee et al[8]获得的香蕉细菌性枯萎病菌插入序列IS Rso19 部分碱基序列(GenBank:AF450275)作为靶标DNA 模板,按照王念武等[9]的方法设计Rs2 的锁式探针。设计合成的锁式探针及引物见表1。

表1 香蕉细菌性枯萎病菌HRCA 的锁式探针和引物Table 1 Primers and padlock probe of HRCA for Rs2
1.4 香蕉细菌性枯萎病菌HRCA 扩增
1.4.1 锁式探针的环化连接 以提取的香蕉细菌性枯萎病菌和其他参试菌株的DNA 为模板,分别与锁式探针PLPrs2 进行环化连接。10 μL 反应体系为:DNA 模板2 μL,40 U·μL-1Taq DNA 连接酶0.15 μL,400 pmol·L-1探针0.2 μL,10 ×Taq DNA 连接酶缓冲液1 μL,无菌去离子水补至10 μL。反应条件为:94℃4 min;94 ℃35 s,65 ℃5 min,15 个循环;95 ℃10 min 灭活剩余连接酶。反应完成后,立即将产物冰浴5 min 用于之后试验,或置于-20 ℃冰箱保存备用。
1.4.2 未环化锁式探针的消化 预先制配10 μL 混合液。混合液构成为:5 U·μL-1核酸外切酶Ⅲ1 μL,10 ×核酸外切酶Ⅲ缓冲液2 μL,10 U·μL-1核酸外切酶Ⅰ1 μL,10 ×核酸外切酶Ⅰ缓冲液2 μL,无菌去离子水4 μL。向环化连接后的产物中加入该10 μL 预混液后,37 ℃水浴2.5 h 消化未环化的锁式探针,再于90 ℃水浴15 min,灭活核酸外切酶。
1.4.3 HRCA 扩增 用扩增引物进行超分支滚环扩增。以锁式探针连接环化、消化后的产物作为模板,反应体系为25 μL(环化连接消化后产物2 μL,10 μmol·L-1padR 0.5 μL,10 mmol·L-1dNTPs 1 μL,10 μmol·L-1padF 0.5 μL,8 U·μL-1Bst DNA 聚合酶0.5 μL,10 ×Bst DNA 聚合酶缓冲液2.5 μL,灭菌去离子水补至25 μL)。63 ℃恒温条件下反应1.5 h。反应结束后每个样取8 μL 进行电泳,琼脂糖凝胶浓度为2.0%,最后经染色、凝胶成像分析反应结果。
1.5 香蕉细菌性枯萎病菌HRCA 扩增的特异性
以供试的香蕉细菌性枯萎病菌等9 种细菌的DNA 为模板,按照上述方法,分别与锁式探针PLPrs2 进行超分支滚环扩增,测试香蕉细菌性枯萎病菌HRCA 扩增的特异性。
1.6 香蕉细菌性枯萎病菌HRCA 扩增的灵敏度
香蕉细菌性枯萎病菌DNA 浓度经测定后,进行系列稀释。之后各取2 μL 作为DNA 模板,按照上述方法,分别与锁式探针PLPrs2 进行超分支滚环扩增,测试香蕉细菌性枯萎病菌HRCA 扩增的最低DNA 检测浓度。
2 结果与分析
2.1 香蕉细菌性枯萎病菌HRCA 扩增检测技术的建立
在香蕉细菌性枯萎病菌HRCA 扩增试验中,分别优化了线性探针的终浓度和扩增的反应温度。结果表明,HRCA 扩增的最适反应温度为63 ℃(图1),线性探针最佳终浓度为5 pmol·L-1(图2);在消化反应中,用6 U 核酸外切酶Ⅲ和10 U 核酸外切酶Ⅰ共同消化连接产物2.5 h,可以消除线性探针的干扰影响。优化后的香蕉细菌性枯萎病菌HRCA 扩增产物经电泳、凝胶成像分析,呈典型的阶梯状条带(图3)。

图1 不同温度下的香蕉细菌性枯萎病菌HCRA 扩增 Fig.1 HRCA under different temperature for Rs2

图2 不同终浓度探针的香蕉细菌性枯萎病菌HCRA 扩增Fig.2 HRCA under different final concentration of padlock probe for Rs2
2.2 香蕉细菌性枯萎病菌HRCA 扩增的特异性
在供试的9 种细菌中,仅香蕉细菌性枯萎病菌出现阶梯状分布条带,检测结果为阳性;其余8 种细菌均未出现阶梯状分布条带,结果为阴性(图4A)。该结果表明,HRCA 扩增检测香蕉细菌性枯萎病菌具有特异性。另外,采用Lee et al[8]提供的引物对供试的9 种细菌进行PCR 扩增,仅香蕉细菌性枯萎病菌的PCR 扩增结果呈阳性,其他则为阴性(图4B)。比较HRCA 扩增和PCR 扩增结果,两种方法扩增结果一致,都具有特异性。因此,建立的香蕉细菌性枯萎病菌HRCA 技术对香蕉细菌性枯萎病菌有特异性,可以用于该菌快速检测。
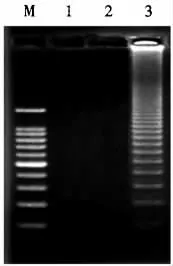
图3 香蕉细菌性枯萎病菌HRCA 扩增电泳图Fig.3 The electrophorogram of HRCA for Rs2
2.3 HRCA 扩增检测香蕉细菌性枯萎病菌的灵敏度
当香蕉细菌性枯萎病菌DNA 浓度≥500 fg·μL-1时,结果为阳性;当DNA 浓度<500 fg·μL-1时,结果呈阴性(图5),最低DNA 检测浓度为500 fg·μL-1。采用Lee et al[8]的PCR 方法,检测香蕉细菌性枯萎病菌的灵敏度,结果表明,最低DNA 浓度为5 pg·μL-1(图5)。比较HRCA 方法和常规PCR 方法可以发现,HRCA 方法具有更高的灵敏度,高出常规PCR 扩增方法一个数量级。

图4 HRCA 法检测香蕉细菌性枯萎病菌的特异性Fig.4 Detected specificity by HRCA for Rs2

图5 HRCA 法检测香蕉细菌性枯萎病菌的灵敏度Fig.5 Detected sensitivity by HRCA for Rs2
3 讨论
细菌快速检测方法各有优势和不足。血清学ELISA 方法灵敏度低,适用于细菌的初筛;常规PCR 方法和实时荧光PCR 方法,有较高的灵敏度,但容易受模板DNA 的污染,而使结果出现误判[10]。HRCA 扩增技术只特异性地扩增锁式探针,模板DNA 只起到让线性锁式探针环化的作用。因此,在HRCA 扩增技术体系中,模板DNA 不会富集,避免了PCR 方法中靶标DNA 富集的影响。
在细菌的分子生物学检测中,常利用细菌相对保守的16S rDNA 碱基序列来设计引物,进行细菌种类的检测和鉴定。对于种间差别较大的细菌可以用该方法检测和鉴定,但对于相似种或是种以下的不同race 型则很难,因为它们的16S rDNA 碱基序列几乎相同。香蕉细菌性枯萎病菌相似的race 型有5 种,因此本研究采用香蕉细菌性枯萎病菌独有的插入序列ISRso19 部分碱基作为模板,用HRCA 扩增技术对该菌进行研究,实现了香蕉细菌性枯萎病菌HRCA 特异性扩增,灵敏度可达500 fg·μL-1,高于常规PCR 一个数量级。
在基层口岸植物检疫实验室,往往都存在着缺乏仪器设备的客观情况,而基于锁式探针的HRCA 扩增技术对仪器设备要求不高,一般的口岸分支植物检疫实验室都能具备,便于口岸应用。另外,锁式探针最大优势是高通量,可以与荧光PCR 技术、反向斑点杂交技术、DNA 芯片等技术结合[11,12],尤其是结合DNA 芯片技术,可同步检测多种病原微生物。在我国进境的植物中,往往存在着多种病原微生物,利用基于锁式探针的扩增技术与DNA 芯片技术结合起来,可实现同种植物产品上多种病原有害生物的同步检测,这对于提高口岸疫情把关,有效防止外来有害生物传入,保护国内农林业和生态安全都具有重要意义。
[1]BUDDENHAGEN I,KELMAN A. Biological and physiological aspects of bacterial wilt caused by Pseudomonas solanacearum[J]. Annual Review of Phytopathology,1964,2:203 -230.
[2]BUDDENHAGEN I W. Strain of Pseudomonas solanacearum in indigenous hosts in banana plantations of Costa Rica,and their relationship to bacterial wilt of bananas[J]. Phytopathology,1960,50(9):660 -664.
[3]BUDDENHAGEN I W,SEQUEIRA L,KELMAN A. Designation of races in Pseudomonas solanacearum[J]. Phytopathology,1962,52:726.
[4]DIATLOFF A,AKIEW E,WOOD B,et al. Characteristics of isolates of Ralstonia solanacearum from Heliconia[J]. Australasian Plant Pathol,1992,21:163 -168.
[5]NILSSON M,MALMGREN H,SAMIOTAKI M,et al. Padlock probes:Circularizing oligonucleotides for localized DNA detection[J]. Science,1994,265(5181):2085 -2088.
[6]SZEMES M,BONANTS P,WEERDT M D,et al. Diagnostic application of padlock probes-multiplex detection of plant pathogens using universal microarrays[J]. Nucleic Acids Research,2005,33(8):70 -74.
[7]LIZARDI P M,HUANG X H,ZHU Z R,et al. Mutation detection and single-molecule counting using isothermal rolling-circle amplification[J]. Nature Genetics,1998,19:225 -232.
[8]LEE Y A,KHOR C N. A novel insertion sequence,ISRso19,isolated from Ralstonia solanacearum and its application to race differentiation[J]. Plant Pathology Bulletin,2003,12:57 -64.
[9]王念武,王婷,沈建国,等.番茄细菌性叶斑病菌超分支滚环扩增快速检测技术[J].植物保护,2014,40(2):90 -94.
[10]WANG B,POTTER S J,LIN Y G,et al. Rapid and sensitive detection of severe acute respiratory syndrome coronavirus by rolling circle amplification[J]. Clinical Microbiology,2005,43(5):2339 -2344.
[11]ZHANG D Y,ZHANG W,LI X,et al. Detection of rare DNA targets by isothermal ramification amplification[J]. Gene,2001,274(1 -2):209 -216.
[12]THOMAS D C,NARDONE G A,RANDALL S K. Amplification of padlock probes for DNA diagnostics by cascade rolling circle amplification or the polymerase chain reaction[J]. Archives of Pathology & Laboratory Medicine,1999,123(12):1170-1176.

