丙型肝炎病毒NS3蛋白的原核表达及多克隆抗体制备
孙涛,杨光文,张金阳,2,夏雪山,2
丙型肝炎病毒NS3蛋白的原核表达及多克隆抗体制备
孙涛1,杨光文1,张金阳1,2,夏雪山1,2
1昆明理工大学生命科学与技术学院,云南昆明 650500 2云南省分子医学研究中心,云南昆明 650500

孙涛, 杨光文, 张金阳, 等. 丙型肝炎病毒NS3蛋白的原核表达及多克隆抗体制备. 生物工程学报, 2015, 31(5): 711–721.Sun T, Yang GW, Zhang JY, et al. Prokaryotic expression of Hepatitis C Virus (HCV) NS3 protein and preparation of polyclonal antibody. Chin J Biotech, 2015, 31(5): 711–721.
提高对丙型肝炎患者实验室检测的灵敏度和特异性,对相关人群进行筛查和早期诊断,是控制丙型肝炎病毒(HCV) 流行与传播的有效措施。为了建立更为可靠的HCV诊断方法,通过采用PCR方法从J6/JFH1 2a型病毒中克隆出HCV基因片段,将其连接到pET-28a载体上,重组载体pET-28a-转化大肠杆菌BL21 (DE3) 后诱导表达,以10% SDS-PAGE进行鉴定,获得表达的NS3重组蛋白分子量为72 kDa。将纯化的NS3蛋白免疫BALB/c小鼠,第4次免疫后采集血液并分离血清进行抗体活性鉴定,小鼠抗体效价为1∶256 000。进一步的Western blotting和间接免疫荧光结果显示,以重组NS3蛋白免疫小鼠制备的多克隆抗体可以很好地识别HCV感染Huh7.5.1细胞中的NS3蛋白,为下一步开展单克隆抗体制备和检测试剂盒研制工作奠定了基础。
丙型肝炎病毒,B细胞表位,NS3蛋白,多克隆抗体
丙型肝炎病毒(Hepatitis C virus,HCV) 是导致丙型肝炎的主要病原体[1]。全球约有2亿人感染HCV病毒[2-3],其中70%的感染者会发展为慢性感染,30%的感染者会通过自身的免疫系统清除体内的病毒,其中在慢性感染者中约有5%的病人最终会死于肝硬化和肝细胞癌[4-8]。丙型肝炎病毒是单股正链RNA病毒,基因组约长9.5 kb,仅有1个开放阅读框,编码约3 000个氨基酸所组成的前体多肽,由宿主蛋白酶和病毒编码的蛋白酶加工成多个成熟蛋白。目前由于HCV病毒的疫苗类制剂尚未成型,也无特效的治疗药物,临床治疗HCV常采用干扰素α (IFN-α) 联合利巴韦林,但应答率较低,尽管目前以依赖RNA的RNA聚合酶 (RNA-dependent RNA polymerase,RdRp) 为靶点的抗病毒药物索非布韦 (Sofosbuvir) 的使用,较大程度地提高了患者持续性病毒学应答率 (SVR),但由于病毒在此区段固然发生的基因变异,仍可能发生耐药性突变,导致该药物使用仍伴有一定的副作用[9-11]。
由于HCV主要是通过输血途径传播,提高对丙型肝炎患者检出的灵敏度和特异性,对相关人群进行筛查和早期诊断,是控制HCV流行与传播的有效措施。目前HCV病毒的检测主要有HCV核酸检测、HCV抗体检测以及HCV抗原检测。患者体内病毒RNA量直接反应病毒复制的活跃程度,检测患者体内的病毒RNA存在和水平可用于确定感染、评价病程与药物治疗效果。但由于核酸检测PCR方法的高灵敏性,这种方法对实验室条件和实验工作人员要求都很高,容易造成污染,同时还存在病毒变异导致引物不能有效结合出现假阴性问题。HCV抗体检测多以HCV核心蛋白、NS3、NS4和NS5非结构蛋白的特异抗体为靶标物,已有金标试纸条和HCV ELISA试剂盒,虽然HCV抗体检测的第3代ELISA技术较大提高了检测灵敏性,但可检测出抗体的窗口期仍需40多天[12]。抗体检测灵敏度低,固然存在的“窗口期”,以及免疫缺陷患者不能有效产生抗体,抗体检测容易漏检和出现假阴性。抗原检测靶标直接针对HCV病毒,检测结果更为直接可靠。国内已有湖南景达生物工程有限公司和山东莱博生物科技有限公司研制的双抗体夹心法检测HCV核心抗原诊断试剂,国际雅培ARCHITECT HCV核心抗原检测试剂也于2012年6月5日获得SFDA认证注册。但由于体内HCV核心抗原的低水平和相关特异性抗体制备的困难,现有的抗原检测技术还存在一些问题。
HCV非结构蛋白NS3分子量为67 kDa,具有丝氨酸蛋白酶和ATP酶及RNA解旋酶活性,是具有重要生物活性的HCV功能蛋白。针对NS3蛋白开发抗体检测试剂,其检测特异性与灵敏性都高于结构蛋白,同时NS3蛋白在HCV感染早期就出现,其与特异性抗体亲和力和特异性也都很强,且持续时间较长,这些都决定了NS3蛋白在丙型肝炎的实验室检测方面具有较大优势[13]。
本研究首先使用生物信息学软件对HCV-NS3蛋白序列的跨膜区和B细胞抗原表位进行分析和预测,用PCR方法从J6/JFH1 2a型病毒中扩增得到HCV基因片段,成功构建了pET-28a-重组载体,在大肠杆菌BL21细胞中成功表达并通过亲和层析得到纯化的NS3蛋白,免疫小鼠结果证实其具有很好的抗原性,为以NS3蛋白及相应抗体为检测靶标的检测试剂的开发奠定了基础。
1 材料与方法
1.1 蛋白质结构分析及抗原表位预测
采用在线网站服务器ExPASy中ProtScale模块 (http://www.expasy.org/cgi-bin/protscale.pl) 预测蛋白质疏水性,使用TMHMM server v.2.0在线软件对HCV-NS3蛋白序列进行跨膜区预测(http://www.cbs.dtu.dk/services/TMHMM/)[14]。使用Swiss-prot蛋白数据库中Swiss-model模块预测NS3蛋白的三级结构,使用DNAstar中Protean模块对HCV-NS3蛋白的抗原表位进行预测[15]。
1.2目的基因扩增与纯化
以J6/JFH1 2a型重组病毒 (GenBank Accession No. AY746460.1) 为扩增模板,根据引物设计原则设计PCR引物 (表1),引物两端分别加上HⅠ和d Ⅲ酶切位点。培养获得的J6/JFH1 2a进行病毒核酸提取,作为模板进行全长基因扩增。PCR扩增条件:94 ℃预变性5 min;94 ℃变性30 s,57 ℃退火30 s,72 ℃延伸90 s,35个循环;72 ℃延伸10 min,4 ℃保存。PCR产物用1.0%的琼脂糖凝胶电泳检测,并回收目的片段进行序列测定确认PCR扩增无误。

表1 扩增ns3基因的引物序列
Notes: the protective bases are marked with bold; the recognition sequences of restriction enzymes are showed in italics;the primers sequences are marked with underline.
1.3 pET-28a-重组载体构建与表达
将回收的目的基因片段双酶切后连接到经同样双酶切的pET-28a载体上,转化DH5α感受态细胞,PCR鉴定确定阳性克隆,并经测序分析进一步验证。阳性菌的小量培养后,用0.1 mol/L IPTG诱导培养4 h,菌体沉淀用1×SDS上样缓冲液悬浮并热处理后,进行10% SDS-PAGE,考马斯亮蓝染色观察蛋白表达。
阳性菌扩大培养,收集菌体,将菌体悬浮于PBS,冰浴条件下超声破碎直至菌体清亮。离心弃上清,包涵体沉淀用8 mol/L尿素裂解过夜,取菌体破碎后上清和包涵体裂解液,进行SDS-PAGE。包涵体裂解液以Ni-NTA进行重组NS3蛋白纯化,并进行SDS-PAGE验证。
1.4 小鼠免疫过程
取纯化后NS3蛋白溶于PBS溶液中,与等量的弗氏完全佐剂充分混匀后,在漩涡振荡器上振荡30 min,保证蛋白溶液与弗氏完全佐剂充分混合。第1次免疫小鼠采用小鼠背部和皮下多点注射,隔14 d免疫1次,除第1次使用弗氏完全佐剂外,第2次和第3次均使用弗氏不完全佐剂进行混合,第4次为加强免疫,只注射纯蛋白液。
1.5 小鼠多抗血清的特异性检测
用J6/JFH1 2a型病毒感染Huh7.5.1细胞,48 h后弃去培养基,PBS洗3次,以固定液 (甲醇:丙酮=1∶1) 于−20 ℃下固定20 min,风干,同时设未感染病毒的细胞为阴性对照。取第3次免疫的血清 (1∶100) 作为一抗孵育2 h (37 ℃),PBS洗5次后,用FITC标记羊抗鼠 (1∶300) 孵育1 h (37 ℃),PBS避光洗涤5次,荧光显微镜下观察荧光反应。
将J6/JFH1 2a型病毒感染Huh7.5.1细胞,感染72 h后,弃去培养液,PBS洗3次,用RIPA裂解细胞,1 200 r/min离心5 min,取上清,进行SDS-PAGE,转膜,取第3次免疫的血清作为一抗 (1∶100) 孵育2 h (37 ℃),HRP标记的羊抗鼠作为二抗孵育1 h (37 ℃),进行Western blotting分析,同时以未感染病毒的细胞作为阴性对照。
1.6 间接ELISA检测与进口试剂盒对比实验
用美国进口试剂盒 (ORTHO) 按其说明书操作对采自泸西的692份临床血清样本进行ELISA检测,之后以本实验室原核表达的NS3蛋白为包被抗原对从中随机筛出的15份阳性样品、5份阴性样品进行检测,由阴性血清样品的均值计算出Cutoff值及A/C (Absorbance at 450 nm/Cutoff value) 值,A/C值>1为阳性,反之则为阴性,计算间接ELISA检测结果与进口试剂盒检测结果的灵敏性及特异性。
2 结果与分析
2.1 蛋白质疏水性分析及跨膜结构预测
通过ExPASy服务器上ProtScale模块进行在线分析 (图1),HCV-基因编码蛋白由632个氨基酸组成,平均疏水性 (Grand average of hydropathicity) 为0.035,结果显示其蛋白序列所表达的蛋白整体不存在强的疏水区,只是蛋白N端几个氨基酸可能存在小的疏水区。为了降低跨膜区对蛋白表达的不利影响,使用在线网站预测HCV-NS3蛋白的跨膜结构 (图2),结果显示HCV-NS3蛋白为632个氨基酸,HCV-NS3蛋白没有典型的跨膜区,HCV-NS3蛋白的1−632个氨基酸都位于细胞膜表面,降低了跨膜区疏水性氨基酸对蛋白折叠的影响,易于表达和纯化。
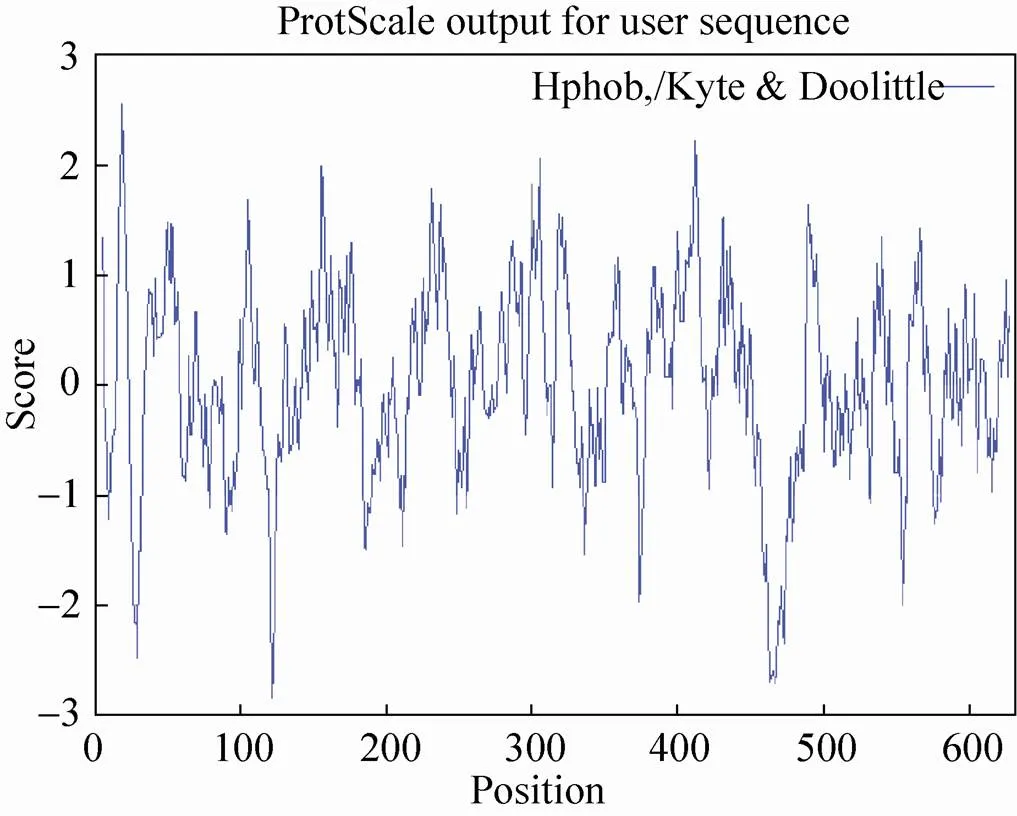
图1 NS3蛋白的疏水性分析
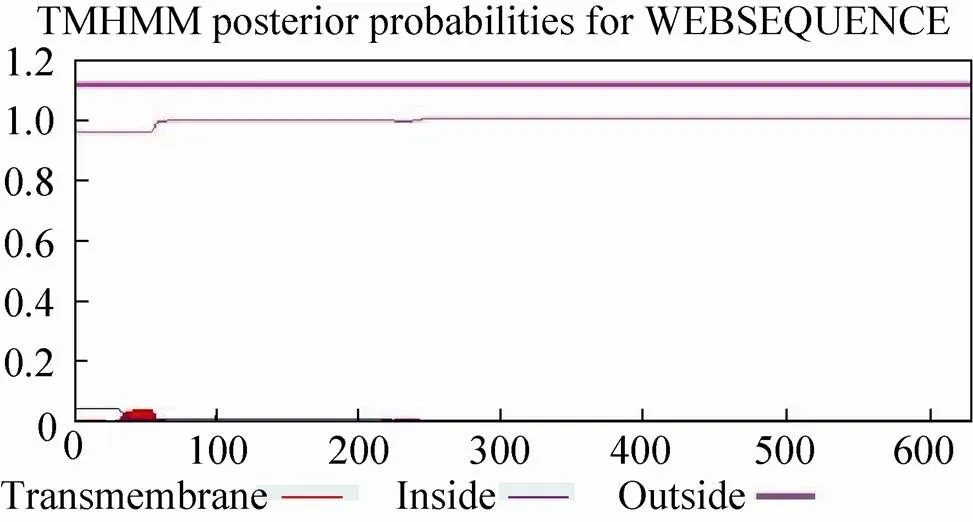
图2 NS3蛋白的跨膜区分析
2.2 HCV-NS3蛋白三级结构及B细胞抗原表位预测
通过Swiss-model在线软件同源建模的方法预测HCV-NS3蛋白三级结构 (图3),HCV NS3蛋白由α-螺旋、β-折叠、β-转角和无规则卷曲等结构组成规则的空间构象。该构象由α-螺旋位于蛋白内部、β-折叠位于蛋白中心,两者构成1个疏水核心区域;β-转角和无规则卷曲位于蛋白外部,它们结构松散,易变性扭曲。使用DNAstar中Protean模块中亲水方案、表面可及性方案、柔性方案、抗原性方案和抗原指数方案对HCV-NS3蛋白质抗原表位进行综合分析发现,NS3蛋白全长序列中具有较为丰富且分布均匀的抗原表位 (图4),主要的抗原氨基酸序列及分布位点 (表2) 所示,有8个优势抗原表位,这些位点的抗原指数较高,且具有强的表面可及性和亲水性位点,因此该蛋白制备抗体具有较强的可行性。
2.3目的基因扩增及重组质粒pET-28a-的构建
以提取、纯化的J6/JFH1 2a型病毒核酸为模板,PCR扩增全长基因 (图5A) 及重组载体pET-28a-双酶切 (图5B) 后经1%琼脂糖凝胶电泳,PCR产物长度为1 893 bp,与预期长度一致。该片段经回收测序,核酸序列准确无误。构建的载体经测序分析,确认插入片段及引物、限制性酶切位点准确无误。
2.4 重组蛋白的表达与纯化
阳性菌经IPTG诱导表达后,SDS-PAGE分析结果发现阳性菌有约72 kDa的特异性条带。重组质粒在N端和C端共表达分子质量约5 kDa 的蛋白,中间的分子质量为67 kDa的HCV NS3蛋白,而未加IPTG的重组菌和BL21菌没有相应的条带 (图6),可初步确定HCV基因在重组表达载体中得到正确表达 (图6)。重组蛋白经大量表达后,用Ni Sepharose Fast Flow进行纯化,使用梯度洗脱 (图7),在洗脱缓冲液:结合缓冲液=6∶4的条件下,纯化效果最佳。
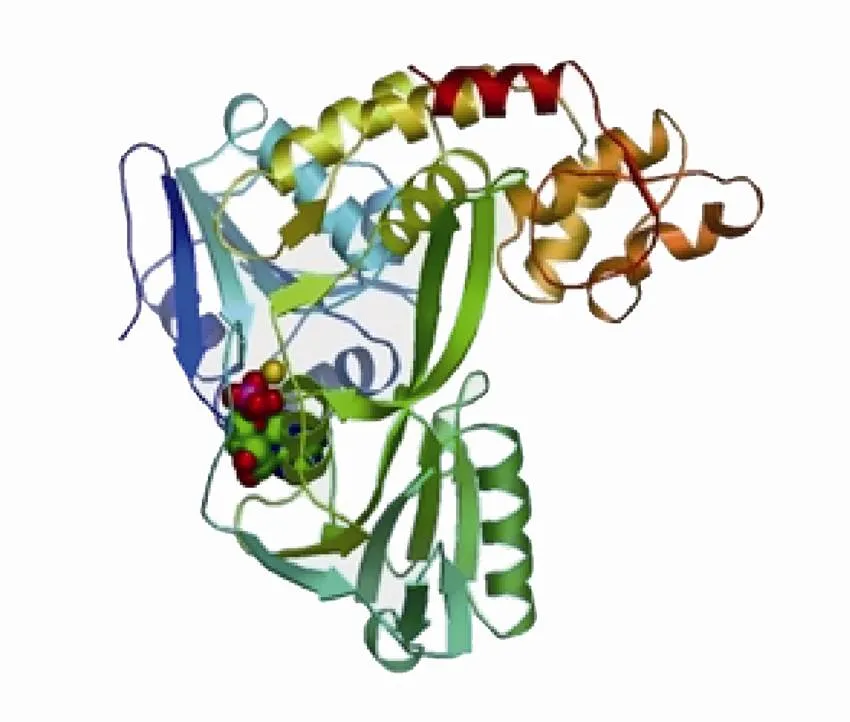
图3 NS3蛋白三级结构
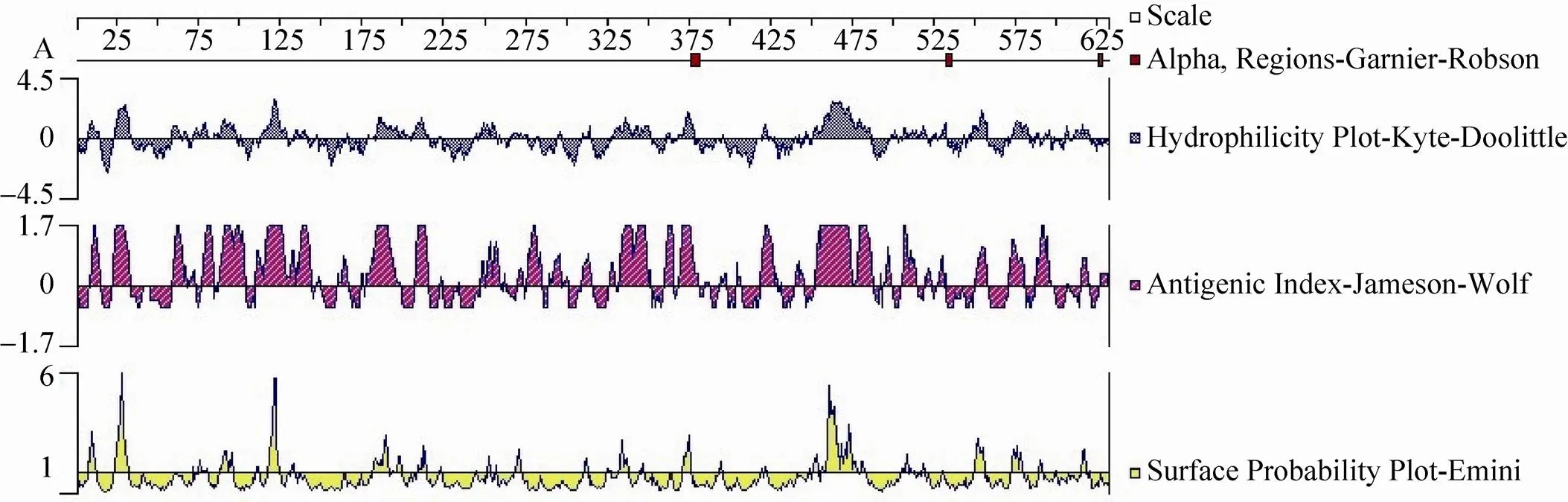
图4 NS3蛋白的特性分析
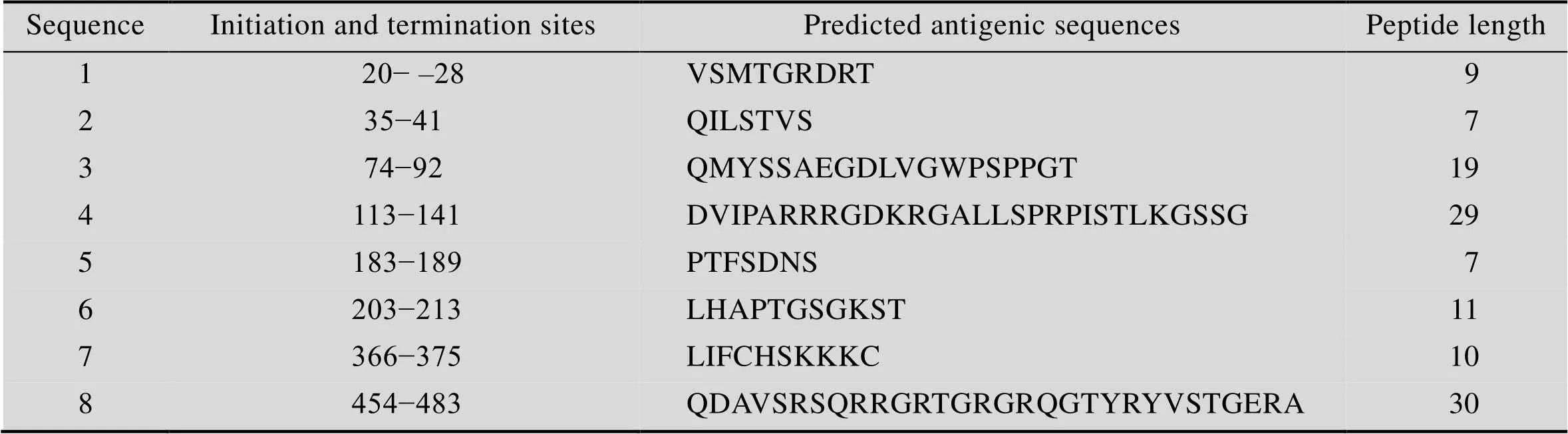
表2 DNAstar预测HCV NS3蛋白优势抗原表位的肽段位置
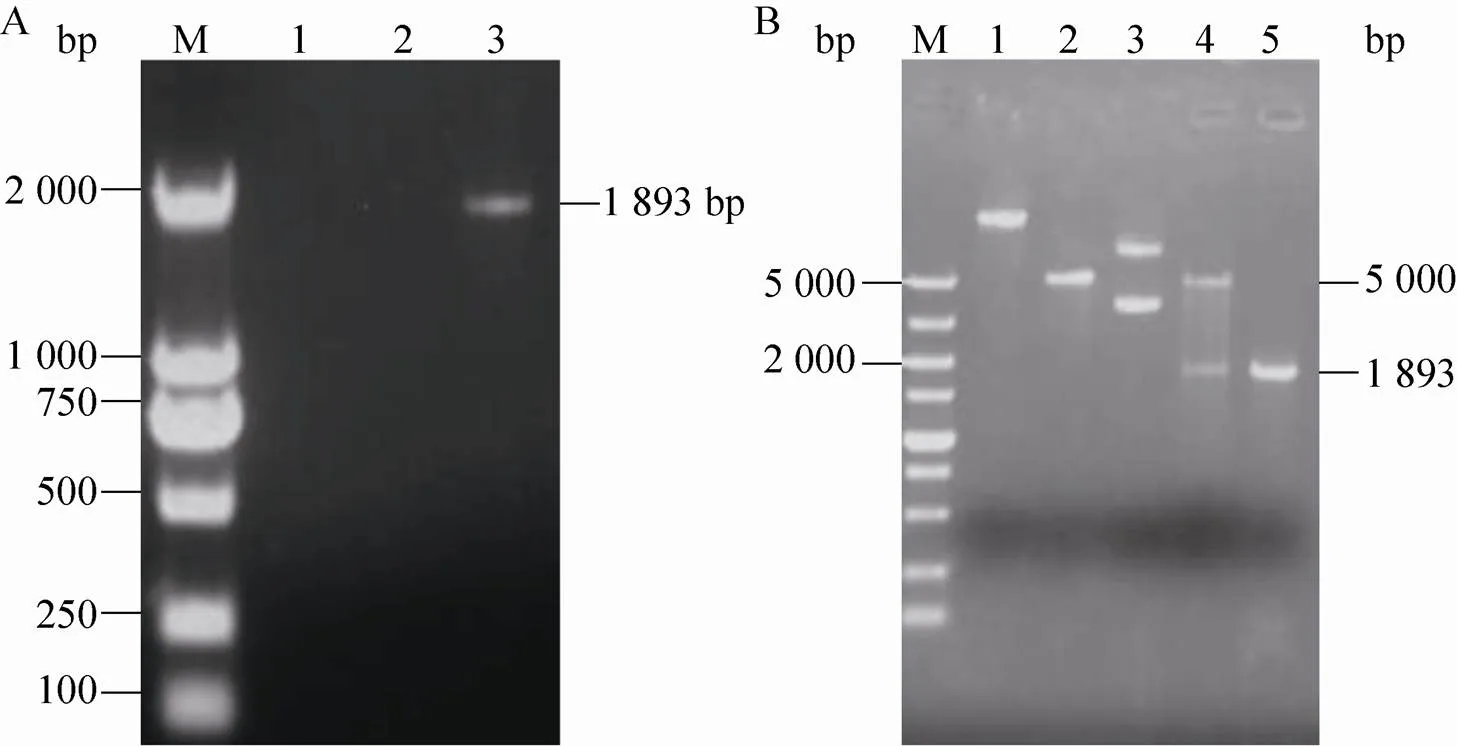
图5 PCR及重组质粒的双酶切鉴定结果
2.5 重组蛋白抗体与NS3蛋白的特异性结合
采集重组蛋白免疫的小鼠血清,间接免疫荧光法IFA测定血清抗体对J6/JFH1 2a型病毒的结合情况。结果表明,J6/JFH1 2a型病毒感染的细胞可以与重组蛋白免疫小鼠抗血清发生阳性反应,产生特异性的绿色荧光,且可见NS3蛋白主要分布于细胞质中 (图8),而未感染病毒的细胞呈阴性反应,无明显的荧光出现,表明重组蛋白免疫小鼠制备的多抗血清具有很好的特异性。
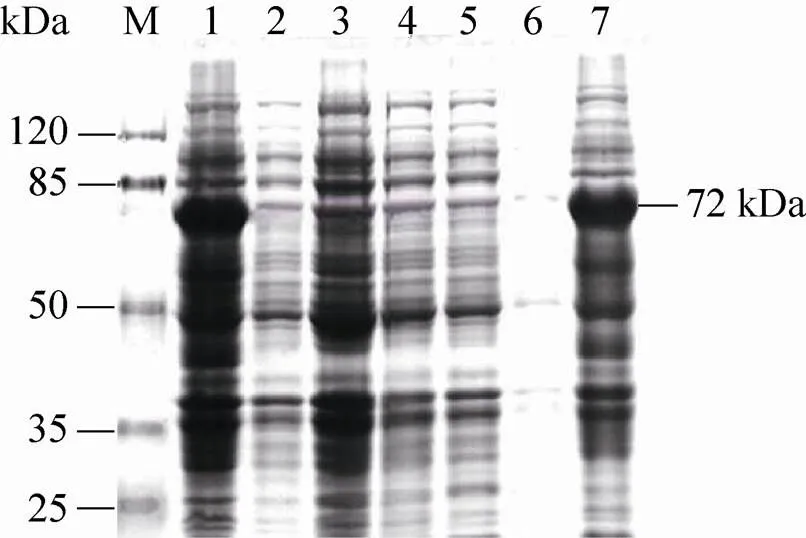
图6 SDS-PAGE检测重组蛋白在E. coli BL21中的表达
2.6 Western blotting分析小鼠多抗血清对病毒编码蛋白的反应性
Huh7.5.1细胞接种J6/JFH1 2a型病毒,病毒感染72 h收集细胞样品。用小鼠多抗血清作为一抗进行Western blotting分析,结果出现与预期分子量大小相符的特异性条带,而未感染病毒的细胞在此位置无相应条带出现,表明重组蛋白制备的小鼠多抗血清对HCV感染细胞中NS3蛋白的线性抗原表位能够很好的识别 (图9)。
2.7 间接ELISA检测与进口试剂盒检测结果比较
以纯化的大肠杆菌表达的NS3蛋白为抗原包被96孔板对随机筛出的15份阳性样品、5份阴性样品进行ELISA检测,450处测吸光值并测定A/C值 (表3),经测定,阴性对照450均值为0.067,根据“阴性对照的平均值≤0.05时,Cutoff=2.1×0.05;当阴性对照的平均值>0.05时,Cutoff=2.1×阴性对照均值”的ELISA测定阈值确定原则,所建立方法的Cutoff值为0.141,A/C值>1为阳性,反之则为阴性。利用该方法对20份样本进行测定,结果14份为阳性,5份为阴性 (表4),以ORTHO试剂盒结果作为参照,HCV NS3 ELISA检测的灵敏性为93.3%,特异性为100%,表明重组蛋白具有很好的灵敏性及特异性。
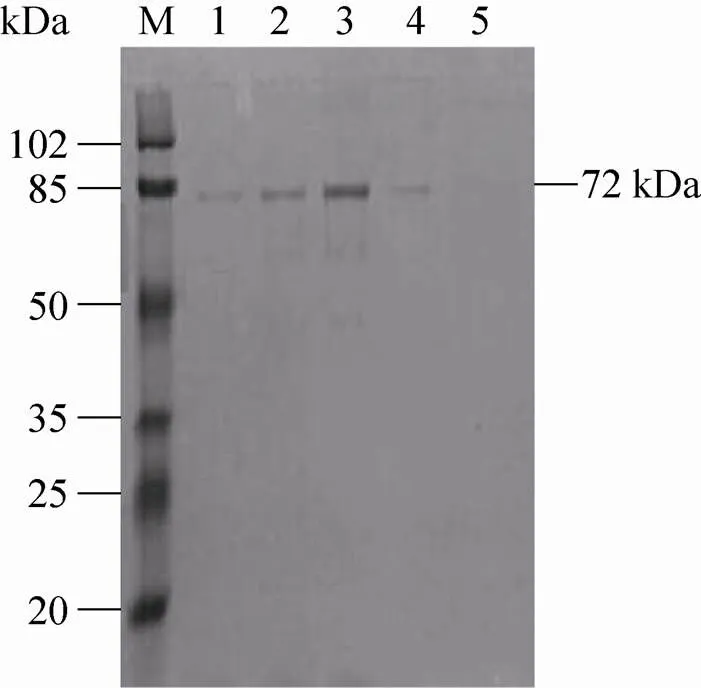
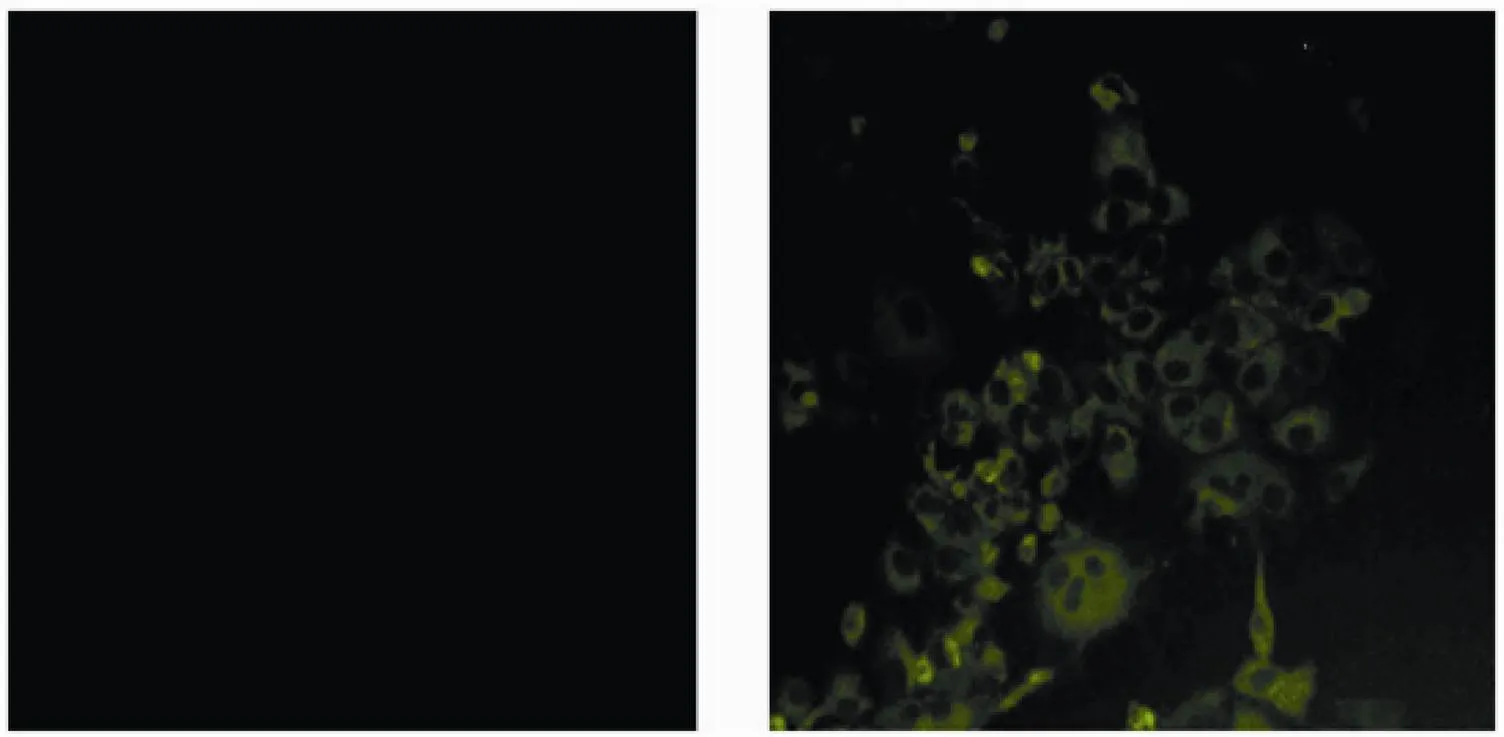
图8 HCV NS3蛋白的IFA分析
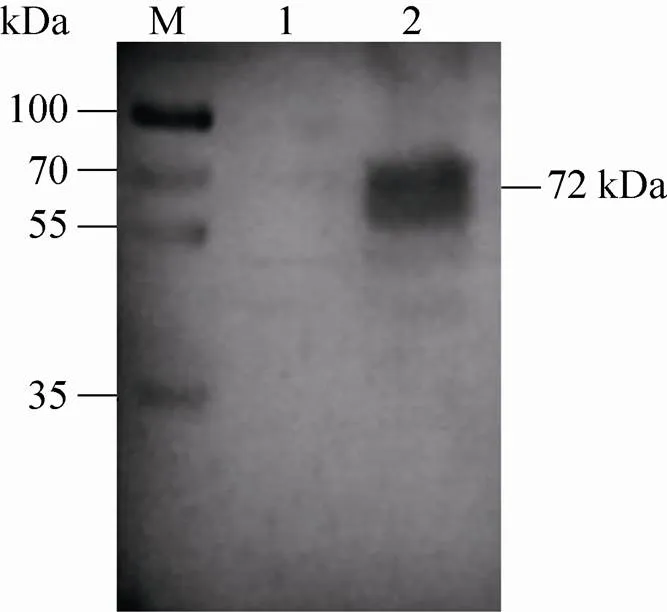
图9 Western blotting分析小鼠多抗血清的特异性
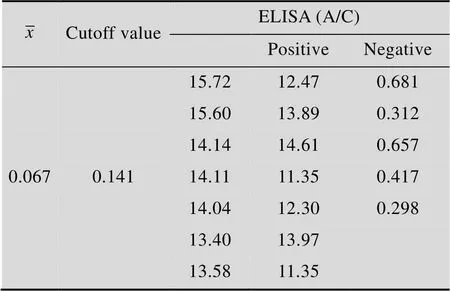
表3 ELISA检测结果的A/C值
3 讨论
丙型肝炎病毒 (HCV) 感染是引起慢性肝病的主要病因,病毒一经感染,超过70%的感染者最终会发展为慢性肝炎,严重危害人类健康。HCV是一种高度变异的病毒,到目前为止,还没有有效的疫苗去预防HCV病毒和有效的药物彻底清除体内的病毒颗粒,并且相关的疫苗和药物研究进展缓慢,及早准确诊断,严格控制HCV的感染源头是控制HCV传播的有效手段,有研究报道[16]针对HCV核心基因的序列构建的M1GS-HCV/C141核酶在体外具有很好的抗HCV活性,也为HCV的后期诊断与治疗提供了一条新的研究途径。
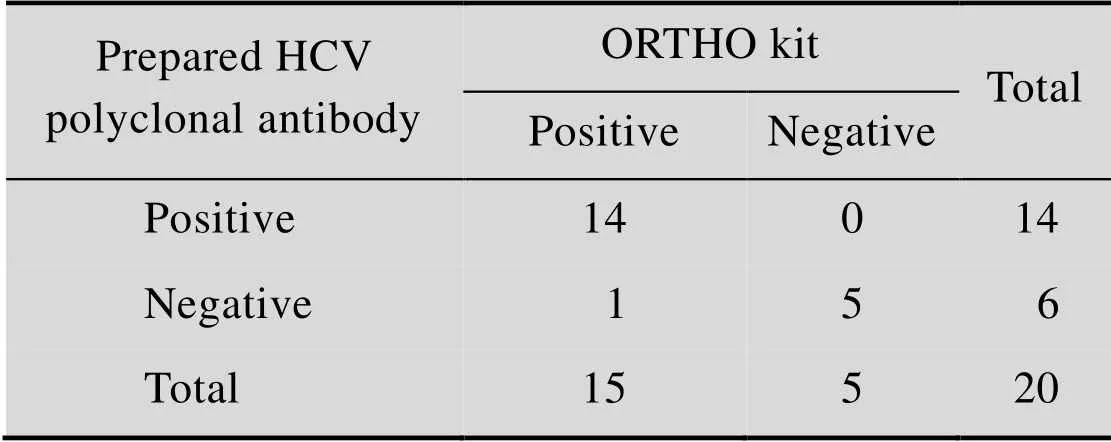
表4 HCV NS3 ELISA检测与ORTHO试剂盒检测结果比较
目前用于HCV ELISA检测所用的抗原主要是多肽合成和基因表达,既有单独使用,也有混合使用,其主要是核心区和NS3非结构区等抗原,但由于我们选择表达所制备的抗原与天然抗原还存在一些差距,故在检测中会出现一些漏检现象,进而影响检测的特异性和灵敏性。在HCV感染者血清中绝大多数都有NS3非结构蛋白抗体的出现且持续存在。NS3蛋白抗原免疫性强,存在多个抗原表位,容易检测患者体内感染的HCV病毒。Murphy等[17]研究发现,基因表达的蛋白分子量很大,具有很强的免疫原性,可以引起机体特异性细胞免疫反应,诱导机体产生大量的干扰素,帮助机体清除病毒和抑制病毒的大量复制。我们研究发现,全长基因很保守,没有较强的疏水区,蛋白存在多个优势抗原表位,且还携带多个目标抗原以及辅助性表位,能够有效地应对病毒的变异和免疫反应中HLA的限制性[18]。再者,其多个优势抗原表位主要存在于N端且又包括了C端除疏水区外的大部分蛋白,故既有利于蛋白的表达与纯化又有利于C端抗原决定簇的存在。抗原表位的覆盖面大,能覆盖更多的基因型和亚型,所设计的药物对绝大部分人群都有用,对诱导细胞免疫反应的强度和广度以及在病毒清除中起着重要作用。由于HCV NS3蛋白还具有NS3丝氨酸蛋白酶、三磷酸核苷酶 (NTPpase) 和螺旋酶 (Helicase) 活性[19-22]。也有研究表明[23],HCV NS3蛋白的NTP酶和解旋酶功能区同样可以诱导产生体液免疫,而且NTPase及Helicase酶能使NS3蛋白结合到HCV基因组的3'末端上,在病毒RNA复制中发挥重要的调节作用[24-26]。所以在HCV感染者筛查方面使用NS3蛋白作为抗原,NS3蛋白的多个优势抗原表位能够充分暴露其抗原决定簇和伸展氨基酸的活性组分,更好地提高检测灵敏度和免疫原性,有效避免HCV感染者的漏检,这也是目前HCV在血清学检测上多选择NS3蛋白作为抗原的原因。
综上所述,本研究成功地将HCV全长基因连接到pET-28a表达载体中,诱导表达出分子质量约为72 kDa的重组蛋白并且通过His标签纯化出蛋白,免疫小鼠后制备出高效价的多克隆抗体。目前关于HCV NS3蛋白的多克隆抗体制备的条件已经很成熟,相关检测试剂盒都已经成熟化,而且单抗试剂盒也已经进入市场,但其都存在一定的缺陷。首先,目前关于NS3多克隆抗体制备的文献报道中,大多是通过ELISA法对其进行特异性及灵敏性验证,由于我们对抗原的选择区域相比于天然抗原还存在一定的差距,故对其灵敏性及特异性的要求就会很高,而本实验主要通过IFA对其进行验证,相比于ELISA法,IFA更能扩大抗体应用范围,而且血清可以通过Huh7.5.1细胞传代进行吸收,大大提高了所制备抗体的特异性;而目前国外单抗检测试剂盒主要以Anti-HCV-NS3单抗检测试剂盒 (Abcam公司) 为主,其特异性及灵敏性都很好,但其反应强度不如多抗,价格昂贵且抗体只针对1种抗原决定簇,比较单一。故本研究通过对HCV NS3蛋白进行生物信息学分析,挖出其优势抗原表位进行表达,并通过IFA方法对所制备的多抗进行验证,更加肯定了其特异性,并准备以该抗体为基础建立竞争性ELISA,进一步提高HCV抗体检测的灵敏性以及为第3代HCV抗体检测试剂的改进和完善提供有力的工具。
REFERENCES
[1] Choo QL, Kuo G, Weiner AJ, et al. Isolation of a cDNA clone derived from a blood-borne non-A, non-B viral hepatitis genome. Science, 1989, 244 (4902): 359–362.
[2] Gravitz I. Introduction: a smouldering public health crisis. Nature, 2011, 474(7350): S2–S4.
[3] Omland LH, Krarup H, Jepsen P, et al. Mortality in patients with chronic and cleared hepatitis C viral infection: a nationwide cohort study. J Hepatol, 2010, 53(1): 36–42.
[4] Hatziapostolou M, Polytarchou C, Aggelidou E, et al. An HNF4α-miRNA inflammatory feedback circuit regulates hepatocellular oncogenesis. Cell, 2011, 147(6): 1233–1247.
[5] Bruix J, Sherman M. Management of hepatocellular carcinoma: an update. Hepatology, 2011, 53(3): 1020–1022.
[6] Yuan P, Chen SSL. Hepatitis C virus and cellular stress response: implications to molecular pathogenesis of liver diseases. Viruses, 2012, 4(10): 2251–2290.
[7] Zaltron S, Spinetti A, Castelli F, et al. Chronic HCV infection: epidemiological and clinical relevance. BMC Infect Dis, 2012, 12 (Suppl 2): 1–7.
[8] Ashfaq UA, Javed T, Rehman S, et al. An overview of HCV molecular biology, replication and immune responses. Virol J, 2011, 8(11): 161–171 .
[9] Gentile I, Borgia F, Buonomo AR, et al. A novel promising therapeutic option against hepatitis C virus: an oral nucleotide NS5B polymerase inhibitor sofosbuvir. Curr Med Chem, 2013, 20(30): 3733–3742.
[10] Herbst DA Jr, Reddy KR. Sofosbuvir, a nucleotide polymerase inhibitor, for the treatment of chronic hepatitis C virus infection. Expert Opin Investig Drugs, 2013, 22(4): 527–536.
[11] Lam AM, Espiritu C, Bansal S, et al. Genotype and subtype profiling of PSI-7977 as a nucleotide inhibitor ofhepatitis C virus. Antimicrob Agents Chemother, 2013, 56(6): 3359–3368.
[12] Kim DW, Gwack Y, Han JH, et al . C-terminal domain of the hepatitis C virus NS3 protein contains an RNA helicase activity. Biochem Biophys Res Commun, 1995, 215(1): 160–166.
[13] Tuke PW, Grant PR, Waite J, et al. Hepatitis C virus window-phase infections: closing the window on Hepatitis C virus. Transfusion, 2008, 48(4): 594–600.
[14] Zhang JY, Sun T, Yang GW, et al. Prediction of B cell epitopes and major characteristics in protein gG of monkey herpes B virus. J Kunming Univ Sci Technol: Nat Sci Ed, 2014, 39(2): 79–85 (in Chinese) .张金阳, 孙涛, 杨光文, 等. 猴B病毒gG蛋白的主要特性与B细胞表位预测. 昆明理工大学学报: 自然科学版, 2014, 39(2): 79–85.
[15] Guo TL, Ye LB, Mao CQ. Expression, purification and usage of truncated HCV NS3 protein. Biotechnology, 2008, 18(6): 20–23 (in Chinese) .郭泰林, 叶林柏, 茆灿泉. 截短的HCV NS3蛋白的表达、纯化及应用. 生物技术, 2008, 18(6): 20–23.
[16] Li XF, Zhang WJ, Huang ZW, et al. Construction of an engineered M1GS-HCV/C141 ribozyme and determination of its antiviral activity. Chin J Biotech, 2013, 29(12): 1786–1795 (in Chinese) .李喜芳, 张文军, 黄志文, 等. M1GS-HCV/C141 核酶的构建及其体外抗病毒活性测定. 生物工程学报, 2013, 29(12): 1786–1795.
[17] Murphy D, Chamberland J, Dandavino R, et al. A new genotype of Hepatitis C virus originating from central Africa. Hepatology, 2007, 46(6): 267–276.
[18] Vider-Shalit T, Raffaeli S, Louzoun Y. Virus-epitope vaccine design: informatic matching the HLA-I polymorphism to the virus genome. Mol Immunol, 2007, 44(6): 1253–1261.
[19] Bartenschlager R, Ahlborn-Laake L, Mous J, et al. Nonstructural protein 3 of the Hepatitis C virus encodes a serine-type proteinase required for cleavage at the NS3/4 and NS4/5 junctions. J Virol, 1993, 67(7): 3835–3844.
[20] Grakoui A, McCourt D W, Wychowski C, et al. Characterization of the hepatitis C virus-encoded serine proteinase: determination of proteinase-dependent polyprotein cleavage sites. J Virol, 1993, 67(5): 2832–2843.
[21] Abian O, Vega S, Neira JL, et al. Conformational stability of hepatitis C virus NS3 protease. Biophys J, 2010, 99(11): 3811–3820.
[22] Fatima K, Mathew S, Suhail M, et al. Docking studies of Pakistani HCV NS3 helicase: a possible antiviral drug target. PLoS ONE, 2014, 9(9): 1–12.
[23] Qu LH, Cheng L, Wang JF. Progress on hepatitis C virus NS3 protease inhibitors. Chin Hepatol, 2007, 12(3): 203–206 (in Chinese) .屈莉红, 陈良, 王介非. HCV NS3蛋白酶及抑制剂的研究进展. 肝脏杂志, 2007, 12(3): 203–206.
[24] de Francesco R, Tomei L, Altamura S, et al. Approaching a new era for hepatitis C virus therapy: inhibitors of the NS3-4A serine protease and the NS5B RNA-dependent RNA polymerase. Antiviral Res, 2003, 58(1): 1–16.
[25] Berdichevsky Y, Zemel R, Bachmatov L, et al. A novel high throughout screening assay for HCV NS3 serine protease inhibitors. J Virol Methods, 2003, 107(2): 245–255.
[26] Yamane D, McGivern DR, Wauthier E, et al. Regulation of the hepatitis C virus RNA replicase by endogenous lipid peroxidation. Nat Med, 2014, 20(8): 927–935.
(本文责编陈宏宇)
Prokaryotic expression of Hepatitis C Virus (HCV) NS3 protein and preparation of polyclonal antibody
Tao Sun1, Guangwen Yang1, Jinyang Zhang1,2, and Xueshan Xia1,2
1 Faculty of Life Science and Technology, Kunming University of Science and Technology, Kunming 650500, Yunnan, China 2 Research Center of Molecular Medicine of Yunnan Province, Kunming 650500, Yunnan, China
ToincreasedetectionsensitivityandspecificityonhepatitisC virus(HCV)isvitalforpreventionand controllingofthedisease.Toestablishamorereliable detection method for HCV diagnosis, the full gene fragment of(non-structural protein of HCV) fromrecombinantplasmid of J6/JFH1 2awasamplifiedandthenconnectedintothepET-28a prokaryoticexpressionvector,andthelatterwassubsequentlytransformedintoBL21 (DE3) to havethetargetproteinexpression. Asaresult,aproteinwith amolecularweightof72kDawasobtainedandvisualized in 10%SDS-PAGE.The purifiedNS3 proteinwasusedasimmunogentoinoculateBALB/c miceandthe serawascollected afterthefourthimmunization.Theantibodytiterofserumisdeterminedtobe about1:256 000withELISA. Western blotting and indirectimmunofluorescenceanalysisshowedthatthe mousepolyclonal antibodycouldreactspecificallywiththenativeNS3protein in Huh 7.5.1 cellsinfectedwithHCV.Thesefindingsmayprovide basisfor further preparation of monoclonal antibodies against NS3 and the development of related detection kit.
Hepatitis C virus (HCV), B cell epitopes, NS3 protein, polyclonal antibodies
September 13, 2014; Accepted:December 10, 2014
Xueshan Xia. Tel: +86-871-65939528; E-mail: oliverxia2000@aliyun.com Jinyang Zhang. Tel: +86-871-65939528; E-mail:zhangjinyangzjy@163.com
Supported by:National Natural Science Foundation of China (No. 81260248), National Science and Technology Surport Program of China (No. 2014BAI01B01), Applied Basic Research Projects of Yunnan Province (No. KKS0201326002), Found for Fostering Talents of Kunming University of Science and Technology (No. KKSY201326085), Construction of Science and Technology Innovation Platform of Yunnan Province (No. 2012DA002).
国家自然科学基金 (No. 81260248),国家科技支撑计划项目 (No. 2014BAI01B01),云南省应用基础研究计划 (No. KKS0201326002),昆明理工大学人才培养基金 (No. KKSY201326085),云南省科技创新平台建设计划 (No. 2012DA002) 资助。

