基于云计算的代谢组学数据处理研究
孙海涛,杨志强,耿 越,孙丰霞
(1.山东师范大学信息技术管理处,山东济南 250014;2.山东师范大学实验室与设备管理处,山东济南 250014;3.山东师范大学生命科学学院,山东济南 250014)
基于云计算的代谢组学数据处理研究
孙海涛1,杨志强2,耿 越3,孙丰霞3
(1.山东师范大学信息技术管理处,山东济南 250014;2.山东师范大学实验室与设备管理处,山东济南 250014;3.山东师范大学生命科学学院,山东济南 250014)
根据代谢组学数据处理的特点,提出基于云计算的代谢组学数据并行处理方法:云平台提供多个安装了开源数据处理软件MZmine的计算资源,一个大规模的数据处理任务按照保留时间分散到多个计算资源上进行并行计算。作为实例,对来源于小鼠血清样本的代谢组学数据进行处理。数据说明,基于云计算的数据处理方法能提高数据处理速度,节约计算成本。
云计算;代谢组学;保留时间;数据处理;并行处理
代谢组学是继基因组学和蛋白质组学之后新发展的生命科学领域的一个分支,通过对生物体内所有代谢物进行定量或定性分析来寻找其与病理变化的关系[1-3]。Nature先后发表了多篇利用代谢组学对人类疾病进行研究的论文[4-5]。我国科研管理部门也发布了关于代谢组学的研究课题[6-7]。核磁共振光谱(NMR)和色谱-质谱(HPLC-GS或GC-MS)联用是代谢组学研究中常用的两种测量手段,每种测量手段都会产生大量的数据,处理、分析这些数据的工作量很大,并且需要专门的数学、统计和信息学工具[8]。
为处理代谢组学实验生成的大量数据,研究者对数据处理和分析方法进行了大量研究,并开发了一些高效率的数据处理软件。在数据预处理阶段,针对不同的测量手段,研究人员做了深入的分析,如基于NMR的软件开发[9]、液相色谱-质谱数据高性能分析研究[10]等;也有对数据的处理方式进行研究的,如开发单机版的代谢数据处理软件[11]和在多核计算机上对数据并行处理的软件[12]。对数据进行并行处理是提高数据处理速度的重要手段,例如质谱数据处理软件X!Tandem实现并行以后,原来在单核计算机上需要处理20 h的数据,在40个核的并行环境下,只需要30 min,处理速度是原来的40倍[13]。
在实验数据处理过程中,笔者发现:利用单机版的代谢组学数据处理软件进行高通量计算需要耗费很长时间;而并行软件又存在着费用高、硬件环境要求高、部署难度大的问题。为此,将云计算与成熟的开源数据处理软件MZmine相结合,根据代谢组学数据处理特点,将一个数据处理任务分解为多个子任务,交由不同的计算资源进行并行计算,最后将结果汇总,既加快了数据处理速度,又从软硬件两方面节约了成本。
1 研究的理论基础
1.1 代谢组学数据处理流程
代谢组学数据处理分为原始数据预处理和数据分析两个步骤。
在数据预处理阶段,样品经过色谱-质谱联用仪检测后得到以谱图的形式显示的原始数据,然后由数据处理软件对原始谱图进行处理,得到样品组分的数据表格。代谢组学数据在经过预处理以后,需要对得到的数据进行分析和挖掘,从而发现有价值的信息。
数据预处理是代谢组学研究的第一步,也是最关键的一步。在质谱检测实验中,质谱仪的高灵敏度产生了大量的原始数据,单个样本文件就有几百MB甚至几GB,这些数据需要经过基线校正、谱峰识别、重叠峰解析、保留时间对齐等处理步骤,才能得到与组分有关的信息。这个过程既耗时,又有一定难度。
为高效而又准确地处理这些原始数据,一些仪器生产商和研究机构开发出多种代谢组学数据处理软件,如Marker Lynx、ChromaTOF、Met Align、MZmine等。本研究选用的数据处理软件MZmine是一款免费的开源软件,能够进行可视化数据处理和数据分析[11],而且具有准确数据处理的能力[14]和友好的用户界面。
1.2 原始数据预处理并行化
并行计算是指同时使用多个计算资源解决计算问题。并行计算具有以下特征:计算任务能够分解成多个部分解决,并行后计算速度有很大提高;由不同的计算资源同时执行多个任务[15]。利用云平台提供的计算资源对代谢组学数据并行处理需要考虑任务的分解和并行的速度问题。
1.2.1 任务的并行分解
在色谱-质谱检测中,被分离样品成分从进样开始到出现组分浓度极大值的时间称为组分保留时间(retention time)。在一定的色谱操作条件下,保留时间被用来作为物质的定性依据,通常以分(min)为单位[16]。依据保留时间对样品进行定性或定量分析是代谢组学数据预处理的重要手段。在不同保留时间段内检出的物质成分和数量相差很大。有研究者曾经对按照保留时间分段的代谢数据分别进行处理,汇总结果证实:分段处理检测出的成分与未分段处理相同[17]。
MZmine提供了对谱图按照保留时间分段检测的功能,参数Retention time可以设置为原始数据的整个保留时间段,如0~12,或者设置为0~3、3~6等不同的分段。随后的数据处理将依据Retention time的设置进行。图1是一个原始质谱图按照保留时间平均划分成4个时间段的示意图。在实际计算中,这种划分可以是随意的,但为了保证检测数据的完整性,要保证分段能覆盖整个保留时间(图1是12 min),同时要处理好保留时间重叠区域在计算完成后的数据去重问题。
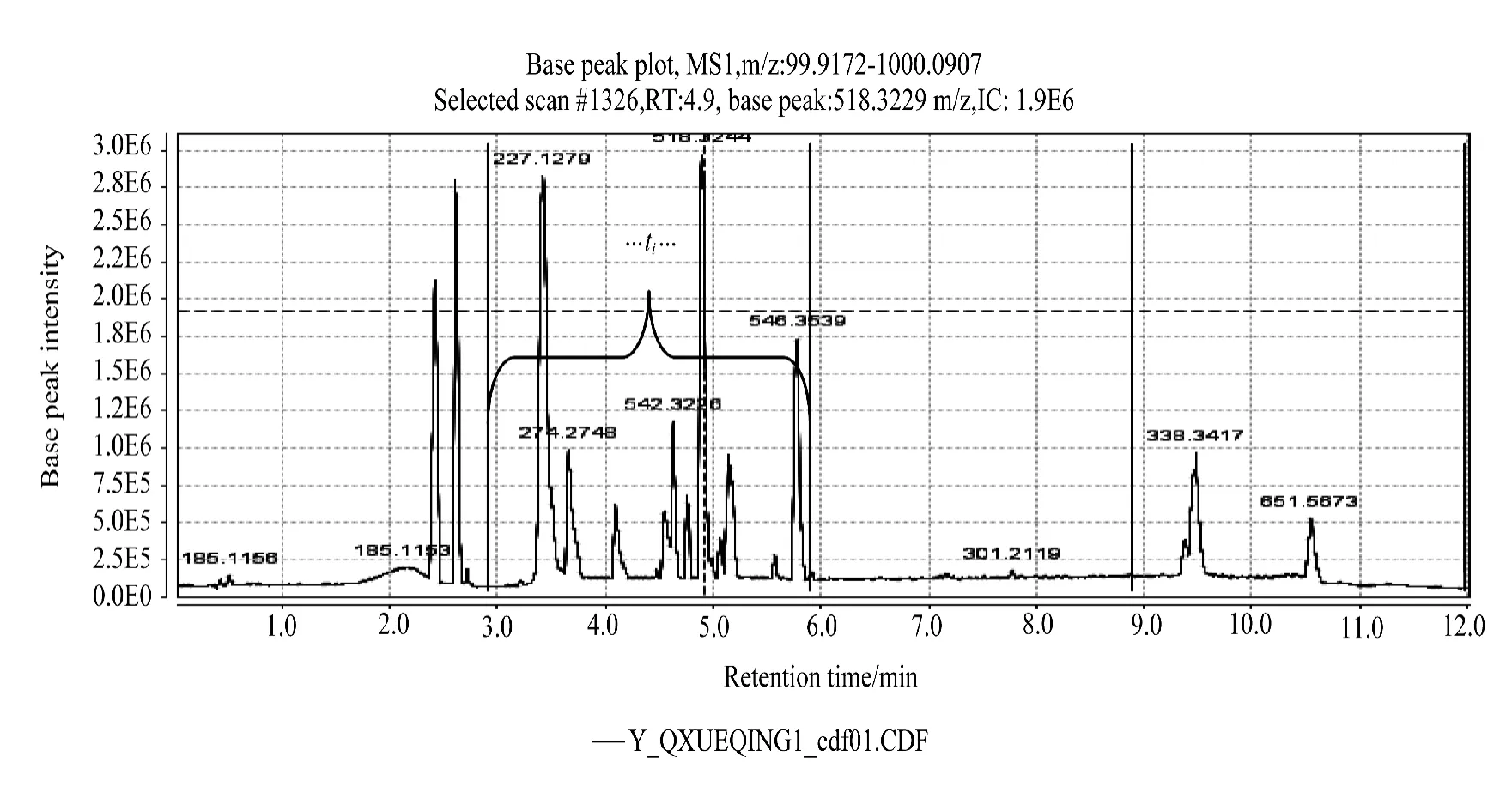
图1 质谱数据分段处理示意图(t2为3.0~6.0)
1.2.2 并行速度
设定处理保留时间为t的原始质谱数据所耗费的时间为ts,则:

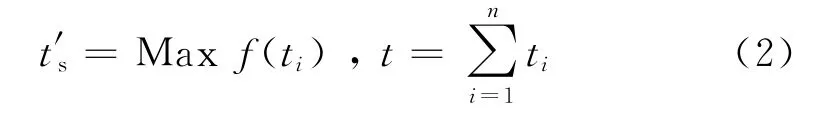
其中ti是两个保留时间之间的时间段;n为计算资源的数目,如t2为3.0~6.0;在只有一个计算资源的情况下,t1=t。(2)式的意思是:当数据并行处理以后,完成任务耗费的时间由所有计算中最长耗时决定。需要指出的是:(1)尽管ts=f(t),但实际ts并非由保留时间t决定,而与检测物的组成有关;(2)如果不考虑额外消耗问题,理论上参与的计算资源越多,单个计算资源所分配的任务就越少,花费的计算时间也越少,但这是在计算任务能够均匀分配的前提下完成的。由于代谢组学数据分析的特殊性,要做到任务的均匀分解并不容易,因此并不能简单地认为计算资源n越大,t′s就越小。
1.3 云计算
云计算的概念由IBM公司于2007年提出,是并行计算、分布式计算和网格计算的进一步发展,能够给用户提供可靠的、自定义的资源利用服务,是一种新的分布式计算模式[18-19]。云计算硬件架构在大规模廉价服务器集群之上,相比功能强大的大型机价格更低廉;通过多个廉价服务器的冗余,保证了系统的稳定性[20]。
利用服务器集群进行代谢组学数据处理是一种粗粒度的并行处理模式,该模式有2个主要优点。
(1)可以按需分配计算资源。在样品的组分检测出来之前,虽然可以预测其组成,但是最终耗时仍需计算决定,所以在固定的保留时间内,由于组分的差别,有的可能需要更多的计算资源。因此,可以在计算过程中根据计算进度,随时提高计算资源的配置,如增加CPU或内存等;当一个计算任务完成后,可以释放计算资源。
(2)计算资源的冗余配置能确保计算安全、高效地进行。在单一计算资源环境下,如果计算资源出现故障,则计算过程需重新开始(或从某个固定的时间点开始),而采用多计算资源并行计算,则只需对故障资源进行替换,重新计算部分数据即可。
将云计算与成熟的代谢组学处理软件相结合以及实现数据处理的并行化,是由云计算与代谢组学数据特点共同决定的。首先是数据处理过程可以并行化,在提高处理效率的同时又不影响结果的准确性;其次是云计算架构在廉价的服务器集群上,集群中的每一个计算资源虽然单独处理超大规模的数据有困难,但是却能完成分解后的小规模的数据处理任务;再就是利用成熟的单机版数据处理软件可以准确而迅速地完成分解后的任务,而且相比并行数据处理软件更容易
当数据处理并行化以后,记数据处理时间为t′s,则:部署和掌握。
2 数据处理
对代谢组学数据进行并行化处理,目的是为了提高数据处理的速度,使一个计算规模很大的数据处理任务能在较短的时间内完成。这可以通过以下实验得到验证。
2.1 实验数据与并行环境
并行处理的数据来源于小鼠血清样本。经过UPLC-QTOF-MS检测得到的原始谱图数据,共有原始谱图50个,数据保留时间都为12 min。从质谱仪得到的数据经过格式转换后由Mzmine2软件完成数据的预处理工作。为了便于比较并行时间,本次实验所使用的计算资源是4个配置相同且满足Mzmine2安装要求的硬件平台。
2.2 数据处理
为检验并行速度,首先要对样本原始数据进行处理,得到数据处理时间,然后进行比较。数据处理主要分以下2个过程。
(1)基准时间tb的确定。为了便于讨论,文中不以每次计算的具体时间做比较,而是设定一个基准时间,其余的实际计算时间与之对比得到相对计算时间,这就使计算结果比较直观,并排除了样品本身性质的影响,从而使处理结果具有普遍意义。本文的基准时间设定为tb=1,是所有原始数据在一个计算资源上一次性计算完成所需要的时间(注:对上述样本数据处理大约用了27 h);
(2)并行时间的确定。将原始数据按照保留时间分段,确定不同的分段规则并记录所花费的时间,然后将计算时间进行归一化处理。表1是对同一原始数据用不同的任务分解法所用计算时间的统计。

表1 同一数据按不同时间段分解所用计算时间
3 实验结果分析
表1说明,第一次数据并行处理所需要的时间t′s=0.47,第二次t′s=0.27,第二次的并行速度更快一些,大约是单一计算资源完成整个数据处理时间的1/4左右。表1还说明,在数据并行处理过程中,不同的任务分解方法并行时间可能不同,按照保留时间平均分解处理任务,并行速度未必是最快;设想在极端的情况下,如果代谢物的组分集中在保留时间的最后一刻,而仍采取平均分配计算资源的方法,则结果是多计算资源并行时间与单机计算时间差不多,并行处理并未加快速度。
按保留时间平均分配计算任务,并行时间是由质谱仪的工作原理和谱图的构成决定的,即在不同的保留时间段内,所检测到的物质组成成分和数量都不相同,因此花费的处理时间也不同。如时间段t2(3~6) 与t1(0~3)相比,检出的组分数目与所耗费的时间都要多。至此,基于云计算的代谢组学并行速度除了与计算资源的配置、数量有关外,更与任务的分解方式有关。在同样的计算规模、同样的计算资源条件下,计算式为

式中N为代谢物检出的成分数目。
按照式(3)来分配计算资源,能使得并行速度最快,但问题是检测结束之前N是未知的。实验证实:根据谱峰和保留时间来进行任务分解,并行速度能得到很大提高;Par Jonsson等人的实验即是按照谱峰划分不同的时间窗口分别进行计算,从而提高了计算速度[17]。
4 结束语
随着代谢组学研究的开展,高效而准确地处理实验中产生的大量数据是一个艰难的任务。本文提出了基于云计算的代谢组学数据并行处理模式,将云计算与成熟的代谢组学数据处理软件相结合。云计算可以按需提供大量的廉价计算资源,成熟的开源软件既能保证数据得到准确的处理,又能降低研究费用。通过对并行任务的分解进行的讨论可知,以保留时间作为参数,以谱图的具体构成作为任务分解的依据,可使并行处理速度最快。实验也证实,对高通量的代谢组学实验数据,采用基于云计算的并行处理方式能显著地提高计算速度。
References)
[1]亓云鹏,胡杰伟,柴逸峰,等.代谢组学数据处理研究的进展[J].计算机与应用化学,2008,25(9):1139-1142.
[2]董继杨,徐乐,曹红婷,等.代谢组学数据分析方法及在糖尿病研究中的应用[J].波谱学杂志,2007,24(4):381-393.
[3]张高勤,王玫,王媛,等.海洛因滥用大鼠尿液同体纵向对照模型的代谢组学研究[J].中国药物依赖性杂志,2013,22(2):85-94.
[4]Clayton T A,Lindon J C,Cloarec O,et al.Pharmaco-metabonomic phenotyping and personalized drug treatment[J].Nature,2006,440 (7087):1073-1077.
[5]Holmes E,Loo R L,Stamler J,et al.Human metabolic phenotype diversity and its association with diet and blood pressure[J].Nature,2008,453(7193):396-400.
[6]许赟.美国空军未来15年科技发展重点[J].航空科学技术,2014, 25(1):1-10.
[7]科技部.国家重点基础研究发展计划和重大科学研究计划2014年重要支持方向[EB/OL].(2013-02-01)[2014-09-10].http:// www.most.gov.cn/tztg/201302/t20130201_99485.htm.
[8]want E J,Nordström A,Morita H,et al.From exogenous to endogenous:the inevitable imprint of mass spectrometry in metabolomics[J].Journal of Proteome Research,2007,6(2):459-468.
[9]董继扬,周玲,Cheng Kain-kai,等.Metaproc:一种基于NMR的代谢组学数据处理软件[C]//第十七届全国波谱学学术会议论文摘要集.2012:145-146.
[10]李灵巧.GC-MS数据高性能分析算法研究[D].桂林:桂林电子科技大学,2011.
[11]Pluskal T,Castillo S,Villar briones A,et al.MZmine 2:Modular framework for processing,visualizing,and analuzing mass spectrometry-based molecular profile data[J].BMC Bioinformatics, 2010,11(1):395-405.
[12]Lommen A,Kools H J.Met Align 3.0:performance enhancement by efficient use of advances in computer hardware[J].Metabolomics,2012,8(4):719-726.
[13]Dexter Duncan and Andrew Link,Vanderbilt University School of Medicine,Parallel Tandem[EB/OL].[2014-09-06].http:// www.thegpm.org/prallel/.
[14]Koh Y,Pasikanti K K,Yap C W,et al.Comparative evaluation of software for retention time alignment of gas chromatography/ time-of-flight mass spectrometry-based metabonomic data[J].Journal of chromatography:A,2010,1217(52):8308-8316.
[15]陈国良,孙广中,徐云,等.并行计算的一体化研究现状与发展趋势[J].科学通报,2009,54(8):1043-1049.
[16]张良晓.气相色谱-质谱定性定量分析新方法研究[D].长沙:中南大学,2011.
[17]Jonsson P,Gullberg J,Nordstrom A,et al.A strategy for identifying differences in large series of metabolomic samples analyzed by GC/MS[J].Anal ytical chem,2004,76(6):1738-1745.
[18]Sims K.IBM introduces ready-to-use cloud computing collaboration services get clients started with cloud computing[EB/OL].[2014-09-06].http://www-03.ibm.com/press/us/en/pressrelease/22613.wss.
[19]李乔,郑啸.云计算研究现状综述[J].计算机科学,2011,38(4): 32-37.
[20]陈康,郑纬民.云计算:系统实例与研究现状[J].软件学报,2009, 20(5):1337-1348.
Study on metabonomic data processing based on cloud computing
Sun Haitao1,Yang Zhiqiang2,Geng Yue3,Sun Fengxia3
(1.Information Management Department,Shandong Normal University,Jinan 250014,China; 2.Administrative Office of Laboratory and Equipment,Shandong Normal University,Jinan 250014,China; 3.School of Life Sciences,Shandong Normal University,Jinan 250014,China)
Metabonomics is a new study branch of life science research after genomics and proteomics.It is looking for the relationships between metabolites of a creature and its pathological changes.Data processing and analysis are the key link of the metabonomics study.According to the data processing characteristic of metabonomics,a new parallel data processing method based on cloud computing is proposed.A large computing task is divided into several small tasks according to the retention time.An open source software named MZmine is used to analyze these small tasks separately with the computing resources provided by the cloud computing platform.The method could improve the speed of data processing and save the cost.
cloud computing;metabonomics;retention time;data processing;parallel processing
TP393
A
1002-4956(2015)4-0171-04
2014-09-19
山东省高等学校科技计划项目(J14LN56)
孙海涛(1979—),男,山东济南,硕士,实验师,主要研究方向为云计算、数据处理和支持向量机.
E-mail:sunht@sdnu.edu.cn

