海岛棉GbCBF6基因克隆及其在逆境胁迫下的表达分析
李月,孔丽颖,李才运,李 翔,代培红,刘晓东
(新疆农业大学 农学院/农业生物技术重点实验室,乌鲁木齐830052)
植物在生长发育过程中会面临各种逆境胁迫, 其中非生物胁迫(低温、干旱和高盐)是影响植物生长发育、导致作物减产的主要因素。植物作为固着生物,不能趋利避害,为了克服这些不利的环境因素,其体内形成了一套复杂的调控机制,涉及生理、生化和与胁迫信号感知、信号传导、基因表达相关的分子调控过程,最终通过代谢调控阻止细胞发生损伤,产生环境适应性[1]。转录因子是一类调控蛋白,通过特异序列组成的DNA 结合功能域,结合下游抗逆功能基因的启动子区,诱导或抑制相应基因的表达,参与生命体不同生理生化途径的调控。根据转录因子的初级结构或三维结构的相似性和多聚化结构域的结构特点,可以分为不同的家族。目前,研究表明在植物中存在至少64个转录因子家族[2-3]。其中,NAC、C2H2 锌 指 蛋 白、bZIP 和WRKY 转 录因子家族是研究最多而且广泛参与非生物胁迫的几个大家族[4-5]。
CRT/DRE (C-repeat/dehydration-responsive element)顺式作用元件结合因子CBF/DREB,属于AP2/ERF亚家族的DNA 结合蛋白,能够特异性结合胁迫诱导基因启动子区域的CRT/DRE 序列,激活靶基因的表达[6]。如干旱和高盐等非生物胁迫,能诱导CBF基因的表达,进而激活COR基因的表达,通过基因产物的作用增强植物的抗逆性[7]。该家族首先在拟南芥中被鉴定出来,是目前已报道的重要的参与胁迫应答的转录因子。在拟南芥中,响应非生物胁迫的6个CBF/DREB1基因,分别参与不同的逆境胁迫。CBF1/DREB1C、CBF2/DREB1B和CBF3/DREB1A受冷胁迫诱导,CBF4/DREB1D、DREB1E/DDF2 和DREB1F/DDF1 响 应 渗 透 胁迫[6,8]。目前,已经在许多植物中鉴定克隆出CBF/DREB基因,如番茄、油菜、玉米、水稻、大麦和小麦等[9-14]。不同植物克隆出的这些同源基因具有高度的保守性,表现出CBF 转录因子典型的结构特性,即C端有酸性激活区,N 端有核定位信号区,中间包含1个由60多个氨基酸组成的AP2结构域和2个CBF 特 征 基 序(PKKR/PAGRxKFxETRHP 和DSAWR)。AP2结构域含有3 个反向平行β折叠和1个α-螺旋结构,参与调控基因的上游启动子区结合,第2个β-折叠的第14位缬氨酸和第19位谷氨酸,起着识别特异DNA 顺式作用元件的作用,PKKP/RAGR 基序是核定位信号(NLS)[15]。根据氨基酸结构特征,该转录因子被分为6个亚类[16]。
棉花作为重要的纤维和油料作物,在全球范围内广泛种植,与水稻、小麦、玉米等主要作物相比,棉花具有较强的抗旱、耐盐性。尽管如此,由于全球气候变化和环境污染,非生物胁迫已成为影响棉花正常生长和产量的主要限制因子[17]。因此,揭示棉花耐逆的分子机理、克隆与逆境胁迫相关的重要功能基因对于棉花耐逆性和广适应性的基因工程改良,扩大棉花生产具有重要的战略意义。本研究利用生物信息学和PCR 的方法,从海岛棉‘新海16’中克隆了1 个新的CBF基因GbCBF6,并对其序列特征、在高盐、干旱和低温胁迫下的表达模式进行了研究,为进一步了解CBF转录因子基因家族功能及其调控的分子机制提供理论依据,为利用基因工程手段提高棉花抗逆性提供重要信息。
1 材料与方法
1.1 材 料
实验材料为海岛棉品种‘新海16’,由新疆农业大学农学院作物遗传育种室提供。选取(25±2)℃培养间生长一致的15d苗龄的棉花幼苗,参照Li等[18]已报道的棉花胁迫处理方法,分别进行干旱、高盐和4 ℃低温处理。干旱处理:将棉苗置于干净的滤纸上,在(25±2)℃、相对湿度45%、连续光照条件下进行自然干旱处理;高盐处理:将苗的根部浸入200mmol/L NaCl溶液中;冷处理:棉苗置于盛有水(已预冷到4 ℃)的大烧杯中,放置于4 ℃培养箱,持续光照。每种胁迫处理10株棉花。分别于上述各处理的0、1、3、6和12h采集2株真叶叶片样品,迅速置于液氮冷冻,用于总RNA 提取。
1.2 方 法
1.2.1 总RNA的提取和cDNA的制备 采用改良的CTAB法,参照Hu等[19]的方法提取棉花不同胁迫处理时间点叶片的总RNA,按照TaKaRa(公司)RNase-Free DNase I试剂盒的方法消化基因组DNA 污染,按照Promega(公司)M-MLV 反转录试剂盒操作说明合成单链cDNA,于-20 ℃保存,用于基因胁迫表达特征分析。
1.2.2 棉花GbCBF基因的克隆 以拟南芥TAIR(http://www.arabidopsis.org/index.jsp)的AtCBF2基因(AT4G25470.1)的序列作为BlastP查询序列,在棉花EST数据库(http://www.leonxie.com)、植物转录因子数据库(PTFD,http://planttfdb.cbi.edu.cn)和美国国家生物技术信息中心(NCBI,http://www.ncbi.nlm.nih.gov)中搜索AP2/ERF亚家族基因与AtCBF2序列保守的EST序列,以其在已公布的棉花基因组数据库(http://www.phytozome.net/)中比对,获得了1个cDNA序列,然后根据此序列的ORF(Open reading frame)序列设计引物(GhCBF6F:5′-ATGGAGTATTTGGAGATTGATTCT-3′和GhCBF6R:5′-TTAGTAATGTCGCCATAAATCA-3′)。以海岛棉‘新 海16’cDNA 为模板,以高保真聚合酶TransStar KD Plus(北京全式金)进行PCR 扩增。高保真Trans-Star KD Plus(TransGen)扩增反应体系为:5×TransStar KD Plus Buffer 10μL;2 mmol·L-1dNTP 5μL;TransStar KD Plus DNA Polymerase 1μL;模板1μL;正反向引物各1μL,补充水到50 μL。反应条件为:94 ℃2 min;94 ℃10s;60 ℃30s;68 ℃45s,35个循环,68 ℃10min。将扩增产物经质量浓度为1%琼脂糖凝胶电泳检测,回收目的片段连接至Peasy-Blunt Zero克隆载体上(北京全式金),然后测序验证。
1.2.3 棉花GbCBF基因的生物信息学分析 将所得的GbCBF基因的ORF用DNAStar翻译成蛋白序列,并用EXPASy的ProtParam tool在线程序(http://web.expasy.org/cgi-bin/protparam/protparam)预测蛋白的分子量和等电点。用Psort(http://www.psort.org/)在线工具完成蛋白亚细胞定位预测。以AtERF1 的DNA 结合域(AtERF1-DNA-binding domain,1gcc)做模板,利用SWISSMODEL(http://swissmodel.expasy.org/workspace/)软件预测棉花CBF 家族蛋白AP2/ERF 结构域的蛋白质三级结构模型。利用NCBI的BlastP对棉花蛋白序列进行氨基酸同源序列比对分析;由DNAMAN 软件完成多重序列比对分析;用Clustal X 进行系统进化树分析,采用邻接法(Neighbor-Joining,NJ)方法构建系统发生树,分支的可靠性评价采用靴带分析(Bootstrap,1000),采用MEGA5对系统树进行作图。
1.2.4 棉花GbCBF基因的半定量RT-PCR 以GbCBF基因的GbCBF6F和GbCBF6R 引物为半定量RT-PCR 引物,以棉花GhACT2(基因登录号:AY305724.1)为内参基因(ACT2F:5′-CGTACAACAGGTATTGTGCTGG-3′,ACT2R:5′-GAAATCCACATCTGCTGGAAGGTG-3′),以干旱、高盐和4 ℃低温处理不同时间点的海岛棉‘新海16’的叶片cDNA 为模板,进行RT-PCR 扩增。RT-PCR反应体系为20μL,包括0.5~1μL cDNA、正反向引物(10μmol·L-1)各0.5μL、2μL 10×PCR 缓冲液(含Mg2+)、0.5μL dNTPs(10 mmol·L-1each)和0.5μL EasyTaq DNA 聚合酶(北京全式金),用灭菌超纯水补至20μL。在Eppendorf PCR仪上进行,反应条件为94 ℃预变性5min;98 ℃变性30s,55~60 ℃退火30s,72 ℃延伸30s,27~33个循环;72 ℃延伸10 min。PCR 产物用1%琼脂糖凝胶电泳检测,并用Imaging DensitoMeter(Bio-Rad)中的Image Lab软件进行拍照分析。
2 结果与分析
2.1 GbCBF6基因的克隆与序列分析
以‘新海16’叶片cDNA 为模板进行的PCR 扩增结果表明,从海岛棉中克隆1个CBF基因cDNA序列,命名为GbCBF6,提交GenBank 获得基因登陆号KR233255。GbCBF6基因的最大开放阅读框(ORF)为753bp(图1),编码251 个氨基酸,用DNAStar软件预测该蛋白质分子量为27.82kD,等电点为7.68。‘新海16’中的GbCBF6基因的全长ORF序列与其对应基因组中的基因编码序列完全一致,结果表明GbCBF6基因不含有内含子。
用DNAMAN 软件对GbCBF6 基因编码的氨基酸序列进行多重序列比对分析。结果表明,Gb-CBF6蛋白具有CBF 蛋白典型的结构特点,包含1个AP2 结 构 域 和2 个CBF 特 征 序 列,PKKR/PAGR 基序和DSAWR 基序,和1个C-端的酸性结构域(图2)。
目前已在棉花中克隆了6 个CBF同源基因,GhDBP1、GhDBP2、GhDBP3、GhDREB、GhDREB1和GhDREB1L,其GenBank登录号分别是AY174160、AY619718、DQ224382、AF509502、AY920495 和DQ409060,对这6个GhCBF基因与本研究克隆的GbCBF6基因进一步比对分析,同时以拟南芥AtCBF1、AtCBF2 和AtCBF3 基 因 为 参 照。结 果如 表1所 示,GhDBP1 和GhDREB基 因 编 码 的 蛋白 氨 基 酸 序 列 的 相 似 性 为1 0 0%;GhDREB1和GhDREB1L之间相似性为98.2%,应该为同一基因。另外这6个基因与已克隆的GbCBF6基因相似性均低于38.5%,而且除GhDREB1 和GhDREB1L外,GbCBF6 与 拟 南 芥AtCBF1、AtCBF2、AtCBF3基因的相似性要高于已克隆的棉花其他CBF同源基因。
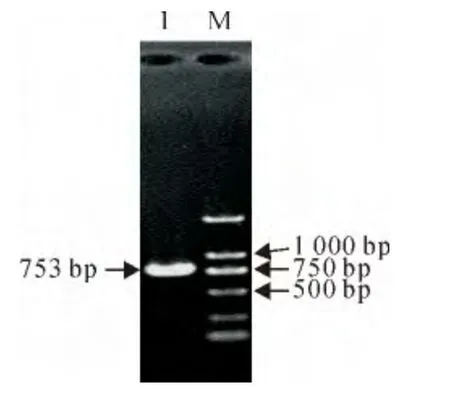
图1 棉花GbCBF6基因PCR 产物的电泳分析Fig.1 Electrophoresis of PCR product of cotton CBFgenes

图2 不同植物CBF/DREB蛋白氨基酸序列比对Fig.2 Alignment of amino acid sequences of CBF/DREB from different plants

图3 GbCBF6蛋白与其他物种CBF蛋白的系统进化树Fig.3 Phylogenetic tree of GbCBF6and CBF proteins from other species
2.2 棉花GbCBF6蛋白系统进化树分析

图4 GbCBF6蛋白三维结构预测Fig.4 Three-dimensional structure prediction of GbCBF6protein
Sakuma等[16]根据CBF/DREB 转录因子的结构特点,把该类蛋白分为A-1~A-6共6个亚组,我们以此为基础,选取拟南芥CBF/DREB不同亚类的基因,并从其他的单双子叶植物包括水稻(Oryza sativa)、大麦(Hordeumvulgare)、玉米(ZeamaysL.)、棉 花(Gossypiumhirsutum)、大 豆(Glycine max)中选取已克隆并鉴定的CBF/DREB转录因子基因序列,用于构建系统进化树(图3)。图3显示,GbCBF6蛋白和A-1 亚组成员AtCBF1、AtCBF2、AtCBF3和HvCBF1聚为一类,属于CBF/DREB转录因子A-1 亚类。与拟南芥AtCBF1、AtCBF2、AtCBF3蛋白相似性在40%以上(表1)。
利用AtERF1 DNA 结 合 域(AtERF1-DNAbinding domain,1gcc)做模板,利用SWISS-MODEL(http://swissmodel.expasy.org/workspace/)软件对棉花CBF 家族蛋白AP2/ERF 结构域进行蛋白质三级结构模型预测。结果如图4所示,GbCBF6 蛋白的三维结构与AtERF1(PDB ID:1gcc)的三级结构模型图相似,均含有CBF 转录因子典型结构特点,即含有3个β折叠和1个α-螺旋结构,进一步证实获得的GbCBF6 基因属于CBE/DREB转录因子家族。此外,利用在线软件Psort(http://www.psort.org/)预测GbCBF6 蛋白定位在细胞核中,属于核定位转录因子。

表1 棉花CBF 基因相似性分析Table1 Similarity analysis of CBFgenes cloned in cotton
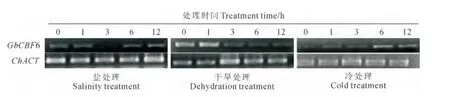
图5 各种非生物胁迫下GbCBF6基因的表达谱Fig.5 Expression profiles of GbCBF6under various abiotic stresses
2.3 GbCBF6基因的诱导表达分析
用高盐、干旱和低温3种非生物胁迫处理棉花,可以较全面地反映GbCBF6 基因在非生物胁迫中的应答机制。RT-PCR 结果(图5)表明,GbCBF6基因对以上3种逆境胁迫均有应答,但是表达模式存在明显差异。在盐处理下,GbCBF6基因在处理3h时表达量最低,几乎检测不到,在0、1、6和12h的表达量基本相同。干旱处理下,GbCBF6基因随着胁迫处理时间的延长,表达量逐渐下降,在12h时最低;而在低温处理下,GbCBF6基因随着胁迫处理时间的延长,基因表达量逐渐升高。
3 讨 论
由于全球耕地面积的减少,粮棉争地矛盾加剧,积极开发旱地、盐碱地植棉潜力,将成为棉花生产保持相对稳定的重要应对策略,因此培育多抗棉花品种已是当前棉花育种的重要目标。转录因子在植物耐逆改良中发挥着关键作用。近年来,从棉花植株分离耐逆关键转录因子基因,应用基因操作技术创制棉花耐逆新材料已成为研究重点。如CCCHtype的锌指结构基因GhTZF1、WRKY 转录因子基因GhWRKY39-1,它们过表达,均能增强植物的抗逆性[20-21]。
本研究利用生物信息学和PCR 的方法在海岛棉中克隆了1 个与拟南芥CBF2 同源的GbCBF6基因。和已报道的双子叶植物CBF基因一样,该基因无内含子,其预测的编码产物大小与拟南芥CBF转录因子相似,表现出CBF转录因子的主要结构特点。GbCBF6蛋白在N 端区域有1 个碱性氨基酸链[PKK(R)RAGRK(R)K(V)FR(Q/K)],与拟南芥AtCBF1、AtCBF2、AtCBF3 中的核定位信号一致[6]。GbCBF6蛋白结构的高度保守性表明,它们可能和其他植物CBF 蛋白一样在植物抗逆调控中扮演重要角色。
研究表明,将拟南芥植物暴露在低温环境下,通过组成型表达AtCBF1、AtCBF2 或AtCBF3 基因增强植物的耐逆性,包括抗冻性[22]。近年来,利用RNA 干扰和反义技术分别下调AtCBF1 和AtCBF3基因表达,导致冷处理的植物抗冻性下降25%~40%[23],这些结果表明拟南芥AtCBF基因参与植物响应和耐受低温胁迫。目前已经在多种植物中鉴定出响应低温胁迫的CBF基因[24]。一般认为蛋白质具有相似的结构域可能具有相似的生物学功 能,GbCBF6 蛋白和A-1亚组成员AtCBF1、AtCBF2、AtCBF3聚为一类,与它们的相似性均在40%以上,推测可能发挥相同的功能。因此本研究分析了GbCBF6基因在高盐、干旱和低温逆境胁迫处理下基因的表达情况。结果表明,GbCBF6基因受4℃低温上调表达,受干旱胁迫下调表达,而高盐处理下其表达量先是下降,然后再增加,推测Gb-CBF6基因除参与棉花低温信号传导,也参与了棉花渗透胁迫信号传递调控网络,在海岛棉抵抗不同逆境中可能扮演重要的角色。
关于棉花CBF/DREB基因,分别报道了6 个CBF基因[25],本研究经过比对发现,其中有2对基因相似性高达98%以上,应该为同一基因。因此,认为棉花上已有4个CBF基因被鉴定出来。它们分别属于DREB家族的A-1亚组、A-4亚组、A-5亚组和A-6亚组。本研究的GbCBF6蛋白与这4 个棉花CBF蛋白氨基酸相似性均低于38.5%,推测为棉花新的CBF基因,而且该基因与拟南芥AtCBF1、AtCBF2、AtCBF3蛋白一起聚类在一个亚组。结果暗示,与已克隆的陆地棉4 个GhCBF基因相比,除属于A-1亚组的1 个陆地棉CBF基因(GenBank 登陆号为DQ409060 和AY920495)外,本研究克隆的GbCBF6 基因与拟南芥AtCBF1、AtCBF2、AtCBF3基因编码的蛋白氨基酸的相似性均高于已克隆的棉花其他CBF同源基因。以上结果显示本研究克隆的GbCBF6 基因为新的未报道的CBF同源基因,而且功能可能与已报道的拟南芥抗逆AtCBFs基因更加接近。
综上,以上研究结果为研究GbCBF6基因的功能以及利用转基因技术培育抗冻棉花新品种奠定坚实的前期研究基础,关于该基因的具体功能和调控机理是未来研究工作的重点。
[1] PAGTER M,ARORA R.Winter survival and deacclimation of perennials under warming climate:physiological per spectives[J].PhysiologiaPlantarum,2013,147(1):75-87.
[2] PEREZ-RORDIGUEZ P,RIANO-PACHON D M,CORREA L G,etal.PlnTFDB:updated content and new features of the plant transcription factor database[J].NucleicAcidsResearch,2010,38(Database issue):D822-D827.
[3] ALVES M S,DADALTO S P,GONCALVES A B,etal.Plant bZIP transcription factors responsive to pathogens:a review[J].InternationalJournalofMolecularScience,2013,14(4):7 815-7 828.
[4] LLORCA C M,POTSCHIN M,ZENTGRAF U.bZIPs and WRKYs:two large transcription factor families executing two different functional strategies[J].FrontiersinPlantScience,2014,5:169.
[5] MAO X G,CHEN S S,LI A,etal.Novel NAC Transcription factorTaNAC67confers enhanced multi-abiotic stress tolerances inArabidopsis[J].PLoSOne,2014,9(1):e84359.
[6] NOVILLO F,MEDINA J,RODRIGUEZ-FRANCO M,etal.Genetic analysis reveals a complex regulatory network modulatingCBFgene expression and Arabidopsis response to abiotic stress[J].JournalofExperimentalBotany,2012,63(1):293-304.
[7] DIETZ K J,VOGEL M,VIEHHAUSER A.AP2/EREBP transcription factors are part of gene regulatory networks and integrate metabolic,hormonal and environmental signals in stress acclimation and retrograde signalling[J].Protoplasma,2010,245(1-4):3-14.
[8] PARK S,LEE C M,DOHERTY C J,etal.Regulation of theArabidopsisCBF regulon by a complex low-temperature regulatory network[J].ThePlantJournal,2015,82(2):193-207.
[9] KIM Y H,YANG K S,RYU S H,etal.Molecular characterization of a cDNA encoding DRE-binding transcription factor from dehydration-treated fibrous roots of sweet potato[J].PlantPhysiologyBiochemistry,2008,46(2):196-204.
[10] CONG L,CHAI T Y,ZHANG Y X.Characterization of the novel geneBjDREB1Bencoding a DRE-binding transcription factor fromBrassicajunceaL.[J].BiochemicalandBiophysicalResearchCommunications,2008,371(4):702-706.
[11] LIU S,WANG X,WANG H,XIN H,etal.Genome-wide analysis ofZmDREBgenes and their association with natural variation in drought tolerance at seedling stage ofZeamaysL.[J].PLoSGenetics,2013,9(9):e1003790
[12] DUBOUZET J G,SAKUMA Y,ITO Y,etal.OsDREBgenes in rice,OryzasativaL.,encode transcription activators that function in drought-,high salt-and cold-responsive gene expression[J].ThePlantJournal,2003,33(4):751-763.
[13] SKINNER J S,ZITZEWITZ J,SZUCS P,etal.Structural,functional,and phylogenetic characterization of a large CBF gene family in Barley[J].PlantMolecularBiology,2005,59(4):533-551.
[14] KUME S,KOBAYASHI F,ISHIBASHI M,etal.Differential and coordinated expression ofCBFandCor/Leagenes during long-term cold acclimation in two wheat cultivars showing distinct levels of freezing tolerance[J].GenesandGeneticSystems,2005,80(3):185-197.
[15] AKHTAR M,JAISWAL A,TAJ G,etal.DREB1/CBF transcription factors:their structure,function and role in abiotic stress tolerance in plants[J].JournalofGenetics,2012,91(3):385-395.
[16] SAKUMA Y,LIU Q,DUBOUZET J,etal.DNA-binding specificity of the ERF/AP2domain of Arabidopsis DREBs,transcription factors involved in dehydration-and cold-inducible gene expression[J].BiochemicalandBiophysicalResearchCommunications,2002,290(3):998-1 009.
[17] QIN F,SHINOZAKI K,YAMAGUCHI-SHINOZAKI K.Achievements and challenges in understanding plant abiotic stress responses and tolerance[J].PlantandCellPhysiology,2011,52(9):1 569-1 582.
[18] LI Y(李月),SUN J(孙 杰),CHEN SH Y(陈受宜),etal.Cloning and transcription function analysis of cotton transcription factorGhGT30gene[J].ActaAgronomicaSinica(作物学报),2013,39(5):806-815(in Chinese).
[19] HU G H(胡根海),YU SH X(喻树迅).Extraction of high-quality total RNA in cotton leaf with improved CTAB method[J].CottonScience(棉花学报),2007,19(1):69-70(in Chinese).
[20] ZHOU T,YANG X,WANG L,etal.GhTZF1regulates drought stress responses and delays leaf senescence by inhibiting reactive oxygen species accumulation in transgenicArabidopsis[J].PlantMolecularBiology,2014,85(1-2):163-177.
[21] SHI W,HAO L,LI J,etal.TheGossypiumhirsutumWRKYgeneGhWRKY39-1promotes pathogen infection defense responses and mediates salt stress tolerance in transgenicNicotianabenthamiana[J].PlantCellReports,2014,33(3):483-498.
[22] JAGLO K R,KLEFF S,AMUNDSEN K L,etal.Components of theArabidopsisC-repeat/dehydration-responsive element binding actorcold-response path way are conserved inBrassicanapusand other plant species[J].PlantPhysiology,2001,127(3):910-917.
[23] LEE C M,THOMASHOW M F.Photoperiodic regulation of the C-repeat binding factor(CBF)cold acclimation pathway and freezing tolerance inArabidopsisthaliana[J].ProceedingsoftheNationalAcademyofSciencesoftheUnitedStatesofAmerica,2012,109(37):15 054-15 059.
[24] KIDOKORO S,WATANABE K,OHORI T,etal.Soybean DREB1/CBF-type transcription factors function in heat and drought as well as cold stress-responsive gene expression[J].ThePlantJournal,2015,81(3):505-518.
[25] GAO S Q,CHEN M,XIA L Q,etal.A cotton(Gossypiumhirsutum)DRE-binding transcription factor gene,GhDREB,confers enhanced tolerance to drought,high salt,and freezing stresses in transgenic wheat[J].PlantCellReports,2009,28(2):301-311.

