蒺藜苜蓿LEA基因家族全基因组分析
刘志敏,刘文献,贾喜涛,张正社,王彦荣
(草地农业生态系统国家重点实验室,兰州大学草地农业科技学院,甘肃 兰州 730020)
蒺藜苜蓿LEA基因家族全基因组分析
刘志敏,刘文献,贾喜涛,张正社,王彦荣
(草地农业生态系统国家重点实验室,兰州大学草地农业科技学院,甘肃 兰州 730020)
胚胎发育晚期丰富蛋白(LEA)是一类在种子胚胎发育后期特异表达并受发育阶段及脱水信号调节的脱水保护蛋白,在响应植物干旱、低温、高盐等逆境胁迫中具有重要功能。本研究利用生物信息学手段,在全基因组水平对蒺藜苜蓿(Medicagotruncatula)LEA基因家族进行分析,并对其进化、基因结构、进化压力、染色体定位及基因表达模式进行了系统分析。本研究共筛选出23个蒺藜苜蓿LEA家族基因,可分为8个亚家族。基因定位结果表明,23个蒺藜苜蓿LEA基因分布于除6号染色体外的其他7条染色体上,但分布不均匀;家族成员外显子数目都不超过两个,结构简单。不同组织表达谱分析结果显示,LEA家族基因具有不同的组织表达模式,并受干旱逆境胁迫调控。本研究结果可为蒺藜苜蓿LEA基因家族的功能分析奠定基础。
蒺藜苜蓿;LEA基因家族;系统进化;表达分析
胚胎发育晚期丰富蛋白(Late Embryogenesis Abundant Proteins,LEA)最早发现于棉花(Gossypiumhirsutum)子叶中,因其在植物胚胎发育后期大量表达而得名[1-2]。LEA基因家族分布广泛,不仅存在于被子植物、裸子植物和藻类中[3-4],在线虫(Aphelenchusavenae)[5]、轮足虫(Rotifer)[6]和蓝细菌(Cyanobacteria)[7]中也普遍存在。根据蛋白质序列同源性及其特殊的基元序列,可将LEA蛋白分为不同类群[8-11]。研究发现,不同LEA蛋白都具有高亲水性和热稳定性,并富含甘氨酸或其他氨基酸类(如丙氨酸、丝氨酸和苏氨酸)[4]。LEA基因家族系统进化分析最先在拟南芥(Arabidopsisthaliana)中被报道,其共有51个成员[12],随后在水稻(Oryzasativa)[13]、大豆(Glycinemax)[14]和杨树(Populus)[15]中也发现分别含有34、35和40个LEA基因。
植物LEA蛋白及其转录本累积水平主要在种子胚胎发育后期、种子脱水之前显著提高;当植物处于渗透胁迫、脱水以及低温胁迫条件下,该类蛋白也会在植物营养组织中高水平累积,与植物耐脱水性密切相关,具有保护植物组织免受水分胁迫伤害的功能[16-19]。前人研究结果显示,LEA基因家族受多种逆境胁迫调控表达[20]。例如,在干旱和低温胁迫条件下,青杨(P.cathayana)LEA2、LEA3基因在叶片中的表达量显著提高[21]。在干旱条件下,拟南芥营养组织中LEA蛋白累积量升高[22];当拟南芥AtLEA4基因在拟南芥中过表达后,其抗旱性相比野生型显著提高,AtLEA4基因表达量降低则导致拟南芥对干旱胁迫更为敏感[23]。应用RNA干扰技术诱导拟南芥LEA14及其同源脱水蛋白(LEA34和LEA51)表达量降低后,种子贮存寿命显著降低,说明LEA蛋白对种子贮存具有十分重要的作用[24]。小麦(Triticumaestivum)TaLEA2和TaLEA3分别在酵母细胞中过表达后,相比之对照,转化酵母细胞的抗干旱和耐低温胁迫能力都显著增强[25]。在中华羊草(Leymuschinensis)中过表达TaLEA3后,转基因植株的抗旱性显著提高[26]。小麦的PMA1959(LEA1)和PMA80(LEA2)基因在水稻中过表达可明显提高转基因植株在干旱胁迫下的叶绿素含量和根系的重量[27]。另外,在草莓(Fragaria)中过表达小麦LEA(WCOR410)基因后能显著提高草莓叶片的抗寒性[28],而将大麦(Hordeumvulgare)LEA(HVA1)分别在水稻和小麦中过表达后能同时提高转基因植物的抗旱性和耐盐性[29-30]。
蒺藜苜蓿(Medicagotruncatula)是紫花苜蓿(M.sativa)的近缘种,具有生长周期短、自花授粉、基因组小且遗传转化效率高等特点,使其成为研究豆科,特别是苜蓿属植物,基因组学和生物学的模式植物[31]。蒺藜苜蓿全基因组测序已经完成,使得利用生物信息学手段研究该物种基因家族系统演化及其功能成为可能,同时借鉴蒺藜苜蓿研究及其基因资源对促进豆类作物遗传研究具有重要意义[32]。截至目前,在全基因组水平对蒺藜苜蓿LEA基因家族的研究尚未见报道。本研究利用生物信息学手段,从全基因组水平对蒺藜苜蓿LEA基因家族的结构特征、进化关系、染色体位置及表达模式进行系统分析,以期为进一步研究LEA蛋白家族功能和豆科牧草作物遗传改良奠定基础。
1 材料与方法
1.1 材料
蒺藜苜蓿基因组序列下载于JCVI数据库(http://jcvi.org/medicago/index.php),拟南芥LEA基因序列和蛋白质序列下载于Phytozome数据库(http://phytozome.jgi.doe.gov/pz/portal.html)[33]。蒺藜苜蓿芯片探针数据来自Affymetrix公司(http://www.affymetrix.com/analysis/index.affx)[34],所得探针于诺贝尔基金会(Noble Foundation)数据库(http://mtgea.noble.org/v3/)[35-36]搜索LEA基因在各器官中的表达量,进行基因表达分析。
1.2 蒺藜苜蓿LEA基因家族成员鉴定
以蒺藜苜蓿全基因组序列构建本地BLAST数据库,利用已鉴定出的51个拟南芥LEA基因家族成员执行本地BLAST(P-value=le-003)搜索同源序列。利用Pfam数据库[37]对获得的蒺藜苜蓿同源LEA基因进行蛋白结构预测,筛选含有LEA典型结构域的蛋白序列。利用Expasy蛋白质组数据库(http://web.expasy.org/protparam/)对蒺藜苜蓿LEA蛋白进行蛋白质分子量、理论等电点和总平均亲水性预测[38]。
1.3 蒺藜苜蓿LEA基因染色体定位
利用Phytozome植物全基因组数据库(http://phytozome.jgi.doe.gov/pz/portal.html)确定蒺藜苜蓿LEA基因染色体位置,利用Mapchart 2.2工具[39]进行染色体定位作图。
1.4 蒺藜苜蓿LEA基因家族成员系统进化树构建及基因结构分析
利用MEGA 6.0[40]对蒺藜苜蓿LEA蛋白进行多序列联配比对分析,采用邻接法构建系统进化树。系统进化树选用p-distance模型,校验参数Bootstrap设为1 000次重复。利用在线工具GSDS 2.0(http://gsds.cbi.pku.edu.cn/)[41]分析LEA基因家族成员基因结构。
1.5 蒺藜苜蓿LEA基因进化压力分析
利用在线工具Clustal Omega[42]和PAL2NAL[43]计算核苷酸的同义替换率Ks(synonymous substitution rate or synonymous substitutions per synonymous site)、非同义替换率Ka(nonsynonymous substitution rate or nonsynonymous substitutions per nonsynonymous site)及两者比率Ka/Ks。
1.6 蒺藜苜蓿LEA基因表达分析
利用Affymetrix基因探针数据库搜索蒺藜苜蓿LEA基因相匹配的探针,从蒺藜苜蓿基因芯片平台(http://mtgea.noble.org/v3/)获得对应探针表达量信息,并使用MeV4[44]软件对蒺藜苜蓿LEA基因在不同组织、不同时期、不同处理的表达芯片数据进行数据聚类分析和作图。
2 结果与分析
2.1 蒺藜苜蓿LEA基因家族的鉴定和分类
通过BLAST比对分析,本研究共初步筛选出32个候选蒺藜苜蓿LEA基因。将所得序列在Pfam网站进行基因结构域分析并删除不含LEA结构域的基因,最终得到23个蒺藜苜蓿LEA基因。根据结构域可将23个LEA基因分为8个亚家族(表1),包括4个LEA1、2个LEA2、2个LEA3、5个LEA4、1个LEA5、2个LEA6、4个SMP和3个Dehydrin。利用在线工具ExPASy对蒺藜苜蓿LEA蛋白质分子量、理论等电点系数和总平均亲水性进行分析发现,23个LEA蛋白中分子量最大为55 384.9 D(LEA4-5),最小为6 740.2 D(LEA4-4);等电点的范围从4.54(LEA7-4)到9.55(LEA1-4)不等;所有LEA蛋白除LEA2-2外均表现为亲水性(表1)。
2.2 蒺藜苜蓿LEA基因家族染色体定位
蒺藜苜蓿LEA基因染色体定位结果显示,23个基因分布于除第6条染色体以外的其他7条染色体上,但分布不均一(图1)。其中,第2、4和7染色体上分布最多,各有5个LEA基因;其次为第1和第3染色体,各3个;第5和第8染色体上最少,各1个。LEA1-2/LEA1-3/LEA1-4、LEA4-2/LEA4-3、LEA7-2/LEA7-3及LEA8-1/LEA8-2在染色体上连锁在一起,属于旁系同源基因,其中LEA7-2/LEA7-3的步长值最高(100)。
2.3 蒺藜苜蓿LEA家族基因系统进化及其基因结构分析
为研究蒺藜苜蓿LEA基因系统进化关系,对筛选出的蒺藜苜蓿LEA蛋白进行了系统进化树构建(图2)。分析结果显示,23个蒺藜苜蓿LEA基因分布在6个无根系统进化树,其中LEA1、LEA5和LEA8三个亚家族分布在同一分支上,并明显地分为3个次亚家族;同时,其他5个亚家族也可明显分为2个次亚家族且含有不同基因数。
利用GSDS在线工具对LEA基因结构进行作图分析。结果显示,蒺藜苜蓿LEA基因结构相对简单,所含基因内含子的数量均不多于两个。其中LEA6-1、LEA6-2和LEA8-3不含有内含子,LEA4-4、LEA7-1和LEA7-3均各含有两个内含子,剩余17个基因只含有1个内含子(图3)。
2.4 蒺藜苜蓿LEA基因进化选择压力分析
同义替换(Synonymous Substitution)和非同义替换(Nonsynonymous Substitution)是生物进化过程中核苷酸替换的两种形式,Ka、Ks及Ka/Ks能反映出基因进化过程中受到的选择压力。对6对旁系同源基因的分析结果显示(表2),6对旁系同源基因Ka/Ks值在0.041 9到0.614 8之间,均小于1。其中,Ka/Ks最大值为LEA4-2和LEA4-3(0.614 8);Ka/Ks最小值为LEA3-1和LEA3-2(0.041 9),表明蒺藜苜蓿LEA基因进化时存在负向选择作用[45]。
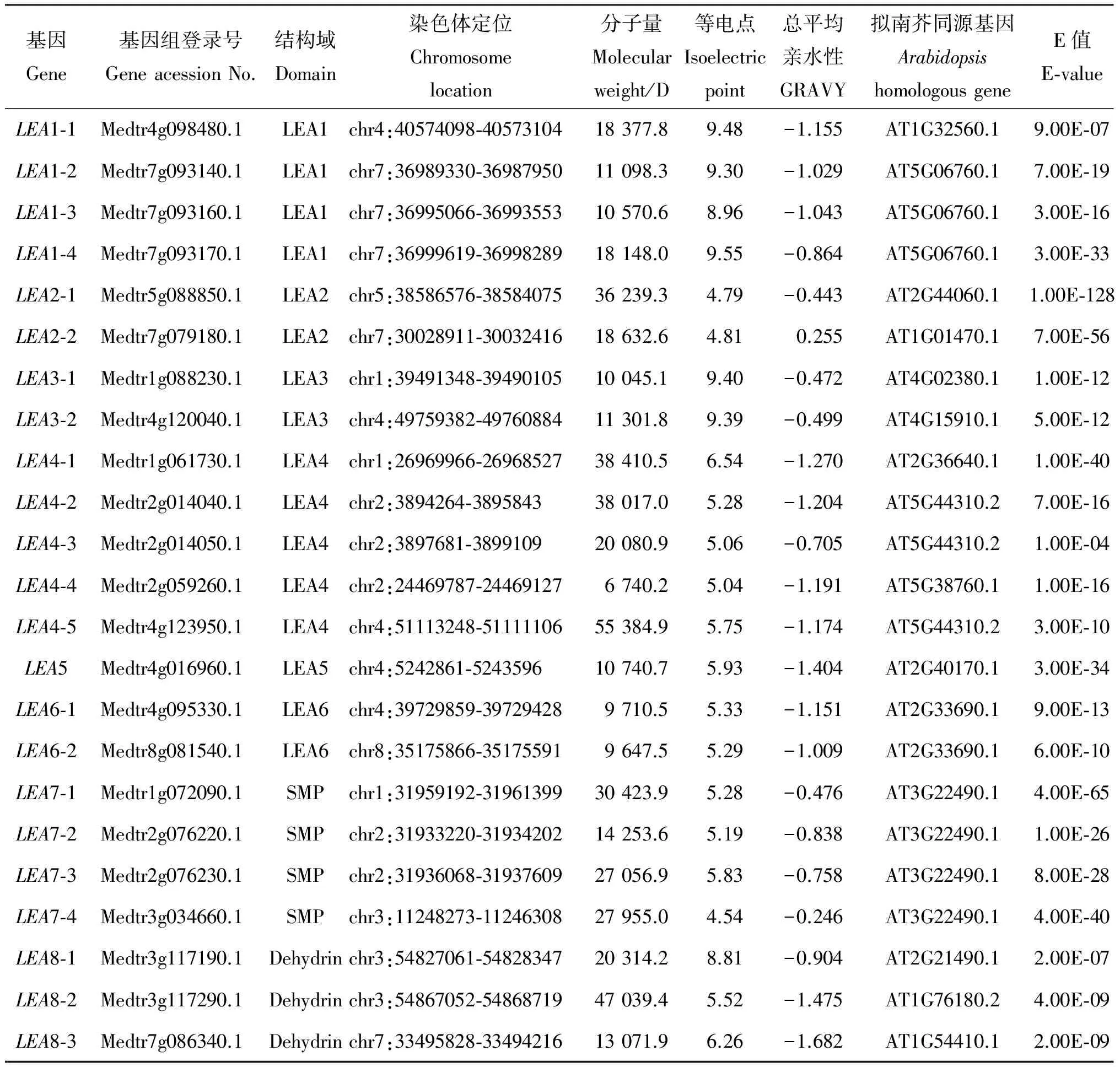
表1 蒺藜苜蓿LEA基因家族信息
2.5 蒺藜苜蓿LEA基因表达模式分析
利用MeV软件对蒺藜苜蓿LEA家族基因表达模式进行分析,绿-黑-红3个颜色表示基因的表达强度。绿色越强代表表达量越小,红色越强代表表达量越大,黑色为中间值。本研究对蒺藜苜蓿LEA基因在不同组织(叶、叶柄、茎、花、果荚、种衣和根)、不同时期及不同水分胁迫处理的31个表达芯片数据进行了分析。
研究结果显示,除LEA4-3仅在成熟种子中表达外,其他LEA基因在种子和种衣中都有较高的表达量,并具有随着种子成熟度的增加表达量呈增高的趋势(图4)。其中,LEA1-2、LEA2-1、LEA2-2、LEA3-1、LEA4-4、LEA7-4、LEA8-2和LEA8-3在叶、叶柄、茎、花、果荚、种衣、根和种子中组成型表达,但LEA1-2在种衣和根,LEA3-1在果荚以及LEA7-4在根中表达量相对较低。而其他15个LEA基因仅在种衣和种子中特异表达。
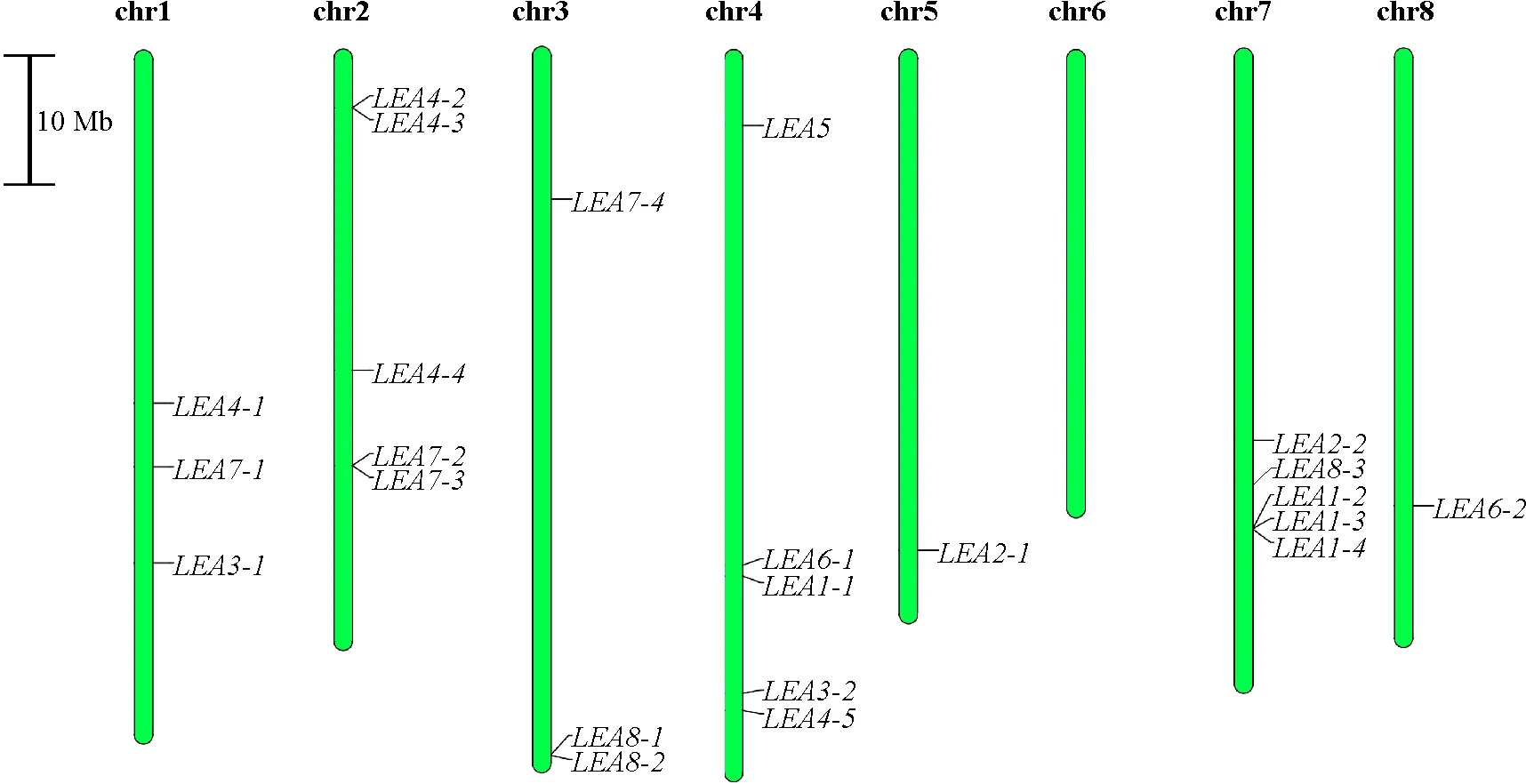
图1 蒺藜苜蓿LEA基因染色体定位Fig.1 The chromosome location of the LEA genes in Medicago truncatula
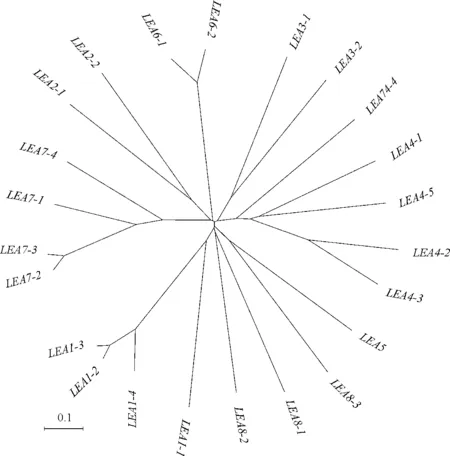
图2 蒺藜苜蓿LEA基因系统进化树Fig.2 The unrooted dendrogram of all Medicago truncatula LEA genes
对水分胁迫处理的幼苗和根进行基因表达芯片数据分析的结果显示,LEA5、LEA7-1、LEA7-2、LEA7-3、LEA1-1、LEA3-1、LEA2-1、LEA2-2、LEA8-2、LEA8-3、LEA4-3和LEA4-4表达量均无明显变化,LEA4-2、LEA1-3、LEA4-5、LEA7-4和LEA8-1基因在幼苗和根中的表达量随着干旱胁迫的加强表达量升高,恢复灌水后表达量急剧下降,LEA1-1、LEA1-4、LEA1-2、LEA4-1、LEA3-2、LEA6-1和LEA6-2仅在根中的表达受水分胁迫的调控,恢复灌水后呈明显的下降趋势;而LEA3-1在幼苗和根中随着干旱胁迫处理和恢复灌水后,基因表达量表现为先下降后升高的趋势。综合分析蒺藜苜蓿LEA基因在不同组织和不同胁迫处理后的表达量表明,LEA5、LEA7-1、LEA7-2和LEA7-3在根和幼苗中表达量极低,却不受干旱胁迫的调控,部分基因,如LEA2-1、LEA4-4、LEA2-2、LEA8-2和LEA8-3表达量无明显变化;而部分基因表达量变化较大,例如在幼苗期干旱处理和根的形态建成过程中水分胁迫时,LEA3-2、LEA1-4、LEA6-1、LEA6-2、LEA4-2、LEA1-3、LEA4-5、LEA7-4和LEA8-1的表达量均增加,但LEA4-1和LEA1-1只在根形态建成过程中受水分胁迫时表达量增加而在幼苗期干旱处理中表达量减低;此外,LEA4-3在幼苗期干旱处理下特异表达,表明这些基因可能在植株幼苗期对干旱生境的抗逆响应具有重要作用。
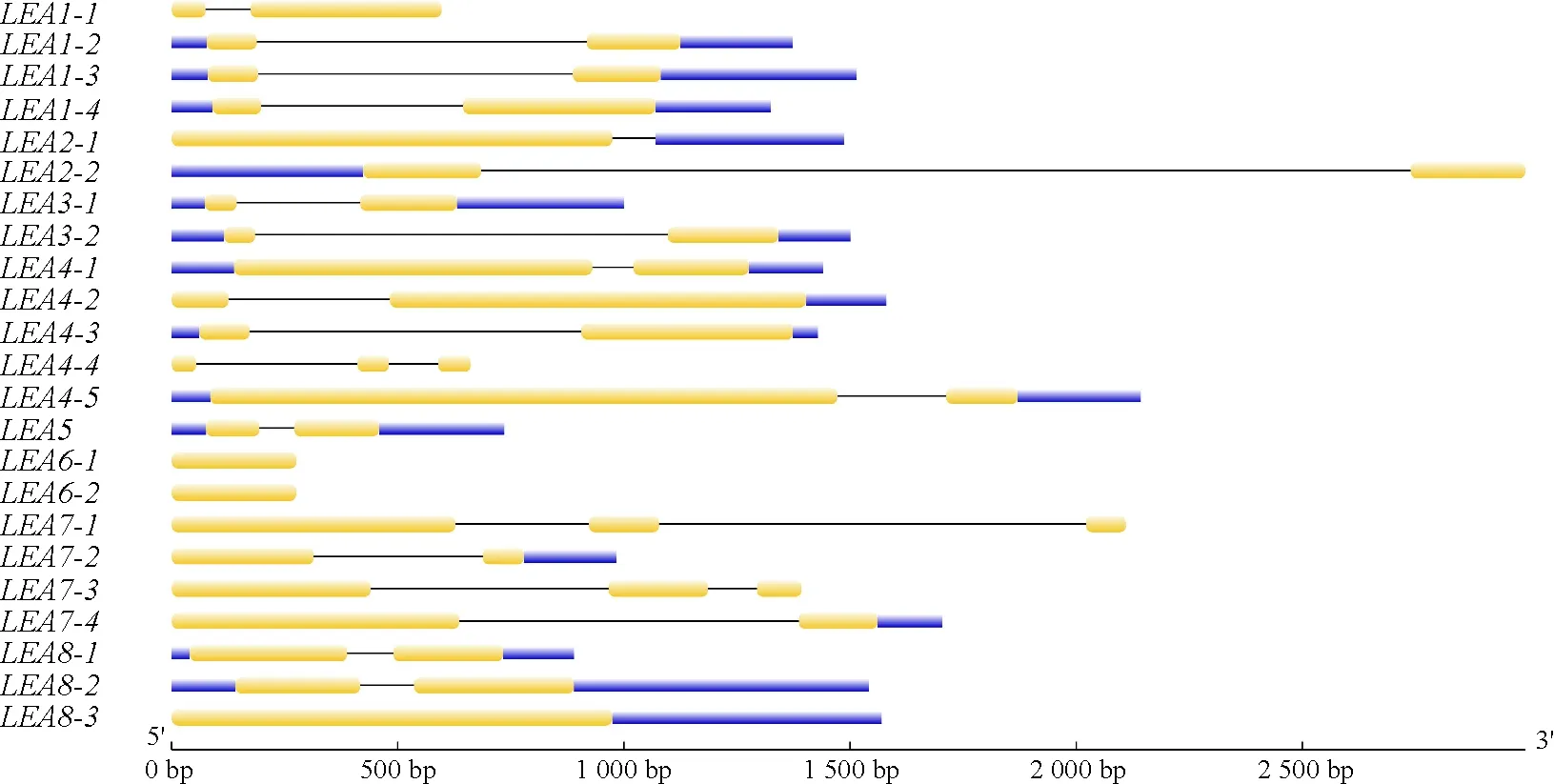
图3 蒺藜苜蓿LEA家族基因结构Fig.3 The gene structure of LEA gene family in Medicago truncatula
注:黄色框、黑色线条、蓝色框分别表示外显子、内含子和非编码区。
Note:The yellow boxes, black lines and blue boxes represent the exon, intron and untranslated region, respectively.
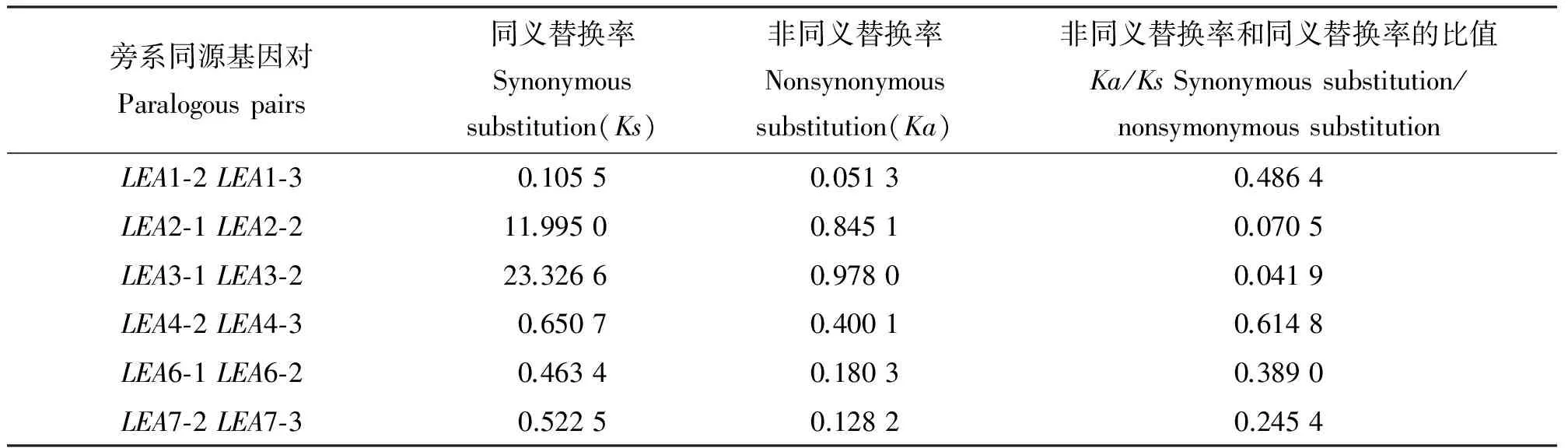
表2 蒺藜苜蓿LEA基因核苷酸替换率
3 讨论与结论
随着植物基因组研究的深入,利用比较基因组学方法分析和预测基因功能已成为目前植物基因功能研究的热点之一。目前,在高等植物中已有的相关研究包括番茄(Lycopersiconesculentum)的LBD基因家族[46]、蒺藜苜蓿WRKY功能基因[47]以及大豆[14]、拟南芥[12]LEA蛋白家族分析等,对其种类、数目和进化特点的研究为各基因家族功能分析奠定了理论基础。LEA蛋白是植物体内广泛存在的一系列蛋白质,在植物抵御干旱、冻害等非生物胁迫方面发挥着重要作用,但蒺藜苜蓿LEA基因家族及其功能分析的研究尚未见报道。本研究通过生物信息学方法分析LEA家族基因,共鉴定出8个亚家族23个蒺藜苜蓿LEA蛋白家族基因。在拟南芥中,已鉴定出9个亚家族51个基因[12],而水稻LEA蛋白家族基因则被分为7个亚家族34个基因[13]。在大豆中,已鉴定的LEA基因分为8个亚家族35个基因[14],与蒺藜苜蓿中的基因分类数目一致。另外,LEA基因结构分析表明,多数蒺藜苜蓿LEA基因只含有一个或不含有内含子,只有3个LEA基因含两个内含子。大豆LEA基因中也只含有较少的内含子,其中,只有两个基因含有多个内含子,其他都只含有一个或者无内含子[14],表明LEA基因家族的基因结构相对较简单。
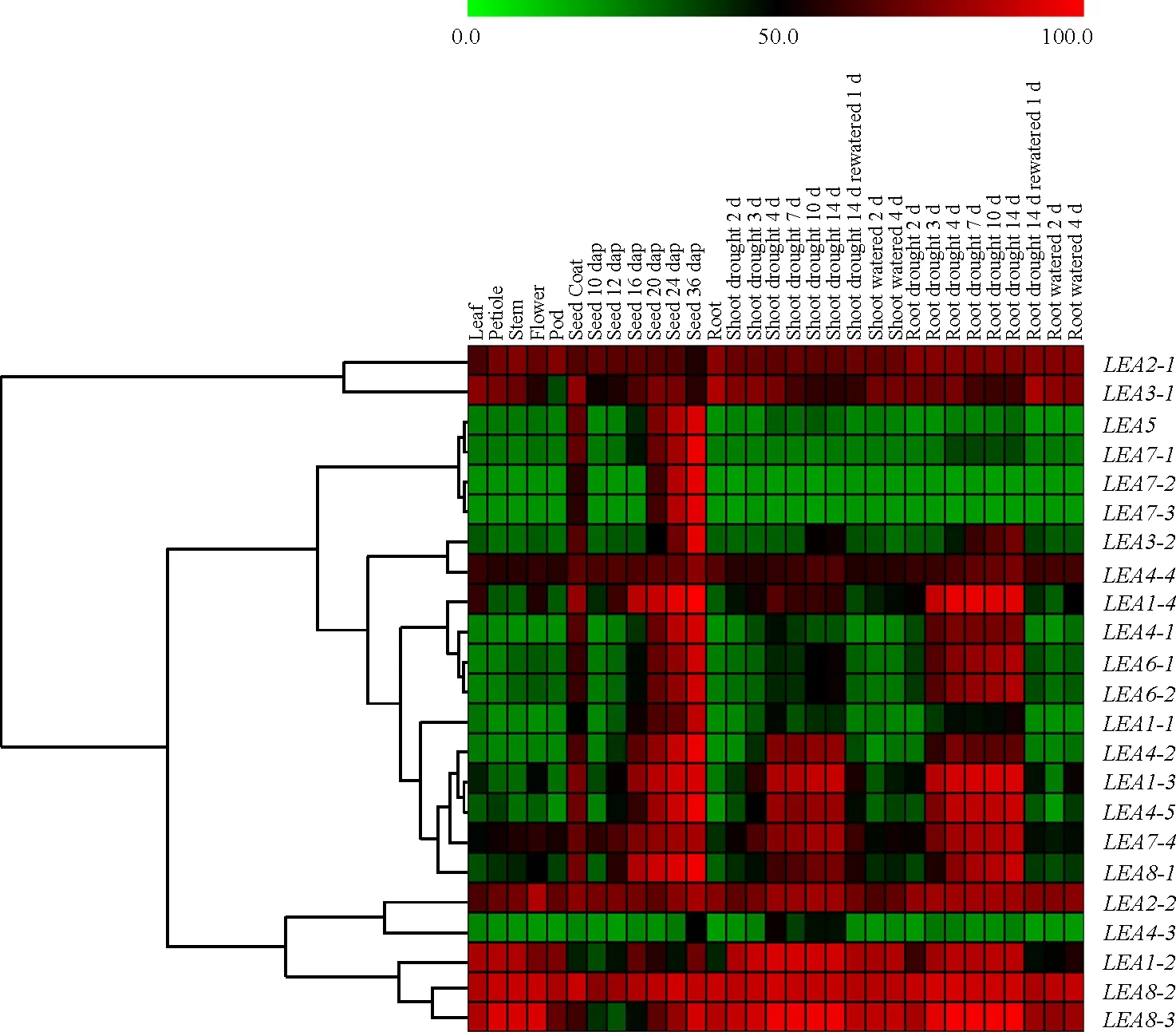
图4 蒺藜苜蓿LEA基因表达模式Fig.4 The expression profiles of Medicago truncatula LEA genes
注:图中用绿-黑-红三色代表基因表达水平。绿色越亮表达越弱,红色越亮表达越强。
Note:The gene expression level is displayed with green-black-red scheme in the picture. The brighter the green, the weaker the expression; the brighter the red, the stronger the expression.
蒺藜苜蓿中LEA家族基因分布差异较大,其中LEA4基因数最多,含有5个;而LEA5基因数最少,仅有1个。基因定位数据显示,第6条染色体上无LEA基因,最多的第2、4和7条染色体上各含有5个LEA基因。染色体定位和系统进化分析表明,有6对旁系同源基因于染色体上紧密连锁在一起,可能是基因组串联复制的结果[48]。另外,通过系统进化树分析得出,LEA基因在进化上表现出差异性,暗示其基因功能的多样性[49-50]。
根据蒺藜苜蓿LEA基因表达信息的分析结果发现,该LEA家族基因在组织表达上具有一定的特异性,且与耐旱性密切相关。15个LEA基因在种子发育时期特异表达,在种皮中也有大量表达,说明LEA基因家族对植物的种子发育具有较大影响;另外5个基因(LEA2-1、LEA2-2、LEA4-4、LEA8-2和LEA8-3)则在蒺藜苜蓿所有的组织中都表达,而LEA1-3和LEA1-4在叶和花中的表达量相较之其他非种子的表达量较高,从而使得该家族的基因功能表显示出一定的多样性和复杂性。有研究表明,LEA基因受干旱胁迫调控表达。例如,水稻OsLEA19a基因在幼苗中不表达,当受到干旱和高盐处理后,其表达量快速增加,并在处理6 h后达到最高值[51]。与此对应,在干旱胁迫下,13个基因(LEA1-2、LEA1-3、LEA1-4、LEA3-1、LEA3-2、LEA4-1、LEA4-2、LEA4-3、LEA4-5、LEA6-1、LEA6-2、LEA7-4和LEA8-1)有表达上调和下调的变化,说明其可能在植物响应干旱胁迫中具有重要作用。
运用生物信息学手段,对蒺藜苜蓿LEA基因家族全基因组进行分析,共鉴定出23个家族基因。23个家族基因可分为8个亚家族,并分布在除第6条染色体外的其他7条染色体上,且其基因中内含子数目较少,结构简单。基因组织表达谱分析结果显示,LEA家族基因组织表达模式差异较大,并受干旱逆境胁迫调控。
[1] Dure L,Greenway S C,Galau G A.Developmental biochemistry of cottonseed embryogenesis and germination-changing messenger ribonucleic-acid populations as shown by in vitro and in vivo protein-synthesis[J].Biochemistry,1981,20(14):4162-4168.
[2] Galau G A,Hughes D W,Dure L.Abscisic-acid induction of cloned cotton late embryogenesis-abundant (LEA) messengerrnas[J].Plant Molecular Biology,1986,7(3):155-170.
[3] Campos F,Zamudio F,Covarrubias A A.Two different late embryogenesis abundant proteins fromArabidopsisthalianacontain specific domains that inhibitEscherichiacoligrowth[J].Biochemical and Biophysical Research Communications,2006,342(2):406-413.
[4] Velten J,Oliver M J.Tr288:A rehydrin with a dehydrin twist[J].Plant Molecular Biology,2001,45(6):713-722.
[5] Solomon A,Salomon R,Paperna I,Glazer I.Desiccation stress of entomopathogenic nematodes induces the accumulation of a novel heat-stable protein[J].Parasitology,2000,121(4):409-416.
[6] Tunnacliffe A,Lapinski J,Mcgee B.A putative LEA protein,but no trehalose,is present in anhydrobiotic bdelloid rotifers[J].Hydrobiologia,2005,546:315-321.
[7] Shinozaki K,Yamaguchi-Shinozaki K.Gene networks involved in drought stress response and tolerance[J].Journal of Experimental Botany,2007,58(2):221-227.
[8] Bray E A.Molecular responses to water-deficit[J].Plant Physiology,1993,103(4):1035-1040.
[9] Wise M J.LEAping to conclusions:A computational reanalysis of late embryogenesis abundant proteins and their possible roles[J].BMC Bioinformatics,2003,4:52.
[10] Dure L,Crouch M,Harada J,Ho T,Mundy J,Quatrano R,Thomas T,Sung Z R.Common amino-acid sequence domains among the lea proteins of higher-plants[J].Plant Molecular Biology,1989,12(5):475-486.
[11] Dure L.A repeating 11-mer amino-acid motif and plant desiccation[J].Plant Journal,1993,3(3):363-369.
[12] Hundertmark M,Hincha D K.LEA (Late Embryogenesis Abundant) proteins and their encoding genes inArabidopsisthaliana[J].BMC Genomics,2008,9(1):118.
[13] Wang X,Zhu H,Jin G,Liu H,Wu W,Zhu J.Genome-scale identification and analysis ofLEAgenes in rice (OryzasativaL.)[J].Plant Science,2007,172(2):414-420.
[14] 李乐,许红亮,杨兴露,李雅轩,胡英考.大豆LEA基因家族全基因组鉴定、分类和表达[J].中国农业科学,2011,44(19):3945-3954.
[15] Lan T,Gao J,Zeng Q.Genome-wide analysis of the LEA (late embryogenesis abundant) protein gene family inPopulustrichocarpa[J].Tree Genetics & Genomes,2013,9(1):253-264.
[16] Steponkus P L,Uemura M,Joseph R A,Gilmour S J,Thomashow M F.Mode of action of theCOR15agene on the freezing tolerance ofArabidopsisthaliana[J].Proceedings of the National Academy of Sciences of the United States of America,1998,95(24):14570-14575.
[17] Grelet J,Benamar A,Teyssier E,Avelange-Macherel M H,Grunwald D,Macherel D.Identification in pea seed mitochondria of a late-embryogenesis abundant protein able to protect enzymes from drying[J].Plant Physiology,2005,137(1):157-167.
[18] Skriver K,Mundy J.Gene expression in response to abscisic acid and osmotic stress[J].Plant Cell,1990,2(6):503-512.
[19] Close T J.Dehydrins:A commonality in the response of plants to dehydration and low temperature[J].Physiologia Plantarum,1997,100(2):291-296.
[20] 王康,朱慧森,董宽虎.植物LEA蛋白及其基因家族成员PM16研究进展[J].草业科学,2008,25(2):97-102.
[21] 鲁松.青杨(PopuluscathayanaRehd.)对逆境胁迫的反应及lea基因的表达分析[D].成都:中国科学院研究生院,2009.
[22] Bies-Etheve N,Gaubier-Comella P,Debures A,Lasserre E,Jobet E,Raynal M,Cooke R,Delsenyet M.Inventory,evolution and expression profiling diversity of the LEA (late embryogenesis abundant) protein gene family inArabidopsisthaliana[J].Plant Molecular Biology,2008,67(1-2):107-124.
[23] Olvera-Carrillo Y,Campos F,Reyes J L,Garciarrubio A,Covarrubias A A.Functional analysis of the group 4 late embryogenesis abundant proteins reveals their relevance in the adaptive response during water deficit inArabidopsis[J].Plant Physiology,2010,154(1):373-390.
[24] Hundertmark M,Buitink J,Leprince O,Hincha D K.The reduction of seed-specific dehydrins reduces seed longevity inArabidopsisthaliana[J].Seed Science Research,2011,21(3):165-173.
[25] Yu J N,Zhang J S,Shan L,Chen S Y.Two new group 3LEAgenes of wheat and their functional analysis in yeast[J].Journal of Integrative Plant Biology,2005,47(11):1372-1381.
[26] Wang L,Li X,Chen S,Liu G.Enhanced drought tolerance in transgenicLeymuschinensisplants with constitutively expressed wheatTaLEA3[J].Biotechnology Letters,2009,31(2):313-319.
[27] Cheng Z Q,Targolli J,Huang X Q,Wu R.WheatLEAgenes,PMA80 andPMA1959,enhance dehydration tolerance of transgenic rice (OryzasativaL.)[J].Molecular Breeding,2002,10(1-2):71-82.
[28] Houde M,Dallaire S,N'Dong D,Sarhan F.Overexpression of the acidic dehydrinWCOR410 improves freezing tolerance in transgenic strawberry leaves[J].Plant Biotechnology Journal,2004,2(5):381-387.
[29] Sivamani E,Bahieldin A,Wraith J M,Al-Niemi T,Dyer W E,Ho T,Qu R D.Improved biomass productivity and water use efficiency under water deficit conditions in transgenic wheat constitutively expressing the barleyHVA1 gene[J].Plant Science,2000,155(1):1-9.
[30] Xu D P,Duan X L,Wang B Y,Hong B M,Ho T,Wu R.Expression of a late embryogenesis abundant protein gene,HVA1,from barley confers tolerance to water deficit and salt stress in transgenic rice[J].Plant Physiology,1996,110(1):249-257.
[31] 魏臻武,盖钧镒.豆科模式植物——蒺藜苜蓿[J].草业学报,2008,17(1):114-120.
[32] 屠德鹏,魏臻武,武自念,雷艳芳,张栋,邱伟伟.蒺藜苜蓿EST-SSRs分布特征及标记的开发[J].草业科学,2011,28(5):746-752.
[33] Goodstein D M,Shu S,Howson R,Neupane R,Hayes R D,Fazo J,Mitros T,Dirks W,Hellsten U,Putnam N,Rokhsar D S.Phytozome:A comparative platform for green plant genomics[J].Nucleic Acids Research,2012,40(D1):D1178-D1186.
[34] Liu G,Loraine A E,Shigeta R,Cline M,Cheng J,Valmeekam V,Sun S,Kulp D,Siani-Rose M A.NetAffx:Affymetrix probesets and annotations[J].Nucleic Acids Research,2003,31(1):82-86.
[35] He J,Benedito V A,Wang M,Murray J D,Zhao P X,Tang Y,Udvardi M K.TheMedicagotruncatulagene expression atlas web server[J].BMC Bioinformatics,2009,10:441.
[36] Benedito V A,Torres-Jerez I,Murray J D,Andriankaja A,Allen S,Kakar K,Wandrey M,Verdier J,Zuber H,Ott T,Moreau S,Niebel A,Frickey T,Weiller G,He J,Dai X,Zhao P X,Tang Y,Udvardi M K.A gene expression atlas of the model legumeMedicagotruncatula[J].The Plant Journal,2008,55(3):504-513.
[37] Finn R D,Mistry J,Schuster-Bockler B,Griffiths-Jones S,Hollich V,Lassmann T,Moxon S,Marshall M,Khanna A,Durbin R,Eddy S R,Sonnhammer E L,Bateman A.Pfam:Clans,web tools and services[J].Nucleic Acids Research,2006,34:D247-D251.
[38] Artimo P,Jonnalagedda M,Arnold K,Baratin D,Csardi G,de Castro E,Duvaud S,Flegel V,Fortier A,Gasteiger E,Grosdidier A,Hernandez C,Ioannidis V,Kuznetsov D,Liechti R,Moretti S,Mostaguir K,Redaschi N,Rossier G,Xenarios I,Stockinger H.ExPASy:SIB bioinformatics resource portal[J].Nucleic Acids Research,2012,40:W597-W603.
[39] Voorrips R E.MapChart:Software for the graphical presentation of linkage maps and QTLs[J].The Journal of Heredity,2002,93(1):77-78.
[40] Tamura K,Stecher G,Peterson D,Filipski A,Kumar S.MEGA6:Molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30(12):2725-2729.
[41] 郭安源,朱其慧,陈新,罗静初.GSDS:基因结构显示系统[J].遗传,2007,29(8):1023-1026.
[42] Sievers F,Wilm A,Dineen D,Gibson T J,Karplus K,Li W,Lopez R,Mcwilliam H,Remmert M,Soding J,Thompson J D,Higgins D G.Fast,scalable generation of high-quality protein multiple sequence alignments using Clustal Omega[J].Molecular Systems Biology,2011,7:539.
[43] Suyama M,Torrents D,Bork P.PAL2NAL:Robust conversion of protein sequence alignments into the corresponding codon alignments[J].Nucleic Acids Research,2006,34:W609-W612.
[44] Saeed A I,Bhagabati N K,Braisted J C,Liang W,Sharov V,Howe E A,Li J,Thiagarajan M,White J A,Quackenbush J.TM4 microarray software suite[J].Methods in Enzymology,2006,411:134-193.
[45] Yang Z,Bielawski J P.Statistical methods for detecting molecular adaptation[J].Trends in Ecology & Evolution,2000,15(12):496-503.
[46] 王小非,刘鑫,苏玲,孙永江,张世忠,郝玉金,由春香.番茄LBD基因家族的全基因组序列鉴定及其进化和表达分析[J].中国农业科学,2013,46(12):2501-2513.
[47] 江腾,林勇祥,刘雪,江海洋,朱苏文.苜蓿全基因组WRKY转录因子基因的分析[J].草业学报,2011,20(3):211-218.
[48] Nei M.Gene duplication and nucleotide substitution in evolution[J].Nature,1969,221:5175-5177.
[49] 周丽,姬向楠,何非,潘秋红,段长青.植物胚胎发育晚期丰富蛋白的结构与功能[J].热带生物学报,2012,3(2):191-196.
[50] 陈雷,李磊,李金花,李永春.植物LEA蛋白及其功能[J].中国农学通报,2009(24):143-146.
[51] 胡廷章,吴应梅,陈再刚,黄小云.水稻OsLEA19a基因的克隆、表达及生物信息学分析[J].华北农学报,2011,26(6):73-78.
(责任编辑 武艳培)
Genome-wide analysis ofLEAgene family inMedicagotruncatula
LIU Zhi-min, LIU Wen-xian, JIA Xi-tao, ZHANG Zheng-she, WANG Yan-rong
(State Key Laboratory of Grassland Agro-ecosystems, College of Pastoral Agriculture Science and Technology, Lanzhou University, Lanzhou 730020, China)
Late embryogenesis abundant (LEA) family proteins are expressed abundantly at the late stage of embryonic development of plant seeds, and play important roles in responding to various environmental stress, such as drought, low temperature and high salinity. At present, the phylogenetic studies ofLEAgene family inMedicagotruncatulahave not been reported yet. In this study, with the application of bioinformatics methods, theLEAfamily genes ofM.truncatulawere identified through the whole genome, and the system evolution, gene structure, evolutionary pressure, chromosomal location and gene expression patterns were further analyzed. A total of 23LEAgenes were systematically identified fromM.truncatulaand classified into 8 subfamilies. Gene location results showed that these 23LEAgenes were distributed unevenly on 7 chromosomes; The exon numbers of all the genes were no more than two, indicating a simple gene structure of this gene family. The expression profiles ofM.truncatulaLEAgenes showed a characteristic of temporal and tissue specific, and regulated by drought stress. This study provides a theoretical foundation for verifying the function ofLEAgenes inM.truncatula.
Medicagotruncatula;LEAgene family; system evolution; expression analysis
LIU Wen-xian E-mail: liuwx@lzu.edu.cn
10.11829j.issn.1001-0629.2014-0418
2014-09-16 接受日期:2014-12-11
国家草品种区域试验项目——苜蓿品种“三性”测试(农财发[2013]49号);长江学者和创新团队发展计划(IRT13019);兰州大学中央高校基础研究基金项目(lzujbky-2013-82)
刘志敏(1990-),男,江苏赣榆人,在读硕士生,主要从事草类种质资源与育种研究。E-mail:liuzhm2013@lzu.edu.cn
刘文献(1981-),男,河南开封人,副教授,硕导,博士,主要从事草类种质资源和分子生物学研究。E-mail:liuwx@lzu.edu.cn
S551+.703;Q943.2
A
1001-0629(2015)03-0382-10
刘志敏,刘文献,贾喜涛,张正社,王彦荣.蒺藜苜蓿LEA基因家族全基因组分析[J].草业科学,2015,32(3):382-391.
LIU Zhi-min,LIU Wen-xian,JIA Xi-tao,ZHANG Zheng-she,WANG Yan-rong.Genome-wide analysis ofLEAgene family inMedicagotruncatula[J].Pratacultural Science,2015,32(3):382-391.

