42份紫花苜蓿种质资源遗传多样性的SSR分析
陈立强,师尚礼
(1.塔里木大学动物科学学院,塔里木畜牧科技兵团重点实验室,新疆 阿拉尔 843300; 2.甘肃农业大学草业学院,甘肃 兰州 730070)
植物生产层
42份紫花苜蓿种质资源遗传多样性的SSR分析
陈立强1,师尚礼2
(1.塔里木大学动物科学学院,塔里木畜牧科技兵团重点实验室,新疆 阿拉尔 843300; 2.甘肃农业大学草业学院,甘肃 兰州 730070)
为了补充现有苜蓿(Medicagosativa)种质资源的遗传多样性信息,采用SSR分子标记技术对1份野生紫花苜蓿种质和41份栽培紫花苜蓿品种的遗传多样性进行了研究。15对引物在供试苜蓿材料中共获得231条扩增带,其中163条具有多态性。引物的多态位点百分率、Nei’s基因多样性指数和Shannon信息指数的平均值分别为71.55%、0.210 0和0.326 3。供试材料间的相似系数介于0.641~0.913,平均为0.791,栽培品种间的平均相似系数相对较大,野生种质资源与栽培品种间的平均相似系数相对较小。聚类分析表明,供试材料在相似系数0.778处可聚为五大类,单独聚为类的陇东野生紫花苜蓿、CW 200和CW 787都表现出了与其他材料间较远的亲缘关系。基于主成分分析图,可把供试材料分为四大类,其中第Ⅰ、Ⅱ类与第Ⅲ、Ⅳ类材料间的亲缘关系较远。综上,供试苜蓿种质资源具有较丰富的遗传多样性,部分种质资源表现出了相对独立的遗传特性。
苜蓿;遗传多样性;亲缘关系;SSR
紫花苜蓿(Medicagosativa)产量高、品质好、适应性强,是我国栽培历史最久、分布最广的豆科牧草,长期以来在生态治理、畜牧业发展和种植业结构调整中发挥着重要作用[1-2]。近年来,随着奶牛养殖业的发展,苜蓿在奶业中的作用也日益突出,种植面积不断扩大。然而,现阶段我国苜蓿优良品种数量少,国产苜蓿无论在数量上还是品质上,均无法满足生产的需求,导致国外品种在我国苜蓿生产中占具主要地位[3]。另外,由于受到栽培区生态条件的影响,引进品种的生产性能都会受到不同程度的抑制,潜在的产量和品质特性不能完全得到开发。因此,利用现有种质资源,培育适合栽培区种植的新品种将成为苜蓿产业高效发展的基础和保障,也将成为我国苜蓿产业走出困境,摆脱危机的必由之路。
遗传多样性是植物育种和遗传改良的基础。目前已有大量研究从分子水平对紫花苜蓿种质资源及其与近缘植物的遗传多样性进行了研究,涉及的材料包括地方品种、育成品种、引进品种[4-5],野生、逸生种质资源[6-7],抗旱种质资源[8],抗病种质资源[9]、抗虫种质资源[10],耐盐种质资源[11],不同秋眠型种质资源[12],杂交子代及亲本[13],紫花苜蓿和黄花苜蓿(M.falcata)[14],紫花苜蓿、黄花苜蓿、多变苜蓿(M.varia)[15],紫花苜蓿、黄花苜蓿、葫芦巴属(Trigonella)植物[16],紫花苜蓿、黄花苜蓿、蒺藜苜蓿(M.truncatula)、天蓝苜蓿(M.lupulina)、蜗牛苜蓿(M.scutellata)、海滨苜蓿(M.littoralis)、刺球苜蓿(M.sphaerocarpos)、南苜蓿(M.polymorpha)[17],紫花苜蓿、胡卢巴(T.foenum-graecum)、网脉胡卢巴(T.cancellata)、黄花苜蓿、多变苜蓿、天蓝苜蓿、花苜蓿(M.ruthenica)、毛荚苜蓿(M.edgeworthii)、阔荚苜蓿(M.platycarpos)、南苜蓿、青海苜蓿(M.archiducis-nicolai)[18]等,其结果为苜蓿种质资源研究、保护和利用提供了依据。但紫花苜蓿种质资源种类多、数量大、分布广,各研究所涉及的材料也都非常有限且不尽相同,特别是随着新资源数量的增加,遗传多样性研究需要不断的完善和补充,以满足苜蓿育种领域的需求。
DNA分子标记技术是近年来发展起来且被广泛应用的遗传多样性研究手段,主要包括RAPD、AFLP、SNPs、ISSR、SSR、SCAR、CAPS、RAMP、SRAP、TRAP、SSCP、REMAP、IRAP、IMP、RFLP等[19],其中SSR标记具有多态性高、重复性好、方法简便等优势[20],是植物品种间遗传多样性研究的理想分子标记,目前已被广泛应用于苜蓿遗传多样性的研究领域[21-24]。因此,本研究选用SSR标记对1份野生紫花苜蓿种质资源和41份栽培紫花苜蓿品种的遗传多样性进行检测,以期为苜蓿品种的遗传改良以及杂交亲本的选配提供依据。
1 材料和方法
1.1 试验材料
试验材料包括野生紫花苜蓿种质资源1份,栽培紫花苜蓿品种41份(表1)。陇东野生紫花苜蓿采自甘肃清水县灌丛草原地带,根系发达,具水平或斜生的根茎,无主根;茎纤细且平卧或半平卧生长,分枝能力强;叶片小且数量少;花序短且紧簇,小花较少;具有抗寒、抗旱、耐牧的特性[25]。Pick 3006、Pick 8925、CW 787、CW 200由中国农业科学院兰州畜牧与兽药研究所提供,其余品种由甘肃农业大学草业学院提供。
1.2 DNA的提取
每品种随机选取100粒种子,置于铺有两层滤纸的培养皿中,在培养箱内发芽(光照9 h,黑暗15 h,温度27 ℃),至第8 天,随机剪取40个单株苗的嫩叶,混合后用CTAB法提取DNA[26]。DNA提取后加入200 μL TE缓冲液进行稀释,并用琼脂糖凝胶电泳技术对其质量进行检测。合格样品置于-20 ℃冰箱保存备用。
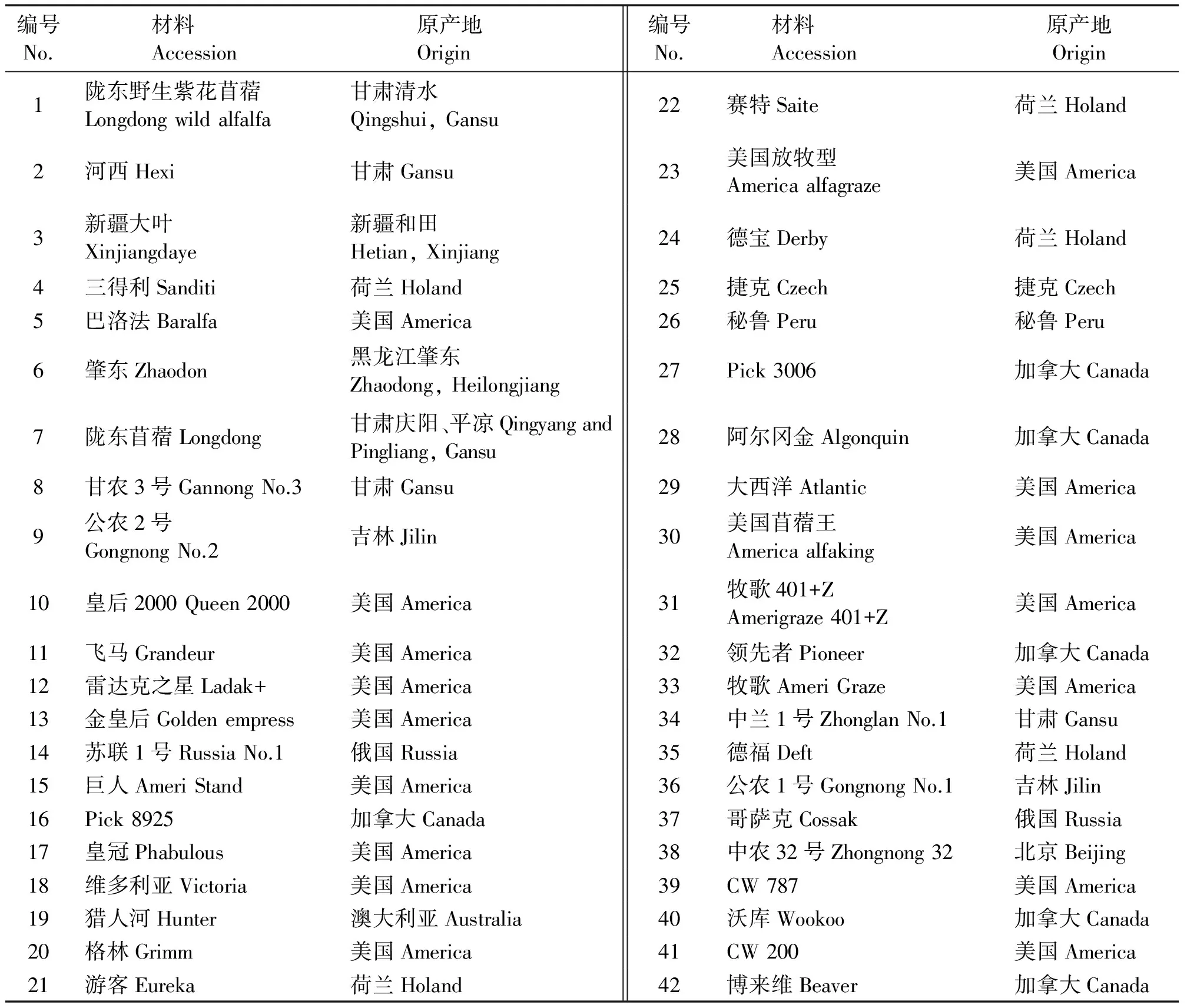
表1 供试紫花苜蓿材料及原产地
1.3 PCR扩增及电泳
结合条带的清晰度、多态性和稳定性,在前人研究[27-29]的基础上挑选出了15对SSR引物(表2),由上海生工生物工程技术服务有限公司合成。PCR反应体系总体积为10 μL,含3 μL 模板DNA,3 μL引物,0.24 μL dNTP(0.20 mmol·L-1),1.5 μL 10× Buffer,0.9 μL Mg2+(2.0 mmol·L-1),0.15 μL TaqDNA聚合酶(1 U),ddH2O补足。扩增程序:94 ℃预变性3 min;94 ℃变性30 s,52 ℃退火1.5 min,72 ℃延伸1.5 min,30个循环;最后72 ℃保温8 min,4 ℃保存[17]。扩增产物在30%的聚丙烯酰胺凝胶上进行电泳分离,胶板厚1.5 mm,上样量10 μL,以6×DNA loading buffer(溴酚蓝,二甲苯青)为前沿指示剂,1× TBE为电极缓冲液,恒压电泳,电压100 V,约5 h,指示剂移至距玻璃板末端2 cm时停止电泳,用硝酸银染色显影后进行凝胶成像。
1.4 数据统计与分析
以扩增条带在相对迁移位置的有无,记数为“1”或“0”,建立原始数据矩阵。计算各引物的多态位点百分率(Percentage of Polymorphic Bands,PPB)。用POPGENE VERSION 1.31 软件统计分析Nei’s多样性指数和Shannon信息指数。用NTSYSpc 2.1软件计算供试材料间的相似系数(Genetic Similarity,GS);用SHAN程序,以非加权类平均法(Unweighted Pair Group Method with Arithmetic Mean,UPGMA)进行聚类分析,构建聚类图;基于相似系数进行主成分分析,构建二维分析图。

表2 SSR引物序列及名称
2 结果
2.1 SSR标记多态性分析
用SSR标记对供试苜蓿材料的遗传多样性进行研究,引物Afl4的扩增及电泳检测结果见图1。15对引物在42份苜蓿材料中共获得231条扩增带,其中163条具有多态性。每对引物扩增的总条带数介于12~22,平均15.40条,多态性条带数介于6~14,平均10.87条。多态位点百分率介于50.00%(w6019)~100.00%(AFca1),平均为71.55%(表3)。引物Nei’s基因多样性指数的变幅为0.125 7(Alf1)~0.383 1(AFca1),平均为0.210 0;Shannon信息指数的变幅为0.203 3(Alf1)~0.563 8(AFca1),平均为0.326 2(表3)。

图1 引物Afl4对42份苜蓿种质资源的扩增结果Fig.1 Amplification products of 42 alfalfa accessions by primer Afl4
注:图中材料编号与表1相同,表4、图2、图3同。M,Marker。
Note:Numbers of the accessions refer to Table 1,the same in Table 4,Fig.2 and Fig.3. M, marker.
2.2 相似系数
基于原始数据,用软件计算出供试材料间的相似系数矩阵(表4)。相似系数是用来比较群体或个体间相似程度的度量参数,材料间相似系数越小,则亲缘关系越远。结果表明,42份苜蓿种质资源861对材料间的相似系数值介于0.641~0.913,平均为0.791,其中有31对材料间的相似系数介于0.641~0.697,占3.60%,亲缘关系相对较远;有422对材料间的相似系数介于0.701~0.797,占49.01%;有404对材料间的相似系数介于0.801~0.896,占46.92%,有4对材料间的相似系数介于0.900~0.913,占0.46%,亲缘关系相对较近。甘农3号(8)和CW 200(41)间的相似系数最小(GS=0.641),亲缘关系最远;大西洋(29)和牧歌401+Z(31)间的相似系数最大(GS=0.913),亲缘关系最近。栽培品种间的相似系数介于0.641~0.913,平均为0.795。陇东野生紫花苜蓿(1)与栽培品种间的相似系数介于0.649~0.788,平均为0.711,表明野生紫花苜蓿与栽培品种间的亲缘关系相对较远;CW 200(41)与其他材料间的相似系数介于0.641~0.801,平均为0.720,相似系数较小,与其他材料间亲缘关系相对较远。

表3 SSR标记基于42份紫花苜蓿材料的遗传多样性参数
2.3 聚类分析
对42份苜蓿种质资源进行聚类分析,并构建聚类图(图2),供试材料在相似系数为0.778 处可聚为5类。第Ⅰ类由陇东野生紫花苜蓿组成。第Ⅱ类由8份材料组成,在相似系数为0.795处可聚为两个亚类,第一亚类由新疆大叶、哥萨克、三得利组成;第二亚类由公农2号、秘鲁、沃库、德宝、德福组成。第Ⅲ类由CW 200组成。第Ⅳ类由31份材料组成,在相似系数为0.812处可聚为4个亚类,第一亚类由河西组成;第二亚类由巴洛法、陇东苜蓿、公农1号、博来维、赛特、美国苜蓿王、雷达克之星、巨人、Pick 8925、维多利亚、猎人河、阿尔冈金、皇冠、肇东、大西洋、牧歌401+Z、中牧32号、金皇后、苏联1号、皇后2000、格林、游客、牧歌、中兰1号、飞马、Pick 3006、领先者组成;第三亚类由美国放牧型苜蓿组成;第四亚类由甘农3号和捷克组成。第Ⅴ类由CW 787组成。单独聚为类的陇东野生紫花苜蓿、CW 200和CW 787,以及单独聚为亚类的河西、美国放牧型苜蓿都表现出了与其他材料间较远的亲缘关系。
2.4 主成分分析
用NTSYSpc 2.1软件对供试材料进行主成分分析,并根据第1和第2主成分构建主成分分析二维图(图3),图中材料间位置相互靠近者表示亲缘关系较近,远离者表示亲缘关系较远。根据材料在图中的位置分布特征,可明显地把供试材料划分为四大类,第Ⅰ类包括陇东野生紫花苜蓿和CW 200;第Ⅱ类包括哥萨克、德福、新疆大叶、公农2号、三得利、德宝、沃库和秘鲁;第Ⅲ类包括CW 787、捷克和甘农3号;第Ⅳ类包括雷达克之星、河西、美国放牧型、猎人河、Pick 8925、公农1号、阿尔冈金、美国苜蓿王、赛特、陇东苜蓿、巴洛法、皇冠、巨人、博来维、中兰1号、游客、皇后2000、格林、维多利亚、牧歌、中农32号、肇东、金皇后、飞马、领先者、Pick 3006、牧歌401+Z、大西洋、苏联1号。第Ⅰ类和第Ⅱ类之间、第Ⅲ类和第Ⅳ类之间在图中的位置相对较近,亲缘关系相对较近,但第Ⅰ、Ⅱ类与第Ⅲ、Ⅳ类在图中的位置相对较远,亲缘关系相对较远。主成分分析从不同角度更直观地展示了种质资源间的亲缘关系,其结果与聚类分析基本一致。
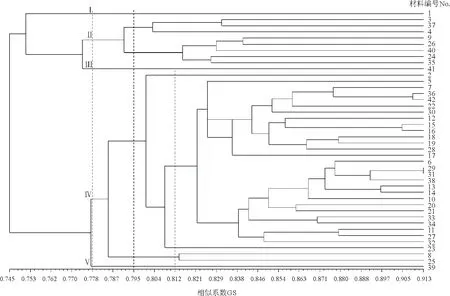
图2 42份苜蓿材料基于15对SSR引物的UPGMA聚类图Fig.2 UPGMA dendrogram of 42 alfalfa accessions based on 15 SSR primer pairs
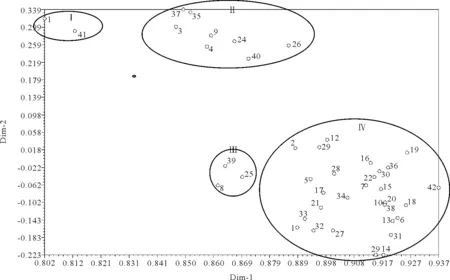
图3 42份苜蓿材料基于15对SSR引物的主成分分析图Fig.3 Principal component analysis of 42 alfalfa accessions based on 15 SSR primer pairs
3 讨论与结论
3.1 SSR标记的检测效率及材料的遗传多样性分析
本研究用15对SSR引物在42份紫花苜蓿材料中检测到了163条多态性扩增带,每对引物平均10.87条,多态位点百分率平均为71.55%;陈斐等[30]用16对SSR引物在82份苜蓿材料中检测到了190条多态性扩增带,每对引物平均11.88条,多态位点百分率平均为83.05%;李景欣等[31]用8对SSR引物在6个苜蓿品种(品系)中检测到了106条多态性扩增带,每对引物平均13.25条,多态位点百分率为82.81%;王森山等[10]用6对SSR引物在9个抗蚜苜蓿品种(系)中检测到了63条多态性扩增带,每对引物平均10.50条,多态位点百分率为91.30%。由于试验所选材料和引物不同,所得结果也不尽相同,但引物的多态性条带平均值及其百分率均相对较高,远高于Talebi等[32]用14对SRAP引物对4个伊朗紫花苜蓿种群的研究结果、俞金蓉[33]用10条ISSR引物对30个苜蓿品种的研究结果及康俊梅等[8]用48条RAPD引物对9个苜蓿品种的研究结果,表明SSR标记能有效检测苜蓿材料的遗传多样性。基因型不同的品种,基因组内核苷酸序列也存在差异,当用相同的SSR引物对不同基因组进行体外扩增时,由于基因组上与引物互补DNA片段的数目、位点不同,扩增产物的大小,数目也不同,因此,扩增产物的多态性,能反映材料的遗传多样性[20],以此为依据,现有苜蓿种质资源具有较丰富的遗传多样性。与多态位点百分率相比,基于Hardy-Weinberg假设的Nei’s基因多样性指数和基于条带表型频率的Shannon信息指数更能客观地衡量材料的遗传多样性水平[34],其变幅分别为0.125 7~0.383 1、0.203 3~0.563 8,平均为0.210 0和0.326 2,且两指标大小的变化趋势基本一致,进一步表明供试苜蓿种质资源具有一定水平的遗传多样性。
3.2 供试材料间的亲缘关系分析
42份苜蓿种质资源861对材料中,有31对材料的相似系数介于0.641~0.697,与Tucak等[35]对欧洲、澳洲、南美及北美品种遗传多样性的研究结果相近,表明现有紫花苜蓿种质资源中,有部分材料间的亲缘关系相对较远,遗传背景丰富。聚类分析表明,单独聚为类的陇东野生紫花苜蓿、CW 200、CW 787及单独聚为亚类的河西、美国放牧型苜蓿都与其他材料间的亲缘关系相对较远,具有相对独立的遗传特性。在育种领域,亲本间的亲缘关系很大程度上决定着后代群体的选择范围,通常认为两亲本遗传差异越大,其杂种优势也越大,后代分离范围就越广泛,获得优良个体的机会也就越多[36-37]。以上结果将为苜蓿杂交亲本的选配提供科学依据。
供试种质资源中,有404对材料间的相似系数介于0.801~0.896,占46.92%,4对材料间的相似系数介于0.900~0.913,占0.46%,材料间的遗传相似程度较高,亲缘关系较近。李景欣等[31]用8对SSR引物分析苜蓿新品系Dy-2006等6个苜蓿品种(品系)的亲缘关系发现,材料间的遗传距离介于0.033~0.076;张颖娟和王斯琴花[38]用13条ISSR引物分析12份苜蓿材料的遗传多样性发现,材料间的平均遗传距离为0.063;Mandoulakani等[39]用10条IRAP和14条REMAP引物分析8个紫花苜蓿群体的遗传多样性发现,基于两种标记,群体间的遗传距离分别介于0.063~0.151和0.048~0.124,平均为0.110和0.089。表明现有苜蓿种质资源中,也有大部分材料间的亲缘关系相对较近,遗传基础狭窄。因此,在常规育种实践中,开发利用野生种质资源及遗传特性相对独立的材料具有重要意义。
3.3 野生紫花苜蓿种质资源的利用潜力分析
陇东野生紫花苜蓿与栽培品种间的相似系数介于0.649~0.788,平均为0.711,小于栽培品种间的平均相似系数(0.795),聚类分析、主成分分析结果也都表明,陇东野生紫花苜蓿与栽培品种间的亲缘关系较远,这与用同工酶研究陇东野生紫花苜蓿遗传特异性[40-43]时所得结论相同,进一步在分子水平上更准确地验证了陇东野生紫花苜蓿相对独立的遗传特性。在野生苜蓿种质资源研究领域,马向丽和毕玉芬[7]用ISSR标记对云南30份野生和逸生苜蓿资源进行了聚类分析,结果表明,两份野生紫花苜蓿种质资源单独聚为一类;关潇[44]用SRAP标记对42份野生紫花苜蓿种质资源的遗传多样性进行了研究,结果表明,各材料间的相似系数介于0.498~0.882,平均为0.686,相似系数较小。表明野生苜蓿种质资源之间、野生种质资源与栽培品种间的亲缘关系较远。野生种质资源是在某一地区自然条件的作用下,经过长期的自然选择形成的,通常携带着抗病、抗虫、抗旱、抗寒、耐盐、持久等优良基因[36]。野生苜蓿种质资源通常具有持久性、匍匐性、根茎性等特点[45],是新品种培育的宝贵基础材料,然而野生紫花苜蓿资源在世界范围内都是稀有的,因此,保护和利用现有野生苜蓿种质资源具有重要的意义。
[1] 杨青川.苜蓿生产与管理指南[M].北京:中国林业出版社,2003:1-7.
[2] 王雪,李志萍,孙建军,冯长松,李绍钰.中国苜蓿品种的选育与研究[J].草业科学,2014,31(3):512-518.
[3] 孙启忠,玉柱,马春晖,徐春城.我国苜蓿产业过去10年发展成就与未来10年发展重点[J].草业科学,2013,30(3):471-477.
[4] 杨晓莉,陈丽,班霆,韩鹏,王晓娟.甘肃省苜蓿种质资源遗传多样性RAPD分析[J].草地学报,2008,16(2):129-134.
[5] 强海平,余国辉,刘海泉,高洪文,刘贵波,赵海明,王赞.基于SSR标记的中美紫花苜蓿品种遗传多样性研究[J].中国农业科学,2014,47(14):2853-2862.
[6] Bagavathiannan M V,Julier B,Barre P,Gulden R H,Van Ackern R C.Genetic diversity of feral alfalfa (MedicagosativaL.) populations occurring in Manitoba,Canada and comparison with alfalfa cultivars:An analysis using SSR markers and phenotypic traits[J].Euphytica,2010,173:419-432.
[7] 马向丽,毕玉芬.云南野生和逸生苜蓿资源遗传多样性的ISSR分析[J].中国草地学报,2010,32(6):34-38.
[8] 康俊梅,杨青川,樊奋成.RAPD技术分析不同抗旱性苜蓿品种DNA的多态性[J].生物技术,2005,15(6):37-40.
[9] Obert D E,Skinner D Z,Stuteville D L.Association of AFLP markers with downy mildew resistance in autotetraploid alfalfa[J].Molecular Breeding,2000,6:287-294.
[10] 王森山,唐守嵘,朱亚灵,贺春贵.抗蚜苜蓿品种(系)SSR标记的遗传多样性分析[J].草业科学,2010,27(7):78-83.
[11] 姜健,杨宝灵,夏彤,于淑梅,乌云娜.紫花苜蓿耐盐种质资源的遗传多样性分析[J].草业学报,2011,20(5):119-125.
[12] 何庆元,王吴斌,杨红燕,向仕华,周丽英,王松华.利用SCoT标记分析不同秋眠型苜蓿的遗传多样性[J].草业学报,2012,21(2):133-140.
[14] 张宇,于林清,慈忠玲.利用SRAP标记研究紫花苜蓿和黄花苜蓿种质资源遗传多样性[J].中国草地学报,2012,34(1):72-76.
[15] 李飞飞,崔大方,羊海军,邓超宏,李庆艳.中国新疆紫花苜蓿复合体3个种的遗传多样性及亲缘关系研究[J].草业学报,2012,21(1):190-198.
[16] 刘磊,王宗礼,李志勇,周国栋,师文贵,李鸿雁,蔡丽艳.利用ISSR标记解析紫花苜蓿、黄花苜蓿和胡卢巴属植物的亲缘关系[J].安徽农业科学,2012,40(25):12393-12395.
[17] 杨占花,魏臻武,雷艳芳,张丽芳.两类SSR对苜蓿属种质遗传多样性和亲缘关系的比较研究[J].草地学报,2008,16(6):559-564.
[18] 李飞飞,羊海军,崔大方.利用SSR及ISSR分子标记研究苜蓿属及其近缘植物的亲缘关系[J].中山大学学报(自然科学版),2014,53(1):113-120.
[19] 黄映萍.DNA分子标记研究进展[J].中山大学研究生学刊(自然科学.医学版),2010,31(2):27-36.
[20] 段永红,渠云芳.遗传标记在植物研究中的应用[M].北京:中国农业科学技术出版社,2010:210-221.
[21] Flajoulot S,Ronfort J,Baudouin P,Barre P,Huguet T,Huyghe C,Julier B.Genetic diversity among alfalfa (Medicagosativa) cultivars coming from a breeding program,using SSR markers[J].Theoretical and Applied Genetics,2005,111:1420-1429.
[23] Falahati-Anbaran M,Habashi A A,Esfahany M,Mohammadi S A,Ghareyazie B.Population genetic structure based on SSR markers in alfalfa (MedicagosativaL.) from various regions contiguous to the centres of origin of the species[J].Journal of Genetics,2007,86:59-63.
[24] 苏东,周延林,于林清,常秀清.利用SSR分析中国北方野生黄花苜蓿种群的遗传多样性[J].中国草地学报,2010,32(5):85-90.
[25] 王亚玲,师尚礼,焦亮.陇东野生紫花苜蓿的生态特征[J].草业科学,2008,25(1):55-58.
[26] Jobin-Décor M P,Graham G C,Henry R J,Drew R A.RAPD and isozyme analysis of genetic relationships betweenCaricapapayaand wild relatives[J].Genetic Resources and Crop Evolution,1997,44:471-477.
[27] 刘志鹏.不同耐盐性状紫花苜蓿SSR分子标记研究[D].杨凌:西北农林科技大学,2004:20-25.
[28] 魏臻武.利用SSR、ISSR和RAPD技术构建苜蓿基因组DNA指纹图谱[J].草业学报,2004,13(3):62-67.
[29] 魏臻武,符昕,耿小丽,赵艳,曹致中,胡自治.苜蓿遗传多样性和亲缘关系的SSR和ISSR分析[J].草地学报,2007,15(2):118-123.
[30] 陈斐,魏臻武,李伟民,潘玲,郑曦,刘倩,张栋,屠德鹏.基于SSR标记的苜蓿种质资源遗传多样性与群体结构分析[J].草地学报,2013,21(4):759-768.
[31] 李景欣,鲁平,赵娜,崔国文.苜蓿新品系Dy-2006亲缘关系的SSR分析[J].中国草地学报,2011,33(2):116-120.
[32] Talebi M,Hajiahmadi Z,Rahimmalek M.Genetic diversity and population structure of four iranian alfalfa populations revealed by sequence-related amplified polymorphism(SRAP) markers[J].Journal of Crop Science and Biotechnology,2011,14(3):173-178.
[33] 俞金蓉.苜蓿品种间遗传多样性的ISSR分析[D].重庆:西南大学,2007.
[34] 刘晓生,郑道序,周春娟,马瑞君,詹潮安,杨海东,杨培奎,庄东红.潮汕余甘子种质资源遗传多样性与亲缘关系的ISSR分析[J].中国南方果树,2014,43(1):18-22.
[36] 云锦凤.牧草及饲料作物育种学[M].北京:中国农业出版社,2001:56-58.
[37] 严华,张冬梅,罗玉兰,鲁琳.38种国兰亲缘关系的ISSR分析[J].分子植物育种,2010,8(4):736-741.
[38] 张颖娟,王斯琴花.不同苜蓿种质材料的ISSR分析及遗传多样性研究[J].中国草地学报,2014,36(3):35-39.
[39] Mandoulakani B A,Piri Y,Darvishzadeh R,Bernoosi I,Jafari M.Retroelement insertional polymorphism and genetic diversity inMedicagosativapopulations revealed by IRAP and REMAP markers[J].Plant Molecular Biology Reporter,2012,30:286-296.
[40] 陈立强,师尚礼,满元荣.陇东野生紫花苜蓿的同工酶分析[J].草原与草坪,2010,30(1):24-32.
[41] 陈立强,师尚礼,王彩芬.陇东野生紫花苜蓿过氧化物酶同工酶分析[J].草地学报,2009,17(6):772-778.
[42] 陈立强,师尚礼,马春晖.野生及栽培苜蓿种质资源遗传多样性的同工酶分析[J].草业科学,2014,31(6):1070-1079.
[43] 张雪婷,师尚礼.陇东野生紫花苜蓿的遗传特异性分析[J].草地学报,2009,17(3):343-348.
[44] 关潇.野生紫花苜蓿种质资源遗传多样性研究[D].北京:北京林业大学,2009.
[45] Prosperi J M,Jenczewski E,Angevain M,Ronfort J.Morphologic and agronomic diversity of wild genetic resources ofMedicagosativaL. collected in Spain[J].Genetic Resources and Crop Evolution,2006,53:843-856.
(责任编辑 武艳培)
Genetic diversity of 42 alfalfa accessions revealed by SSR markers
CHEN Li-qiang1, SHI Shang-li2
(1.College of Animal Science, Tarim University; Key Laboratory of Tarim Animal Husbandry Science and Technology, Xinjiang Production & Construction Corps, Ala 843300, China; 2.Paracultural College of Gansu Agricultural University, Lanzhou 730070, China)
In order to reveal the genetic diversity of alfalfa germplasm resources, the genetic diversity of one wild and 41 cultivated alfalfa accessions were analyzed using SSR markers. The results showed that there were totally 231 alleles and 163 polymorphic alleles generated from these 42 alfalfa accessions by 15 SSR primers. The mean values of polymorphic percentage, Nei’s gene diversity and shannon’s information index for these 15 primer pairs were 71.55%, 0.210 0 and 0.326 3, respectively. The similarity coefficient of accessions ranged from 0.641 to 0.913 with an average of 0.791, and the mean value of similarity coefficient between wild alfalfa accessions and cultivars was lower than that among cultivars. Cluster analysis classified accessions into five defined groups at the genetic similarity value of 0.778, among which Longdong wild alfalfa, CW 200 and CW 787 were clustered into three independent groups, indicating that these accessions had relatively distant genetic relationship with the others. According to principal component analysis, the accessions can be divided into four groups, and the accessions in the Ⅰ and the Ⅱ groups had relatively distant genetic relationship with those in the Ⅲ and the Ⅳ groups. In conclusion, the alfalfa accessions used in the present study had relatively high genetic diversity, and some of which had relatively independent genetic characteristics.
alfalfa; genetic diversity; genetic relationship; SSR
SHI Shang-li E-mail:shishl@gsau.edu.cn
10.11829j.issn.1001-0629.2014-0333
2014-07-14 接受日期:2014-09-29
新疆生产建设兵团塔里木畜牧科技重点实验室开放课题(HS201103);新疆生产建设兵团塔里木畜牧科技重点实验室开放课题(HS201405);农业部全国畜牧总站“牧草种质资源保种”项目
陈立强(1981-),男,甘肃会宁人,讲师,硕士,研究方向为牧草种质资源与育种。E-mail:clqdky@126.com
师尚礼(1962-),男,甘肃会宁人,教授,博士,研究方向为牧草种质资源与育种。E-mail:shishl@gsau.edu.cn
S812;S541+.102;Q943
A
1001-0629(2015)03-0372-10
陈立强,师尚礼.42份紫花苜蓿种质资源遗传多样性的SSR分析[J].草业科学,2015,32(3):372-381.
CHEN Li-qiang,SHI Shang-li.Genetic diversity among 42 alfalfa accessions revealed by SSR markers[J].Pratacultural Science,2015,32(3):372-381.

