拟南芥DNA甲基化及去甲基化研究进展
卓婉清 腊红桂
摘 要 DNA甲基化及去甲基化是表观遗传的重要标志,广泛存在于细菌、动物、植物中,在控制转座子转录沉默、调控基因表达(X染色体失活,基因印迹,副突变)、维持植物发育等方面发挥重要作用。DNA表观修饰不改变基因序列,仅改变碱基修饰,并通过有丝分裂或者无丝分裂在后代中遗传。DNA甲基化是在甲基转移酶作用下,以S腺苷甲硫氨酸为甲基供体,使DNA胞嘧啶发生甲基化。研究发现,DNA去甲基化普遍存在于生物体内,在植物中,DNA糖基化酶介导的碱基切除修复机制被发现。基于此,对模式植物拟南芥中存在的DNA甲基化及去甲基化机制进行综述。
关键词 DNA甲基化;DNA去甲基化;表观遗传学
中图分类号:Q943 文献标志码:A 文章编号:1673-890X(2015)09-129-05
表观遗传学(Epigenetics)的词根最早出现于2000多a前,亚里士多德提出后生理论(The Theory of Epigenesis)。直到1975年,Holiday.R对表观遗传学进行了较为准确的解释,他认为,表观遗传学不仅在发育过程,而且应该在成体阶段研究可遗传的基因表达改变[1]。经典遗传学认为,核酸是遗传的分子基础,生命的遗传信息都是由核酸序列决定的。然而随着现代分子遗传学的发展,人们发现,不仅DNA序列包含遗传信息,核苷酸、组蛋白、染色质水平的修饰也会造成基因表达模式的改变,并可以在后代中稳定表达。
DNA甲基化是最常见的表观遗传现象,常见的DNA甲基化发生在DNA链上的胞嘧啶第五位碳原子和甲基间的共价结合,胞嘧啶被修饰为5甲基胞嘧啶(5mC)。与之相反,DNA去甲基化则是指5甲基胞嘧啶(5mC)被胞嘧啶取代的过程。
1 拟南芥DNA甲基化模式
在拟南芥中,DNA甲基化包括从头开始甲基化及维持性甲基化。从头开始甲基化是指DNA两条链都没有甲基化,通过甲基转移酶(DNMTs)先使其中一条链甲基化,然后再使另一条链甲基化。维持性甲基化指DNA中已有一条链发生了甲基化,在DNA复制过程中通过甲基转移酶来维持甲基化状态[2]。DNA甲基化主要发生在对称的CG,CHG序列和不对称CHH序列(H代表A,G,C,T)。其中CG甲基化的维持由DNA甲基转移酶MET1( DNMT1-like enzyme )调控,CHG由CMT3植物特异甲基转移酶来维持[3,4],通过与KYP(H3K9me2甲基化转移酶)形成回路,维持CHG甲基化[5]。不对称的CHH甲基化在复制过程中不能保持,由RdDM(RNA介导的DNA甲基化)通道的甲基转移酶DRM1和DRM2 发挥作用而形成[6]。新的研究发现,在met1和cmt3缺失突变体中,CHH基因组水平甲基化有更大的影响,相较于drm1和drm2突变体,这说明CMT3和MET1对于CHH甲基化可能有一定作用。此外维持CG,CHG甲基化可能也需要DRM1和DRM2作用[7]。拟南芥甲基化位点中,1/3位点与siRNAs 相关[8],剩下2/3位点独立于siRNAs。甲基转移酶可以靶向依赖siRNA(siRNA-dependent)通道和不依赖siRNA (siRNA-independent)通道实现CG或者非CG甲基化。
1.1 MET1介导的CG甲基化
MET1具有与哺乳动物DNMTs相同的结构域,在DNA复制过程中,发挥DNA甲基转移酶作用维持植物CG甲基化。此外,met1突变体表现出明显的花期延迟现象[9],且通过全基因组甲基化分析发现其对于非CG甲基化水平也有一定影响。
1.2 CMT3介导的CHG甲基化
CMT家族含有染色质结合结构域编码植物特异甲基转移酶,具有3个成员——CMT1、CMT2和CMT3,主要参与维持CHG甲基化。拟南芥中,通过对CMT3及KYP组蛋白甲基转移酶进行结构分析,揭示CMT3与KYP形成协同反馈回路共同维持CHG甲基化及TE转座子沉寂[10]。KYP被招募到CHG甲基化位点通过SET和SRA结构域;同时,CMT3的BAH结构域识别H3K9me2甲基化位点,招募CMT3形成CHG甲基化[11,12]。
1.3 RdDM介导的CHH甲基化
在拟南芥中,24个核苷酸大小的小干扰RNA (24-nt siRNAs)和长链非编码RNA指导的重新开始DNA甲基化及转录沉寂过程即为RNA指导的DNA甲基化(RdDM)。RNA介导的转录基因沉寂是一个保守的现象,在真菌、植物、动物中都会发生。对于许多细胞内功能如转座子沉寂,基因组稳定性,细胞保持等功能维持具有一定作用[13,14]。RdDM模型目前认为:RNA聚合酶Pol IV 产生单链RNAs 作为siRNAs前体,Pol IV与RNA依赖的RNA聚合酶II(RDR2)紧密联系,RDR2对Pol IV的转录产物加工成双链RNA,这个过程借助ATP依赖的染色质重塑因子CLSY1 作用。dsRNAs 被DCL3酶切成 24-nt siRNAs小片段,通过HEN1作用使siRNAs甲基化。单链siRNAs与AGO4或者AGO6[15]形成RdDM 效应因子复合物。AGO4复合物结合于RNA聚合酶Pol V ;同时,Pol V转录需要蛋白复合物DDR(包括DRD1、DMS3、RDM1)。其中,DRD1 是ATP依赖的染色质调控因子,DMS3 是黏连蛋白类似物, RDM1 具有单链DNA结合活性。RDM1是RdDM 效应因子复合物的一部分,既与AGO4 相联系又与重新开始的甲基转移酶DRM2 一起催化CG, CHG和CHH甲基化的形成。Pol V转录物的进一步扩增需要一些RNA结合蛋白,其中SPT5L/KTF1被招募到Pol V 转录独立于AGO4因子,起着稳定效应因子复合物作用[16]。IDN蛋白复合物也结合于 Pol V 转录物招募 SWI/SNF染色质重塑复合物,一起参与异染色质形成。而最近又发现,DTF1识别组蛋白H3K9甲基化,与CLSY1 相互作用在RdDM途径上游发挥作用[17]。
2 拟南芥DNA去甲基化模式
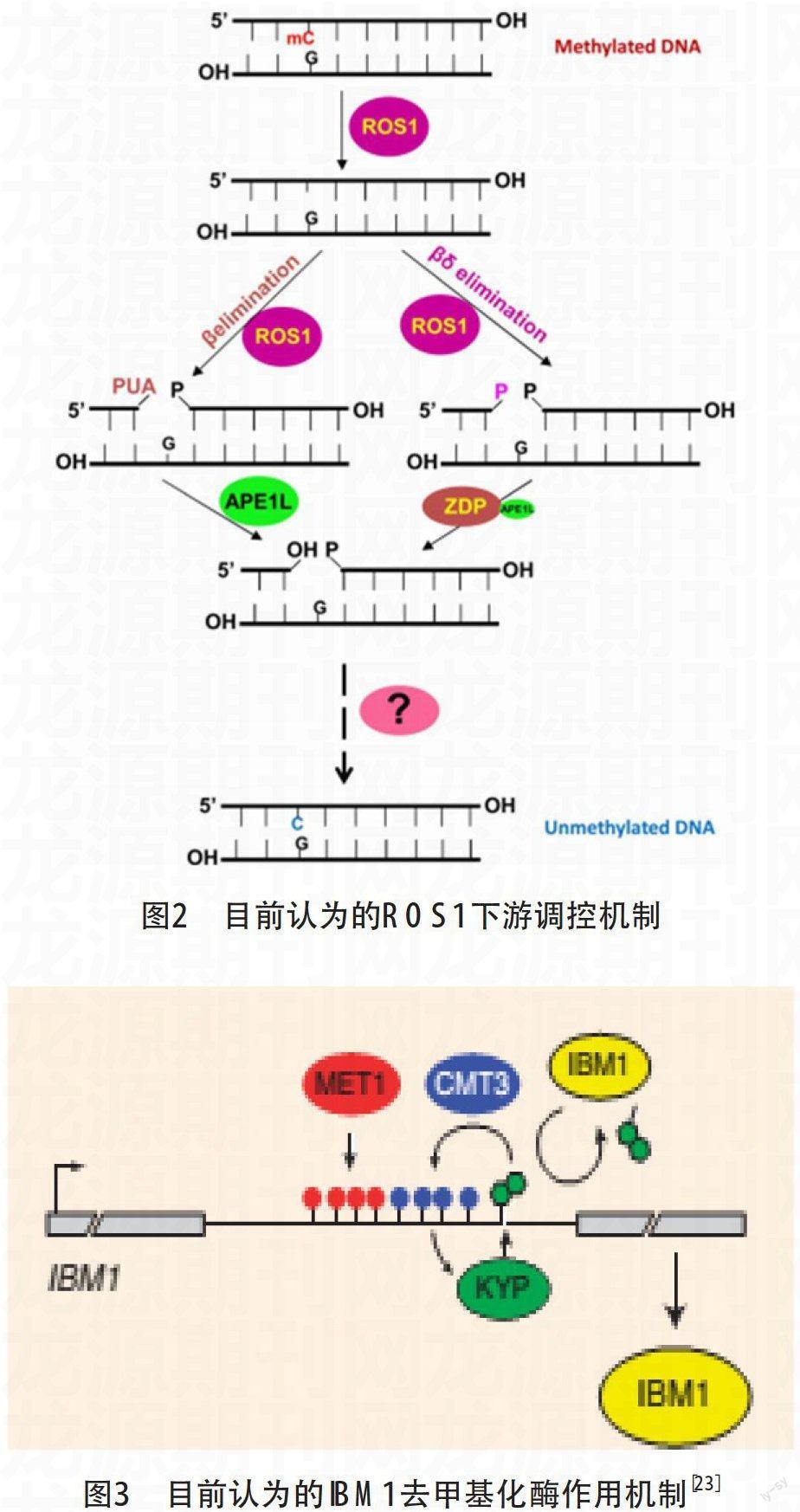
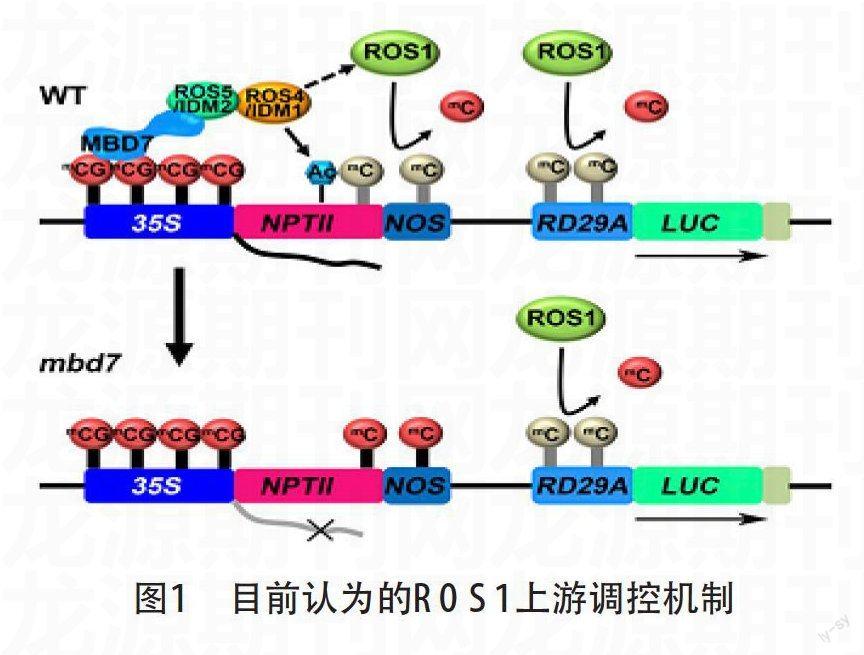
DNA去甲基化包括被动去甲基化和主动去甲基化。被动去甲化发生在DNA复制过程中,DNA甲基转移酶不活跃,导致未被甲基化的位点保留在新合成的链中。而活跃的去甲基化独立于DNA复制,存在5种可能的机制DNA糖基化酶初始的碱基切除修复机制、脱氨基作用及G/T碱基错配修复机制、核酸切除修复机制、氧化去甲基化机制及水解去甲基化[18]。在拟南芥中研究进展主要集中于DNA糖基化酶初始的碱基切除修复及氧化去甲基化两个机制上。其中,DNA糖基化酶家族中最先发现ROS1基因,之后DML2 和DML3等与ROS1功能类似的基因被发现,也发挥着去甲基化作用。目前认为 ROS1双官能团酶切除甲基,切割碳链暴露缺口,产生脱碱基位点,ZDP和APE1L将3'磷酸变成3'羟基,最后通过DNA聚合酶和DNA连接酶进行缺口修复,使甲基化位点脱甲基化[18-20]。ROS1是该通道中发挥作用的主要功能蛋白[19],那么ROS1是怎么靶向特异的位点?最近研究表明,甲基结合蛋白MBD7特异结合于高度甲基化的CG位点,并通过与IDM2和IDM3的相互作用,招募组蛋白乙酰转移酶IDM1催化H3K18和H3K23的乙酰化[21,22],进而招募去甲基化酶ROS1和其他去甲基化酶来限制DNA甲基化的延伸,防止基因沉默。氧化去甲基化机制揭示JmjC 蛋白对组蛋白H3K9和H3K36甲基化进行氧化去甲基化。在拟南芥中,JmjC 组蛋白去甲基化酶相关蛋白家族的成员 IBM1被发现,使组蛋白H3K9去甲基化,阻止DNA中BNS(BONSAI ——位点高度甲基化[23,24] ,研究还发现负责可变剪切的EDM2等蛋白[25]通过调节H3K9去甲基化酶IBM1的表达参与DNA去甲基化。
3 拟南芥DNA去甲基化相关的酶
3.1 ROS1(Repressor Of Silencing 1)
ROS1基因编码DNA去甲基化酶,是一个双官能团蛋白,既糖基化DNA又发挥裂解酶作用裂解DNA,对于维持转基因及同源内源基因的表达非常重要。ROS1通过抑制启动子甲基化来抑制RD29A-LUC转基因沉寂及内源基因沉寂[19]。在ROS1活性缺失的情况下,RdDM介导特异位点高度甲基化并使转录沉寂。
3.2 IDM1(Increased DNA Methylation 1)
IDM1,拟南芥DNA去甲基化的调控因子,对于防止ROS1调控的同源多拷贝基因及重复序列基因的高度甲基化非常重要。通过亚硫酸盐测序发现在idm1突变体中造成拟南芥基因组DNA甲基化升高的位点与ros1突变体产生的甲基化位点有较高的重合度,推测IDM1参与ROS1介导的去甲基化调控通路。 IDM1 结合于染色质中缺乏组蛋白H3K4二甲基及三甲基位点,使组蛋白H3乙酰化创造一个染色质环境为后续ROS1发挥作用。IDM1 是一个组蛋白H3乙酰基转移酶,具有三个结构域。通过DNA甲基化识别MBD结构域识别CG甲基化位点,植物同源PHD结构域识别未被甲基化的组蛋白H3K4表观遗传特征,组蛋白乙酰基转移酶HAT结构域使组蛋白H3K18和H3K23乙酰化[26]。由于IDM1与ROS1不能共定位,推测一个未知的与ROS1相互作用的蛋白可能识别乙酰化的H3K18 和H3K23 标志,招募ROS1靶向特异位点进行DNA去甲基化作用。
3.3 IDM2及IDM3(Increased DNA methylation 2 and Increased DNA methylation 3)
IDM2 属于高度保守的ACD蛋白家族,ACD蛋白最早在动物中发现,在各种细胞内进程及抵抗逆境病害等方面有着重要作用[27,28,29],在植物当中大部分ACD蛋白属于热休克蛋白[30] (sHSPs).大量研究表明ACD蛋白作为分子伴侣发挥作用[27] ,IDM2是一个编码α-晶体结构域的核定位点白其转录水平并不受热激影响,与IDM1具有相互作用证明它可能作为分子伴侣与IDM1一起参与组蛋白H3K18 乙酰化,在DNA活跃去甲基化及阻止转录沉寂中发挥作用[31]。IDM3与IDM2类似,具有α-晶体结构域且与MBD7具有相互作用[21]。
3.4 MBD7(Methyl-CpG-Binding Domain)
之前研究表明,DNA活跃去甲基化主要是由ROS1蛋白发挥主要作用,但是ROS1蛋白怎么靶向特异的甲基化位点并不清楚,而研究发现MBD7在特异识别甲基化位点发挥作用。MBD7蛋白的N端为甲基化识别结构域特异性识别CG甲基化位点,C端有很强的染色质识别能力。与IDM2及IDM3具有相互作用,进一步招募IDM1[26] 及其他一些蛋白进行组蛋白H3K18 和H3K23乙酰化,为ROS1发挥作用创造染色质环境(图1)。mbd7 突变体导致NPTII转基因沉寂 但不影响ProRD29A:LUC转基因沉寂,全基因组甲基化位点分析mbd7突变体甲基化位点与ros1突变体造成的甲基化位点有部分重合,推测MBD7可能是ROS1调控通道中的蛋白[22],且MBD7靶向靠近染色体中心高密度DNA甲基化位点, 同时影响Gypsy类型反转座子活动。
3.5 ZDP(Zinc Finger DNA 3Phosphoesterase)
ZDP突变体导致DNA高度甲基化及报告基因转录沉寂,全基因组甲基化分析zdp突变体使上百个内源基因位点高度甲基化,并且发现DNA磷酸化酶ZDP与ROS1具有相互作用,是ROS1活跃去甲基化分支的下游蛋白[32]。ROS1作用位点后产生单核苷酸缺口,ZDP将 3'磷酸变为3'羟基,通过单链核苷酸切除和长链DNA合成[33,34] ,使后续发生DNA聚合及连接反应修复缺口,实现去甲基化过程。最近又发现ROS1作用后的位点并不继续β,δ裂解而仍然是β裂解产物,产生3'不饱和醛基,不依赖于ZDP活动,可能通过AP 核酸内切酶 (APEs)发挥作用。
3.6 APE1L(Apurinic/Apyrimidinic Endonuclease)
APE1L,拟南芥AP核酸内切酶的一种,与ROS1蛋白在体内共定位。ROS1蛋白β裂解产生3'不饱和醛基,随后APE1L将不饱和醛基转换成3'羟基。生化数据表明APE1L有微弱的3'磷酸活性,证明APE1L属于ROS1调控通道另一分支的DNA活跃去甲基化下游蛋白,独立于ZDP分支通道(图2)。通过对突变体ape1l全基因组高度甲基化检测[20]。推测APE1L是一个多功能酶,可能与ZDP一起作用于DME糖基化酶调控通道,控制胚乳中印记基因及种子的生长。
3.7 IBM1(Increase in BONSAI Methylation 1)
IBM1是组蛋白H3K9me2去甲基化酶,在拟南芥中一方面,甲基转移酶CMT3与组蛋白甲基化酶KYP产生CHG甲基化及组蛋白H3K9me2;另一方面,由存在的CG甲基化招募IBM1发挥去除KYP产生的H3K9me2作用(图3)。IBM1调控的甲基化位点位于IBM1基因的一段很长且高度保守的内含子上(BONSAI 位点),编码两种mRNA变体,长链变体(IBM1-L)和短链变体(IBM1-S)。IBM-L编码有功能的IBM1蛋白包含jmjC 结构域[24],有实验证明在met1突变体中由于CG甲基化缺失,短链变体(IBM1-S) 表达量不受影响,长链变体(IBM1-L) 的表达量下调,导致H3K9me2的聚集。同时,ROS1去甲基化酶的活性也受到影响[23,24]。因此,通过控制H3K9去甲基化酶及DNA去甲基化酶活性,形成自我调控的回路维持CG甲基化及组蛋白修饰。
3.8 EDM2(Enhanced Downy Mildew 2)
染色质调控因子EDM2在阻止转录沉寂和DNA高度甲基化方面起作用,包含植物同源结构域识别活跃的或者受抑制的组蛋白甲基化标志。EDM2通过促进mRNA转录产生3'ployA末端来调控组蛋白H3K9去甲基化酶基因IBM1的表达,一起控制GHG甲基化和转录基因沉寂。edm2突变体与ibm1及DNA糖基化酶三突变体rdd重合上百个高度甲基化区域,其中包括与ROS1靶向区域[25]。研究认为类似于IDM1,EDM2和IBM1可能一起创造合适的染色质环境以便后续ROS1发挥作用,同时还发现EDM2在一些转座子的沉寂方面也有着一定的作用。
4 结语与展望
DNA甲基化、组蛋白修饰、非编码RNA调控、染色质结构重塑、RNA干扰、基因组印迹、基因沉默、X染色体不活跃等都属于表观遗传学范畴。其中,DNA甲基化是表观遗传学重要的标志之一,发展了各种方法检测DNA甲基化水平。在拟南芥中主要应用了甲基敏感酶进行核酸杂交(Southern blotting)及酶切PCR(CHOP PCR)检测,单个位点重亚硫酸盐测序(Bisulfite sequencing),染色质免疫共沉淀序列分析(ChIP-seq),全基因组重亚硫酸盐测序(Whole genome bisulfite sequencing)等,为进行相关研究提供了极大便利。目前,研究DNA甲基化对于控制转座子活动、植物生长发育、调节转录基因表达、如X染色体不激活、基因印迹等方面起着重要的生物学作用。对于DNA甲基化机制的研究也逐渐趋于完善,但是对于DNA甲基化与其他表观遗传调控机制如组蛋白修饰,染色质结构等方面的关系仍然不是很清楚,这将是今后研究的一个重点。
与DNA甲基化相反,DNA去甲基化在阻止转录基因沉寂,防止特异位点高度甲基化等方面发挥重要作用。目前,对于DNA去甲基化的研究上不够完善,如ROS1糖基化酶的碱基切除修复机制的下游机制如何进行?是否存在其他独立于ROS1的活跃去甲基化机制及其如何发挥作用等方面尚不清楚。
DNA甲基化的水平主要由甲基化和去甲基化这两个途经来协同调控。因此,有关DNA甲基化及去甲基化研究必将有力地促进表观遗传学的研究进程,进一步提高人们对于表观遗传学的认识,为其将来的应用奠定坚实的基础。
参考文献
[1]薛京伦,汪旭.表观遗传学:原理、技术与实践[M].上海:科学技术出版社,2006:5-6.
[2]李娜,张旸.植物DNA甲基化进展[J].植物生理学报,2012,48(11):1027-1036 .
[3]He XJ,Chen TP,Zhu JK.Regulation and Function of DNA Methylation in Plants and Animals[J].Cell Res,2011,21(3):442-465.
[4]Law JA,Jacobsen SE.Establishing,Maintaining and Modifying DNA Methylation Patterns in Plants and Animals[J].Nat Rev Genet,2010,11(3):204-220.
[5]Henderson IR,Jacobsen SE.Epigenetic Inheritance in Plants[J].Nature,2007(447):418-424.
[6]Huettel B,Kanno T,Daxinger L,et al.Endogenous Targets of RNA-directed DNA Methylation and Pol IV in Arabidopsis[J].EMBO J,2006(25):2828-2836.
[7]Zhu JK.Epigenome Sequencing Comes of Age[J].Cell,2008(133):395-397.
[8]Lister R,O'Malley RC,Tonti-Filippini J,et al.Highly Integrated Single-base Resolution Maps of the Epigenome in Arabidopsis[J].Cell,2008(133):523-536.
[9]Kankel MW,Ramsey DE,Stokes TL,et al.Arabidopsis MET1 Cytosine Methyltransferase Mutants[J].Genetics,2003(163):1109-1122.
[10]Du J,Zhong X,Bernatavichute YV,et al.Dual Binding of Chromomethylase Domains to H3K9me2-containing Nucleosomes Directs DNA Methylation in Plants[J].Cell,2012(151):167-180.
[11]Johnson LM,Bostick M,Zhang X,et al.The SRA Methyl-cytosine-binding Domain Links DNA and Histone Methylation[J].Curr Biol,2007(17):379-384.
[12]Cui X,Cao X.Epigenetic Regulation and Functional Exaptation of Transposable Elements in Higher Plants[J]. Curr Opin Plant Biol,2014(21):83-88.
[13]Zhang H,He X,Zhu JK.RNA-directed DNA Methylation in Plants:Where to start?[J].RNA Biol,2013(10):1593-1596.
[14]Zhang HM,Zhu JK.RNA-directed DNA methylation[J].Curr Opin Plant Biol,2011,14(2):142-147.
[15]Zheng X,Zhu J,Kapoor A,et al Role of Arabidopsis AGO6 in siRNA Accumulation,DNA Methylation and Transcriptional Gene Silencing[J].EMBO J,2007(26):1691-1701.
[16]He XJ,Hsu YF,Zhu S,et al.An Effector of RNA-directed DNA Methylation in Arabidopsis is an ARGONAUTE 4- and RNA-binding Protein[J].Cell,2009(137):498-508.
[17]Zhang H,Ma ZY,Zeng L,et al. DTF1 is a Core Component of RNA-directed DNA Methylation and May Assist in the Recruitment of Pol IV[J].Proc Natl Acad Sci U S A,2013(110):8290-8295.
[18]Zhu JK.Active DNA demethylation Mediated by DNA Glycosylases[J].Annu Rev Genet,2009(43):143-166.
[19]Gong Z,Morales-Ruiz T,Ariza RR,et al. ROS1,a repressor of transcriptional gene silencing in Arabidopsis, encodes a DNA glycosylase/lyase[J].Cell,2002(111):803-814.
[20]Li Y,Cordoba-Canero D,Qian W,et al. An AP Endonuclease Functions in Active DNA Dimethylation and Gene Imprinting in Arabidopsis[J].PLoS Genet,2015(11):1004905.
[21]Lang Z,Lei M,Wang X,et al.The Methyl-CpG-Binding Protein MBD7 Facilitates Active DNA Demethylation to Limit DNA Hyper-Methylation and Transcriptional Gene Silencing[J].Mol Cell,2015(57):971-983.
[22]Wang C,Dong X,Jin D,et al.Methyl-CpG-Binding Domain Protein MBD7 Is Required for Active DNA Demethylation in Arabidopsis[J].Plant Physiol,2015(167):905-914.
[23]Rigal M,Kevei Z,Pelissier T,et al.DNA Methylation in an Intron of the IBM1 Histone Demethylase Gene Stabilizes Chromatin Modification Patterns[J].EMBO J,2012(31):2981-2993.
[24]Fan D,Dai Y,Wang X,et al.IBM1,a JmjC Domain-containing Histone Demethylase,is Involved in the Regulation of RNA-directed DNA Methylation Through the Epigenetic Control of RDR2 and DCL3 Expression in Arabidopsis[J].Nucleic Acids Res,2012(40):8905-8916.
[25]Lei M,La H,Lu K,et al.Arabidopsis EDM2 Promotes IBM1 Distal Polyadenylation and Regulates Genome DNA mMethylation Patterns[J]. Proc Natl Acad Sci U S A,2014(111):527-532.
[26]Qian W,Miki D,Zhang H,et al.A Histone Acetyltransferase Regulates Active DNA Demethylation in Arabidopsis[J].Science,2012(336):1445-1448.
[27]Basha E,O'Neill H,Vierling E.Small Heat Shock Proteins and Alpha-crystallins:Dynamic Proteins with Flexible Functions[J].Trends Biochem Sci,2012(37):106-117.
[28]Hilton GR,Lioe H,Stengel F,et al.Small Heat-shock Proteins:Paramedics of the Cell[J].Top Curr Chem,2013(328):69-98.
[29]Welsh MJ,Gaestel M.Small Heat-shock Protein Family:Function in Health and Disease[J].Ann N Y Acad Sci,1998(851):28-35.
[30]Scharf KD,Siddique M,Vierling E.The Expanding Family of Arabidopsis Thaliana Small Heat Stress Proteins and A New Family of Proteins Containing Alpha-crystallin Domains(Acd proteins)[J].Cell Stress Chaperones,2001(6):225-237.
[31]Qian W,Miki D,Lei M,et al.Regulation of Active DNA Demethylation by an α-crystallin Domain Protein in Arabidopsis[J]. Mol Cell,2014(55):361-371.
[32]Martinez-Macias MI,Qian W,Miki D,et al. A DNA 3' Phosphatase Functions in Active DNA Demethylation in Arabidopsis[J].Mol Cell,2012(45):357-370.
[33]Cordoba-Canero D,Morales-Ruiz T,Roldan-Arjona T,et al.Single-nucleotide and Long-patch Base Excision Repair of DNA Damage in Plants[J]. Plant J,2009(60):716-28.
[34]Cordoba-Canero D,Dubois E,Ariza RR,et al.Arabidopsis Uracil DNA Glycosylase (UNG)is Required for Base Excision Repair of Uracil and Increases Plant Sensitivity to 5-fluorouracil[J].J Biol Chem,2010(285):7475-7483.
(责任编辑:刘昀)

