一种高效的葡萄球菌基因敲除方法的建立与应用
朱 涛,段 芹,李朝品,瞿 涤
一种高效的葡萄球菌基因敲除方法的建立与应用
朱 涛1,段 芹2,李朝品1,瞿 涤3
目的 建立一种高效构建葡萄球菌基因敲除突变株的方法。方法 通过融合PCR将vraSR操纵子上、下游同源片段连接起来;通过基于位点特异性重组的Gateway克隆技术将融合PCR产物连接入温度敏感型穿梭质粒pKOR1,转化修饰缺陷型大肠杆菌DC10B,获得同源重组质粒pKOR1-vraSR;电转化表皮葡萄球菌RP62A株;在高温和氯霉素双重选择压力下,质粒通过第1次同源重组整合到表皮葡萄球菌染色体上;转移到无抗生素的培养基中继续培养,发生第2次同源重组;将菌液涂布于含1 μg/mL脱水四环素的培养基上进行反向筛选,那些未发生重组的整合菌株因表达必需基因secY的反义RNA而无法生存,最后,挑取菌落通过PCR进行鉴定。结果 成功构建了难以转化的表皮葡萄球菌RP62A株的vraSR基因敲除突变株。结论 利用大肠杆菌DC10B和穿梭质粒pKOR1对葡萄球菌进行基因敲除的方法简便高效,适用的范围更广泛。
基因敲除;葡萄球菌;反向选择;限制修饰系统
Supported by the Open Research Fund Program of Key Laboratory of Medical Molecular Virology, Ministry of Education and Health(No.201406),the Research Fund of Anhui Province Key Laboratory of Biological Macro-molecules Research (No.LAB201408),the Key Program of Educational Commission of Anhui Province of China (No .KJ2015A218), and the Provincial Training Program of Innovation and Entrepreneurship for Undergraduates (No.AH201310368111) Corresponding author: Li Chao-pin, Email: cpli001@163.com
应用遗传操作鉴定病原菌毒力因子的原则被Stanley Falkow称为分子郭霍法则。基本的前提是通过精确的遗传操作,使编码潜在毒力因子的基因失活[1]。基因敲除是实现基因失活的重要手段之一。现阶段葡萄球菌基因敲除的基本策略是将目的基因上游片段、抗生素抗性基因、下游片段分别连接入大肠杆菌/葡萄球菌穿梭质粒构建同源重组质粒;然后导入限制性缺陷的金黄色葡萄球菌RN4220中进行修饰,最后导入目标菌株;在温度和抗生素的双重选择压力下,重组质粒与染色体发生两次单交换,抗生素抗性基因取代目的基因,突变株最终通过其对抗生素的抗性而被筛选出来[2-3]。
然而,由于同源重组以及质粒丢失的几率都很低,因此突变株的筛选就成为了一项费时费力的工作。而pKOR1质粒则通过引入反向选择改进了突变株的筛选工作。该质粒在诱导条件下可表达secY基因的反义RNA,secY是葡萄球菌的必需基因,携带或整合了该质粒的菌株在筛选时将无法生存,因此极大地提高了筛选的效率。另外,由于在质粒中引入了重组位点,因此可运用Gateway克隆技术快速构建同源重组质粒[4]。此外,由于葡萄球菌存在强大的限制修饰系统,因此对其基因敲除只能集中在少数可转化的金黄色葡萄球菌和表皮葡萄球菌实验室菌株上。近年来研究发现IV型限制修饰系统是阻止质粒DNA从大肠杆菌转移至葡萄球菌的主要屏障。IV型限制性内切酶识别胞嘧啶甲基化的DNA序列[5]。Monk等人建立了大肠杆菌克隆菌株DH10B的dcm突变株DC10B,发现分离自DC10B的质粒DNA能够直接转化金黄色葡萄球菌的大多数克隆复合体和表皮葡萄球菌[6-7]。
因此,本研究拟采用大肠杆菌DC10B和穿梭质粒pKOR1构建一株难以转化的表皮葡萄球菌的vraSR基因敲除突变株,vraSR基因编码的双组分调节系统在葡萄球菌的耐药性调控方面发挥着重要作用[8],以期建立一种高效的葡萄球菌基因敲除的新方法,为后续的基因功能研究提供强有力的技术支撑。
1 材料和方法
1.1 菌株和质粒 表皮葡萄球菌35 984株由本实验室保存,大肠杆菌DC10B由复旦大学许涛博士惠赠,穿梭质粒pKOR1由复旦大学丁百兴博士惠赠。
1.2 主要试剂和培养基 溶葡萄球菌素(Lysostaphin)、氯霉素、SanPrep柱式DNA胶回收试剂盒购自生工生物工程(上海)股份有限公司;脱水四环素为中国药品生物制品检定所产品;细菌基因组提取试剂盒、2×Pfu PCR MasterMix购自天根生化科技(北京)有限公司;BP ClonaseTMII Enzyme Mix购自Invitrogen公司;质粒提取试剂盒购自Qiagen公司;引物合成和测序由生工生物工程(上海)股份有限公司完成;TSB( Tryptic Soy Broth) 为美国BD公司产品;B2培养基成分为:10 g/L干酪素水解物,25 g/L酵母提取物,25 g /L氯化钠,1 g/L磷酸氢二钾,5 g/L葡萄糖。
1.3 表皮葡萄球菌基因组DNA的提取 将过夜培养的表皮葡萄球菌RP62A株按1∶100接种于4 mL TSB培养基中,37 ℃振荡培养4~5 h,取1 mL菌液12 000 g离心收集菌体,重悬于180 μL TE缓冲液,加入20 μL溶葡萄球菌素溶液(1 mg/mL),37 ℃孵育30 min以使菌体充分裂解(以澄清透明为准)。后续实验依照试剂盒使用说明书进行,最后基因组DNA溶解于60 μL超纯水中(pH7.0)。
1.4vraSR基因上下游同源片段的扩增及融合PCR反应 基因敲除突变株的构建流程和基本原理如图1所示。以表皮葡萄球菌基因组DNA为模板,以引物vraSR-UF和vraSR-UR扩增上游同源片段,以引物vraSR-DF和vraSR-DR扩增下游同源片段,切胶回收。具体引物序列见表1,小写字母为上下游同源片段之间的互补序列,下划线为attB1和attB2位点。参照李敏等人的文献[9],在50 μL的体系中加入等摩尔的上下游同源片段共3 μL(DNA总质量约600 ng),2×Pfu PCR MasterMix 25 μL,超纯水18 μL进行互补延伸,以形成全长的融合PCR产物(中间产物)。反应条件如下:94 ℃ 2 min;94 ℃ 20 s,55 ℃ 20 s,72 ℃ 2 min,10 cycles;72 ℃ 10 min。在上述中间产物中加入引物vraSR-UF和vraSR-DR各2 μL,依照下列反应条件:94 ℃ 5 min;94 ℃ 30 s,55 ℃30 s,72 ℃ 2 min,30 cycles;72 ℃ 7 min进行融合片段的全长扩增。
1.5 同源重组质粒pKOR1-vraSR的构建Gateway克隆 取融合PCR产物4 μL(约300 ng),pKOR1质粒1 μL(约150 ng),TE缓冲液3 μL,BP ClonaseTMII Enzyme Mix 2 μL,混和均匀后置于25 ℃孵育18 h。加入1 μL 2 μg/μL蛋白酶K,置于37 ℃ 孵育10 min以停止BP 反应。将上述BP 反应产物转化大肠杆菌DC10B感受态细胞,转化后的全部菌液涂布于含100 μg/mL氨苄青霉素的LB平板上,37 ℃培养过夜。从LB平板上挑取5个菌落进行质粒小量提取并对获得的质粒进行PCR扩增验证。PCR反应条件如下:94 ℃ 5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 2 min,30 cycles;72 ℃ 7min。
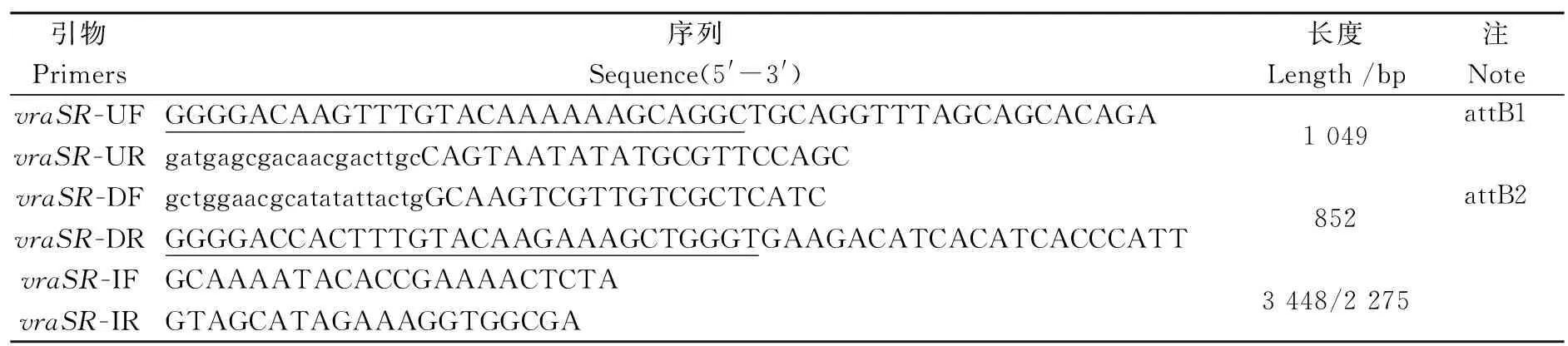
表1 用于基因敲除突变株构建和验证的PCR引物
1.6 表皮葡萄球菌感受态制备及电转化实验 取对数生长早期的表皮葡萄球菌6 mL,1/2体积的超纯水洗涤2次,1/5和1/10体积的10%甘油各洗涤一次,最后重悬于80 μL 10%甘油,即得感受态细胞。向感受态细胞中加入中量提取的重组质粒10 μg,混合均匀后转移至1 mm间隙的电击杯中进行电击。电转条件:电压2.1 kV,电容25 μF ,电阻100 Ω。立即加入800 μL B2培养基,30 ℃振荡培养3 h。全部菌液涂布至含10 μg/mL氯霉素的TSB平板上,30 ℃温箱培养48 h。
1.7 基因敲除突变株的筛选与鉴定 挑取PCR鉴定正确的单菌落接种至3 mL 含10 μg/mL氯霉素的TSB培养基(TSBCm10),30 ℃培养过夜。将菌液1∶100稀释到50 mLTSBCm10培养基中,42 ℃培养过夜。将菌液1∶100 稀释到50 mL TSBCm5培养基中,42 ℃培养过夜。菌液稀释涂布至TSBCm10平板上,42 ℃培养过夜。挑取单个菌落接种至5 mL TSB中,30 ℃培养过夜以促进质粒丢失。将菌液104倍稀释涂布至含1 μg/mL脱水四环素(ATc)的TSB平板上,37 ℃培养24 h。挑取50个菌落分别接种普通TSB平板和TSBCm10平板,37 ℃培养过夜。选择10个仅在TSB平板上生长的菌落进行基因组DNA提取,以上游同源片段外侧的引物vraSR-IF和下游同源片段外侧的引物vraSR-IR进行PCR扩增(如图1所示),并以野生株基因组作为对照,PCR反应条件为:94 ℃ 5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 3 min 30 s,30 cycles;72 ℃ 7 min。若能扩增出目的大小的DNA片段,则说明vraSR基因已被成功敲除。
2 结 果
2.1 同源区域融合片段的获得 如图2所示,以表皮葡萄球菌RP62A株基因组DNA为模板,通过PCR扩增分别获得了1 049 bp和852 bp大小的vraSR基因上、下游同源片段,进一步通过融合PCR反应将两段连接起来,获得了1 901 bp大小的融合产物。

UF: upstream fragment; DF: downstream fragment.
Fig.1 Schematic chart for the construction of thevraSRknockout mutant

UF: upstream fragment, DF: downstream fragment, UD: upstream + downstream
图2vraSR操纵子上、下游同源片段的融合PCR反应
Fig.2 Fusion PCR product of fragments upstream and downstream of thevraSRoperon
2.2 同源重组质粒pKOR1-vraSR的鉴定 采用引物vraSR-UF和vraSR-DR对挑取的5个转化后的DC10B菌落提取质粒进行PCR扩增,结果显示从5个菌落均扩增出了长度为1 901 bp的特异性DNA片段(图2),表明通过Gateway克隆技术成功构建了同源重组质粒pKOR1-vraSR。
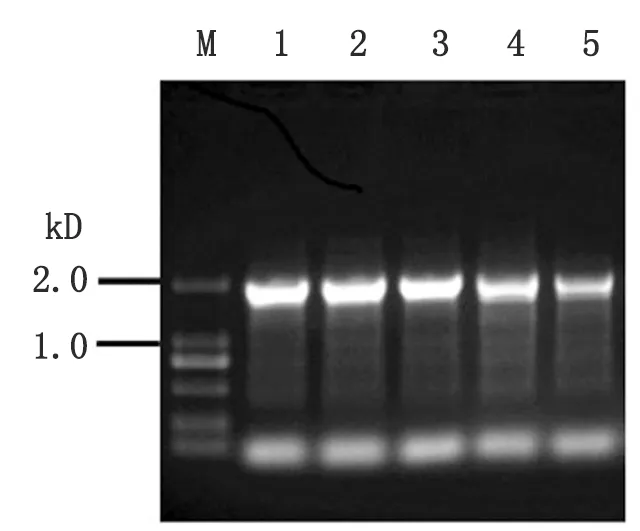
M: D2 000 marker; Lanes 1-5: five transformants.
2.3 表皮葡萄球菌vraSR基因突变株的鉴定 以上、下同源片段外侧区域设计的引物vraSR-IF和vraSR-IR,对挑选的10个可在脱水四环素诱导条件下生长、在含氯霉素培养基上不能生长的菌落和野生株分别提取基因组DNA进行PCR扩增,结果显示3、4、7、8、9号菌落的PCR产物长度与野生株的一致,在3.5 kb左右,而1、2、5、6、10号菌落的PCR产物长度不到2.5 kb(图3),表明1、2、5、6、10号菌落的vraSR基因被成功敲除。

M: 1 kb DNA marker; Lanes 1-10: ten colonies picked from TSB agar containing ATc; wt: wild type strain.
图4 表皮葡萄球菌vraSR基因突变株的PCR筛选鉴定
Fig.4 PCR screening forvraSRknockout mutants ofS.epidermidis
3 讨 论
CA-MRSA的出现以及表皮葡萄球菌医院分离株耐甲氧西林的高发生率,亟须我们在基因水平上更好地理解这些医学重要葡萄球菌[10-11]。然而,由于无法对大多数的临床分离株进行包括基因敲除在内的遗传操作,金黄色葡萄球菌和表皮葡萄球菌的功能基因组研究受到了很大的限制。现阶段对金黄色葡萄球菌的功能研究主要通过突变实验室菌株Newman、8325-4,以及近年来通过突变一些社区获得性耐甲氧西林USA300菌株来进行。而对于表皮葡萄球菌,可转化的菌株1457和O-47是遗传研究的主体,但是这些菌株的基因组目前尚未被测定[7]。本研究利用Gateway克隆技术构建同源重组质粒,经大肠杆菌DC10B扩增后成功转化了过去难以转化的表皮葡萄球菌RP62A株,通过反向选择成功筛选出了表皮葡萄球菌vraSR突变株,建立了一种强大的可在葡萄球菌实验室菌株和先前无法被转化的菌株上通过同源重组构建突变株的方法。
该方法简便快捷,适用范围广泛,首先体现在同源重组质粒的构建方面,基于位点特异性重组的Gateway技术,相较于传统的克隆构建省去了酶切连接的步骤。其次,同源重组质粒经DC10B扩增后无须经过金黄色葡萄球菌RN4220的修饰便可直接转化目标菌株,不仅简化了实验步骤和节约了时间和试剂成本,因为从RN4220中提取质粒不但需要昂贵的溶葡萄球菌酶,而且往往由于裂解效率低导致质粒的浓度和纯度都不高,还克服了经RN4220修饰的质粒仅能转化一小部分亲缘关系较近的金黄色葡萄球菌或表皮葡萄球菌分离株的不足。大肠杆菌DC10B的建立与应用使得之前难以对其进行遗传操作的葡萄球菌的研究工作能够得以开展。最后,反向选择技术的应用既能提高筛选的效率,又能获得无抗生素标记的突变株。本研究中作者挑取了50个菌落进行筛选,最后鉴定到5个菌落的基因被成功敲除,突变株的获得率不低于10%;而作者曾经使用pBT2穿梭质粒构建双组分调节系统基因lytSR和arlSR的突变株,需要挑取200~400个菌落,突变株的获得率通常不足1%[12]。穿梭质粒pMAD虽然通过引入蓝白斑筛选很大程度上提高了筛选效率,但由于需要利用抗生素抗性作为筛选标记,因此会在突变株中引入外源基因。
[1]Falkow S. Molecular Koch's postulates applied to microbial pathogenicity[J]. Rev Infect Dis, 1988, 10(Supplement 2): S274-S276.
[2]Bruckner R. Gene replacement inStaphylococcuscarnosusandStaphylococcusxylosus[J]. FEMS Microbiol Lett, 1997, 151(1): 1-8. DOI: 10.1111/j.1574-6968.1997.tb10387.x
[3]Arnaud M, Chastanet A, Debarbouille M. New vector for efficient allelic replacement in naturally nontransformable, low-GC-content, gram-positive bacteria[J]. Appl Environmental Microbiol, 2004, 70(11): 6887-6891. DOI:10.1128/AEM.70.11.6887-6891.2004
[4]Bae T, Schneewind O. Allelic replacement inStaphylococcusaureuswith inducible counter-selection[J]. Plasmid, 2006, 55(1): 58-63.
[5]Corvaglia AR, Francois P, Hernandez D, et al. A type III-like restriction endonuclease functions as a major barrier to horizontal gene transfer in clinicalStaphylococcusaureusstrains[J]. Proc Nat Acad Sci USA, 2010, 107(26): 11954-11958. DOI: 10.1073/pnas.1000489107
[6]Monk IR, Foster TJ. Genetic manipulation ofStaphylococci--breaking through the barrier[J]. Front Cellul Infect Microbiol, 2012,2:49. DOI: 10.3389/fcimb.2012.00049
[7]Monk IR, Shah IM, Xu M, et al. Transforming the untransformable: application of direct transformation to manipulate geneticallyStaphylococcusaureusandStaphylococcusepidermidis[J].mBio,2012,3(2):e00277-11.DOI:10.1128/mBio.00277-11
[8]Qureshi NK, Yin S, Boyle-Vavra S. The role of the staphylococcal VraTSR regulatory system on vancomycin resistance and vanA operon expression in vancomycin-resistantStaphylococcusaureus[J]. PLoS One, 2014, 9(1): e85873. DOI:10.1371/journal.pone.0085873
[9]Li M, Yang Q. A rapid method for generation of homologous recombinant fragments-fusion PCR [J]. China Biotechnol, 2007, 27(8): 53-58.DOI: 10.3969/j.issn.1671-8135.2007.08.010(in Chinese) 李敏,杨谦.一种高效构建同源重组 DNA 片段的方法——融合 PCR[J].中国生物工程杂志, 2007, 27(8): 53-58.DOI: 10.3969/j.issn.1671-8135.2007.08.010
[10]Otto M.Staphylococcusepidermidis--the'accidental'pathogen[J]. Nat Rev Microbiol, 2009, 7(8): 555-567. DOI: 10.1038/nrmicro2182
[11]Chambers HF, DeLeo FR. Waves of resistance:Staphylococcusaureusin the antibiotic era[J]. Nat Rev Microbiol, 2009, 7(9): 629-641. DOI: 10.1038/nrmicro2200
[12]Lou Q, Zhu T, Wang JX, et al. Methods of constructingStaphylococcusepidermidisgene mutants via different plasmids[J]. J Microbes Infect, 2008, 3(1): 11-16. DOI: 10.3969/j.issn.1673-6184.2008.01.005 (in Chinese) 娄强,朱涛,王家学,等.表皮葡萄球菌基因删除突变株构建方法的研究[J].微生物与感染,2008, 3(1): 11-16.DOI: 10.3969/j.issn.1673-6184.2008.01.005
Establishment and application of a rapid and efficient method for gene replacement inStaphylococci
ZHU Tao1,DUAN Qin2,LI Chao-pin1,QU Di3
(1.DepartmentofMedicalParasitology,WannanMedicalCollege,Wuhu241002,China;2.DepartmentofStomatology,WannanMedicalCollege,Wuhu241002,China; 3.KeyLaboratoryofMedicalMolecularVirology,MinistryofEducationandHealth,FudanUniversity,Shanghai200032,China)
We established a rapid and efficient method for creating knockout mutants inStaphylococci. The upstream and downstream homologous regions of thevraSRoperon were combined by fusion PCR, ligated into temperature-sensitive shuttle plasmid pKOR1 by the Gateway cloning technology, and then transformed into a restriction-defectiveE.colistrain DC10B, yielding the recombinant plasmid pKOR1-vraSR. The plasmid was then electroporated intoStaphylococcusepidermidisRP62A strain. Under the selective pressure of high temperature and chloramphenicol, the plasmid was firstly integrated into the chromosome ofS.epidermidisthrough a single crossover event. The integrant strain was then subcultured, and the second crossover occurred subsequently in the absence of antibiotic selection, resulting in either wild-type strain or the mutant, which depended upon where the recombination event occurred. Finally, the culture was plated on the medium containing 1 μg/mL anhydrotetracycline for counter selection, several colonies were picked up and analyzed by PCR to identify the mutant. Result showed that avraSRknockout mutant of the untransformableS.epidermidisRP62A was successfully generated in this study. In conclusion, the method for gene replacement inStaphylococciby usingE.coliDC10B and shuttle plasmid pKOR1 is simple and efficient.
gene knockout;Staphylococci; counter selection; restriction modification system
10.3969/j.issn.1002-2694.2015.12.003
李朝品,Email:cpli001@163.com
1.皖南医学院医学寄生虫学教研室,芜湖 241002; 2.皖南医学院口腔医学院,芜湖 241002; 3.复旦大学教育部/卫生部医学分子病毒学重点实验室,上海 200032
R378.1
A
1002-2694(2015)12-1098-05
2015-04-15;
2015-07-09
教育部医学分子病毒学重点实验室开放课题(No.201406),安徽省重点实验室自助研究课题(No.LAB201408),安徽省教育厅自然科学研究重点项目(No.KJ2015A218),省级大学生创新创业训练计划(No.AH201310368111)

