阿尔茨海默症miRNA靶基因预测研究
孔 薇, 张 昕, 牟晓阳
(1. 上海海事大学信息工程学院, 上海 201306; 2. 美国罗文大学生物化学系, 新泽西 08028)
阿尔茨海默症miRNA靶基因预测研究
孔 薇1, 张 昕1, 牟晓阳2
(1. 上海海事大学信息工程学院, 上海 201306; 2. 美国罗文大学生物化学系, 新泽西 08028)
为深入了解阿尔茨海默症的发病机制, 利用匹配的基因表达数据和miRNA表达数据, 对miRNA的靶基因进行预测分析. 首先对选择出的差异表达miRNA进行靶基因预测; 然后利用HCTarget算法验证预测的靶基因; 最后对miRNA及其靶基因构建调控网络. 调控网络中包含11个已确定的与AD有关的miRNA及6个AD致病基因. 通过分析网络发现miRNA及其靶基因在正常与患病两种情况下的活性变化趋势, 生物学分析也证实了它们在AD中的重要作用, 为AD发病机制研究提供了依据.
微小RNA; 阿尔茨海默症; 靶基因; 预测; HCTarget算法
0 引言
阿尔茨海默症(Alzheimer’s disease, AD)是一种典型的中枢神经退行性疾病, 以进行性痴呆为主要临床表现. AD的特征性病理改变为大脑皮层及脑区的纤维蛋白沉积, 即细胞外间隙的β淀粉样蛋白(β-amyloid,β-A)和细胞内多聚Tau蛋白的沉积, 病理形态学上分别表现为老年斑(SP)和神经纤维缠结(NFT)[1-2]. 近年来对AD的生物信息学研究主要集中在基因表达数据的处理、 基因调控网络的构建等方面. 随着miRNA研究的深入, 其在AD发生发展中的作用也渐渐为人们重视. miRNA对AD发生发展的影响涉及到对APP、 BACE1、 神经元的凋亡等多环节的调控作用[3-5]. 融合基因表达数据和匹配的miRNA数据, 进行miRNA靶基因预测并构建miRNA—基因调控网络.
miRNAs(micro RNAs)是一类能在转录水平或转录后水平调控基因表达的长度为19~27个核苷酸的内源性非编码的小分子RNA, 广泛分布于中枢神经系统中, 对神经发育、 分化和成熟起着重要作用[6]. 目前, 对miRNAs的研究主要集中在其mRNA靶基因的识别上, 研究人员从实验和计算机方法两方面对miRNAs和mRNAs靶基因进行识别. 计算机方法成为miRNAs和mRNAs靶基因识别的主要方法. 近年来, 已经有GenMiR++, Talasso等用表达数据来预测miRNA-mRNA相互作用关系的算法. 它们结合序列基本信息和表达数据来获得更可靠的miRNA靶基因. 使用由苏乃芳等人在GenMir++基础上提出的HCtarget算法预测靶基因, 并使用了马尔科夫链(MCMC)对GenMir++进行了改进, 其具备更好的稳健性和准确性[7].
现有的miRNA研究大部分是基于单个miRNA在疾病中的功能及受其调控的基因的研究, 对于多个与疾病相关的miRNA综合研究及其靶基因预测方面的研究相对较少[8-10]. 使用miRNA与靶基因匹配数据, 对与AD相关的miRNAs进行靶基因预测、 验证及调控网络的构建. 首先使用TargetScan、 Pictar、 miRanda等在线分析工具对筛选出的miRNA进行靶基因预测, 取三者的交集, 提高预测靶基因的准确性; 其次用HCtarget算法, 利用基因表达数据和miRNA表达数据, 结合miRNA—靶基因调控关系, 对预测的靶基因进行验证, 找出相互关系概率高的miRNA—靶基因对; 最后对筛选出的miRNA—靶基因对绘制调控网络.
1 算法
1.1 miRNA靶基因预测算法
近年来, 研究者提出了很多miRNA的靶基因预测方法, 其中比较公认的是TargetScan[11]、 Pictar[12]和miRanda[13]三种方法. TargetScan(http://www.targetscan. org/index.html)算法是基于mRNA的3’非编码区搜寻与miRNA的5’端第2~8位核苷酸完全互补的种子序列, 并综合考虑RNAFold软件计算得到的结合位点的热力学稳定性值, 最后选取评分最高的mRNA序列. miRanda(http://www.miRNA.org/miRNA)算法原理依据主要是通过得分矩阵计算出互补程度大小, 寻找互补性最高的3’UTRs, 强调miRNA 与靶基因结合位点的保守性, 同时以miRNA 序5’端搜索靶基因. miRanda 利用Vien2naRNA计算miRNA 靶标复合体热力学稳定性, 并淘汰不能形成双连体的假阳性靶标. Pictar(http://pictar.mdc-berlin.de/)认为基因3’UTR序列是由miRNA绑定点及背景序列组成. PicTar利用Baum-welch算法来计算3’UTR序列是由此隐马尔科夫模型产生的最大似然概率. 但是以上算法由于对完全种子匹配的要求过于严格, 训练数据集不够等导致了较高的假阳性率, 因此使用HCtarget算法结合序列信息和表达数据预测miRNA靶基因.
1.2 HCTarget算法原理
HCtarget用一个线性模型来描述miRNA和mRNA的关系, 并使用马尔科夫链-蒙特卡罗(Markov Chain Monto Carlo, MCMC)算法计算靶标的概率.
1.2.1 模型
假设miRNA-mRNA对中, miRNA个数为M, 基因个数为N, 样本数为T. 输入矩阵Z代表M个miRNA在T个样本中的表达值; 输入C矩阵代表基因与miRNA的调控关系, 受miRNA调控则对应写1(Cij=1), 否则写0(Cij=0)[14]; 输出yit代表N个基因在T个样本中的表达值, 组成表达值矩阵Y. 这里i=1, …,N;j=1, …,M.
mRNA及miRNA表达谱间的关系用公式表示如下:
这里的yit和zjt分别表示mRNA及miRNA在T个样本中表达值.βjt代表miRNA在T个样本中调控强度,βot是样本t的背景影响.
使用R这个潜在的二进制数来表示miRNA与受其调控的靶基因之间的关系. 此算法模型的目标就是求出R. 在矩阵C中, 假设R服从伯努利分布(二项分布), 也就是说, 在Cij=1和rij=0(Cij=0)的情况下,rij~bernoulli(π), 假设R:
这里的π可以作为预测序列的准确性. 此假设可以减少先前预测的假阳性率.
1.2.2 统计推断算法MCMC
基于上面的模型, 假设观察数据概率分布为:
为了估计参数θ= (β,σ2,π)及潜在变量R, 应用贝叶斯方法和MCMC算法. 结合适合的先验假设,R和θ可通过如下迭代使用MCMC算法直接计算得出:
1) 基于更新的潜在变量取参数θ;
2) 基于更新的参数取变量R.
校正参数:
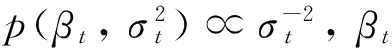
这里v=N-M-1, 且
对于π, 结合先验的π~Beta(a0,b0),π的后验分布为:
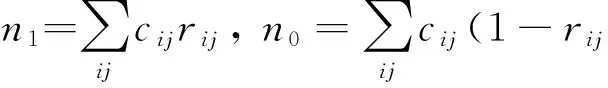
校正潜在变量:
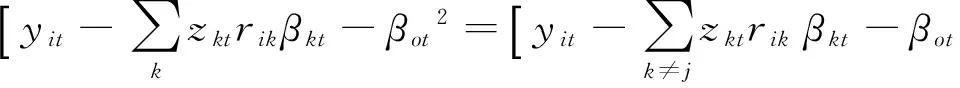

HCTarget算法:
基于上述讨论, 使用一个传统的MCMC方法来反复评估参数和变量:
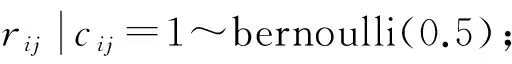
③ 由校正的参数来取样潜在变量rij;
④ 重复上面两个步骤直至收敛.
输出的pij, 作为预测的miRNA调控mRNA的后验概率.miRNA-mRNA对的p值大于某一阈值时, 此miRNA-mRNA对就认为是推断正确的miRNA-mRNA对.
2 实验与结果
2.1 数据来源
所使用的阿尔茨海默症mRNA表达数据及miRNA表达数据来自美国国立生物技术信息中心(NationalCenterforBiotechnologyInformation,NCBI)网站的基因表达综合数据库(geneexpressionomnibus,GEO)中的数据集GSE16759. 基于AffymetrixHumanGenomeU133Plus2.0Array平台的数据. 此数据来自AD患者及年龄相仿的正常实验者大脑皮层的匹配的miRNA和mRNA表达数据. 包括16组AD样本, 其中8个样本为mRNA表达数据, 共54 675个基因, 8个样本为miRNA表达数据, 共940个miRNA值. 两类样本都是按照4组正常对照样本和4组患病样本分类. 阿尔茨海默症的miRNA来自人类小RNA疾病数据库. 该数据库提供实验验证的与人类疾病相关的miRNA, 及其与对应疾病的关系.
2.2 实验数据处理
为了去除基因表达数据中大量的冗余数据和噪声, 首先用T统计对数据进行预处理. 将4个正常样本与4个患病样本的基因表达数据和miRNA数据进行T统计分析, 最终选取t>1.0(p<0.048)的7 447个基因作为差异表达基因. 选取t>2.455(p<0.05)的169个miRNA作为差异表达miRNA. 将此169个差异表达的miRNA与HMDD中与AD相关的miRNA进行匹配, 得到11个miRNA, 这11个miRNA及其与AD的关系如表1所示, 表中Description为对应miRNA在HMDD中得到的与AD关系的描述. 这些描述来自实验方法、 遗传学实验、 表观遗传学实验及miRNA靶标交互实验.
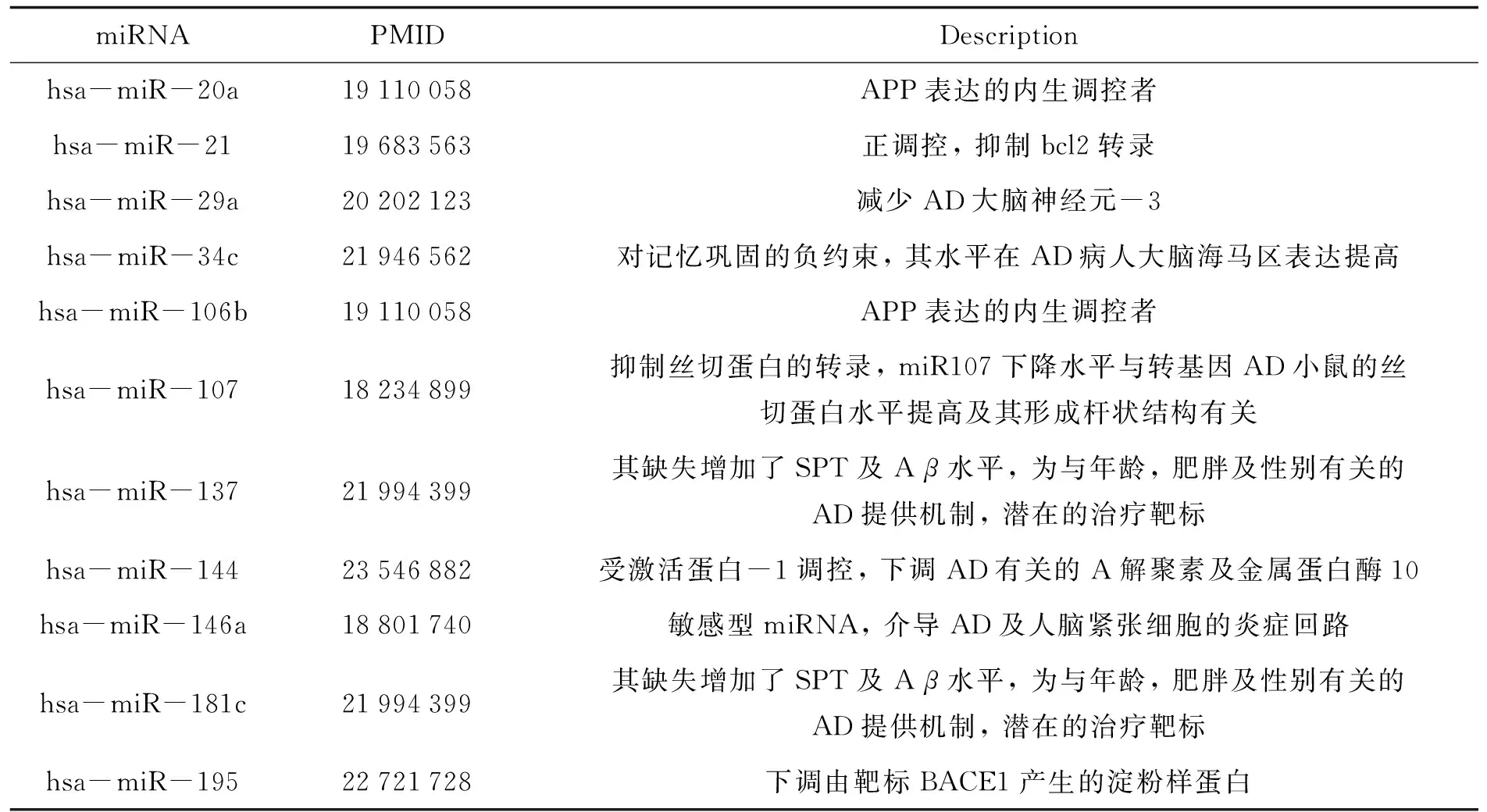
表1 11个miRNA及其与AD的关系
2.3 miRNA靶基因预测
首先, 将上述选取的11个miRNA通过软件TargetScan、 Pictar和miRanda预测它们的靶基因. 将每个miRNA预测的靶基因与上述选取的差异表达基因进行匹配, 得到对应的基因表达数据. 最终得到11个miRNA共调控806个靶基因, 每个miRNA调控的靶基因数目如表2所示.
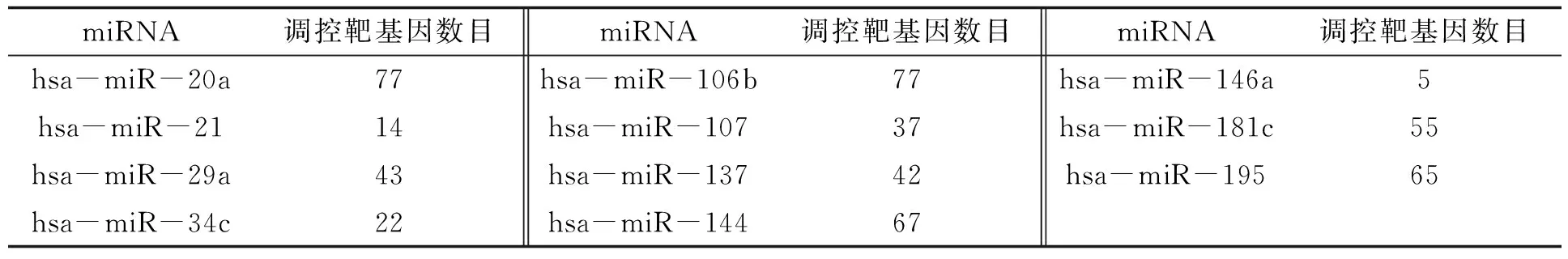
表2 11个miRNA及其调控的靶基因数目
2.4 miRNA调控网络构建及结果
上述得到的miRNA及其靶基因对中, 有单个miRNA调控多个靶基因, 也有同一靶基因受多个miRNA调控的情况. 为了提高预测的准确性, 利用HCTarget算法检验得到的1 374个成对的miRNA-mRNA, 经过取不同阈值进行试验, 在提高预测靶基因的准确性及结果可靠性的前提下, 设定阈值为0.8, 最后得到504对miRNA-mRNA, 其中mRNA共292个. 为了更直观地观测, 用Cytoscape软件绘制出它们之间的调控网络图, 如图1所示. 其中靶基因的表达值由源数据中基因在各样本中的表达值取均值所得, miRNA表达活性由源数据中miRNA在各样本中表达活性取均值所得. 图中圆中心的11个菱形节点代表miRNA, 外层两圈圆形节点代表靶基因; 节点为红色代表表达水平上升, 绿色代表表达水平下降, 颜色越深表示表达水平越高.
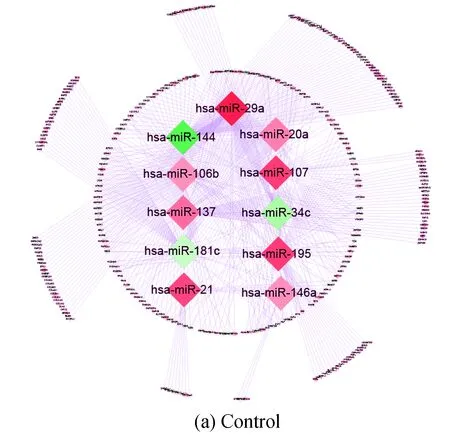

图1 11个miRNA及其靶基因在正常、 患病样本下调控图
2.5 调控网络分析
从调控图可以直观地看出, miR-20a、 miR-29a、 miR-195、 miR-21、 miR-107和miR-137在正常样本和患病样本中表达活性都较高. 与正常样本相比较, 除了miR-107以外的10个miRNA在患病样本中的表达活性都上升. miR-107在早期AD发病进展中在颞叶皮层灰质区表达活性降低, 邻近大脑组织中神经元血小板的计数和神经元纤维缠结数的增加与miR-107的表达水平降低有关[15]. 计算分析表明, 3’UTR区域的mRNA-BACE1受miR-107调控. BACE1是一种叫做淀粉样蛋白前β位分解酶1的酶, 其促进β-淀粉样蛋白生成和调控另一个细胞过程导致记忆丧失而引起AD的发病. miR-29a调控的靶基因NAV3在AD患者大脑中表达上升, 免疫组织化学研究表明, NAV3的表达在AD患者大脑皮层退化锥体神经元中明显增强. miR-29a就是通过增强AD大脑中的神经元NAV3的表达来影响神经退行性过程的[16]. miR-146a调节免疫反应并影响AD的发病. miR-146a参与重要的细胞功能, 其调控的靶基因bcl2控制线粒体功能和细胞老化, 与AD的发生发展有重要关系[17].
为了进一步分析图1中miRNA及其靶基因与AD的关系, 使用在线分析网站DAVID (http://david.abcc.ncifcrf.gov/home.jsp)对预测的靶基因进行KEGG通路、 生物过程、 分子功能分析. 表3为预测靶基因的KEGG通路分析结果.
由表3可以看出, 预测的靶基因中大量基因参与了癌症相关的信号通路, MAPK信号通路, TGF-β信号通路, jak-STAT信号通路, 这些都是与AD相关的通路. 在阿尔茨海默症中, 通过MAPK信号传导通路, 淀粉样纤维激活细胞坏死信号通路. MAPK属于脯氨酸依赖的蛋白激酶的一种, 在病人大脑中诱使tau蛋白过度磷酸化, 继而导致AD. TGF-β诱导生物钟基因的失调, 导致神经通路的改变, 这与AD患者的睡眠—觉醒节律异常是有因果联系的[18]. 与此同时, TGF-β通路在肿瘤的发生发展中有重要的作用, 通过研究其与miRNA机制可以得到新的潜在治疗靶标[19]. jak-STAT信号通路是一条由细胞因子刺激的信号转导通路, 参与细胞的增殖、 分化、 凋亡以及免疫调节等许多重要的生物过程. 虽然没有确切的解答答案, 但是现有的大量研究表明, 癌症和阿尔茨海默症呈现一种负相关的关系. 因此, 在表3中, 预测的靶基因参与了很多癌症相关通路, 这并不表示这些基因与AD无关. 如p53基因是一种重要的抑癌基因, 它的缺失或突变将明显增加恶性肿瘤的易感性. 有研究显示, p53与AD也有着密切的联系. p53的激活对促进细胞老化具有直接作用, AD患者的皮质神经元中大量表达p53[20].
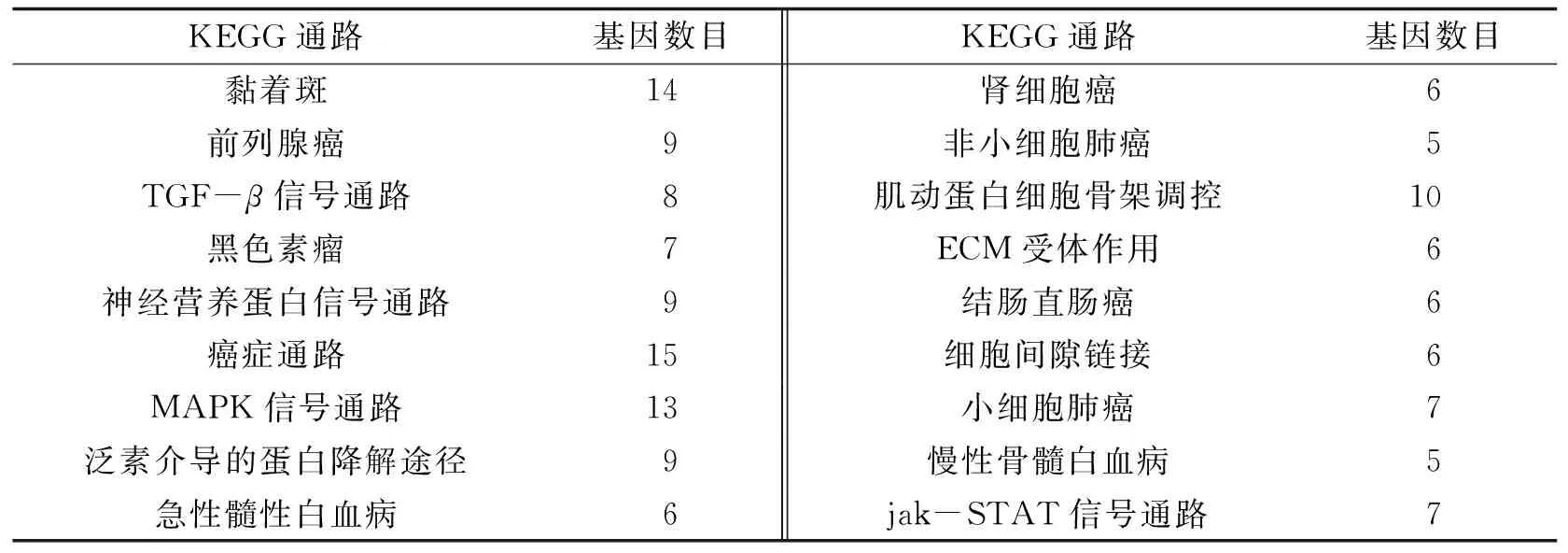
表3 KEGG通路分析结果
以miR-195为例来详细讨论miRNA在调控网络中的作用. 研究表明, miR-195表达水平与BACE1表达水平有关, miR-195表达上升, 则BACE1表达下降; 而miR-195表达下降会导致BACE1的表达上升[21]. 其在网络中调控65个靶基因, 如图2所示. 通过KEGG通路分析, 得出结果如表4.
miR-195调控的基因有几个显著的功能: 蛋白质复合物捆绑(GO:0032403), 蛋白激酶活性(GO:0004672), 转录调节因子粘合物(GO:0030528)和转录因子粘合物(GO:0008134). 这些都是生物的基本功能, 说明miR-195的重要作用.
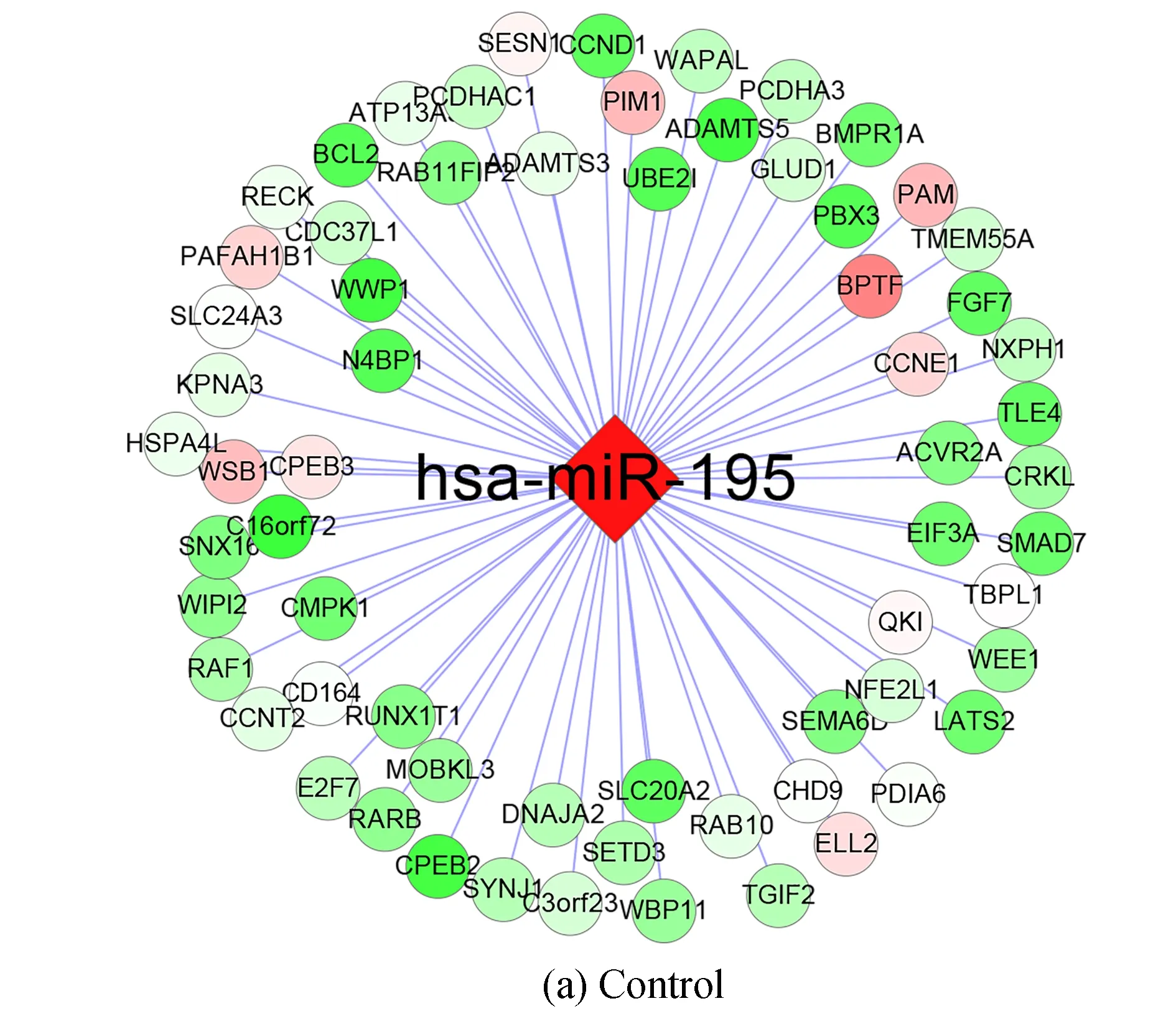
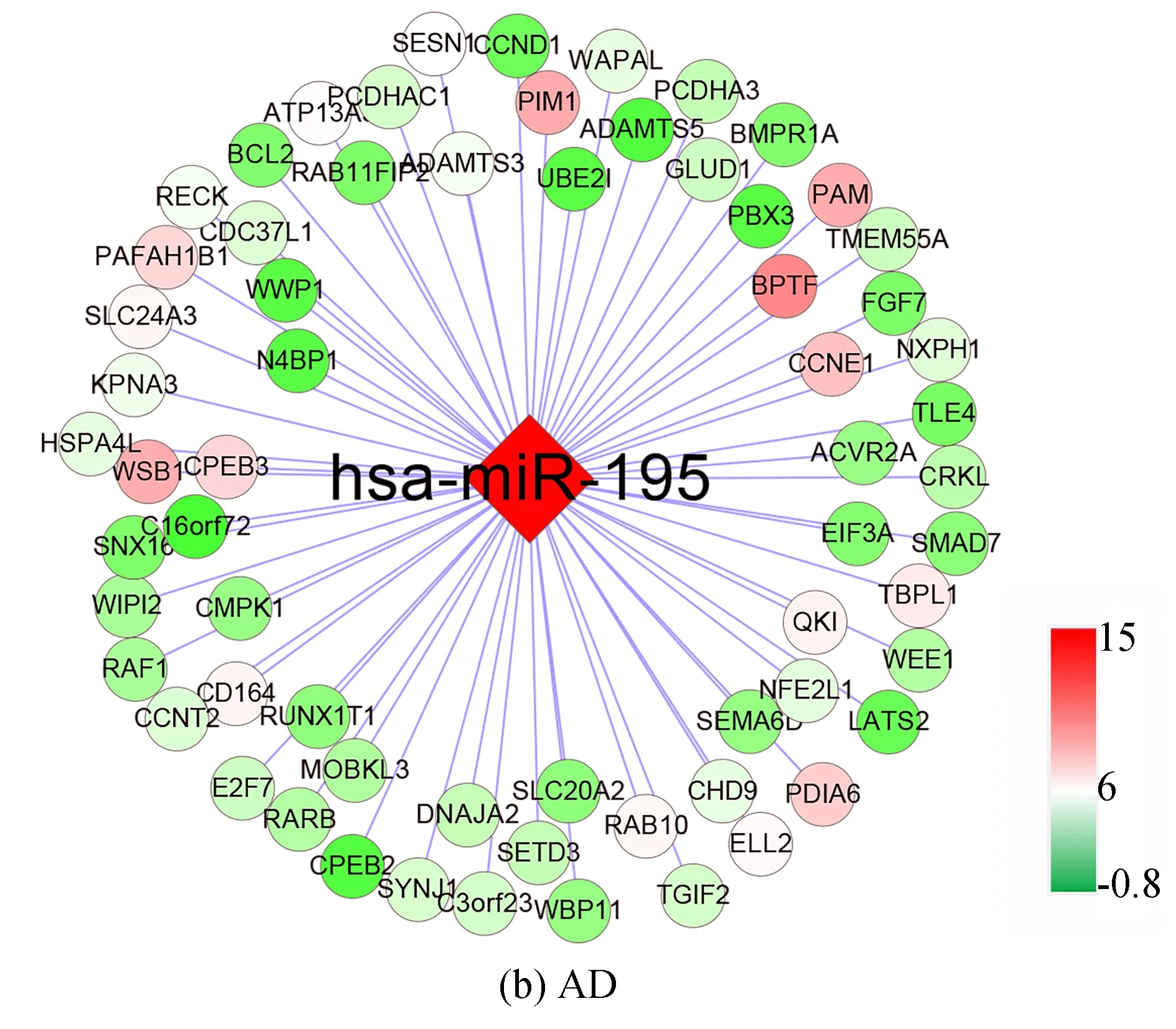
图2 miR-195调控的靶基因
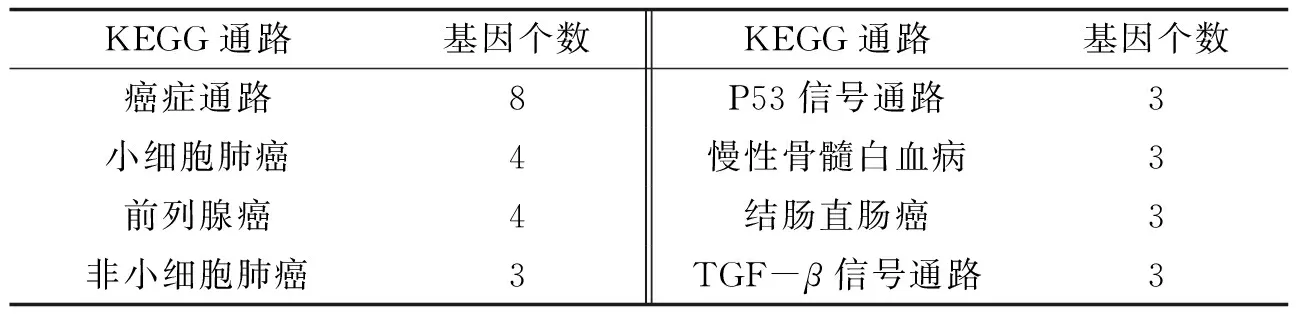
表4 miR-195靶基因KEGG通路分析结果
在此基础上, 对预测靶基因进行了生物过程、 分子功能分析. 结果如表5~6.

表5 GO注释—生物过程(BP)注释
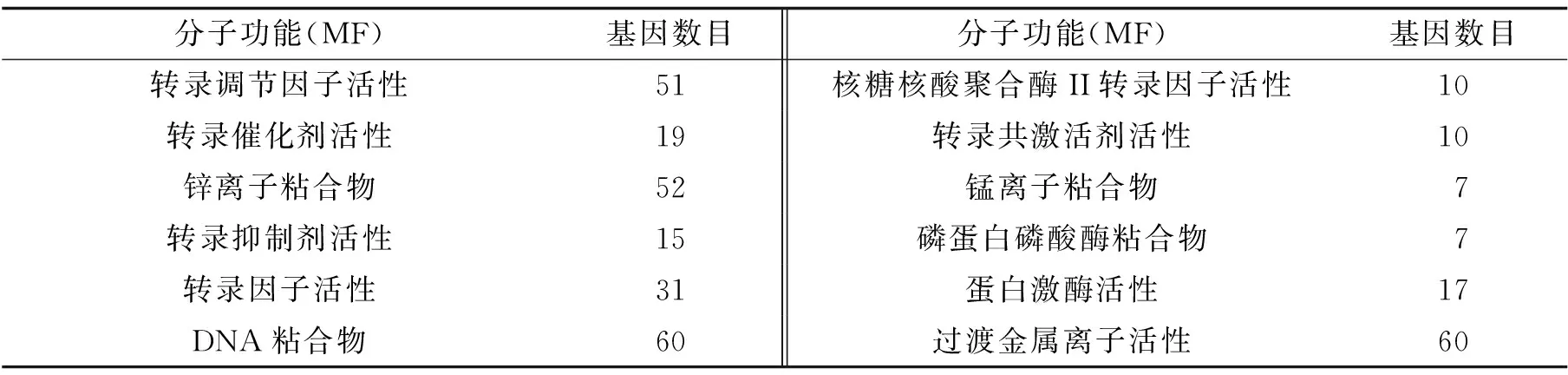
表6 GO注释—分子功能(MF)注释
从上述的生物过程、 分子功能和细胞组成分析可知, 参与生物过程靶基因数目最多的是: 管理细胞增殖、 正调控细胞生物合成过程、 酶链接受体蛋白信号通路、 碱基、 核苷、 核苷酸和核酸代谢过程的正调控等. 说明这些生物过程在AD发生发展过程中都扮演了重要的角色. 主要的分子功能为DNA粘合物、 过渡金属离子活性、 锌离子粘合物、 转录调节因子活性、 转录因子活性等. 这些基因参与的金属离子活性和转录活性等分子功能, 都与AD产生密切相关.
3 结语
采用TargetScan、 miRanda和Pictar三种软件预测miRNA的靶基因, 并采用HCTarget算法对预测的靶基因进行检验, 融合匹配的miRNA表达数据和基因表达数据, 构建miRNA-mRNA调控网络, 分析miRNA和靶基因在AD中的表达活性. 同时利用此算法对AD显著基因进行对应miRNA的预测. 将参与AD的显著基因, 利用预测算法得到调控它们的miRNA, 再结合基因表达数据分析, 从而得到与AD相关的新的miRNAs.
miRNAs通过转录后水平调控细胞蛋白质的表达, 在神经系统的生长发育、 分化及功能执行中发挥重要的作用. 脑组织内miRNA的异常表达可通过多种途径影响阿尔茨海默症的发生和发展. 对于miRNA的研究将有助于深入了解阿尔茨海默症的发病机制. 选取miR-195、 miR-107、 miR-29a等与AD发病密切相关的miRNA, miR-195和miR-107调控的靶基因BACE1通过促进β-淀粉样蛋白生成和调控另一个细胞过程导致记忆丧失而引起AD的发病. 受miR-29a调控的靶基因NAV3影响神经退行性过程, 与AD的发病密切相关. 选取的miRNA贯穿AD的发生发展过程, 为AD的致病机理研究, 临床诊断提供新的方法.
[1] 应侠, 吴振, 雷严, 等. 阿尔茨海默病的发病机制及治疗药物研究进展[J]. 中国药房, 2014, 25(33): 3 152-3 155.
[2] 董贤慧, 柴锡庆. 阿尔茨海默病发病机制研究进展[J]. 中国老年学杂志, 2014(20): 148.
[3] Nunez-Iglesias J, Liu C C, Morgan T E,etal. Joint genome-wide profiling of miRNA and mRNA expression in Aizheimer’s disease cortex reveals altered miRNA regulation[J]. The Public Library of Science One, 2010, 5(2): e8898.
[4] Wang W X, Rajeev B W, Stromberg A J,etal. The expression of microRNA miR 107 decreases early in Alzheimer’s disease and may accelerate disease progression through regulation of beta-site amyloid precursor protein-cleaving enzyme l[J]. The Journal of Neuroscience, 2008, 28(5): 1 213-1 223.
[5] Justin L M, Lahiri D K.MieroRNA 101 downregulates Alzheimer’s amyloid-8 precursor protein levels in human cell cultures and is differentially expressed[J]. Biochemical and Biophysical Research Communications, 2011, 404: 889-895.
[6] Sun E, Shi Y. Micrornas: small molecules with big roles in neurodevelopment and diseases[J]. Experimental Neurology, 2015, 268: 46-53.
[7] Su N, Wang Y, Qian M,etal. Predicting microRNA targets by integrating sequence and expression data in cancer[C]// 2011 IEEE International Conference on Systems Biology (ISB). Zhuhai: IEEE, 2011: 219-224.
[8] Tiribuzi R, Crispoltoni L, Porcellati S,etal. miR128 up-regulation correlates with impaired amyloidβ(1-42) degradation in monocytes from patients with sporadic Alzheimer’s disease[J]. Neurobiology of Aging, 2014, 35(2): 345-356.
[9] Long J M, Ray B, Lahiri D K. MicroRNA-339-5p down-regulates protein expression ofβ-site amyloid precursor protein-cleaving enzyme 1 (BACE1) in human primary brain cultures and is reduced in brain tissue specimens of Alzheimer disease subjects[J]. The Journal of Biological Chemistry, 2014, 289(8): 5 184-5 198.
[10] Lee K H, Lin F C, Hsu T I,etal. MicroRNA-296-5p (miR-296-5p) functions as a tumor suppressor in prostate cancer by directly targeting Pin1[J]. Biochimica et Biophysica Acta (BBA)-Molecular Cell Research, 2014, 1 843(9): 2 055-2 066.
[11] Lewis B P, Burge C B, Bartel D P. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets[J]. Cell, 2005, 120(1): 15-20.
[12] Krek A, Grün D, Poy M N,etal. Combinatorial microRNA target predictions[J]. Nature Genetics, 2005, 37(5): 495-500.
[13] Enright A J, John B, Gaul U,etal. MicroRNA targets in Drosophila[J]. Genome Biology, 2003, 5(1): R1.
[14] Tu K, Yu H, Hua Y J,etal. Combinatorial network of primary and secondary microRNA-driven regulatory mechanisms[J]. Nucleic Acids Research, 2009, 37(18): 5 969-5 980.
[15] Nelson P T, Wang W X. MiR-107 is reduced in Alzheimer’s disease brain neocortex: validation study[J]. Journal of Alzheimer’s Disease, 2010, 21(1): 75-79.
[16] Shioya M, Obayashi S, Tabunoki H,etal. Aberrant microRNA expression in the brains of neurodegenerative diseases: miR-29a decreased in Alzheimer disease brains targets neurone navigator 3[J]. Neuropathology and Applied Neurobiology, 2010, 36(4): 320-330.
[17] Rippo M R, Olivieri F, Monsurrò V,etal. MitomiRs in human inflamm-aging: a hypothesis involving miR-181a, miR-34a and miR-146a[J]. Experimental Gerontology, 2014, 56: 154-163.
[18] Gast H, Gordic S, Petrzilka S,etal. Transforming growth factor‐beta inhibits the expression of clock genes[J]. Annals of the New York Academy of Sciences, 2012, 1261(1): 79-87.
[19] Butz H, Rácz K, Hunyady L,etal. Crosstalk between TGF-βsignaling and the microRNA machinery[J]. Trends in Pharmacological Sciences, 2012, 33(7): 382-393.
[20] 石海滨. 阿尔茨海默病与恶性肿瘤相关性的研究进展[J]. 医药前沿, 2014 (2): 117-118.
[21] Zhu H C, Wang L M, Wang M,etal. MicroRNA-195 downregulates Alzheimer’s disease amyloid-βproduction by targeting BACE1[J]. Brain Research Bulletin, 2012, 88(6): 596-601.
(责任编辑: 洪江星)
Target genes prediction for miRNA in Alzheimer’s disease
KONG Wei1, ZHANG Xin1, MOU Xiaoyang2
(1. Information Engineering College, Shanghai Maritime University, Shanghai 201306, China;2. Department of Chemistry and Biochemistry, Rowan University, New Jersey 08028, USA)
The research of miRNAs will help to further understand the pathogenesis of Alzheimer’s disease. In this paper, we use matching gene expression data and the miRNA expression data to estimate and analyze the miRNAs’ target genes. First we estimate the target genes of the chosen differentially expressed miRNAs. Then we use HCTarget algorithm to validate the forecast target genes. At last we built regulation network of miRNAs and its target genes. The regulation network contains 11 identified miRNAs associated with AD and 6 AD disease-causing genes. By analyzing the network we found the miRNAs and its target genes’ active trend in two cases of AD: normal and disease, biological analysis also confirmed their important role in the AD, this provided the basis for the research of the pathogenesis of AD.
mircoRNA; Alzheimer’s disease; target gene; prediction; HCTarget algorithm
2014-10-22
孔薇(1977-), 教授, 主要从事生物信息学方面的研究, weikong@shmtu.edu.cn
国家自然科学基金资助项目(61271446; 61003093)
10.7631/issn.1000-2243.2015.06.0851
1000-2243(2015)06-0851-08
Q343.1
A

