6份狼尾草属菌草的ITS和叶绿体matK序列分析
林雄杰, 范国成, 林冬梅, 林 辉, 胡菡青, 鲁国东, 林占熺
(1.国家菌草工程技术研究中心,福建 福州 350002;2.福建省农业科学院果树研究所,福建 福州 350013)
6份狼尾草属菌草的ITS和叶绿体matK序列分析
林雄杰1,2, 范国成2, 林冬梅1, 林 辉1, 胡菡青2, 鲁国东1, 林占熺1
(1.国家菌草工程技术研究中心,福建 福州 350002;2.福建省农业科学院果树研究所,福建 福州 350013)
对来自福建农林大学、闽清、闽侯和南平等地的6份狼尾草属菌草的ITS片段和叶绿体matK基因序列进行测定,并用ClustalX 2.0和MEGA 4.1软件对其进行比对分析,构建系统发育树.结果表明,6份菌草的ITS序列长度均为585 bp,其中变异位点0-3个,信息位点2个,遗传距离为0-0.089,平均遗传距离为0.022;matK序列长度为880-881 bp,其中变异位点(信息位点)1个,遗传距离为0-0.016,平均遗传距离为0.010.ITS系统树结果显示6份共菌草聚成四类,其中杂交狼尾草和象草(MQ)聚类在第I类的同一分支,巨菌草和象草聚在第Ⅱ类的不同分支,表明其亲缘关系比较近;matK序列系统树结果除了象草(MQ)外其余5份菌草均聚在了同一分支上,表明叶绿体matK序列无法将供试的6份菌草区分开.
ITS序列;matK基因; 菌草; 序列分析
菌草(JUNCAO)是指适合用于栽培食用菌、药用菌且具有综合开发利用价值的草本植物.它主要涵括了单子叶植物纲、禾本科的类芦属(类芦)、甘蔗属(斑茅、甘蔗)、芦苇属(芦苇)、芒属(五节芒、荻)、菅属(菅)、狼尾草属(象草、巨菌草等)、格兰马属(大米草)、雀稗属(宽叶雀稗)、香根草属(香根草)、高梁属(拟高粱)、玉蜀黍属(玉米)、香茅属(香茅)、芦竹属(芦竹),双子叶植物纲、豆科的苜蓿属(紫苜蓿),菊科(串叶草)和蕨类植物门、里白科的芒萁属(芒萁)等共计20多个属的植物[1].菌草的开发利用不仅解决了食药用菌生产与林业生态平衡之间的“菌林矛盾”及菌业生产与畜牧业生产之间争饲料粮的“菌粮矛盾”,还有效的解决了农民就业和增收问题.同时,高产、优质、光合作用效率高的菌草(如巨菌草等)还可作为能源原料用于发电及作为乙醇、纤维板、纸浆等的工业原料.菌草因其根量大,网络土壤效果好,可以有效固定土壤养分和提高土地蓄水能力,用于水土保持、防风固沙环境修复.
狼尾草属(Pennisetum)菌草多数为热带、亚热带和温带地区的一年生或多年生禾本科植物,多数原产于非洲,其蛋白质含量高,可用作香菇、木耳、灵芝、竹荪等多种食药用菌的培养料.狼尾草属菌草的形态特征均比较相似,随着菌草技术在全国范围内不断推广,各地农民的广泛种植,目前品种比较混乱.由于人们对狼尾草属菌草的种质背景比较模糊,难以区分种植的品种.随着分子生物学技术的日益完善,Hebert et al在2003年提出DNA条码(DNA barcoding)技术,它是指用标准的、有足够变异的、易扩增且相对较短的DNA片段(DNA barcode)自身在物种内的特异性和种间的多样性而创建的一种新的生物身份识别系统,它可以对物种进行快速的自动鉴定.由于其具有操作简便、快速和准确性高等特点,现已成为生物分类学研究中的研究热点和新方向[3].线粒体内的细胞色素C氧化酶Ⅰ(cytochrome c oxidase Ⅰ, CO Ⅰ)基因已广泛应用于许多鸟类、昆虫[5]和鱼类等动物分类和鉴定,已证明是一种较理想的DNA条形码.但在植物中由于线粒体基因进化速度较慢、基因片段变异较小等原因,CO Ⅰ不适合用作条形码片段[7].学者们尝试着从叶绿体基因组和核基因组中寻找理想的DNA条形码.Kress et al[10]通过对整个被子植物类群取样研究,从9个叶绿体基因间隔区(trnC-ycf6、psbM-trnD、trnK-rps16、trnH-psbA、ycf6-psbM、rp136-rps8、atpB-rbcL、trnv-atpE和trnL-F)和叶绿体基因rbcL及核糖体DNA片段ITS中得出ITS和trnH-psbA是较好的选择,并建议采用多基因片段的组合对植物类群的条形码进行鉴定.Newmaste et al在对肉豆蔻科的毛楠属(Compsoneura)研究中,从7个叶绿体DNA片段(accD、UPA、rpoB、matK、rpoC1、rbcL和trnH-psbA)中筛选出较理想的matK和trnH-psbA组合.第三届DNA条形码国际学术大会上,专家们一致建议除rbcL和matK之外,加快进化速率较快的ITS和trnH-psbA在陆地植物中的通用性、分辨率和适合程度研究[7].
为了明确菌草研究所选育的若干优质高产的狼尾属菌草的分类地位,本研究分别对采自福建农林大学和闽侯等地的巨菌草、莱牧1号、象草(Pennisetumpurpureum)等6份狼尾草属菌草的核ITS序列和叶绿体matK基因序列进行测定,并通过序列间遗传距离比较及系统发育分析,从分子手段探讨它们之间的进化关系,从而为狼尾草属菌草的系统发育和遗传多样性研究提供参考.
1 材料与方法
1.1 供试材料
巨菌草(Pennisetumgianteumlin)、莱牧1号(Pennisetumsp .Laimu-1)和象草(P.purpureum)、甜象草(PennisetumpurpureumMH)、象草(P.purpureumMQ)和杂交狼尾草(Pennisetumamericanum×Pennisetumpurpureum)等优质菌草的信息详见表1.样品采集后放置-28 ℃冰箱保存备用.

表1 供试菌草样品信息Table 1 Sample information
1.2 植物基因组DNA的提取
剪取采集好的叶片样品(约0.5 g),放置到冷却的研磨中加入适量液氮迅速研磨成粉末,收集到2 mL离心管中,按植物DNA提取试剂盒(天根生化科技(北京)有限公司)说明提取DNA.
1.3 ITS序列及叶绿体matK基因的扩增及测序
ITS和matK序列的扩增的引物由生工生物工程(上海)股份有限公司合成(表2),反应体系为50 μL,2×TaqMaster Mix 25 μL,10 μmol·L-1的上下游引物各2 μL,模板DNA 2 μL,无菌水19 μL.PCR扩增程序为95 ℃预变性3 min,32个循环[包括94 ℃变性30 s,58 ℃(ITS)或55 ℃(matK)退火30 s,72 ℃延伸1 min],最后72 ℃延伸7 min.PCR产物经1%琼脂凝胶检验纯度和浓度,合格后进行回收纯化,然后送生工生物工程(上海)股份有限公司测序.
1.4 序列比对及遗传距离分析
以测序所得的巨菌草等的ITS序列和matK序列作为参考序列,在NCBI中经BLAST搜索并下载狼尾草属(Pennisetum)的全部ITS序列及matK序列,然后采用ClustalX 2.0软件进行多序列比对,并手工校正,再用Mega 4.1软件分析其遗传距离.

表2 ITS和matK引物信息表Table 2 The primer information of ITS and matK
1.5 系统发育树构建
用Mega 4.1软件的最大简约法(Maximum Parsimony, MP)分别对得到的ITS序列和matK序列构建系统发育树,同时以1000次自举分析(Bootstrap)检测各分支的置信度.
2 结果与分析
2.1 PCR产物检测结果
PCR产物经1%琼脂糖凝胶电泳检测,结果表明,6份菌草的ITS序列和matK序列均获得单一清晰目的条带,无非特异性条带,表明所选择的两引物对6份菌草样品基因组DNA都有很好扩增效果(图1).
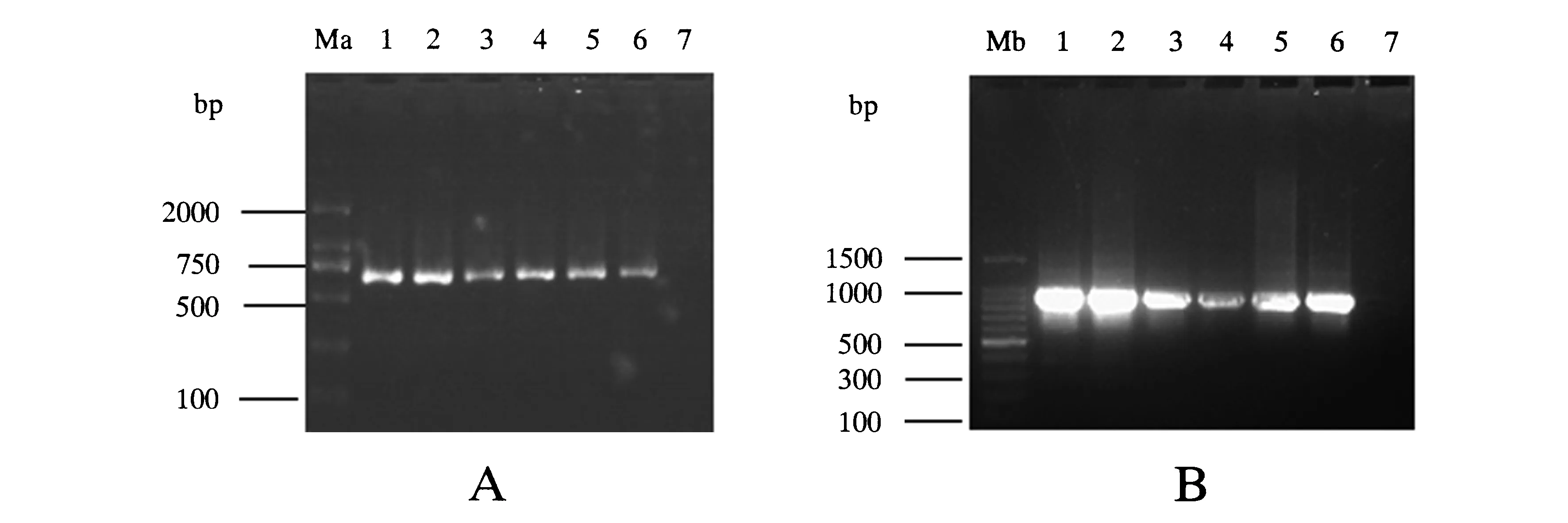
A:ITS序列;B:matk基因序列;Ma:DL2000;Mb:100 bp DNA Ladder;1:巨菌草;2:莱牧1号;3:象草;4:甜象草(MH); 5:象草(MQ);6:杂交狼尾草;7:阴性对照).图1 ITS和matK序列PCR产物电泳检测图谱Fig.1 Electrophoresis of PCR products of ITS and matK sequences
2.2 菌草ITS序列和matK序列分析
将测序所得的6份菌草ITS序列分别去掉18S和26S序列后,序列长度均为585 bp,G/C含量为58.29%-58.63%,变异位点0-3个(表3).用MEGA 4.1软件分析其与NCBI数据库中19条狼尾草属植物ITS序列间的变异情况,结果表明,共有578个保守位点,7个变异位点(2个简约信息位点,5个单突变位点),变异位点比例为1.2%,而信息位点比例达98.8%,说明ITS序列总体变化较小,但具有一定的遗传信息位点,可用作遗传分析.
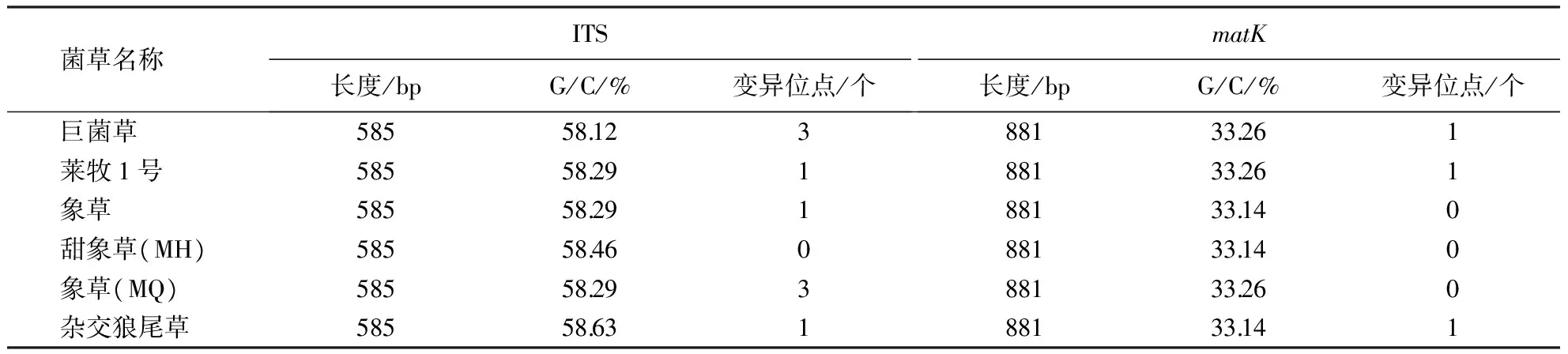
表3 不同菌草ITS和matK序列的长度及G/C含量Table 3 Length and G /C content of ITS and matK sequences of different JUNCAO
对6份菌草matK基因序列的测序结果表明,序列长度均为881 bp,G/C含量为33.14%-33.26%,变异位点0-1个(表3).用MEGA 4.1软件分析6份菌草植物材料与NCBI数据库中10条狼尾草属植物matK序列间的变异情况,结果表明,881个位点中共有880个保守位点,1个变异位点(简约信息位点),变异位点比例为0.1%,可见matK序列非常保守,遗传信息位点较少,不适宜用作狼尾草属植物遗传分析.
2.3 菌草ITS序列和matK序列遗传距离分析
用Mega 4.1软件对25种狼尾草属植物ITS序列的遗传距离分析结果如表4所示,它们间的遗传距离为0-0.089,平均遗传距离为0.022;其中羽绒草Pennisetumsetaceum(AF019833)与象草(N51)(FJ626357)间的遗传距离最大,为0.089;供试的象草(MQ)与细茎杂交狼尾草(FJ626353)和杂交狼尾草(FJ626352)、甜象草(MH)和象草(WK 104)(GQ870179)的遗传距离最小为0(表4).

表4 不同狼尾草属植物ITS序列的种间遗传距离1)Table 4 Genetic distance of ITS sequence among different Pennisetum plants
1)1:P.americanum×P.purpureum闽牧6号(FJ626366);2:P.americanum×P.purpureum皇竹草(FJ626364);3:P.purpureum红象草(FJ626356);4:P.purpureum象草(MQ);5:P.americanum×P.purpureum杂交狼尾草(FJ626352);6:P.americanum×P.purpureum细茎杂交狼尾草(FJ626353);7:P.purpureum象草N51(FJ626357);8:P.americanum×P.purpureum桂牧1号(FJ626362);9:P.americanum×P.purpureum牧草庶(FJ626365);10:P.sp.莱牧1号;11:P.purpureum蒺藜草(GQ870178);12:P.sp. WK104蒺藜草(GQ870179);13:P.purpureum甜象草(MH);14:P.sp.巨菌草;15:P.purpureum紫象草(FJ626354);16:P.americanum×P.purpureum台农2号(FJ626359);17:P.americanum×P.purpureum台湾甜草(FJ626363);18:P.purpureum象草;19:P.americanum×P.purpureum南牧1号(FJ626360);20:P.purpureum(AF345232);21:P.americanum×P.purpureum南牧2号(FJ626361);22:P.americanum×P.purpureum杂交狼尾草;23:P.purpureum象草N85 (FJ626358);24:P.setaceum羽绒草(AF019833);25:P.alopecuroides狼尾草(FJ766172).
对16种狼尾草属植物叶绿体matK序列的遗传距离分析结果如表5所示,它们间的遗传距离为0-0.016,平均遗传距离为0.010;其中以绒毛狼尾草P.villosum(FN908065)与其余序列间的遗传距离最大,为0.012-0.016,本研究供试的6份狼尾草属菌草间的遗传距离均为0(表5).
2.4 ITS序列和matK序列系统发育分析
基于ITS序列以黍Panicummiliaceum(JX576677)作为外类群,通过最大简约法构建MP树,得到的系统发育树(图2).由图2可知,25种狼尾草属植物主要聚为6大类,供试的6份菌草分别聚在第Ⅰ、Ⅱ、Ⅲ、Ⅳ类.其中杂交狼尾草和象草(MQ)聚类在第Ⅰ类的同一分支;巨菌草和象草聚在第Ⅱ类的不同分支;甜象草(MH)和莱牧1号分别聚类于第Ⅲ和Ⅳ类.巨菌草和杂交狼尾草(FJ626352)聚类在同一分支上.
基于matK基因序列以狗牙根Cynodondactylon(AF144584)作为外类群,通过最大简约法构建MP树,得到的系统发育树如图3所示.由图3可知,供试的6份菌草除了象草(MQ)外,其余的均聚类在同一分支.

表5 不同狼尾草属植物matK序列的种间遗传距离1)Table 5 Genetic distance matK sequence among different Pennisetum plants
1)1:P.purpureum象草;2:P.sp.莱牧1号;3:C.purpureus蒺藜草(JQ588783);4:P.caffrum狼尾草(HE586086);5:P.setaceum羽绒草(GU135021);6:P.purpureum甜象草(MH);7:C.purpureus象草(GU135069);8:C.purpureus象草(JQ588780);9:C.purpureus象草(JQ588782);10:P.americanum×P.purpureum杂交狼尾草;11:C.purpureus象草(GU134990);12:C.purpureus象草(JQ588781);13:P.villosum羽绒狼尾草(FN908065);14:P.sp.巨菌草;15:P.purpureum象草(MQ);16:C.purpureus象草(JQ588784).
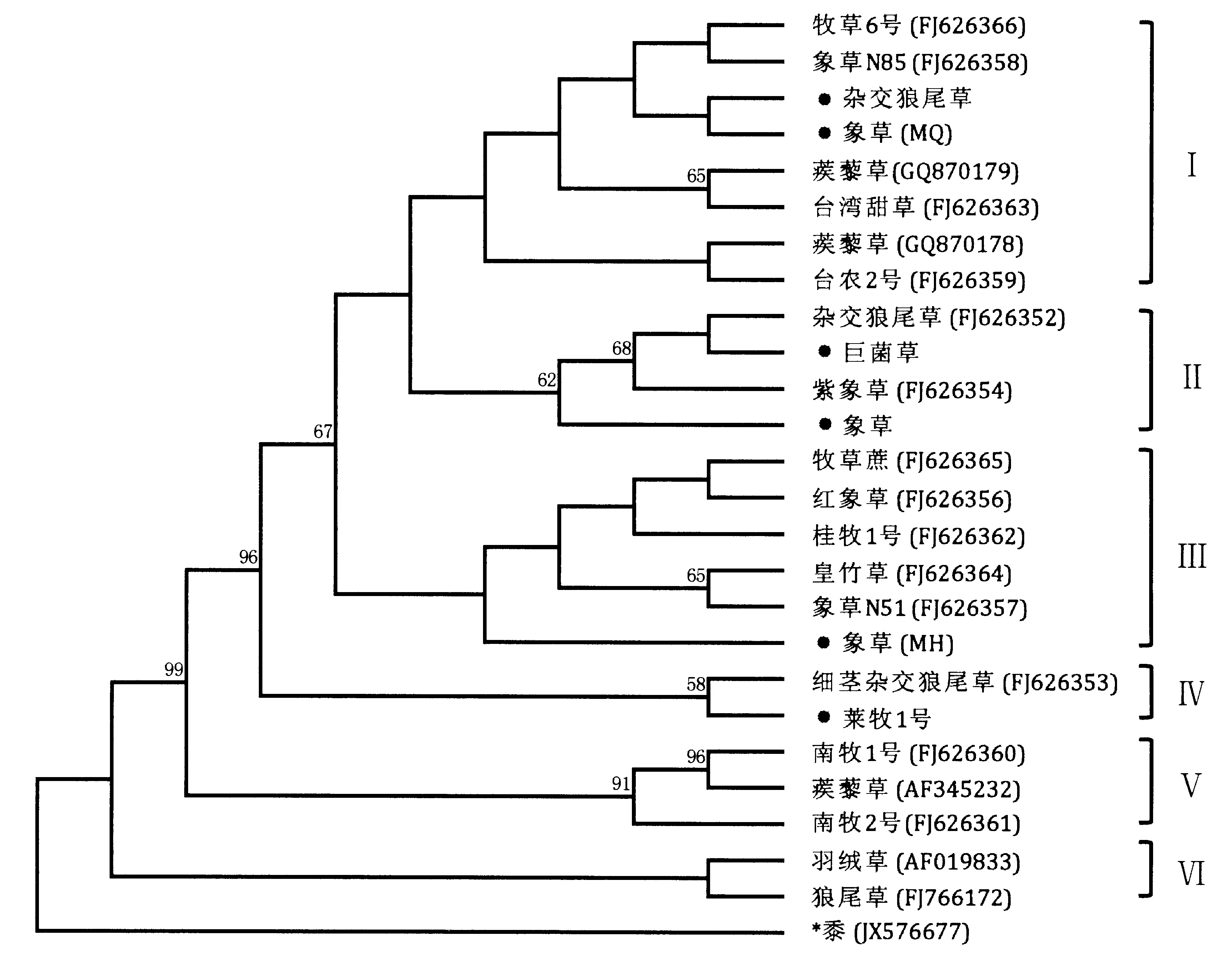
数字表示各分支自展数据支持率,“●” 表示本研究供试植物材料,“*” 表示外类群.图2 最大简约法构建的ITS序列系统发育树Fig.2 ITS sequences phylogenetic tree using the maximum parsimony method
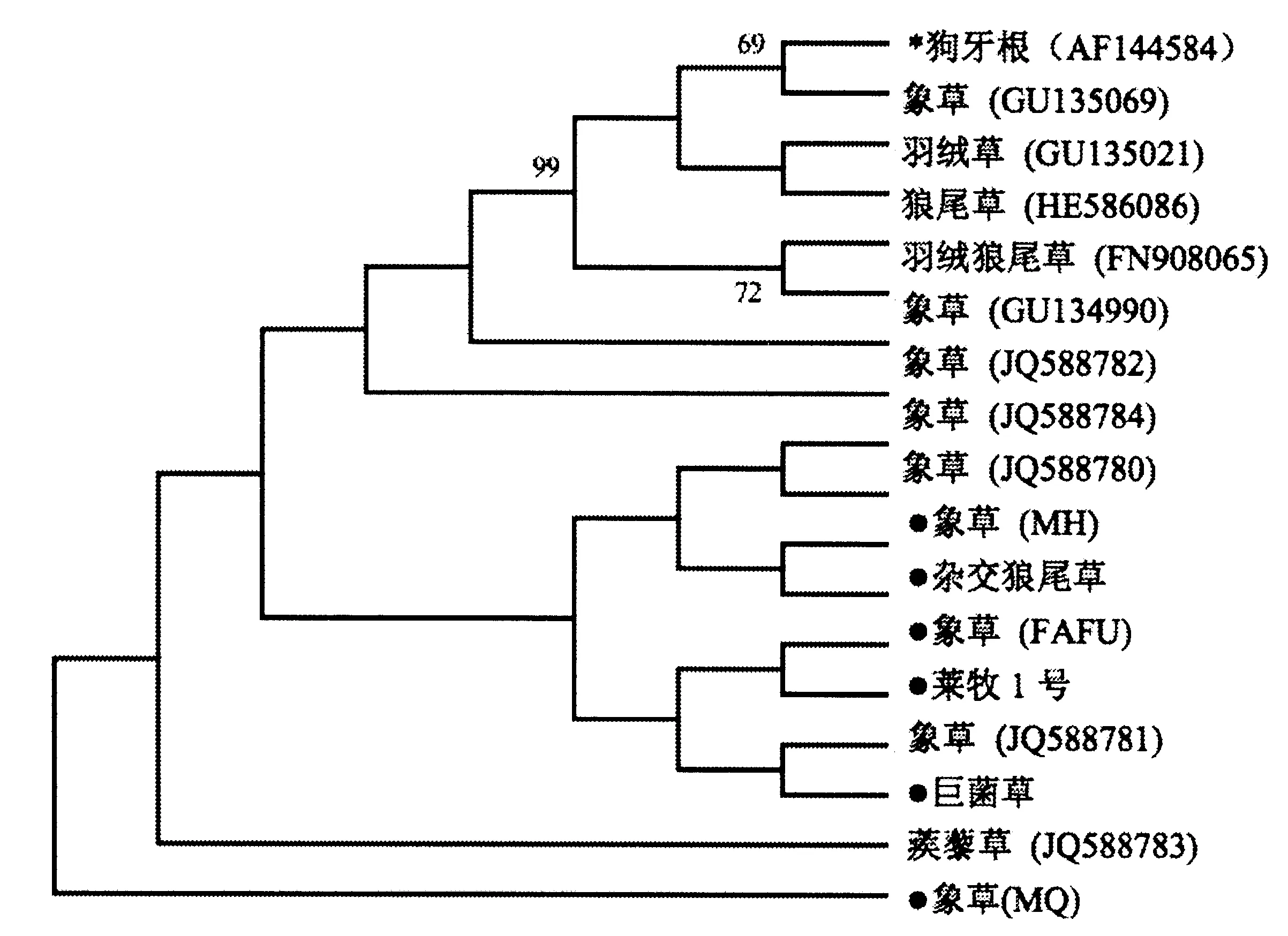
数字表示各分支自展数据支持率,“●” 表示本研究供试植物材料,“*” 表示外类群.图3 最大简约法构建的matK基因系统发育树Fig.3 The matK gene phylogenetic tree using the maximum parsimony method
3 讨论
随着现代分子生物学技术的发展,DNA测序的得到了蓬勃发展,已成为比较分子数据研究中的一个主要手段,它为解决物种分类学、形成与进化及系统发育等领域的研究提供了有力的技术途径.在高等植物中,核核糖体DNA(nrDNA)的编码区序列是高度保守的,序列间的差异主要表现在非编码区上(如ITS、ETS及IGS等).nrDNA的内转录间隔区ITS(Internal transcribed spacer)由ITS1、ITS2和5.8S共3部分组成,在被子植物中,由于ITS1和ITS2非编码区受外界环境因素的影响较小,承受的选择压力较小,因而进化速度较快,核苷酸序列变异较大,且主要是以相互独立的点突变为主,可为属以下水平的研究提供较丰富的信息位点和变异位点等有效信息[16].ITS区的这种核苷酸序列的高度变异但序列长度高度保守的特性,可较好的用于识别那些近缘类群的间隔区的序列,可有效解决较低的分类阶元上(如属间、种间)的植物系统发育问题.
matK基因是叶绿体基因组蛋白编码基因中进化速率最快的基因之一,也是最早被用于植物系统发育学研究的基因序列之一.目前,matk序列分析已成功为科、属级水平类群内部的系统重建提供了大量的信息并获得较高的支持率.同时matK序列还成功应用于某些类群的种间、种内的系统进化研究[19-21].
本研究通过测定6份狼尾草属菌草ITS序列和叶绿体基因matK序列的,并与NCBI数据库中已有的序列进行比对分析,结果表明,ITS序列可以较好的区分这6份菌草,而matK序列无法将其区分开.根据构建ITS序列的系统发育树可知,与巨菌草亲缘关系最近的是杂交狼尾草(FJ626352),它们的ITS序列仅在第216位上存在1个碱基差异,这与序列测定结果是相符的.巨菌草与陈志彤等研究的紫象草(FJ626354)在同一分支,表明它们的亲缘关系较近,推测可能是它们的近缘种或亚种.供试的莱牧1号与象草(MH)聚在同一分支,虽然它们的地域差别很大,但这并不影响它们间的亲缘关系,它们间的ITS序列仅在第564位上存在1个碱基差异.6份菌草在matK序列构建的系统发育树上除了象草(MQ)外,其余5份均聚在一起,这与之前的遗传距离分析相吻合,推测可能是狼尾草属植物matK序列变异太少,不适合用于种间和种内分析.由于狼尾草属植物基本的染色体数目,基因组大小或倍性水平多样性广泛存在,关于基因组结构进化趋势的仍有一些疑问尚待解决[22].因而对于狼尾草属植物种属间亲缘进化关系上的判定,需要综合应用多种有效手段,如采用多种DNA条形码相结合等进行分析,从而使试验结果更加准确、合理.本研究为进一步明确供试6份菌草材料的分类地位提供依据.
[1] 林占熺.菌草学[M].3版.北京:国家行政学院,2013.
[2] HEBERT P N, RATNASINGHAM S, DE WAARD J R. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings of the Royal Society of London. Series B: Biological Sciences, 2003,270(S):96-99.
[3] STOECKLE M, WAGGONER P, AUSUBEL J. Identifying species by DNA barcoding life: ten reasons[J]. Consortium for the Barcode of Life, 2004:1-2.
[4] TAVARES E S, BAKER A J. Single mitochondrial gene barcodes reliably identify sister-species in diverse clades of birds[J]. BMC Evolutionary Biology, 2008,8(1):81-94.
[5] BARRETT R H, HEBERT P N. Identifying spiders through DNA barcodes[J]. Canadian Journal of Zoology, 2005,83(3):481-491.
[6] HUBERT N, HANNER R, HOLM E, et al. Identifying Canadian freshwater fishes through DNA barcodes[J]. PLos One, 2008,3(6): e2490. doi: 10.1371/journal.pone.0002490.
[7] 任保青,陈之端.植物DNA条形码技术[J].植物学报,2010,45(1):1-12.
[8] CHASE M W, SALAMIN N, WILKINSON M, et al. Land plants and DNA barcodes: short-term and long-term goals[J]. Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences, 2005,360(1462):1889-1895.
[9] CHO Y, MOWER J P, QIU Y L, et al. Mitochondrial substitution rates are extraordinarily elevated and variable in a genus of flowering plants[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004,101(51):17741-17746.
[10] KRESS W J, WURDACK K J, ZIMMER E A, et al. Use of DNA barcodes to identify flowering plants[J]. PNAS, 2005,102(23):8369-8374.
[11] FAZEKAS A J, BURGESS K S, KESANAKURTI P R, et al. Multiple multilocus DNA barcodes from the plastid genome discriminate plant species equally well[J]. PLoS One, 2008,3(7):e2802.
[12] BALDWIN B G. Phylogenetic utility of the internal transcribed spacers of nuclear ribosomal DNA in plants: An example from the compositae[J]. Molecular Phylogenetics and Evolution, 1992,1(1):3-16.
[13] CUENOUD P, SAVOLAINEN V, CHATROU L W. Molecular phylogenetics of Caryophyllales based on nuclear 18S rDNA and plastidrbcL,atpB, andmatKDNA sequences[J]. American Journal of Botany, 2002,89:132-144.
[14] 陈志彤,黄勤楼,潘伟彬,等.狼尾草属牧草rDNA的ITS序列分析[J].草业科学,2010,19(4):135-141.
[15] 田欣,李德铢.DNA序列在植物系统学研究中的应用[J].云南植物研究,2002,24(2):170-174.
[16] 王建波,张文驹,陈家宽.核rDNA的ITS序列在被子植物系统与进化研究中的应用[J].植物分类学报,1999,37(4):407-416.
[17] PLUNKETT G, SOLTIS D, SOLTIS P. Clarification of the relationship beteen Apiaceae and Araliaceae based onmatKandrbcLsequence data[J]. Am J BOT, 1997,84(4):565.
[18] KRON K A. Phylogenetic relationships of Rhododeen-droirleae (Errcaceae)[J]. American Journal of Botany, 1997,84:973-980.
[19] 刘静,何涛,淳泽.药用石斛的叶绿体matK基因序列分析及鉴别[J].药学学报,2009(9):1051-1055.
[20] GAO X, ZHU Y P, WU B C, et al. Phylogeny of Diascorea sect.Stenofora based on chloroplast matk,rbcL and trnL-Fsequence[J]. Joural of Systenatics and Evolution, 2008,46(3):315-321.
[21] ZHANG Z Y, LI D Z. Molecular phylogeny of section Parrya of Pinus (Pinaceae) based on chloroplastmatKgene sequence data[J]. ACTA Botanica Sinica, 2004,46(2):171-179.
[22] MARTEL E, PONCET V, LAMY F, et al. Chromosome evolution of Pennisetum species (Poaceae): implications of ITS phylogeny[J]. Plant Systematics and Evolution, 2004,249(3-4):139-149.
(责任编辑:吴显达)
Sequence analysis on ITS and chloroplastmatKgene of sixPennisetumJUNCAO
LIN Xiong-jie1,2, FAN Guo-cheng2, LIN Dong-mei1, LIN Hui1, HU Han-qing2, LU Guo-dong1, LIN Zhan-xi1
(1.National JUNCAO Engineering Technology Research Center, Fuzhou, Fujian, 350002, China; 2.Fruit ResearchInstitute, Fujian Academy of Agricultural Sciences, Fuzhou, Fujian 350013, China)
ITS sequences and chloroplastmatKgene of 6PennisetumJUNCAO which were from Fujian Agriculture and Forestry University, Minqing, Minhou and Nanping were sequenced, compared and analyzed with ClustalX 2.0 and MEGA 4.1 softwares and a phylogenetic tree was constructed. It was shown that the length of ITS sequences was 585 bp, including 0 to 3 variable sites and 2 information sites. The genetic distances were 0 to 0.089, averaging 0.022. The length ofmatKgene in 6 JUNCAO was between 880 bp and 881 bp including 1 variable site (information site). The genetic distance was 0 to 0.016, averaging 0.010. Six JUNCAO were clustered into four groups in ITS phylogenetic tree,Pennisetumgianteumlin andPennisetumpurpureum,Pennisetumsp.Laimu-1 andP.purpureum(MQ) were clustered into the group Ⅰ and group Ⅲ, respectively. It was visible from thematKphylogenetic tree that 5 JUNCAO were clustered into the same branch except theP.purpureum(MQ), which indicated that six JUNCAO could not be distinguished by chloroplastmatKsequence.
ITS sequence;matKgene; JUNCAO; sequence analysis
2014-05-28
2014-08-27
国家菌草工程技术研究中心开放基金项目(JCJJ13001)资助.
林雄杰(1979-),男,博士,助理研究员.研究方向:分子植物病理学.Email:linxj_019@163.com.通讯作者林占熺(1943-),男,研究员,博士生导师.研究方向:菌草学.Email:lzxjuncao@163.com.
Q943.2
A
1671-5470(2015)02-0174-07
10.13323/j.cnki.j.fafu(nat.sci.).2015.02.012

