水稻稻瘟病抗病基因Pi-1和Pi-9双重PCR检测体系的建立
林艳秋, 彭江涛, 官华忠, 侯新坡, 陈志伟, 朱秀凤, 黄荣德, 周元昌
(福建农林大学福建省作物设计育种重点实验室,福建 福州 350002)
水稻稻瘟病抗病基因Pi-1和Pi-9双重PCR检测体系的建立
林艳秋, 彭江涛, 官华忠, 侯新坡, 陈志伟, 朱秀凤, 黄荣德, 周元昌
(福建农林大学福建省作物设计育种重点实验室,福建 福州 350002)
利用分别与抗稻瘟病基因Pi-1和Pi-9紧密连锁的SSR标记MRG4766和SNP标记FP1+FP2+RP组成双重PCR体系,通过设置Mg2+、dNTPs和引物浓度梯度,探索双重PCR体系中各反应物的使用量,由此确定最优双重PCR反应体系.在水稻恢复系R1059与R673杂交的BC3F2群体中随机挑选20个单株对该双重PCR反应体系的效果进行验证.结果表明,20个单株的双重PCR扩增结果与2个单一PCR扩增结果相符,并且条带清晰,说明该双重PCR反应体系在利用分子标记进行基因聚合上可提高选择效率和减少费用.
双重PCR; 反应剂浓度; 扩增效果
Chambercian et al[1]在1988年首次提出多重PCR(mutiplex PCR),即在单一反应体系中同时加入两对或两对以上的特异引物进行扩增多个序列的反应过程.多重PCR具有高效率、高产率、高度特异敏感、减少实验成本、加快试验进程等优点,所以自从它诞生后就得到了飞速发展并广泛应用[2,3].其技术原理与单一PCR相同,主要区别在于多重PCR技术加入两对或两对以上的特异引物对到单一反应体系中,因而可以同时在同一反应管中扩增出两条或两条以上的目的DNA片段.由于各种生物体基因组出现连续20-30个碱基完全相同的可能性极小,因而与单个引物对特异互补的模板是高度保守的,同时DNA聚合酶具有极高的复制准确性,所以由引物交叉结合而产生的非特异性扩增在多重PCR反应体系中出现的可能性很小,保证了多重PCR的特异性和敏感性.因此,理论上只要条件符合,多重PCR中引物对的数量是可以不限的,可同时扩增几条目的DNA片段,从而减少了试验成本和加快试验进程等.
目前,多重PCR在病原体鉴别、性别筛选、连锁分析、法医研究、模板定量和遗传疾病诊断等方面应用较多[4-9].在植物生物学方面,主要应用于检测植物病虫害、植物分子育种和植物种质纯度鉴定等方面[10].但近年来,也有令人鼓舞的应用报道,例如Ma et al[11]利用三重PCR(3个引物对分别对应小麦的3个染色体组)对50个澳大利亚主栽小麦品种进行了高分子量麦谷蛋白亚基分析,结果表明,大多数情况下,高分子质量的麦谷蛋白亚基可被同时鉴定,并发现2个品种的高分子质量的麦谷蛋白亚基与前人测定存在差异.该研究结果证明了采用多重PCR反应体系不仅减少试验费用,而且缩短了时间,加快试验进程.另外,我国的育种家利用多重PCR筛选育种材料也有不少收获.易继财等[12]利用多重PCR对空间搭载诱导水稻种子发生的突变进行分子检测,检测出SJ突变体的缺失,同时发现其大片段的缺失比例与LET及剂量有关.
随着研究的深入,多重PCR在植物生物学的各个领域发挥着重要作用.如植物分子育种、基因表达研究、种质纯度鉴定、病虫害检测等方面,并逐渐成为重要且有效的研究方法.Pi-1与Pi-9均是对稻瘟病具有广谱抗性的显性基因,本研究是将与Pi-1紧密连锁的SSR标记MRG4766和与Pi-9紧密连锁的SNP标记FP1+FP2+RP组成双重PCR(double PCR, DPCR),探索适合的DPCR反应体系,建立准确、高效、快速的分析技术,为分子标记辅助选择育种提供依据.
1 材料与方法
1.1 植物材料
水稻恢复系R1059、R673以及R1059与R673回交的BC3F2群体.
1.2 方法
1.2.1 DNA提取 用SDS小量提取法,提取供试材料幼嫩叶片DNA.提取方法参照段远霖等[13]方法.
1.2.2 引物设计 参考陈志伟等[14]报道的与Pi-1基因紧密连锁的SSR标记MRG4766,其中MRG4766上游引物序列为:5′-ATT GCT GCA AAG TGG GAG AC-3′,下游引物序列为:5′-AAG TGG AGG CAG TTC ACC AC-3′.另外,与Pi-9基因紧密连锁的SNP标记FP1+FP2+RP引物序列分别为:FP1:5′-TCC CTG ATT CCA ACC TGC AGC AAG AG-3′;FP2:5′-CCA ACC TGC ACC AGC CT-3′;RP:5′-CCA TGC TGA TAC TTG GGG CA-3′[15].
1.2.3 多重PCR扩增反应 由于本实验采用两对引物的退火温度均为60 ℃,故以此温度为扩增反应的退火温度.单一引物FP1+FP2+RP的PCR反应体系为15 μL,含Sterile water 6.7 μL、FP1 0.6 μL、FP2 1.2 μL、RP1.2 μL、10×buffer 1.5 μL、Mg2+1.5 μL、dNTPs 0.2 μL、5UTap酶0.1 μL、20 ng·μL-1DNA 2 μL,终浓度分别是FP1为0.16 μmol·L-1、FP2 0.32 μmol·L-1、RP0.32 μmol·L-1、buffer为1×buffer、Mg2+为2.5 mmol·L-1、dNTPs为0.13 mmol·L-1、Tap酶为0.033 U·μL-1、DNA为2.67 ng·μL-1;单一引物MRG4766的PCR反应体系为15 μL,含Sterile water 8.2 μL、MRG4766 1.5 μL、10×buffer 1.5 μL、Mg2+1.5 μL、dNTPs 0.2 μL、5UTap酶0.1 μL、20 ng·μL-1DNA 2 μL,终浓度分别是MRG4766为0.4 μmol·L-1、buffer为1×buffer、Mg2+为2.5 mmol·L-1、dNTPs为0.13 mmol·L-1、Tap酶为0.033 U·μL-1、DNA为2.67 ng·μL-1.通过除了buffer、Tap酶、DNA的浓度不变外,分别设置Mg2+、dNTPs和混合引物FP1+FP2+RP、MRG4766(混合引物间的比例不变)的5个浓度梯度,以其中一个反应物为变量,其余反应物的浓度不变,探索双重PCR扩增反应的最佳反应条件.Mg2+的5个终浓度梯度分别为0.83、1.67、2.50、3.33、4.17 mmol·L-1;dNTPs的5个浓度梯度分别为0.07、0.10、0.13、0.17、0.20 mmol·L-1;混合引物FP1+FP2+RP、MRG4766的5个浓度梯度分别为0.11、0.21、0.21、0.27 μmol·L-1,0.13、0.27、0.27、0.33 μmol·L-1,0.16、0.32、0.32、0.40 μmol·L-1,0.19、0.37、0.37、0.47 μmol·L-1,0.21、0.43、0.43、0.53 μmol·L-1.具体方法见文献[16].
PCR程序:第1步94 ℃预变性4 min;第2步94 ℃变性30 s;第3步60 ℃退火30 s;第4步72 ℃延伸30 s;第2步到第4步循环34次;72 ℃终延伸10 min后4 ℃保存.反应在Eppendorf AG扩增仪上进行.
1.2.4 电泳 通过6%的非变性聚丙烯酰胺凝胶垂直电泳进行分离扩增产物.
1.2.5 银染 电泳结束后,将取出的凝胶放入100 mL质量分数为10%酒精与500 μL冰乙酸的混合溶液中固定5 min,再加入500 μL质量分数为20% AgNO3溶液染色5-8 min,用蒸馏水漂洗2次,最后放入100 mL质量分数为3%的NaOH与500 μL甲醛的混合溶液中显影,观察扩增结果.
2 结果与分析
2.1 Mg2+适宜反应浓度的确定
Mg2+浓度通过影响聚合酶的活性,使PCR的扩增效率产生变化,浓度过高会产生非特异性条带,影响观察效果,浓度过低则会降低PCR扩增产量甚至使扩增失败.在本次试验设置5个Mg2+浓度梯度(0.83、1.67、2.5、3.33、4.17 mmol·L-1),结果表明,Mg2+终浓度为3.33和4.17 mmol·L-1的DPCR反应扩增的条带清晰、效果较好,且无显著差异(图1).从节约实验成本出发,故确定3.33 mmol·L-1浓度为双重PCR的适宜Mg2+反应浓度.
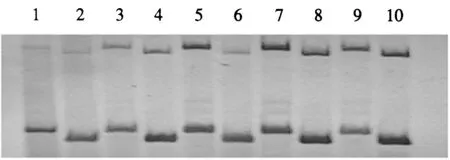
上部分泳带为FP1+FP2+RP扩增的产物,下部分泳带为MRG4766扩增的产物;1、3、5、7、9泳道均为R1059,2、4、6、8、10泳道均为R463; 1、2泳道Mg2+浓度为0.83 mmol·L-1,3、4泳道Mg2+浓度为1.67 mmol·L-1,5、6泳道Mg2+浓度为2.5 mmol·L-1, 7、8泳道Mg2+浓度为3.33 mmol·L-1,9、10泳道Mg2+浓度为4.17 mmol·L-1.图1 不同Mg2+浓度梯度下的DPCR扩增效果Fig.1 DPCR amplification results in different Mg2+ concentrations
2.2 dNTPs适宜反应浓度的确定
dNTPs是PCR的原料,高浓度dNTPs易产生错误掺入,而浓度太低会降低扩增的产量.本试验设置五个dNTPs浓度梯度(0.07、0.10、0.13、0.17、0.20 mmol·L-1),结果表明,dNTPs终浓度为0.17和0.20 mmol·L-1时,DPCR的扩增条带清晰、效果较好,且无明显差异(图2),故确定0.17 mmol·L-1浓度为双重PCR的适宜dNTPs反应浓度.

上部分泳带为FP1+FP2+RP扩增的产物,下部分泳带为MRG4766扩增的产物;1、3、5、7、9泳道均为R1059,2、4、6、8、10泳道均为R463; 1、2泳道dNTPs浓度为0.07 mmol·L-1,3、4泳道dNTPs浓度为0.10 mmol·L-1,5、6泳道dNTPs浓度为0.13 mmol·L-1, 7、8泳道dNTPs浓度为0.17 mmol·L-1,9、10泳道dNTPs浓度为0.20 mmol·L-1.图2 不同dNTPs浓度梯度下的DPCR扩增效果Fig.2 DPCR amplification results in different dNTPs concentrations
2.3 引物适宜反应浓度的确定
引物做为PCR反应的主要成分,其浓度过高易发生错配和产生引物二聚体,过低直接影响扩增产量.本试验设置5个不变混合引物浓度梯度(引物间的比例不变).结果表明,当混合引物FP1、FP2、RP、MRG4766终浓度为0.16、0.32、0.32、0.40 μmol·L-1, 0.19、 0.37、0.37、0.47 μmol·L-1和0.21、0.43、0.43、0.53 μmol·L-1时,其条带清晰、稳定,扩增效果较好,且无明显差异(图3).为了节省材料,因此确定混合引物FP1、FP2、RP、MRG4766适宜引物反应浓度为0.16、0.32、0.32、0.40 μmol·L-1.
2.4 验证反应体系
根据以上试验结果拟确定DPCR反应体系,具体反应体系15 μL,含Sterile water 4.65 μL、FP1 0.6 μL、FP2 1.2 μL、RP1.2 μL、10×buffer 1.5 μL、Mg2+2.0 μL、dNTPs 0.25 μL、5UTap酶 0.1 μL、20 ng·μL-1DNA 2 μL,终浓度分别是FP1为0.16 μmol·L-1、FP2 0.32 μmol·L-1、RP0.32 μmol·L-1、MRG4766 0.4 μmol·L-1、buffer为1×buffer、Mg2+为3.33 mmol·L-1、dNTPs为0.17 mmol·L-1、Tap酶为0.033 U·μL-1、DNA为2.67 ng·μL-1.

上部分泳带为FP1+FP2+RP扩增的产物,下部分泳带为MRG4766扩增的产物;1、3、5、7、9泳道均为R1059,2、4、6、8、10泳道均为R463; 1、2泳道FP1+FP2+RP、MRG4766浓度为0.11、0.21、0.21 、0.27 μmol·L-1,3、4泳道FP1+FP2+RP、MRG4766浓度为0.13、0.27、 0.27、0.33 μmol·L-1,5、6泳道FP1+FP2+RP、MRG4766浓度为0.16、0.32、0.32、0.40 μmol·L-1,7、8泳道FP1+FP2+RP、 MRG4766浓度为0.19、0.37、0.37、0.47 μmol·L-1,9、10泳道FP1+FP2+RP、MRG4766浓度为0.21、0.43、0.43、0.53 μmol·L-1.图3 引物对间比例不变条件下混合引物浓度对DPCR扩增效果的影响Fig.3 Effects of different mixed primers concentrations on DPCR amplification results under the condition of the invariant proportion of primers
在R1059与R673杂交后代BC3F2群体中随机挑选20株单株进行验证,图4为FP1、FP2、RP与MRG4766的双重PCR扩增效果,图5为单一引物FP1+FP2+RP的PCR扩增效果,图6为单一引物MRG4766的PCR扩增效果,双重PCR的检查结果与单一PCR的扩增结果一致,说明可以将拟确定的双重PCR反应体系用于Pi-1和Pi-9基因聚合的标记辅助育种中,以减轻工作量.
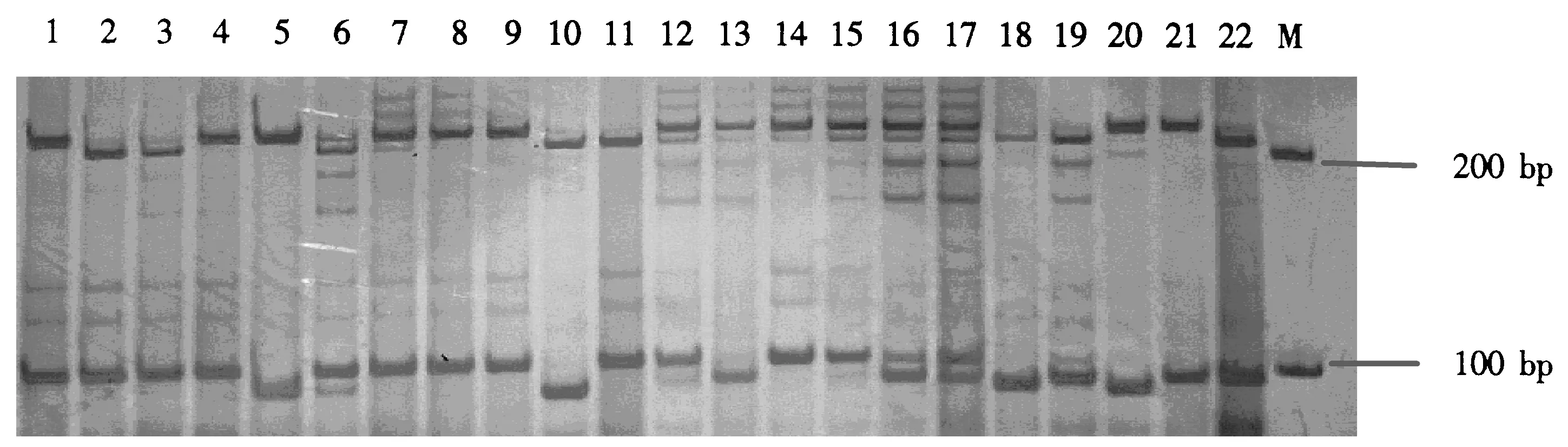
上部分泳带为FP1+FP2+RP扩增的产物,下部分泳带为MRG4766扩增的产物;泳道9为R1059,泳道10为R673, 其余泳道为R1059与R673的BC3F2单株;M:100bp DNA Ladder.图4 BC3F2单株的DPCR扩增效果Fig.4 DPCR amplification results of BC3F2 populations

泳道9为R1059,泳道10为R673,其余泳道为R1059与R673的BC3F2单株;M:100bp DNA Ladder.图5 BC3F2单株的FP1+FP2+RP单一PCR扩增效果Fig.5 FP1+FP2+RP single PCR amplification results of BC3F2 populations

泳道9为R1059,泳道10为R673,其余泳道为R1059与R673的BC3F2单株;M:100 bp DNA Ladder.图6 BC3F2单株的MRG4766单一PCR扩增效果Fig.6 MRG4766 single PCR amplification results of BC3F2 populations
3 讨论
单一PCR扩增方法仅能扩增1个目的基因片段,因此检测不同的目的基因需要重复配置PCR反应液.而多重PCR通过优化单一PCR的反应体系,加入两对或两对以上的特异引物对到同一个反应体系中,便可检测多个目的基因.
为了提高水稻分子标记辅助育种效率,本次研究将与Pi-1紧密连锁的SSR标记MRG4766和与Pi-9紧密连锁的SNP标记FP1+FP2+RP组成双重PCR,设置Mg2+、dNTPs和引物浓度梯度,探索双重PCR体系中各反应物的使用量,并拟确定双重PCR反应体系为15 μL,含4.65 μL Sterile water、1.5 μL 10×buffer、2.0 μL 25 μmol·L-1Mg2+、0.25 μL 10 mmol·L-1dNTPs、1.5 μL 4 μmol·L-1MRG4766、0.6 μL 4 μmol·L-1FP1、1.2 μL 4 μmol·L-1FP2、1.2 μL 4 μmol·L-1RP、0.1 μL 5 UTap酶、2.0 μL 20 ng·μL-1模板DNA,终浓度分别是FP1为0.16 μmol·L-1、FP2 0.32 μmol·L-1、RP0.32 μmol·L-1、MRG4766 0.4 μmol·L-1、buffer为1×buffer、Mg2+为3.33 mmol·L-1、dNTPs为0.17 mmol·L-1、Tap酶为0.033 U·μL-1、DNA为2.67 ng·μL-1.在水稻R1059与R673的杂交后代BC3F2群体中随机挑选20株单株对探索的拟确定反应体系进行验证.结果表明,随机挑选的20株单株的双重PCR扩增结果与两个单一PCR扩增结果相符,并且条带清晰、稳定,说明本次试验探索的双重PCR反应体系可以用于Pi-1和Pi-9基因聚合的标记辅助选择育种中,提高育种选择效率以及减少试验成本.另外,在本次试验发现不同类型的分子标记可以混合进行多重PCR.
[1] CHAMBERCIAN J S, GIBBS R A, RANIER J E, et al. Deletion screening of the Duchenne muscular dystrophy locus via multiplex DNA amplification[J]. Nuclear and Acids Research, 1988(16):1141-1156.
[2] 黄银花,胡晓湘,徐慰倬,等.影响多重PCR扩增效果的因素[J].遗传,2003,25(1):65-68.
[3] 刘正斌,高庆荣,王瑞霞,等.多重PCR技术在植物生物学研究中的应用[J].分子植物育种,2005,3(2):261-268.
[4] BLAIS B W, PHILLIPPE L M, VARY N. Cloth-based hybridization array system for detection of transgenic soy and corn by multiplex polymerase chain reaction[J]. Biotechnology Letters, 2002,24(17):1407-1411.
[5] ARLORIO M, CERETI E, TRAVAGLIA F, et al. Polymerase chain reaction (PCR) of puroindolineb and ribosomal/puroindolineb multiplex PCR for the detection of common wheat (Triticumaestivum) in Italian pasta[J]. European Food Research and Technology, 2003,216(3):253-258.
[6] JAMES D, SCHMIDT A M, WALL E, et al. Reliable detection and identification of genetically modified maize, soybean, and canola by multiplex PCR analysis[J]. J Agric Food Chem, 2003,51(20):5829-5834.
[7] PASTORINO G N, ALCANTARA V M, BALATTI P A. Identification of fast and slow growing rhizobia nodulating soybean (GlycinemaxL., Merr) by a multiplex PCR reaction[J]. FEMS Microbiol Letters, 2003,229(2):153-158.
[8] POLASHOCK J J, VORSA N. Development of SCAR markers for DNA fingerprinting and germplasm analysis of american cranberry[J]. Journal of the American Society for Horticultuiral Science, 2002,127(4):677-684.
[9] LI Y, MUSTAPHA A. Simultaneous detection ofEscherichiacoliO157: H7 andSalmonellaandShigellain apple cider and produce by a multiplex PCR[J]. Journal of Food Protection, 2006,67(1):27-33.
[10] 詹亚光,苏涛,韩梅,等.转基因白桦外源基因的多重PCR快速检测[J].植物研究,2006,26(4):480-485.
[11] MA W, ZHANG W, GALE K. R. Multiplex-PCR typing of high molecular weight glutenin alleles in wheat[J]. Euphytica, 2003,34(1):51-60.
[12] 易继财,庄楚雄,姚涓.空间搭载诱导水稻种子突变的分子标记多态性分析[J].生物物理学报,2002,18(4):478-483.
[13] 段远霖,吴为人,张丹凤,等.水稻幼穗分化受阻突变体lhd的遗传分析与基因定位[J].科学通报,2003,48(15):1662-1665.
[14] 陈志伟,官华忠,吴为人,等.稻瘟病抗性基因Pi-1连锁SSR标记的筛选和应用[J].福建农林大学学报:自然科学版,2005,34(1):74-77.
[15] 蒋云林.利用MAS 技术选育持久抗病杂交水稻亲本的研究[D].福州:福建农林大学,2008.
[16] CHEN D, XEIGLER R S, LEUNG H, et al. Population structure of pyricularia grisea at two screening sites in the Philippines[J]. Phytopatheology, 1995(85):1011-1020.
(责任编辑:吴显达)
Establishment of double PCR detection system for rice blast resistancegenesPi-1 andPi-9
LIN Yan-qiu, PENG Jiang-tao, GUAN Hua-zhong, HOU Xin-po, CHEN Zhi-wei,ZHU Xiu-feng, HUANG Rong-de, ZHOU Yuan-chang
(Fujian Provincial Key Laboratory of Crop Breeding by Design, Fujian Agriculture and Forestry University,Fuzhou, Fujian 350002, China)
For the purpose of improving the efficiency of molecular maker-assisted selection, a double PCR (DPCR) system with a SSR marker MRG4766 closely linked to the rice blast-resistant genePi-1 and a SNP markerFP1+FP2+RPclosely linked to the rice blast-resistant genePi-9 was established by setting the Mg2+, dNTPs and primers concentration gradients. 20 plants derived from rice restorer lines R1059/R673//R1059 BC3F2populations were selected to verify the effect of the DPCR system. The results showed that the stripes by means of the DPCR amplification were clear and were consistent with those by using of two single PCR, respectively. It was indicated that the application of the DPCR system in pyramiding genes could improve breeding selection efficiency and reduce cost.
double PCR; concentration of reactants; amplification result
2014-05-07
2014-08-27
“十二五”农村领域国家科技计划课题(2013BAD01B0107);福建省科技厅科技重点项目(2012N0007);福建省自然科学基金(2012J01091).
林艳秋(1990-),女,硕士.研究方向:水稻遗传育种.Email:linyanqiu116@126.com.通讯作者周元昌(1963-),男,研究方向:水稻遗传育种.Email:zwy_2002@163.com.
S511
A
1671-5470(2015)02-0169-05
10.13323/j.cnki.j.fafu(nat.sci.).2015.02.011

