微生物群落结构与偶氮染料之间对应关系的研究
花 莉, 解井坤, 朱 超, 易维洁
(1.陕西科技大学 资源与环境学院, 陕西 西安 710021; 2.贵州大学 农学院, 贵州 贵阳 550025)
微生物群落结构与偶氮染料之间对应关系的研究
花 莉1, 解井坤1, 朱 超1, 易维洁2
(1.陕西科技大学 资源与环境学院, 陕西 西安 710021; 2.贵州大学 农学院, 贵州 贵阳 550025)
为探究偶氮染料结构与微生物群落结构之间的关系,利用T-RFLP技术对经不同偶氮染料驯化后的微生物群落结构进行了分析.通过分析表明,微生物群落经过偶氮染料驯化后发生了较大演替,而且不同的偶氮染料对应形成的微生物群落结构也不相同.因此,偶氮染料的结构对于微生物群落结构的形成起着重要作用,其经偶氮染料驯化后都能形成与之相对应的以优势种群为主、其它微生物种群为辅的复杂微生物群落结构.采用典范对应分析法(Canonial Correspondence Analysis,CCA)分析了不同驯化处理生境的相似度以及与变量因子的关系,发现脱色率对样本生境分布的贡献率最大,Shannon多样性指数的贡献率最小.这说明微生物群落结构的形成取决于偶氮染料,经过驯化处理更倾向于形成以优势种群为主的特定微生物群落结构,微生物群落多样性在偶氮染料的脱色作用中不是主要的决定因素.
微生物群落; 脱色; 偶氮染料; 微生物燃料电池
0 引言
偶氮染料是一类由偶氮发色基团(-N=N-)联接的芳香类化合物,因其功能多样而被广泛地应用于纺织、制革、塑料制造、印染、造纸、制药和化妆品制造等产业[1,2].由于偶氮染料分子的高毒性和致癌性,其工业源排放对公共健康造成了极大地威胁[3,4].
偶氮键的断裂是偶氮染料分子细菌降解的前提,细菌通过偶氮还原酶来转移电子攻击偶氮键使其断裂[5].由于单一纯种菌株只能对某一种或某一类偶氮染料表现出较好的脱色效果,故对于含有复杂结构的偶氮染料废水而言,其脱色作用并不明显[6].
微生物群落中不同菌株所产生的酶的种类和数量远远高于纯培养菌株,从而使微生物群落脱色偶氮染料的效果较之纯培养菌株具有明显的优势[7].目前,在微生物群落脱色偶氮染料的研究中,有关微生物群落多样性以及微生物群落结构的研究正逐渐成为研究热点[8].
然而,对于偶氮染料与微生物群落结构之间是否存在着对应关系、不同的偶氮染料是否具有各自的特定高效脱色微生物群落等,在这方面的研究目前还鲜有报道.
本研究选用了6种不同的偶氮染料(如表1所示),采用目前分子生物学中较为先进的手段T-RFLP,对经过不同偶氮染料驯化后的脱水污泥中的微生物群落结构进行了分析,并和可分离培养的微生物群落结构进行了对比,为构建具有高效脱色偶氮染料的微生物群落提供了理论参考.
1 材料与方法
1.1 可分离培养的微生物群落
采取序批式间歇补料运行方式,利用脱水污泥对甲基橙、甲基红、金橙I、橙黄G、肼黄、茜黄素R钠盐以及它们的混合染料等进行脱色.待染料脱色完全,将上清液倒掉,加入新的染料驯化.培养液进行新一轮驯化.反应器为模拟的Winogradsky柱,即高为99.0 cm,直径为5.0 cm,厚度为1.0 cm的有机玻璃柱[9].
用无菌移液管吸取10 mL驯化污泥置于90 mL无菌水中(内含玻璃珠),移液管吹洗3次,摇动10 min混合均匀,即为10-1浓度混合液;取10-1浓度的菌液1 mL于9 mL无菌水中,移液管吹洗3次,摇匀,即为10-2浓度菌液;同法依次稀释到10-8.
从稀释倍数为10-6、10-7、10-8等的试管中分别取0.5 mL菌液,涂布于固体分离培养基中,35 ℃恒温培养24 h,选取不同形态的菌落进行计数,通过计数的菌种数以及每一菌种所具有的菌落数获得可分离培养的微生物群落.

表1 选用偶氮染料
1.2 T-RFLP分析方法
(1)DNA提取
利用北京康为世纪生物科技有限公司的土壤DNA试剂盒,对经不同偶氮染料驯化后的脱水污泥进行DNA的提取.
(2)细菌16SrDNA片段的PCR扩增
以样品基因组DNA为模板,采用细菌通用引物27F/1492R进行16SrRNA的基因扩增.
25μL体系为:模版1μL,混合液12.5μL 2×Taq Plus PCR MasterMix,无菌水9.5μL ddH2O,引物 (27F/1492R)各1μL.PCR反应条件为:95 ℃ 5 min、94 ℃ 1 min、60 ℃ 45 s、72 ℃ 1 min,循环25次;72 ℃延伸1 min.所得PCR产物用DNA回收试剂盒进行纯化,然后利用限制性内切酶HhaI在37 ℃孵育3 h,最后将酶切后的样品在65 ℃条件下处理20 min使限制性内切酶失活.
利用ABI基因分析仪3130XL(生物系统,美国),对处理后的样品进行毛细血管凝胶电泳;利用Genemarker(HID V1.7),分析毛细血管电泳后的基因片段的大小、高度、面积等;通过去除引物峰(小于50 bp的T-RFLP)和杂峰(相对丰度小于0.5%的T-RFLP),使得T-RFLP图谱得到修正.
单个T-RFLP的相对峰面积按如下公式计算:
Ap=Ni/N×100
式中:Ni—单个T-RFLP的峰面积;N—总T-RFLP的峰面积.
T-RFLP用MiCA3 PAT分析后移交http://mica.ibest.uidaho.edu/pat.php,进行进一步地分析,再根据所得数据构建微生物群落结构.
2 结果与讨论
2.1 可分离培养的微生物群落的结构
通过驯化后,从7个Winogradsky柱中分离出来9种细菌,并通过DNA序列分析和生理及形态观察对所得细菌进行了命名.每个柱中的细菌结构分布图见图1所示.
PseudomnonasgeniculatestrainKa38(变形菌门) 是甲基橙脱色过程的优势菌,占可分离培养细菌的50%;对于甲基红、金橙I、茜黄素R钠盐而言,Bacilluscereusstrain1FFF (厚壁菌门)是其优势菌,所占比例分别为30.4%、60%、38.63%,其次是KlebsiellavariicolastrainRVEV3 (变形菌门) 和ExiguobacteriumaestuariistrainYS-6 (厚壁菌门);橙黄G、肼黄、混合染料的优势菌为KlebsiellavariicolastrainRVEV3 (变形菌门),所占比例分别为31.82%、37.04%、34.63%,其次是ExiguobacteriumaestuariistrainYS-6 (厚壁菌门)和Bacilluscereusstrain1FFF (厚壁菌门).
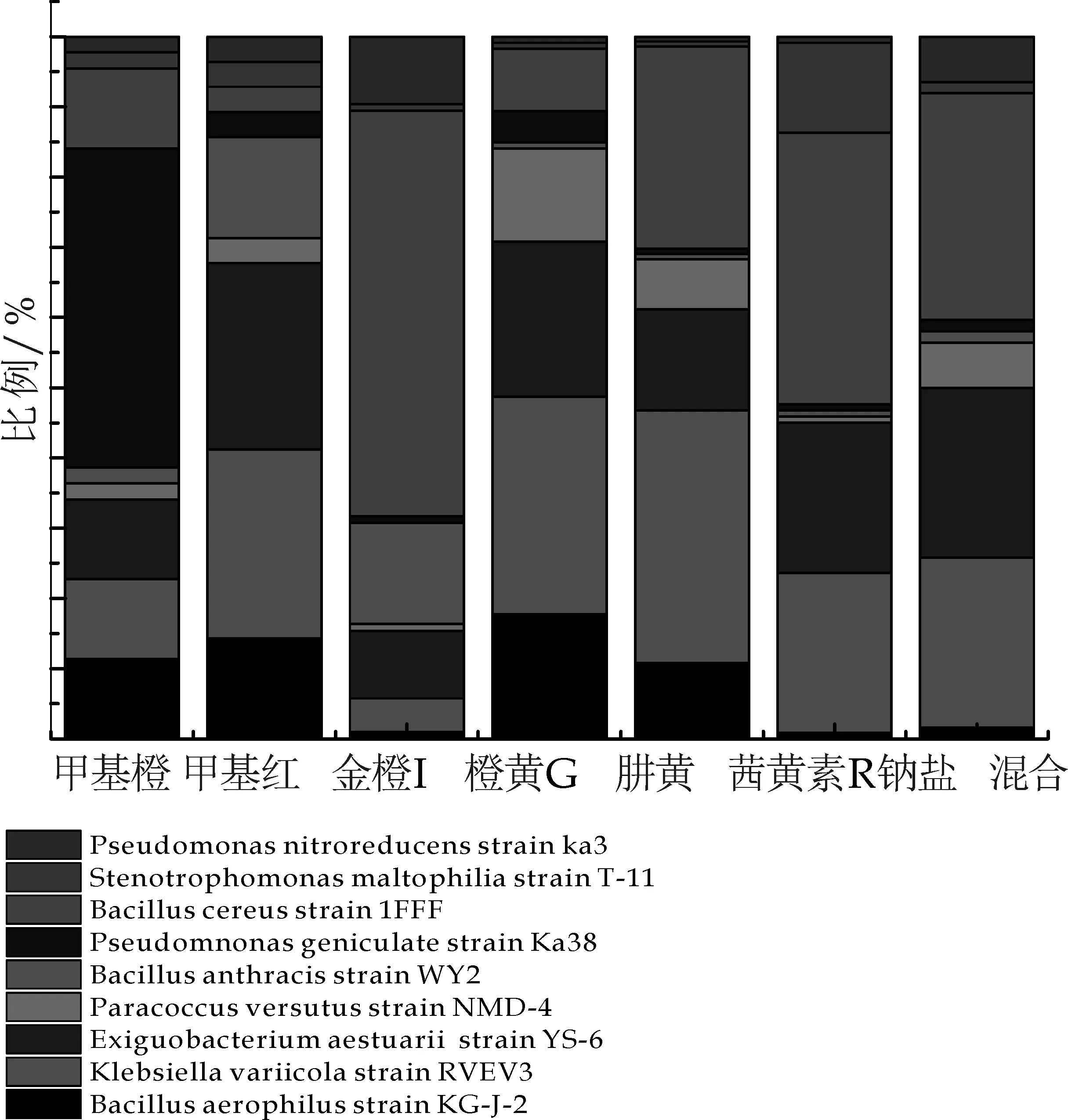
图1 可分离培养的微生物群落结构
2.2 T-RFLP技术分析微生物群落
分别从7个柱子中的驯化污泥中,提取基因组DNA用于T-RFLP分析.T-RFLP分析结果表明,经过驯化,每个柱子中都形成了各自独特的微生物群落结构,如图2所示.
2.2.1 菌门水平
从菌门水平看,原始脱水污泥中含有13种菌门,其中,变形菌门、拟杆菌门、厚壁菌门作为优势菌门,分别占有35.9%,17.7% 和17.6%,不可培养的菌群占有13.2%.经过偶氮染料驯化后,微生物群落发生了明显的演替,不可培养的菌群在所有的柱子中都占有了较大比例.
除了不可培养的菌群之外,在经过甲基橙、甲基红、金橙I、茜黄素R钠盐和混合染料驯化后的微生物群落中,拟杆菌门成为优势菌门,其次为变形菌门和厚壁菌门(混合染料).然而,经橙黄G和肼黄驯化过的菌门分布和其它处理具有明显的不同.橙黄G对应的优势菌门为变形菌门,其次为拟杆菌门;肼黄对应的优势菌门为放线菌门,其次为变形菌门.
从偶氮染料结构方面可知,肼黄和橙黄G具有较为复杂的分子结构,而甲基橙、甲基红、金橙I、茜黄素R钠盐结构较为简单,且拟杆菌门在其驯化的微生物群落中作为优势菌门出现,说明拟杆菌门在结构简单的偶氮染料的脱色过程中起着重要作用.放线菌门和变形菌门则可能具有特异性的脱色肼黄和橙黄G的能力,从而为寻找高效脱色复杂结构的偶氮染料的菌群提供了参考.
由于染料结构的不同,相应地在驯化过程中所形成的菌门结构和丰度也各不相同.例如,甲基橙和甲基红具有相似的分子结构,在经甲基橙和甲基红驯化后的微生物群落具有相似的菌门结构.厚壁菌门同时出现于金橙I和橙黄G的驯化柱中,可归因于在其分子结构中都有萘环的出现.和其它驯化处理相比,在橙黄G、茜黄素R钠盐、肼黄驯化柱中,变形菌门表现出了较大丰度.从三者的结构上看,其具有相似的结构,即分子上都连有两个磺酸基团,说明变形菌门在含有磺酸基团的偶氮染料的脱色过程中起着重要作用.而且,和单一染料的驯化相比,经混合染料驯化后,变形菌门、放线菌门、拟杆菌门等的丰度都在一定程度上有所降低,可能是因为混合染料的毒性明显高于单一染料,使得微生物的活性降低,从而使丰度下降[10].
2.2.2 种属水平
在种属水平,除不可培养的菌群外,原始脱水污泥中的优势菌种为Prevotella(拟杆菌门),占有7.8%的比例,其次为Bacillus(厚壁菌门)占5.1%.经过偶氮染料驯化后的微生物种群结构发生明显变化:对于混合染料,其优势种群为Clostridium(厚壁菌门,8.4%) 和Cytophaga(拟杆菌门,5.5%);对于甲基橙,其优势种群为Flavobacterium(拟杆菌门, 7.9%)、Cytophaga(拟杆菌门,7.0%)、Capnocytophaga(拟杆菌门,6.1%)、Prevotella(拟杆菌门,5.4%);对于甲基红,其优势种群为Flavobacterium(拟杆菌门,10.4%)、Cytophaga(拟杆菌门,6.9%)、Chlorobium(绿菌门,6.8%),Prevotella(拟杆菌门,6.0%);对于金橙I,其优势种群为Prevotella(拟杆菌门,28.9%)、Bacteroides(拟杆菌门,8.5%);对于橙黄G,其优势种群为Neisseria(变形菌门,6.9%)、Marinobacter(变形菌门,5.1%);对于茜黄素R钠盐,其优势种群为Cytophaga(拟杆菌门,7.7%)、Flavobacterium(拟杆菌门,5.1%);对于肼黄,其优势种群为Microbacterium(放线菌门,12.7%).
由图2可知,原始污泥的微生物群落多样性明显高于经过偶氮染料处理的微生物群落.在种属水平,和初始污泥相比,经过驯化处理后的微生物种属发生明显的变化,而且不同的处理所得到的微生物群落结构也有很大差异,这说明在偶氮染料分子结构上的微小变化能够引起微生物群落结构的较大改变[11].
并且在不同偶氮染料的驯化处理中,都能形成各自相应的以丰度较大的优势种群为主的特定微生物群落结构.在所形成的微生物群落中,都有丰度较低种类各异的种属存在,它们有的不参与偶氮染料的脱色,有的则参与偶氮染料的脱色并具有传递电子或产生诱导酶的作用.因此,由于微生物群落中微生物之间的协同作用,使得微生物群落较之纯培养菌株,具有更为高效的脱色性能[12].



内圆为菌门水平分布;外圆为种属水平分布
2.3 典范对应分析(CCA)
采用典范对应分析法(Canonial Correspondence Analysis,CCA)[13],分析了不同驯化处理生境的相似度以及与变量因子的关系.排序中,变量因子包括pH、COD、脱色率(Decolorization Rate)、均匀度(Eveness)和Shannon多样性指数[14](Shannon Diversity Index)等5个.CCA排序采用国际通用软件CANOCO4.5完成.
采用CCA对所研究的原始和7个驯化处理样本,以及5个变量因子等进行了关联分析(如图3所示).箭头表示变量因子;箭头连线的长短表示不同变量因子对不同样本生境相似度分布的贡献大小,即相关性大小;箭头连线与排序轴夹角的大小表示变量因子与排序轴相关性的大小,夹角越小说明关系越密切;箭头所处的象限表示变量因子与排序轴之间的正负相关性[15].
与第一排序轴相关系数较高的是第一次脱色率(1st DCr)、第二次脱色率(2nd DCr)、初始pH(pHi)、末端pH(pHe)、初始COD(CODi)、末端COD(CODe)、Shannon指数,表明在不同的pH条件下微生物对偶氮染料的脱色、COD的去除等表现出了较强的生理生化代谢强度,同时在驯化后微生物的多样性也有所增加.
与第二排序轴相关性较高的是均匀度(eveness),说明第二排序轴自下而上反映了不同处理样品所形成的均匀度增加.较之第二排序轴,第一排序轴对样本生境的影响较大.同时,由箭头的长短可知,对样本生境相似度分布贡献较大的是脱色率和初始pH,其次是末端pH(pHe)、初始COD(CODi)、末端COD(CODe)和均匀度(eveness),而Shannon多样性指数的贡献率则最小,说明经过驯化处理后的样品可能更倾向于形成具有特定优势种群的微生物群落结构[16].

DCr=脱色率,pHi=初始pH,pHe=末端pH,CODi=初始COD,CODe=末端COD,Shannon=Shannon多样性指数,CK=初始脱水污泥,D1-D7是经不同偶氮染料驯化后的生境

图4 种群分布和经不同偶氮染料驯化后的生境之间CCA分析的双轴图
图4表明了微生物群落分布和样本生境的关系.微生物被分成5个组群,分别命名为组1、组2、组3、组4、组5等.其中,组群2反映了空白样本生境的微生物群落组成;组群1处在多种偶氮染料驯化的样本生境中具有最大的物种多样性和种群密度,说明其对于偶氮染料的存在具有较好的适应能力,可能具有高效脱色偶氮染料的能力,这为寻找高效脱色偶氮染料的功能菌群提供了参考;组群3作为一个特殊的微生物群落,处在肼黄驯化处理的样本生境中,和组1、组2表现出了较大的差异性,说明经肼黄的驯化改变了样本的生境,使得其中的微生物向着适应生境的方向演替,形成了具有肼黄特异性适应性和脱色效能的微生物群落;另外,组群4和组群5可视为过渡性微生物群落,在微生物群落的演替过程中起着过渡性作用.
微生物群落的演替路线可能为:组2—组4—组1,组1—组5—组3.
3 结论
本研究采用分离培养技术和T-RFLP技术对偶氮染料驯化后的微生物群落进行了分析.通过分离培养,得到了2个菌门的9种菌种,其数量和种类都远远低于T-RFLP技术.然而,自然界中的多数微生物不能被分离培养[17],而且由于富集和分离的方法具有很强的选择性,使得经过培养的微生物不能真实地反映出自然状态下的微生物群落[18].
经过偶氮染料驯化后的微生物群落发生了较大演替,和原始污泥相比,经过驯化后的污泥中微生物群落的多样性在一定程度上有所下降,然而脱色性能却有所增强.在驯化过程中,不可培养的微生物菌群快速增加,逐渐成为微生物群落中的优势菌种,可能是由于偶氮染料作为难降解的外源化合物改变了微生物生境,使得微生物产生变异从而适应新的生存环境而具有脱色偶氮染料的能力.
从CAA分析可知,脱色率对样本生境分布的贡献率最大,从而也反应了偶氮染料对微生物群落结构的影响;而Shannon多样性指数的贡献率较小,说明经过驯化处理更倾向于形成以优势种群为主的特定微生物群落结构,微生物群落多样性在偶氮染料的脱色作用中不是主要因素.
由微生物种群分布于微生物生境的分布图(如图4所示)可知,组群1具有最大的种群密度和丰度,其集中分布于多种偶氮染料的生境中,其在偶氮染料的脱色过程中可能起着重要作用;经过肼黄处理后的生境与其它生境相比,表现出了较大差异,经过特异性的选择作用形成了具有脱色肼黄能力的微生物种群组群3.
[1] Blumel S.,Busse H.J.,Stolz A.,et al.Xenophilus azovoransgen.nov.,sp.nov.,a soil bacterium able to degrade azo dyes of the Orange II type[J].Int J Syst Evol Bacteriol,2001,51(5):1 831-1 837.
[2] Suzuki Y.,Yoda T.,Ruhul A.,et al.Molecular cloning and characterization of the gene coding for azoreductase from Bacillus sp.OY1-2 isolated from soil[J].J Biol Chem,2001,276(12):9 059-9 065.
[3] Bisschops I.,Spanjers H.Literature review on textile wastewater charactreisation[J].Environ Technol,2003,24(11):1 399-1 411.
[4] Weisburger J.H.Comments on the history and importance of aromatic and heterocyclic amines in public health[J].Mutat Res,2002,507:9-20.
[5] 朱 超,解井坤,花 莉,等.产自Bacillus sp.的热稳定偶氮还原酶结构模型研究[J].陕西科技大学学报(自然科学版),2014,32(6):33-38.
[6] Saratale R.G.,Saratale G.D.,Kalyani D.C.,et al.Enhanced decolorization and biodegradation of textile azo dye Scarlet R by using developed microbial consortium-GR[J].Bioresour.Technol.,2009,100(9):2 493-2 500.
[7] Saratale R.G.,Saratale G.D.,Chang J.S.,et al.Bactreial decolorizaiton and degradation azo dyes:a review[J].J.Taiwan Inst.Chem.Eng.,2011,42(1):138-157.
[8] Myrna S.,Aida S.,Hermminial I.P.,et al.Microbial decolouration of azo dyes:A review[J].Process Biochemistry,2012,47(12):1 723-1 748.
[9] 解井坤,朱 超,花 莉.脱水污泥中脱色偶氮染料功能菌群的驯化分离[J].微生物学通报,2014,41(12):2 432-2 441.
[10] 刘志培,杨惠芳.偶氮染料的微生物降解[J].环境污染与防治,1987(2):2-5.
[11] 杜小明, 刘厚田.偶氮染料分子结构特征与其生物降解性的关系[J].环境化学,1991,10(6):12-18.
[12] Liu W.T.,Marsh T.L.,Cheng H.,et al.Characterization of microbial diversity by determining terminal restriction fragment 1ength po1ymorphisms of genes encoding 16SrRNA[J].Applied Environmental Microiology,1997,63(11):4 516-4 522.
[13] TerBraak C.J.F.,Verdonschot P.F.M.Canonical correspondence analysis and related multivariate methods in aquaticecology[J].Aquatic Sciences,1995,57(3):255-289.
[14] 蔡立哲,马 丽,高 阳,等.海洋底栖动物多样性指数污染程度评价标准的分析[J].厦门大学学报,2002,41(5):641-646.
[15] Mohsen B.B.,Salehi M.H.,Jose A.M.C.,et al.Using canonical correspondence analysis (CCA) to identify the most important DEM attribtutes for digital soil mapping applications[J].Catena,2011,86(1):66-74.
[16] 李国芳.偶氮染料降解菌的群落结构分析及其环境降解初步探究[D].哈尔滨:东北林业大学,2012.
[17] Amann R.I,Ludwig W.,Schleifer K.H.Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J].Microbiol.Rev,1995,59(1):143-169.
[18] Tom Petersen A.,Leser T.D.,Marsh T.L.,et al.Effects of copper amendment on the bacterial community in agricultural soil analyzed by the T-RFLP technique[J].FEMS Microb.Ecol,2003,46(1):53-62.
The exploration of the relationship between azo dyesand microbial community structure
HUA Li1, XIE Jing-kun1, ZHU Chao1, YI Wei-jie2
(1.College of Resources and Environment, Shaanxi University of Science & Technology, Xi′an 710021, China; 2.College of Agriculture, Guizhou University, Guiyang 550025, China)
In this study the relationship between azo dyes structure and microbial community structure was explored and the microbial community structure in acclimated sludge of different azo dyes was analyzed using the T-RFLP technology.The analysis showed that the microbial community after domestication had a drastic shift,and different azo dyes corresponded to different microbial community structures.Therefore,the formation of the microbial community was depended on the structures of azo dyes.And a complex microbial community structure including the dominant population with a large abundance and other population with small abundance would form after acclimating by each of azo dyes.Also Canonial Correspondence Analysis (CCA) was used to analyze the relationship between the environment similarity after acclimating and variables.It had found that the decolorization rate had the most contribution rate for the environment distribution and the Shannon diversity index least,which indicated that the formation of the microbial community structure was determined by azo dyes,and the effect of the microbial community diversity may not be notable.
microbial community; decolorization; azo dyes; microbial fuel
2015-03-24
陕西省教育厅专项科研计划项目(2013JK072); 陕西科技大学博士科研启动基金项目(BJ12-29)
花 莉(1978-),女,贵州兴义人,副教授,博士,研究方向:环境生物技术
1000-5811(2015)03-0001-06
X172
A

