SSR分子标记在花生杂种鉴定中的应用
王 辉, KHERA Pawan, 李双铃, 任 艳, 袁 美, 庄伟建, VARSHNEY Rajeev K, 郭宝珠, 谢联辉
(1.福建农林大学植物保护学院,福建 福州 350002; 2.Department of Plant Pathology, University of Georgia,Tifton, GA 31793, USA; 3.International Crops Research Institute for the Semi-Arid Tropics, Patancheru, Andhra Pradesh 502324, India; 4.山东省花生研究所,山东 青岛 266100; 5.福建农林大学作物科学学院,福建福州 350002; 6.Crop Protection and Management Research Unit, USDA-ARS, Tifton, GA 31793, USA)
SSR分子标记在花生杂种鉴定中的应用
王 辉1,2, KHERA Pawan2,3, 李双铃4, 任 艳4, 袁 美4, 庄伟建5, VARSHNEY Rajeev K3, 郭宝珠6, 谢联辉1
(1.福建农林大学植物保护学院,福建 福州 350002; 2.Department of Plant Pathology, University of Georgia,Tifton, GA 31793, USA; 3.International Crops Research Institute for the Semi-Arid Tropics, Patancheru, Andhra Pradesh 502324, India; 4.山东省花生研究所,山东 青岛 266100; 5.福建农林大学作物科学学院,福建福州 350002; 6.Crop Protection and Management Research Unit, USDA-ARS, Tifton, GA 31793, USA)
以24份花生材料为亲本,配制22个杂交组合,并利用40对SSR引物对24份亲本材料进行多态性筛选,运用多态性引物鉴定92个F1代杂种的真伪.结果表明,不同组合亲本间多态性差异较大,差异最大的两亲本(GT-C20-D和TamrunOL07)的多态性引物比例为65%.SSR标记法鉴定出49个单株为真杂种;而田间表型鉴定出62个单株为真杂种,与标记鉴定结果差异较大.本研究证实利用分子标记鉴定真假杂种非常必要,且准确可行.同时,本试验证明40对SSR引物具有较高的多态性,可直接用于花生遗传多样性、杂种鉴定以及遗传图谱构建等的研究.
花生; SSR; 杂种鉴定; 分子标记
花生是世界范围内广泛栽培的重要经济与油料作物,其遗传育种研究一直备受重视[1].花生属严格的自花授粉作物,通过人工操作即可进行杂交育种,但由于诸多不可控因素的影响,其自交产生假杂种的机率很高.及时准确地鉴别杂交F1代的真假杂种,是杂交育种成功的关键.传统真假杂种的鉴定主要通过观察田间的表型性状,但随着花生育成品种间遗传基础的日益狭窄以及杂交亲本间越来越小的形态表型差异,传统方法鉴别杂种的难度增大.因此,发展高效和可靠的真假杂种鉴别方法十分必要.
分子标记辅助育种(marker-assisted selection,MAS),将现代分子生物学技术与传统育种方法相结合,借助分子标记对育种材料从DNA水平上进行选择,从而改良作物产量和品质等性状.同时,随着各种标记体系的建立,分子标记被越来越多地应用于现代作物遗传育种研究的各个方面,并取得了很多成果[2-5].分子标记用于杂种鉴定和纯度估计性研究时具有周期短、不受环境条件影响、真实、准确、可靠等优点.近年来很多研究表明,SSR标记在花生栽培种间有较高的多态性,带型稳定,操作简单,能检测出微小的变异,是目前分析花生栽培种遗传多样性和鉴定花生杂种真伪的理想标记之一[6-9].因此,本研究选择多态性较高的40对SSR引物对花生24份亲本进行多态性筛选,并运用得到的多态性引物鉴定22个杂交组合的F1代杂种的真伪,为花生杂交育种工作提供依据.
1 材料与方法
1.1 供试材料
以24份花生材料为亲本(由美国佐治亚大学提供),设计22个杂交组合,2013年夏季于佐治亚大学的温室中进行人工杂交,获得22个组合的杂交F1代种子(表1).2014年5月将亲本及收获的F1代种子种植于佐治亚大学的实验农场.苗期剪取幼叶, 置于冰箱备用.在收获前期根据亲本和F1单株形态表型判断真假杂种.
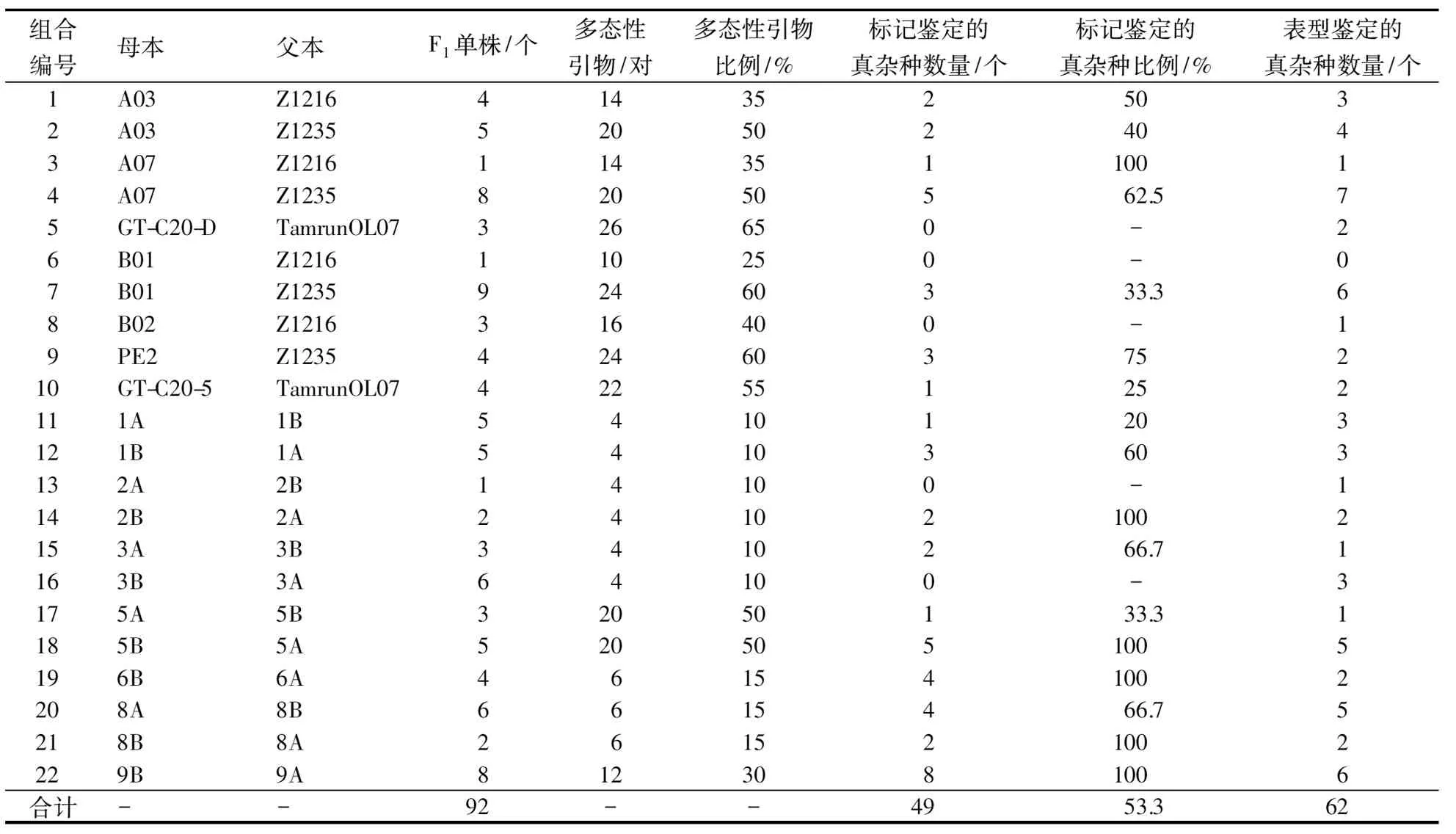
表1 供试材料和亲本间的多态性引物以及杂种的分子标记与表型鉴定结果
1.2 DNA提取与鉴定所用引物
DNA提取采用小量的CTAB法[10],略做改良.通过1%琼脂糖凝胶检测DNA质量,并用 NanoDrop-1000分光光度计(Nano Drop Technologies, USA)测量浓度后,将所有DNA稀释至25 ng·μL-1待用.杂种鉴定中,选用40对多态性高的花生栽培种基因组SSR引物(表2),筛选亲本DNA 多态性.这些引物均来自佐治亚大学实验室,序列设计参照文献[11-17].
1.3 SSR标记分析
PCR反应体系15 μL,包含10×PCR反应缓冲液1.5 μL(含2.5 mmol·L-1MgCl2)、dNTPs(2.5 mmol·L-1)1.2 μL、正向和反向引物(5 μmol·L-1)各1.0 μL、Taq聚合酶(5 U·μL-1)0.2 μL、50 ng DNA.扩增反应在DNA Engine Tetrad 2 Peltier Thermal Cycler(BioRad Laboratories, Hercules, CA, USA)和Eppendorf Mastercycler Pro(Eppendorf North America, USA)PCR仪上完成.SSR 扩增反应程序:95 ℃变性4 min;94 ℃变性45 s,53 ℃退火45 s,72 ℃延伸1 min,35个循环;72 ℃延伸10 min,4 ℃保存.扩增产物采用6.0%非变性聚丙烯酰胺凝胶电泳,电泳槽型号DYCZ-30(六一牌,北京),电泳缓冲液1×TBE,电压140 V,时间2 h,0.1% AgNO3染色,BIORAD凝胶成像系统观察拍照.
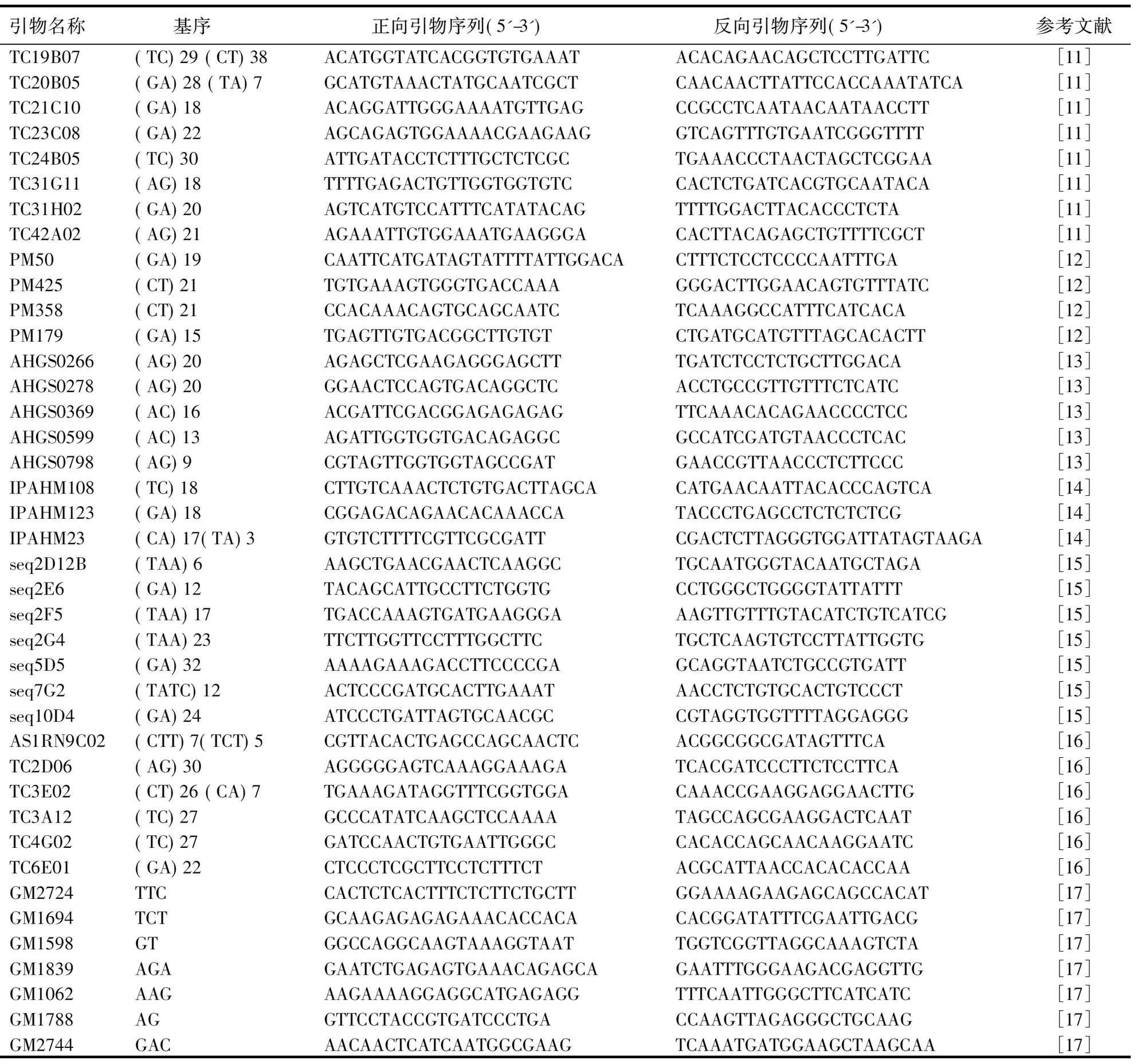
表2 本研究所用40对SSR引物的信息
2 结果与分析
2.1 杂交F1代田间表型鉴定
若父母亲本形态表型差异大,其F1代真假杂种较易鉴别;若父母亲本形态表型高度相似,F1后代虽有杂种优势,但根据形态表型仍无法完全鉴别其真伪.根据田间表型鉴定的结果见表1.
2.2 亲本间的SSR标记多态性
利用40对SSR引物检测杂交亲本的多态性,40对引物均获得扩增产物.每个组合中检测出的多态性引物的数目和比例各不相同(表1).多态性引物数目最多的为“GT-C20-D×TamrunOL07”组合,有26对引物在两亲本中表现多态,多态性比例为65%;多态性比例超过50%的有8个组合,占所有组合的36.4%;仅有4对引物表现多态的组合有6个,占所有组合的27%.由引物IPAHM123和IPAHM23在24份亲本中的扩增图谱(图1)可以看出,前12个亲本在这2个引物中的多态性较好,后12个亲本在这2个引物中的差异较小,与表1结果一致.
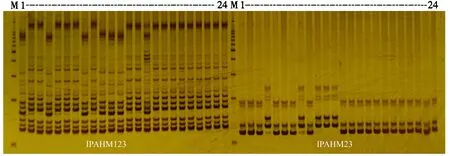
M.Marker (100 bp DNA Ladder, Promega);1-24为24份亲本,依次为B02、Z1216、A03、Z1235、A07、B01、PE2、GT-C20-D、
2.3 真假杂种分子鉴定结果
选择扩增带型好,且表现为共显性的引物进行F1真假杂种的分子鉴定.每个组合至少选用2对引物,以确保鉴定的准确性.以SSR引物扩增F1单株,根据带型鉴别杂种真伪,其中表现父本带型或同时出现父母本带型的为真杂种,仅出现母本带型的为伪杂种.由表1可以看出,共对92个F1单株进行真假杂种鉴定,其中分子标记鉴定的真杂种的单株数为49个,比例为53.3%;表型鉴定的真杂种数为62个.可见,分子标记和表型鉴定的结果存在一定差异,仅有7个组合经2种方法的鉴定结果完全一致.
由图2可以看出,经引物IPAHM123和IPAHM23扩增,F1单株中1、2、5的带型均与母本A07一致,因此可以判定其为假杂种;3、4、6、7、8的带型均是父母亲本的混合带型,可以认定为真杂种.而在图3中,组合19(6B×6A)的4个F1单株在引物AHGS0266中表现为父本带型,在引物AHGS0599中则是父母亲本的混合带型,也可以认定为真杂种.
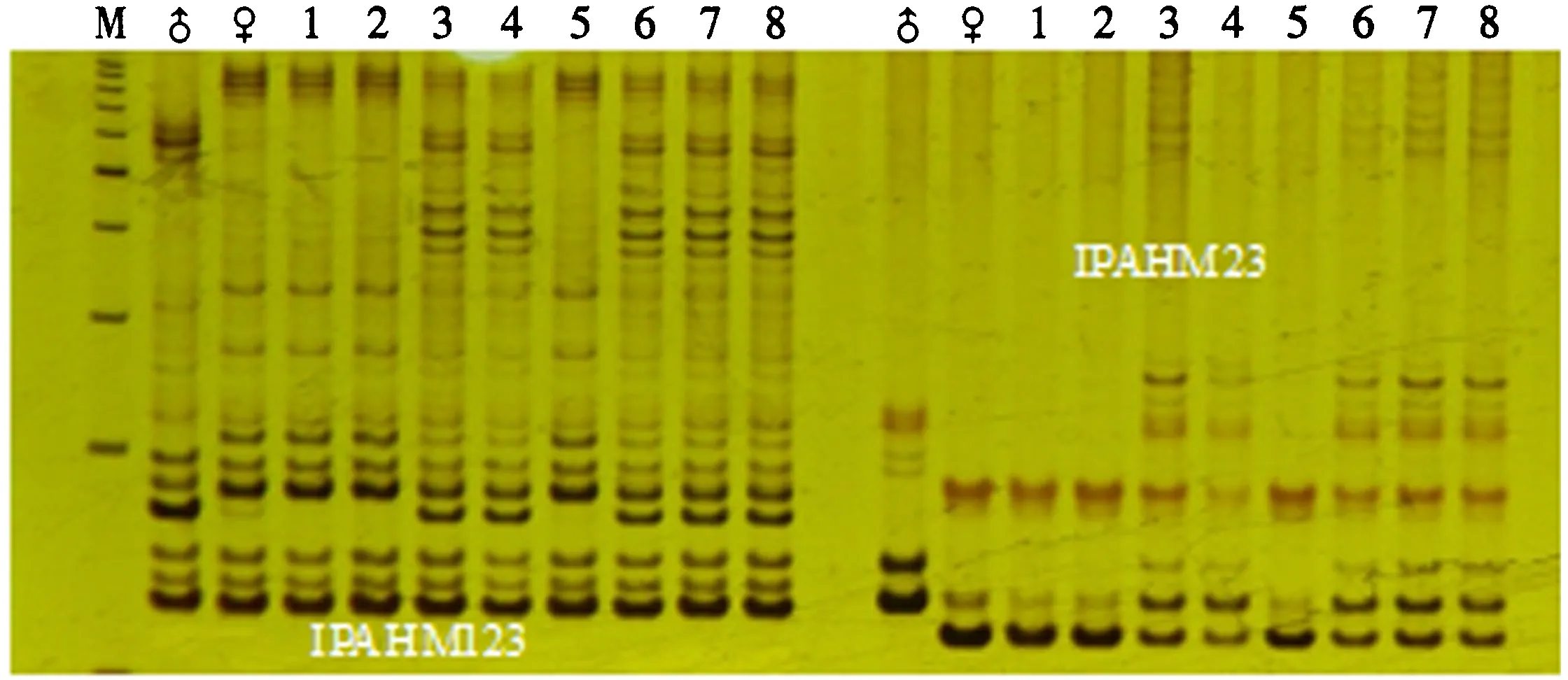
M.Marker (100 bp DNA Ladder, Promega);♂.父本Z1235;♀.母本A07;1、2、5.带型与母本一致,为假杂种;
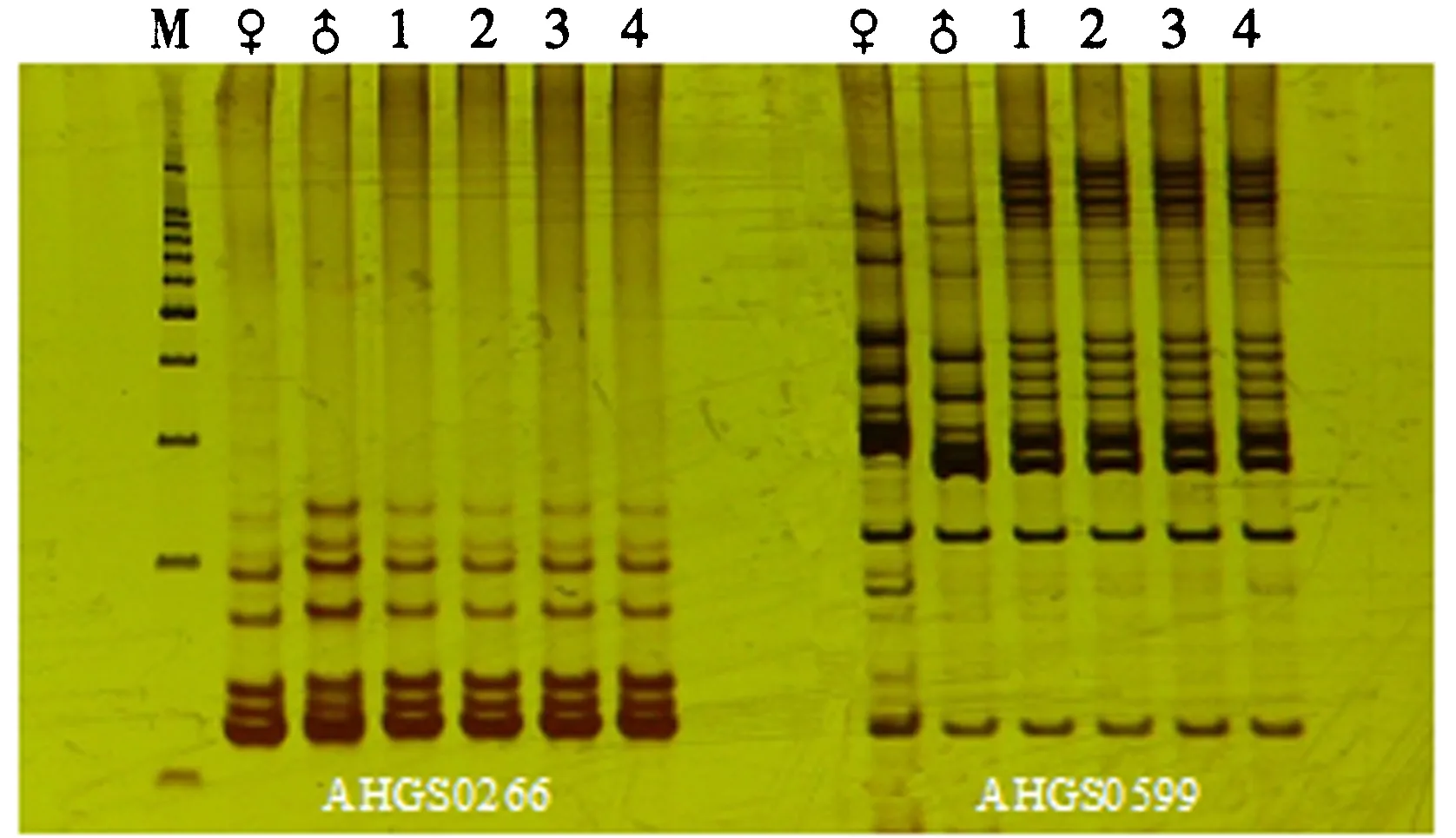
M.Marker (100 bp DNA Ladder, Promega);♀.母本6B;♂.父本6A; 1、2、3、4.带型与父本一致或出现父母本杂合带型,为真杂种
3 讨论
SSR标记可从DNA水平上鉴定杂种真伪,具备周期短、重复性高、不受时间和生长环境影响等优点.利用该方法辅助选择杂种,可提高真杂种的选择效率,有助于促进育种进程.另外,SSR标记具备鉴定苗期真杂种的特点, 所以利用SSR标记辅助创建遗传群体, 可以确保所创建群体遗传来源的可靠性[18].本研究利用SSR标记准确鉴定出22个杂交组合的F1代杂种的真伪,矫正了形态表型鉴定的误差,为后续的育种工作奠定了基础.
用分子标记进行真假杂种鉴定时,亲本间的多态性分析非常重要,找到差异明显的多态性标记是第一步.本研究根据2000多对SSR引物在多个群体中筛选的结果,选择多态信息含量高的40对SSR引物,对所有组合的亲本进行了筛选.各个组合中的多态性引物数目各不相同,这在一定程度上反映了亲本间遗传差异的大小.其中,多态性引物比例最高的为65%;8个组合的多态性比例超过50%;在少数几个组合中多态性比例为10%,可能是由于亲本间的遗传差异太小引起.因此,本研究选用的40对SSR引物具有较高的多态性,可以直接用于花生的遗传多样性、杂种鉴定以及遗传图谱的构建等研究.
[1] 李成军.花生分子育种研究概况与前景展望[J].中国种业,2009(6):15-17.
[2] 杨清岭,张少泽,甄志高,等.分子标记在花生育种中的应用[J].中国农学通报,2007,23(5):79-82.
[3] 苏君伟,于树涛,史普想,等.花生分子标记的研究进展[J].安徽农业科学,2011,39(1):5704-5705.
[4] GUO B, CHEN C Y, CHU Y, et al. Advances in genetics and genomics for sustainable peanut production[M]∥BENKEBLIA N. Sustainable Agriculture and New Biotechnologies. Boca Raton: CRC Press, 2012:341-367.
[5] GUO B, PANDEY M K, HE G, et al. Recent advances in molecular genetic linkage maps of cultivated peanut (ArachishypogaeaL.)[J]. Peanut Science, 2013,40:95-106.
[6] JIANG H, HUANG L, REN X, et al. Diversity characterization and association analysis of agronomic traits in a Chinese peanut (ArachishypogaeaL.) mini-core collection[J]. Journal of Integrative Plant Biology, 2014,56(2):159-169.
[7] KOILKONDA P, SATO S, TABATA S, et al. Large-scale development of expressed sequence tag-derived simple sequence repeat markers and diversity analysis inArachisspp.[J]. Molecular Breeding, 2012,30(1):125-138.
[8] REN X, JIANG H, YAN Z, et al. Genetic diversity and population structure of the major peanut (ArachishypogaeaL.) cultivars grown in China by SSR markers[J]. PLoS ONE, 2014,9(2):e88091.
[9] ROOMI S, SABIHA B, IQBAL A, et al. SSR based genetic diversity analysis in a diverse germplasm of groundnut (ArachishypogaeaL.) from Pakistan[J]. Australian Journal of Crop Science, 2014,8(1):55-61.
[10] GRATTAPAGLIA D, SEDEROFF R R. Genetic linkage maps ofEucalyptusgrandisandEucalyptusurophillausing a pseudo-testcross: mapping strategy and RAPD markers[J]. Genetics, 1994,137:1121-1137.
[11] MACEDO S E, MORETZSOHN M C M, LEAL-BERTIOLI S C, et al. Development and characterization of highly polymorphic long TC repeat microsatellite markers for genetic analysis of peanut[J]. BMC Research Notes, 2012,5:86.
[12] HE G H, MENG R H, NEWMAN M, et al. Microsatellites as DNA markers in cultivated peanut (A.hypogaeaL.)[J]. BMC Plant Biology, 2003,3:1-3.
[13] SHIRASAWA K, HIRAKAWA H, TABATA S, et al. Characterization of active miniature inverted-repeat transposable elements in the peanut genome[J]. Theoretical and Applied Genetics, 2012,124:1429-1438.
[14] CUC L M, MACE E S, CROUCH J H, et al. Isolation and characterization of novel microsatellite markers and their application for diversity assessment in cultivated groundnut (ArachishypogaeaL.)[J]. BMC Plant Biology, 2008,8:55.
[15] FERGUSON M E, BUROW M D, SCHULZE S R, et al. Microsatellite identification and characterization in peanut (A.hypogaeaL.)[J]. Theoretical and Applied Genetics, 2004,108:1064-1070.
[16] MORETZSOHN M C, LEOI L, PROITE K, et al. A microsatellite-based, gene-rich linkage map for the AA genome of (Arachisfabaceae)[J]. Theoretical and Applied Genetics, 2005,111(6):1060-1071.
[17] NAGY E D, CHU Y, GUO Y, et al. Recombination is suppressed in an alien introgression in peanut harboring Rma, a dominant rootknot nematode resistance gene[J]. Molecular Breeding, 2010,26:357-370.
[18] 陈静,胡晓辉,石运庆,等.花生品种间杂种F1代的SSR标记分析[J].核农学报,2009,23(4):617-620.
(责任编辑:杨郁霞)
Application of SSR markers in peanut F1hybrid identification
WANG Hui1,2, KHERA Pawan2,3, LI Shuang-ling4, REN Yan4, YUAN Mei4, ZHUANG Wei-jian5,VARSHNEY Rajeev K3, GUO Bao-zhu6, XIE Lian-hui1
(1.College of Plant Protection, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China; 2.Department of Plant Pathology, University of Georgia, Tifton, GA 31793, USA; 3.International Crops Research Institute for the Semi-Arid Tropics, Patancheru, Andhra Pradesh 502324, India; 4.Shandong Peanut Research Institute, Qingdao, Shandong 266100, China; 5.College of Crop Science, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002,China; 6.Crop Protection and Management Research Unit, USDA-ARS, Tifton, GA 31793, USA)
The objective of this study was to screen highly informative genic and genomic SSR markers and to identify true hybrids from 22 crosses between 24 parents. 24 diverse peanut parental lines were used to make hybrid combinations. There were a total of 92 F1seeds produced that were planted in the field for identification of true hybrids. The results showed that the traditional morphological method identified 62 "true" F1s and the molecular method identified 49 "true" F1s. The traditional morphologic method often resulted in false positive. In contrast, the molecular marker method gave accurate identification. Forty SSR markers showed highly polymorphic information content (PIC) among the parental lines. Among the cross combinations, "GT-C20-D×Tamrun OL07" had the highest polymorphism (65%). These SSR markers could be very useful not only for evaluating the genetic diversity of breeding materials, but also for selection of parental lines for genetic mapping population development.
peanut (ArachishypogaeaL.); SSR; hybrid identification; molecular marker
2014-12-11
2015-04-06
青岛市科技计划(14-2-4-12-jch);国家863计划(2013AA102602);国家自然科学基金(31471533);山东省农业资源创新利用项目;山东省农业科学院科技创新重点项目(2014CXZ10-4).
王辉(1978-),女,助理研究员,博士研究生.研究方向:花生植物病理和遗传育种.Email: huixu@uga.edu.通讯作者谢联辉(1935-),男,教授.研究方向:植物病毒学.Email: fjxlh@126.com.
S336
A
1671-5470(2015)04-0350-05
10.13323/j.cnki.j.fafu(nat.sci.).2015.04.003

