泥蚶人工选育群体的微卫星分析
田 野 , 邵艳卿, , 肖国强, , 滕爽爽, , 柴雪良 , , 张炯明, , 方 军,
(1.温州医科大学 检验医学院 生命科学学院, 浙江 温州 325005; 2.浙江省海洋水产养殖研究所, 浙江 温州 325005; 3.浙江省近岸水域生物资源开发和保护重点实验室, 浙江 温州 325005)
泥蚶(Tegillarca granosa)属软体动物门(Mollusca)、双壳纲(Bivalvia)、蚶目(Arcoida)、蚶科(Arcidae)动物,广泛分布于我国南北各沿海地区及韩国、越南、马来西亚等, 为我国传统四大养殖经济贝类之一。因其具有独特的风味及丰富的营养价值而深受人们喜爱。20世纪90年代初期, 浙江省海洋水产养殖研究所等单位率先突破了泥蚶规模化生产性人工育苗技术, 自此以后, 泥蚶养殖获得快速发展, 泥蚶养殖所用苗种也绝大部分由人工苗种所替代[1]。浙江乐清湾一带也成为国内最主要的泥蚶人工苗种产地。
但近年来研究发现, 因长期以来生产性养殖的泥蚶, 主要通过人工育苗提供苗种, 苗种繁育所用亲贝均为人工养成, 经过多年自繁自养和亲贝的随意选留, 加剧了泥蚶的近交衰退, 已不同程度地出现了遗传多样性减低、杂合度下降、生长速度减缓、抗逆性差、性状退化等问题[2]。微卫星标记(simple sequence repeat, SSR), 具有多态性丰富、共显性遗传、重复性好、操作分析简单等优点, 已被广泛用于群体遗传多样性评价、遗传连锁图谱构建、分子标记辅助育种等领域[3]。进行选育时, 因近交机率增加和有效亲本数的减少, 可能导致选育群体的遗传多样性下降, 进而引起选育群体的性状衰退[4], 虽然一些研究报道了贝类养殖群体的遗传多样性与野生群体相比未发生明显变化[5-8], 但贝类的苗种培育和群体选育过程中, 常因亲本数量过少、雌雄比例不当、亲本贡献不均、配子质量差异、配子竞争、人工选优和选型交配等因素, 引起有效群体数量的减少,从而降低选育群体的遗传多样性[9-14]。
目前国内对于贝类选育群体遗传多样性的分析已有不少报道: 王庆志等[15]对长牡蛎人工选育群体的微卫星分析; 王学颖等[16]对马氏珠母贝金黄壳色系和基础群体遗传结构进行比较; 本研究利用微卫星标记对泥蚶连续 3代选育群体的遗传多样性进行分析, 与基础群体进行比较, 考查随着选育代数的进行群体各种遗传指数的变化规律, 为进一步优化育种方案提供参考。
1 材料与方法
1.1 样本的采集
本研究采集的泥蚶样品为经浙江省海洋水产养殖研究所连续多年选育的F3代, F4代, F5代速生群体, 选育策略为群体内个体选择结合家系/亚群选择,同时以最早保存的未经选育的基础群体作为对照组。F3, F4, F5代个体均从群体中随机选取65个, 于–70℃冰箱中保存备用, 基础群体35个于2007年保存在–70℃冰箱备用。
1.2 DNA提取
采用常规的蛋白酶 K 消化, 酚-氯仿-异戊醇抽提, 无水乙醇沉淀的方法提取基因组 DNA; 用琼脂糖凝胶电泳检测后, 用紫外分光光度计检测DNA纯度与浓度, 选取电泳条带较亮且浓度较高的DNA稀释成统一浓度, 保存于4℃冰箱中用于后续实验。
1.3 PCR扩增及产物检测
从39对泥蚶微卫星引物中[17]筛选出12对作为本研究使用的引物, 并对引物退火温度进行优化。引物由上海生工公司合成, 其编号及序列见表1。

表1 引物编号及序列Tab.1 Primer number and sequences
PCR反应体系为15 µL, 包含100 ng模板DNA,1×PCR缓冲液, 0.4UTaKaRa Taq DNA聚合酶, 2.5 mmol/L MgCl2, 0.25 mmol/L dNTP混合液, 上下游引物各0.7 µmol/L。PCR扩增条件为: 94℃ 预变性5 min,然后30个循环, 每个循环包括: 94℃ 30 S, 49~56℃退火30 S, 72℃延伸45 S。PCR产物在6%变性聚丙烯酰胺凝胶上电泳, 50 bp DNA ladder (Takara)作为Marker检测产物位置, 硝酸银法染色, 条带结果由人工读取、统计。
1.4 数据统计与分析
统计每个位点的等位基因数量(Observed number of allele,No), 用 Popgene 32(1.32)分析数据, 计算有效等位基因数(Effective number of allele,Ne),Shannon遗传多样性指数(Shannon genetic ranged index,Rs), 近交系数(Wright's fixation index,Fis), 观测杂合度(Observed heterozygosity,Ho), 期望杂合度(Expected heterozygosity,He)以及哈迪-温伯格平衡(Hardy-Weinberg equilibrium, HWE)。PIC_Calc 0.6(http://www.bbioo.com/download/58-247-1.html)计算多态信息含量(Polymorphism information content,PIC)。
2 结果
2.1 遗传多样性
泥蚶基础群体和选育群体的遗传参数结果如表2所示。12个位点均表现出多态性: 基础群体各位点有效等位基因数1.69~6.30个, 平均3.16个; F3代为1.52~3.88个, 平均2.71个; F4代1.15~4.75个, 平均2.73个; F5代1.57~5.17个, 平均2.58个; 表明随着群体选育的进行, 等位基因数总体呈现下降趋势。所有群体12个位点观测杂合度(Ho)均小于期望杂合度(He)。观测杂合度平均范围为 0.4195~0.4725, 期望杂合度平均范围为 0.5700~0.6323, 表明各位点有一定程度的杂合子缺失。在 48个群体-位点组合中有32个群体-位点组合显著偏离平衡(P<0.05), 约占总数 66.67%。Shannon遗传多样性指数平均值为1.06~1.22。各位点多态信息含量范围0.1119~0.8209。
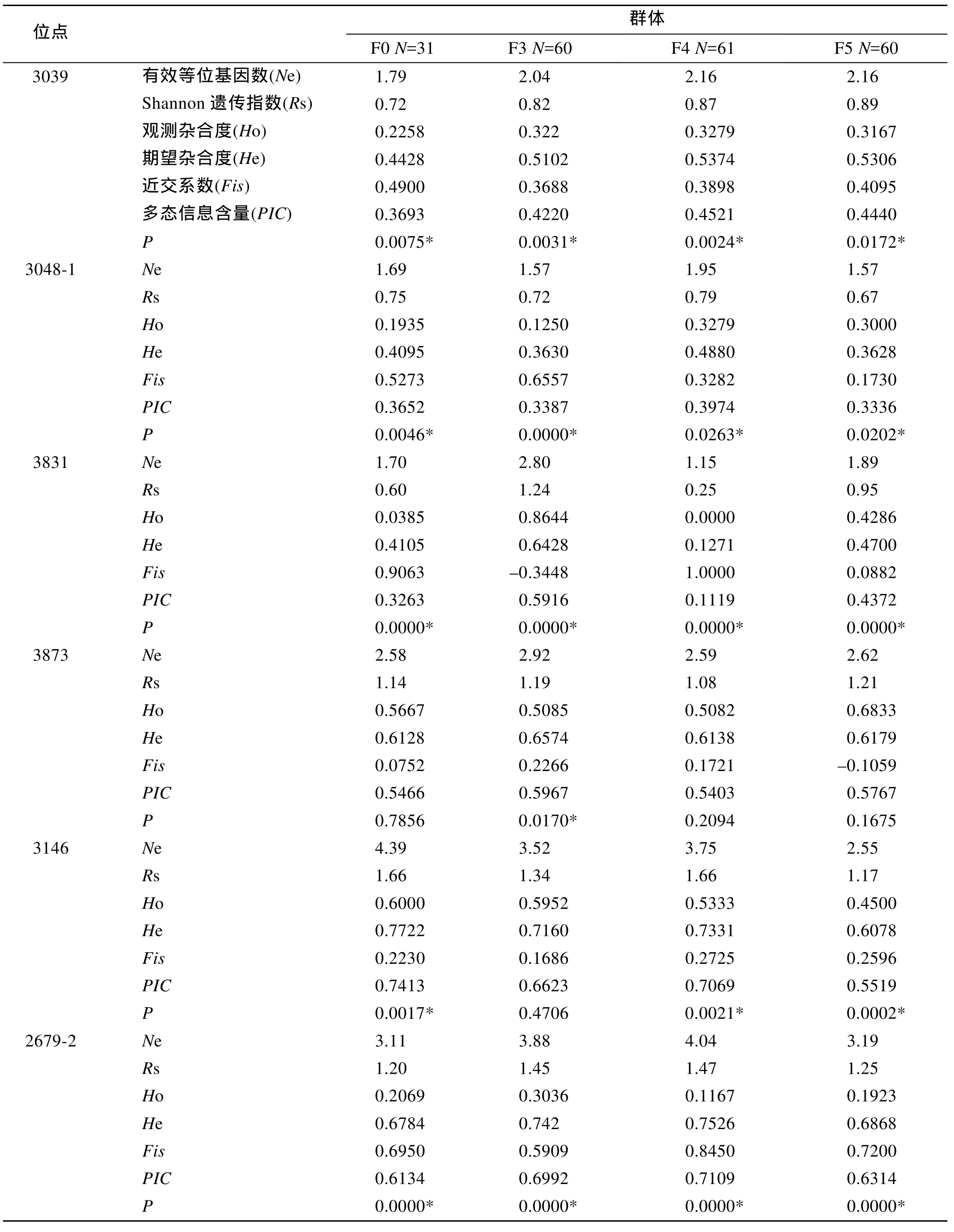
表2 泥蚶4个群体的遗传多样性分析Tab.2 Genetic variability analysis of 4 blood clam T.granosa populations
2.2 遗传分化
4个群体间不同位点的遗传分化系数 Fst值从0.0347~0.4549, 平均值 0.1930, 相关文献认为遗传分化程度比较大(处于 0.15~0.25之间); 基因流Nm值为 0.2996~6.9617, 平均值 2.3367; 群体近交系数Fis范围0.0052~0.7135, 平均值0.2468, 表明群体的近交程度较高。
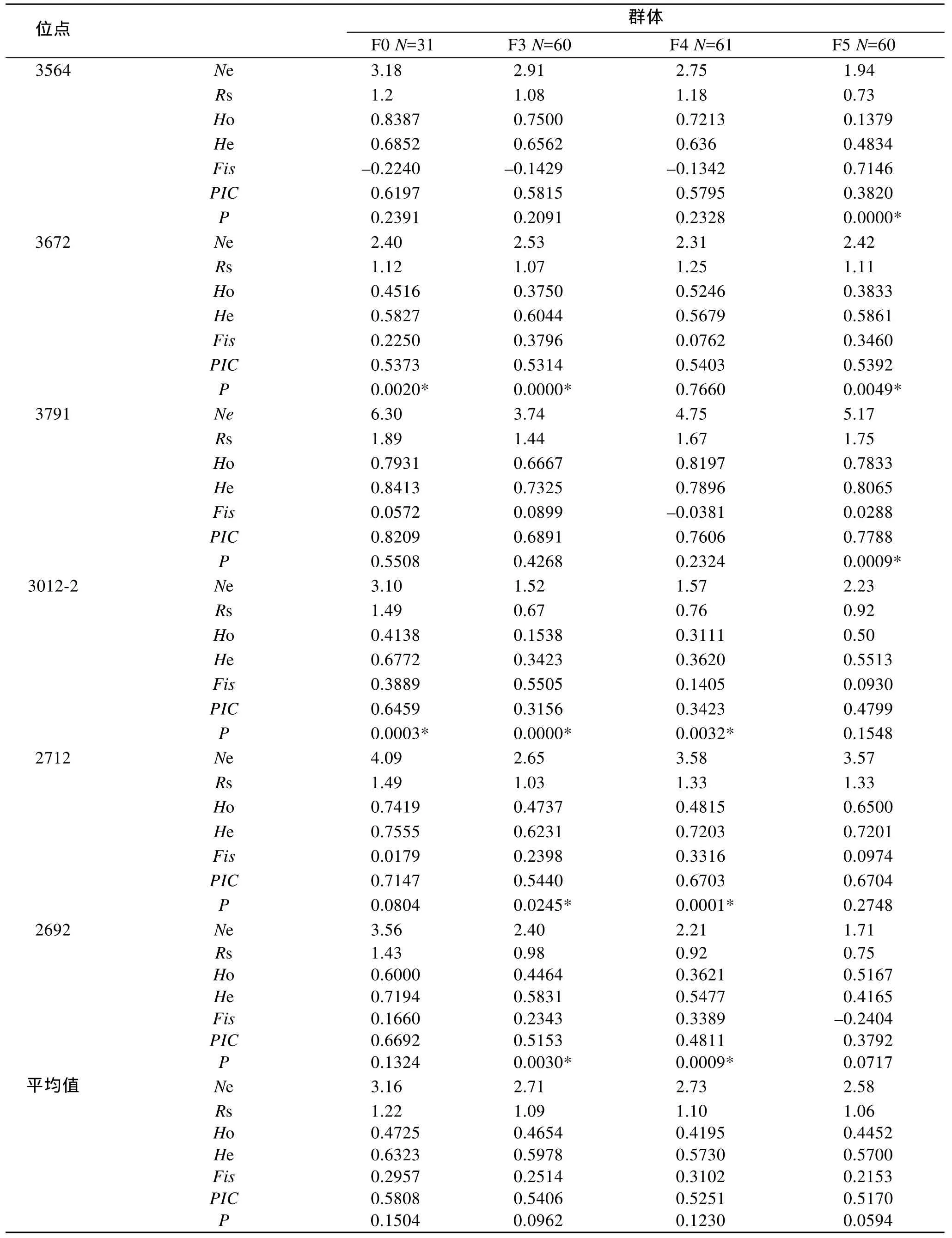
续表

表3 4个泥蚶群体的基因分化与基因流Tab.3 Gene differentiation and gene flow of 4 blood clam T.granosa populations
3 讨论
遗传育种、保护种质资源等相关工作能顺利开展的前提和基础是维持群体的遗传多样性, 群体遗传多样性的高低与其适应能力、生存能力和进化潜力密切相关。遗传多样性降低可导致其适应能力降低、有害隐性基因表达增加及经济性状衰退, 最终导致物种退化[18]。因此, 为了保证选育工作的顺利进行并获得高质量的选育新品种, 保持群体的遗传多样性是非常重要的。
3.1 泥蚶群体的遗传多样性分析
多态信息含量、等位基因数、遗传多样性指数都是体现群体遗传多样性的量度参数。文献中认为PIC>0.5时为高度多态性位点, 0.25 Shannon遗传多样性指数(Rs)在3个选育群体中的变化不大, 而与基础群体相比较略有下降, 在王庆志[15]对长牡蛎的报道中的数据比较发现结果是相似的。说明随着选育的进行, 遗传多样性指数趋于稳定。3代连续选育的群体平均有效等位基因数与基础群体相比有所下降, 但选育群体之间变化不大, 基本稳定。12个位点PIC平均值: F3、F4、F5代分别为0.5406、0.5251、0.5170; 基础群体PIC平均值为0.5808。以上结果表明与基础群体相比较, 选育的遗传多样性虽有一定程度的下降, 但是随着选育的进行, 群体多样性各指标趋于稳定。 遗传分化指数(Fst)反映群体间遗传分化程度的重要参数。从本研究的结果来看: 4个群体中12个位点的Fst值范围0.0347~0.4549, 平均值为0.1930。表明有19.3%的变异是由群体分化导致的, 而80.7%的变异来源于群体内, 群体间的遗传变异程度处于一般水平。Wright[21]认为, 基因流Nm大于1时, 能够发挥其均质化作用, 从而抑制由遗传漂变引起的群体间分化。而在本研究中, 12个位点中有8个位点的Nm值大于1, 说明群体间的交流程度比较高。马春艳等[22]对凡纳滨对虾引进群体和 2个养殖群体遗传变异的微卫星分析中得到的Fst值为0.0500~0.1657,王学颖等[16]对马氏珠母贝金黄壳色系F3和基础群体遗传结构比较研究中得到的Fst值范围为 0.0031~0.1478, 李云霞等[23]对刺参群体遗传结构分析及与经济性状的相关性研究中的Fst值为0.0130~0.1805,汤健等[24]对马氏珠母贝家系遗传结构的微卫星分析中的Fst值 0.0375~0.2733, 这些结果都比本研究的结果要低, 产生此差异的原因可能是由于引物设计的不同产生结果差异, 由于引物定位的位置不同,生物群体在该位置发生不同变异。 本研究结果表明, 在经过连续的人工选育过程,对群体的遗传多样性与基础群体相比有一定程度的降低, 但仍处于较高水平。这与我们育种选取的方式有关: 因为采用群体选育方式确保了有效繁育群体数量所以遗传多样性下降较慢, 但针对某个性状选育的进展较慢, 若采用家系选育方式, 虽然就某个性状而言选育进展较快, 但遗传多样性也下降较快。因此, 我们采用群体选育结合家系选择的方式, 虽然有一定程度的下降, 但下降程度不显著。从研究的结果来看, 经连续选育的泥蚶群体遗传分化程度还比较低, 并且其遗传多样性和遗传分化程度逐渐趋于稳定。所以, 泥蚶育种仍有非常大的潜力进行优良性状的筛选。但今后应避免近交繁殖对选育群体的优良性状产生的不良影响, 确保泥蚶育种工作的持续健康进行。 [1] 刘博, 滕爽爽, 柴雪良, 等.泥蚶遗传连锁图谱的构建[J].海洋与湖沼, 2012, 43(5): 983-991. [2] 吕振明, 李太武, 苏秀榕.泥蚶遗传多样性的研究[J].高技术通讯, 2005, 15(12): 104-110. [3] Wright J M, Bentzen P.Microsatellites: genetic markers for future[J].Reviews in Fish Biology and Fisheries, 1994, 4(3): 384-388. [4] Beaumont A, Boudry P, Hoare K.Biotechnology and genetics in fisheries and aquaculture[M].Singapore:Wiley-Blackwell, 2010. [5] Durand P, Wada K T, Blanc F.Genetic variation in wild and hatchery stocks of the black pearl oyster,Pinctada margarififera, from Japan[J].Aquaculture,1993, 110(1) : 27-40. [6] English L J, Maguire G B, Ward R D.Genetic variation of wild and hatchery populations of the Pacific oyster,Crassostrea gigas(Thunberg), in Australia[J].Aquaculture,2000, 187(3-4): 283-298. [7] Li Q, Yu H, Yu R H.Genetic variability assessed by microsatellites in cultured populations of the Pacific oyster(Crassostrea gigas) in China[J].Aquaculture,2006, 259(1-4): 95-102. [8] Zhao C, Li Q, Kong L F.Inheritance of AFLP markers and their use for genetic diversity analysis in wild and farmed scallop (Chlamys farreri)[J].Aquaculture, 2009,287(1-2): 67-74. [9] Hedgecock D, Sly F.Genetic drift and effective population sizes of hatchery-propagated stocks of the Pacific oyster,Crassostrea gigas[J].Aquaculture, 1990, 88(1): 21-38. [10] Boudry P, Collet B, Cornette F, et al.High variance in reproductive success of the Pacific oyster (Crassostrea gigas, Thunberg)revealed by microsatellite-based parentage analysis of multifactorial crosses[J].Aquaculture,2002, 204 (3-4): 283-296. [11] Taris N, Ernande B, McCombie H, et al.Phenotypic and genetic consequences of size selection at the larval stage in the Pacific oyster(Crassostrea gigas)[J].Journal of Experimental Marine Biology and Ecology,2006, 333(1): 147-158. [12] Appleyard S A, Ward R D.Genetic diversity and effective population size in mass selection lines of Pacific oyster(Crassostrea gigas)[J].Aquaculture, 2006,254(1-4): 148-159. [13] Lind C E, Evans B S, Knauer J, et al.Decreased genetic diversity and a reduced effective population size in cultured silver-lipped pearl oysters(Pinctada maxima)[J].Aquaculture, 2009, 286(1-2): 12-19. [14] Li R H, Li Q, Yu R H.Parentage determination and effective population size estimation in mass spawning Pacific oyster(Crassostrea gigas)based on microsatellite loci analysis[J].Journal of the World Aquaculture Society, 2009, 40(5): 667-677. [15] 王庆志, 李琪, 孔令锋.长牡蛎3代人工选育群体的微卫星分析[J].水产学报, 2012, 36(10): 1529-1536. [16] 王学颖, 高远镇, 杜晓东, 等.马氏珠母贝金黄壳色系 F3和基础群体遗传结构比较[J].海洋通报, 2012,31(3): 324-328. [17] Liu B, Teng S S, Shao Y Q, et al.Isolation and characterization of 39 novel polymorphic EST-SSR loci for the blood clam,Tegillarca granosa[J].Conservation Genet Resour, 2012, 4: 375-378. [18] Beaumont A, Boudry P, Hoare K.Biotechnology and genetics in fisheries and aquaculture[M].Singapore:Wiley-Blackwell, 2010. [19] Botstein D, White R L, Skolnick M, et al.Construction of a genetic linkage map in mall using restriction fragment length polymorphisms[J].Am J Hum Genet,1980, 32: 314-331. [20] 刘博, 邵艳卿, 王侃, 等.4个缢蛏群体遗传多样性和系统发生关系的微卫星分析[J].海洋科学, 2013, 37(8),96-102. [21] Wright S.Evolution in Mendelian population[J].Genetics, 1931, 16: 91-159. [22] 马春艳, 马洪雨, 马凌波, 等.凡纳滨对虾引进群体和2个养殖群体遗传变异的微卫星分析[J].海洋渔业,2011, 33(1): 1-8. [23] 李云霞, 李娇, 丁君, 等.基于微卫星标记的刺参群体遗传结构分析及与经济性状的相关性研究[J].大连海洋大学学报, 2013, 28(5): 438-444. [24] 汤健, 管云雁, 刘文广, 等.马氏珠母贝家系遗传结构的微卫星分析[J].海洋科学, 2013, 37(8): 35-41.3.2 泥蚶群体遗传分化分析

