基于18S rRNA、COⅡ基因的鮸鱼(Miichthysmiiuy)系统发育分析
陈莹莹, 苗 亮, 李明云, 张晓丽, 郭晓飞, 潘 娜, 陈 炯
(宁波大学 应用海洋生物技术教育部重点实验室, 浙江 宁波 315211)
基于18S rRNA、COⅡ基因的鮸鱼(Miichthysmiiuy)系统发育分析
陈莹莹, 苗 亮, 李明云, 张晓丽, 郭晓飞, 潘 娜, 陈 炯
(宁波大学 应用海洋生物技术教育部重点实验室, 浙江 宁波 315211)
为了解鮸鱼的系统发育地位,探索18S rRNA、COⅡ分子标记对石首鱼科系统分类的适用性,对鮸鱼的18S rRNA基因、线粒体COⅡ基因进行克隆、序列分析和构建NJ系统进化树。结果表明:扩增得到鮸鱼18S rRNA基因1305 bp,A+T含量为46.2%;COⅡ基因序列为854 bp,A+T含量为53.5%,有明显的反G偏倚(15.2%),含一个699 bp的ORF,编码233个氨基酸;基于18S rRNA序列构建的系统树显示,鲈形目鱼类聚为一个紧密的簇,该目中各科鱼类间18S rRNA序列的同源性均大于97.8%,无法反映鮸鱼在石首鱼科中的系统发育情况。基于COⅡ序列构建的系统树显示,鲈形目鱼类分为2簇:包括鮸鱼在内的所有石首科鱼类聚为一个大簇,其他各科聚为另一个大簇;其中鮸鱼与黄唇鱼亲缘关系最近,二者序列相似性为97.4%。虽然石首科鱼类聚成的簇中有些分支的自展支持率较低(<50%),个别种类的聚类与传统分类有所差异,但大部分聚类是一致的。结果既能丰富鮸鱼的分子系统学资料,又可为研究鮸鱼的系统发育地位及石首鱼科鱼类的进化关系提供参考资料。
鮸鱼; 18S rRNA; COⅡ; 系统发育
鮸鱼(Miichthysmiiuy)俗称黑鮸、米鱼等,属硬骨鱼亚纲(Osteichthyes)、鲈形目(Perciformes)、石首鱼科(Sciaenidae)、鮸鱼属(Miichthys),单属单种,主要分布于太平洋西岸的近海,是中国重要的经济鱼类之一,具有较高的营养和药用价值[1]。鲈形目是鱼类中最大的一个目,有7000余种;石首鱼科是鲈形目中最大的一个科,有210余种,该科鱼类分布范围广、生活环境多种多样、起源与进化关系复杂,在物种分类和进化关系方面仍存在较多分歧[2-4]。许多学者根据形态和解剖特征探讨鮸鱼的分类地位,但存在分歧,如Taniguchi根据脑颅的骨学特征将石首鱼科鱼类划分为毛鲿鱼式型、黄姑鱼式型、叫姑鱼式型、白姑鱼式型和黄鱼式型,其中鮸属、毛鲿鱼属和部分黄姑鱼属物种归毛鲿鱼式型[5];而Taniguchi根据椎骨特征将石首鱼科鱼类分为黄姑鱼式型、叫姑鱼式型、白姑鱼式型、黄鱼式型和梅童鱼式型,其中鮸属和黄姑鱼属归为黄姑鱼式型[6];朱元鼎等[7]和成庆泰等[8]则根据鳔和耳石形态将石首鱼科鱼类分为叫姑鱼亚科、白姑鱼亚科、黄鱼亚科等7个亚科,其中鮸鱼属、黄鱼属和梅童鱼属归属于黄鱼亚科[7-8]。由于鱼类形态特征会受生活环境、年龄、性别等因素的影响,且依据形态特征进行分类也受到样品及测量误差等的限制,因此有必要使用其他方法作为补充。
随着分子生物学的发展,分子标记技术已被广泛应用于生物系统分类中,常用的分子标记有16S、18S核糖体亚基基因(16S rRNA、18S rRNA)、线粒体COⅠ、COⅡ、D-Loop序列等。目前,已有学者克隆了包括鮸鱼在内的石首鱼科鱼类的16S rRNA、COⅠ等基因并进行了系统进化分析[9-10],但鲜少有基于18S rRNA 和线粒体COⅡ研究鮸鱼系统分类地位的文献。本文通过PCR扩增获取鮸鱼的18S rRNA和线粒体COⅡ序列,通过与其他鱼类的序列比对及系统进化分析,探讨其起源情况和进化地位,研究结果可与传统形态分类互为补充,为研究石首鱼科鱼类的种系进化和分子系统分类提供参考材料。
1 材料与方法
1.1 材料
实验用鮸鱼为采自浙江宁波象山港网箱养殖的1龄鱼,麻醉后解剖、剪取背部肌肉,液氮速冻后-80℃保存备用。
1.2 方法
1.2.1 DNA提取与PCR扩增
用常规酚/氯仿法[11]提取鮸鱼肌肉组织DNA,并调整浓度至50 ng/μL。
根据鱼类18 s rRNA序列保守区域设计扩增引物,序列为:上游引物5′-ACGGGTAACGGGGAATCAGG-3′;下游引物5′-ACGGGCGGTGTGTACAAAGG-3′;根据鲈形目鱼类COⅡ基因上、下游保守区域设计扩增引物,序列为:上游引物5′-ACATAACCSCTCTGYCACTTTC-3′;下游引物5′-GGAGYCACCAATCTTTAGCTTAA-3′。引物均由上海立菲生物技术有限公司合成。
PCR反应体系20 μL,包括2×Taq Premix-Dye(上海博彩生物科技有限公司) 10 μL,上、下游引物各1 μL,模板DNA 1 μL,灭菌水 7 μL。反应程序为:94 ℃预变性3 min;94 ℃变性30 s,退火30 s(18S rRNA和COⅡ引物退火温度分别为58℃和52℃),72 ℃延伸1min 30 s,30个循环;72 ℃延伸10 min。PCR产物经1.5%琼脂糖凝胶电泳检测,用Biospin胶回收试剂盒(Bioflux公司)纯化回收目的片段,与pMD19-T 载体(TaKaRa公司)连接后转化入大肠杆菌TGⅠ感受态细胞,经蓝白斑筛选,挑阳性克隆送至上海立菲生物技术有限公司测序。
1.2.2 序列分析与系统树构建
从GenBank中下载22种硬骨鱼纲鱼类18S rRNA基因序列和32种鲈形目鱼类COⅡ基因序列,所用序列的物种名称及登录号见图1和图2。将本实验扩增得到的鮸鱼18S rRNA序列和COⅡ基因序列与GenBank检索序列用Clustal X1.83软件进行多重比对;用MEGA5.0软件、以七鳃鳗(Geotriaaustralis)为外群用邻接法(Neighbor-Joining,NJ法)构建分子系统树,采用Bootstrap(重复数=1000)检验分子系统树各分支的置信度。
2 结果
2.1 鮸鱼18S rRNA和COⅡ基因序列特征
扩增得到鮸鱼18S rRNA基因序列1305 bp,碱基组成A、T、G、C分别为24.1%、22.1%、29.2%和24.6%,A+T含量(46.2%)低于G+C含量(53.8%),编码435个氨基酸。与其他鲈形目11种鱼类(平均值为:A 24.0%、G 29.2% 、T 22.1%、C 24.7%)比对,各碱基含量基本相似。
扩增得到的鮸鱼COⅡ序列854 bp,碱基组成情况A、T、G、C分别为28.5%、25.0%、15.2%和31.3%,A+T含量(53.5%)高于G+C含量(46.5%),碱基组成有明显的偏向性,G的含量明显低于其他3种碱基,包含一个699 bp的开放读码框(Open Reading Frame,ORF),编码233个氨基酸。
2.2 基于18S rRNA基因的分子系统发育分析
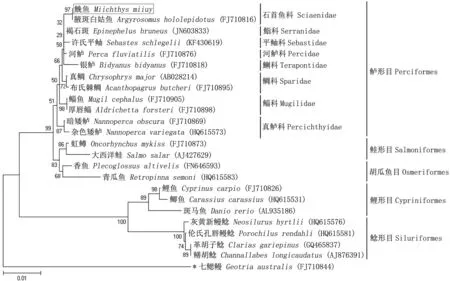
图1 基于18S rRNA构建的硬骨鱼纲鱼类分子系统树(NJ法)
Bootstrap=1000;*表示外群。
采用NJ 法对23种硬骨鱼的18S rRNA基因序列构建分子系统树(图1)。结果显示:鮸鱼与其他硬骨鱼类聚为一个大簇;鮸鱼与同属石首鱼科的腋斑白姑鱼亲缘关系最近,形成一个紧密的簇,序列比对显示二者18S rRNA序列的同源性为100.0%;鲈形目中的各个科均聚在一个大簇中,该目各鱼类18S rRNA序列的同源性范围97.8%~100.0%,提示该科鱼类可能有共同的进化起源;另外,鲈形目鱼类与鲑形目和胡瓜鱼目亲缘关系较近,而与鲤形目和鲶形目亲缘关系较远。
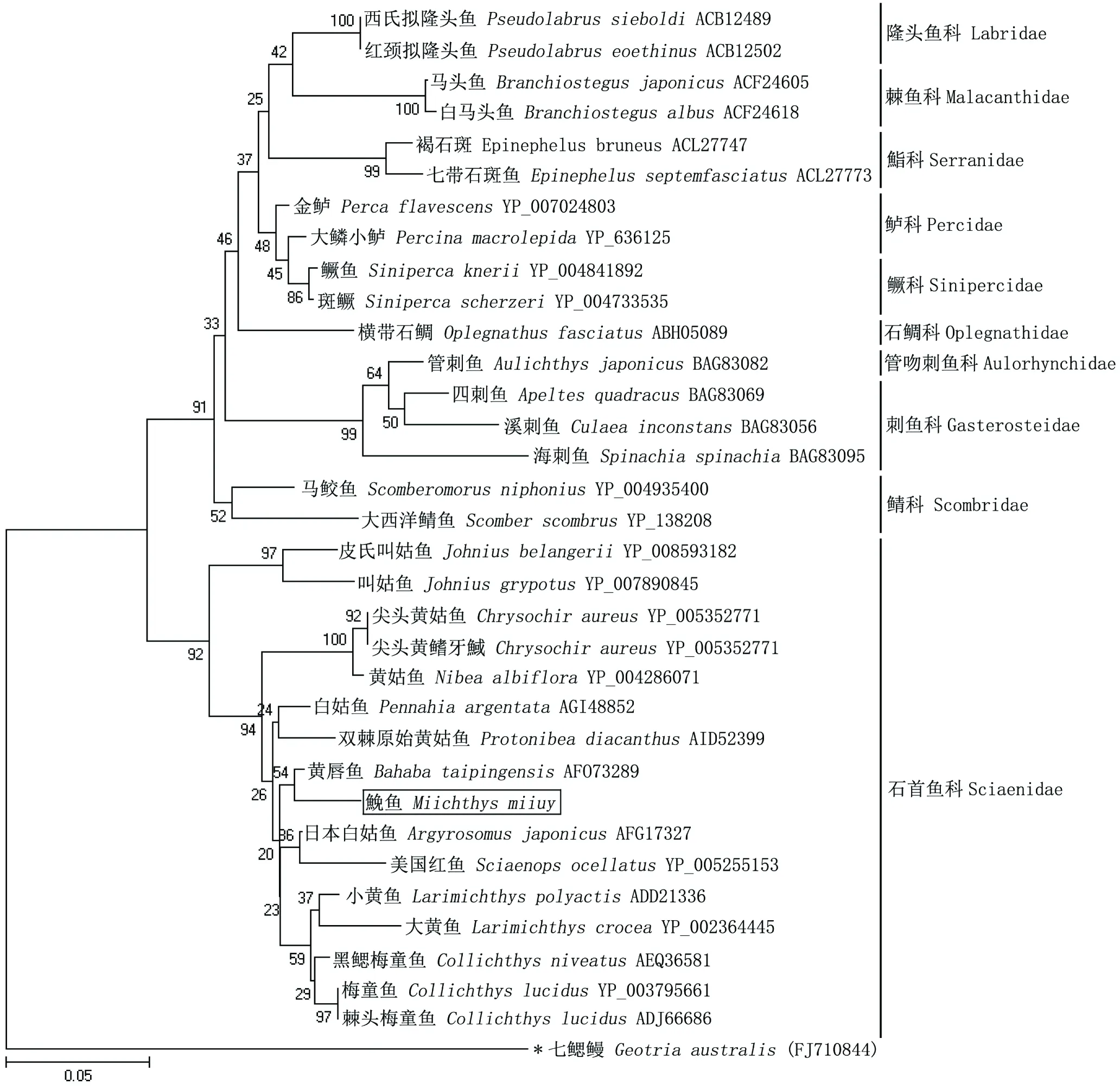
图2 基于COⅡ氨基酸序列的鲈形目鱼类分子系统树(NJ法)
Bootstrap=1000;*表示外群。
2.3 基于COⅡ序列的分子系统发育分析
图2为基于33种鲈形目鱼类COⅡ氨基酸序列构建的分子系统树。结果表明:鲈形目鱼类分为两大系,其中棘鱼科、隆头鱼科、刺鱼科、管吻刺鱼科、石鲷科、鮨科、鲈科、鳜科、鲭科分别相聚后再聚为一个大簇,而石首鱼科单独聚为另一个大簇;石首鱼科中尖头黄姑鱼、尖头黄鳍牙鰄、黄姑鱼、白姑鱼、双棘原始黄姑鱼、黄唇鱼、鮸鱼、日本白姑鱼、美国红鱼、小黄鱼、大黄鱼、黑鳃梅童鱼、梅童鱼和棘头梅童鱼亲缘关系较近,聚为一个大簇,而皮氏叫姑鱼和叫姑鱼则单独聚为一个小簇;鮸鱼与黄唇鱼亲缘关系较近,二者聚为一个小簇,序列比对显示其COⅡ氨基酸序列同源性为97.4%。
3 讨论
18S rRNA基因是编码核糖体小亚基的基因,序列保守性较高,易于用通用引物扩增,较适合科级以上的物种之间的分子分类系统学研究,近年来在生物分子系统演化研究中得到广泛应用,是研究生物高级分类群系统演化最为理想的工具之一[12-15]。本文中扩增得到鮸鱼18S rRNA基因部分序列1305 bp,构建的进化树中显示,鲈形目鱼类聚为一个紧密的簇,序列比对显示鲈形目各鱼类18S rRNA序列的同源性均>97.8%;其中鮸鱼与真鲈科鱼类(杂色矮鲈、暗矮鲈)的亲缘关系较远,其18S rRNA序列同源性却分别也有99.1%和99.2%。程汉良等[16]的研究显示帘蛤科6种贝类间18S rRNA基因序列的差异率均在3%以上,系统发育树分析显示18S rRNA序列适合作为帘蛤科系统发育研究的分子标记。本研究显示18S rRNA序列可用于鲈形目各科间的系统发育研究,但在石首鱼科中,鉴于18S rRNA序列的高保守性和GenBank中可用物种数据较少,18S rRNA不适合该科不同物种间的系统发育分析。要研究鮸鱼与石首鱼科其他鱼类间的系统发育和演化关系,还需使用其他进化速度更快、序列信息更丰富的分子标记。
细胞色素氧化酶(cytochrome oxidase)是由线粒体基因编码的蛋白之一,由3个基因(COⅠ、COⅡ和COⅢ)编码的3个亚基构成。COⅠ、COⅡ和COⅢ 具有进化信息位点多、易扩增且相对较短的特点,是理想的DNA分子标记,被广泛应用于分析亲缘关系较近的种、种以下分类阶元以及地理种群之间的分子系统关系,在水产动物研究中已多有报道,如柳淑芳等[9]和Lakra等[17]都使用COⅠ序列对石首鱼科鱼类进行了系统发育分析,崔文涛等[18]用COⅡ序列分析蛸亚科的系统进化关系。本文扩增得到鮸鱼COⅡ序列854 bp,包含一个699 bp的ORF,编码284个氨基酸。基于COⅡ序列构建的系统树显示:鲈形目33种鱼聚成两个大簇,其中石首鱼科鱼类单独聚为一簇,而其他9个科聚为另一簇。在石首鱼科中叫姑鱼和皮氏叫姑鱼首先与该科的其他鱼类分开,二者单独形成一簇,表明在石首鱼科中皮氏叫姑鱼和叫姑鱼与其他鱼类的分歧时间较早;鮸鱼与黄唇鱼首先聚为一簇,二者的COⅡ序列相似性为97.4%,表明二者亲缘关系较近。
蒙子宁等[10]对中国石首鱼类基于形态学的系统发育研究结果认为,叫姑鱼亚科口小下位、牙齿细小、营标准底栖生活,是最先分化出来的一支。本研究的COⅡ系统树也显示叫姑鱼属与其他石首科鱼类系统位置较远,这与陈泉酶[4]用16S rRNA基因序列对石首鱼科进行分析时得出的叫姑鱼属与其它石首鱼科鱼类的亲缘关系较远的结论相一致。本研究的COⅡ系统树中,鮸鱼与黄唇鱼、白姑鱼、双棘原始黄姑鱼、日本白姑鱼、美国红鱼、小黄鱼、大黄鱼、黑鳃梅童鱼、梅童鱼和棘头梅童鱼聚为一簇,这与马春艳等[19]基于RAG和Cheng等[20]基于Cytb和COⅠ基因对石首鱼科鱼类系统进化关系分析的结果基本一致,也支持朱元鼎等[7]根据传统形态特征将黄鱼属、梅童鱼属和鮸鱼属划分在同一个黄鱼亚科的观点。但在本研究构建的COⅡ系统树中,这一簇中有多个小支的自展支持率偏低(<50%),特别是其中白姑鱼属的日本白姑鱼与同属的白姑鱼距离较远而与拟石首鱼属的美国红鱼优先聚为一簇(自展支持率24%),以及双棘原黄姑鱼与黄姑鱼距离较远而与白姑鱼优先聚为一簇,这些均与传统的分类观点不相一致。因此,要更加全面、准确地分析石首鱼科鱼类的系统发育关系、评估鮸鱼的系统发生和进化地位,一方面有待于数据库中该科鱼类分子标记信息的不断丰富,另一方面也可联合采用其他多个进化速率不同的分子标记(如线粒体D-Loop区、ND基因等)做进一步的分析。
综上所述,本实验克隆了鮸鱼18S rRNA和线粒体COⅡ序列并进行了系统进化分析,研究结果既有与传统分类相互印证的地方,也有一些分歧之处,本研究结果可为探讨鮸鱼的起源、进化和鲈形目及石首鱼科鱼类的系统发生提供参考资料,并可作为传统形态分类学的补充,在此基础上今后仍需做更为深入、全面地研究。
[1]苏锦祥. 鱼类学与海水鱼类养殖(第2版) [M]. 北京:中国农业出版社, 1993: 214-215.
[2]辛 俭, 张玉荣, 徐冬冬, 等. 基于线粒体16sDNA和COⅠ基因探讨中国近海黄姑鱼类的分子系统进化关系[J]. 海洋与湖沼,2014,45(2):307-313.
[3]韩 冰. 黄姑鱼和鮸鱼的形态学特征及黄姑鱼群体的遗传学研究[D]. 青岛:中国海洋大学,2007.
[4]陈泉梅.中国石首鱼类分子系统学研究[D]. 广州:暨南大学,2007.
[5]Taniguchi N. Comparative osteology of the sciaenid fishes from Japan and its adjacent waters-I. Neurocranium[J]. Japa Jour Ich, 1969a, 16(2):55-67.
[6]Taniguchi N. Comparative osteology of the sciaenid fishes from Japan and its adjacent waters-II. Vertebrae I bid[J]. Japa Jour Ich, 1969b, 16(4):153-156.
[7]朱元鼎, 罗云林, 伍汉霖. 中国石首鱼类分类系统的研究和新属新种的叙述[M]. 上海:上海科学技术出版社, 1963.
[8]成庆泰, 郑葆珊. 石首鱼科. 中国鱼类系统检索(上册)[M]. 北京: 科学出版社, 1987. 317-324.
[9]柳淑芳, 陈亮亮, 戴芳群,等. 基于线粒体COⅠ基因的DNA条形码在石首鱼科(Sciaenidae)鱼类系统分类中的应用[J]. 海洋与湖沼, 2010,41(2):223-232.
[10]蒙子宁, 庄志猛, 丁少雄,等. 中国近海8种石首鱼类的线粒体16S rRNA基因序列变异及其分子系统进化[J]. 自然科学进展,2004,14(5):514-521.
[11]Sambrook J, Ffitsch E F, Maniatis T. Molecular cloning: A Laboratory Manual, 2nd ed [C]. New York: Cold Spring Harbor Laboratory Press. 1989.
[12]郝家胜, 杨 群. 后生动物起源与进化的分子系统学研究进展[J]. 安徽师范大学学报:自然科学版,2001,24(4):378-382.
[13]Giribet G, Okusu A, Lindgren A R, et al. Evidence for a clade composed of molluscs with serially repeated structures: Monoplacophorans are related to chitons[J]. Proc Natl Acad Sci, 2006, 103(20): 7723-7728.
[14]Passamaneck Y J, Schander C, Halanych K M. Investigation of molluscan phylogeny using large subunit and small-subunit nuclear rRNA sequences[J]. Mol Biol Evol, 2004, 32(1): 25-38.
[15]Martin J A, Pashey D P. Molecelar systematic analysis of butterfly family and some subfamily relationships (Lepidopterat Papilionidae)[J]. Ann Ent Soc Am, 1992,85:127-135.
[16]程汉良, 彭永兴, 王 芳, 等. 6种帘蛤科贝类18S rRNA基因全序列比较分析[J]. 中国水产科学, 2008,15(4):559-567.
[17]Lakra W S, Goswami M, Gopalakrishnan A. Molecular identification and phylogenetic relationships of seven Indian Sciaenids (Pisces: Perciformes, Sciaenidae) based on 16S rRNA and cytochromecoxidase subunit I mitochondrial genes[J]. Mol Biol Rep, 2009,36(5): 831-839.
[18]崔文涛, 刘立芹, 李红梅, 等. 基于COⅡ基因序列的中国近海12种蛸亚科动物的分子系统进化研究[J]. 渔业科学进展,2013,34(3):21-27.
[19]马春艳, 马凌波, 倪 勇, 等. 基于RAG基因的中国近海13种石首鱼科鱼类系统进化关系[J]. 水产学报, 2012,36(1):9-16.
[20]Cheng Y, Xu T, Shi G, et al. Complete mitochondrial genome of the miiuy croakerMiichthysmiiuy(Perciformes, Sciaenidae) with phylogenetic consideration[J]. Mar Gene, 2010, 3:201-209.
Phylogenetic analysis of miiuy croaker (Miichthysmiiuy) base on 18S rRNA and COⅡ
CHEN Ying-ying , MIAO Liang, LI Ming-yun, ZHANG Xiao-li,GUO Xiao-fei, PAN Na, CHEN Jiong
(Key Laboratory of Applied Marine Biotechnology, Ministry of Education, Ningbo University, Ningbo 315211, China)
In this study, the 18S ribosomal RNA (18S rRNA) and cytochrome oxidase Ⅱ(COⅡ) of miiuy croaker,Miichthysmiiuywere sequenced by PCR and were analyzed for evaluating the phylogenetic position ofM.miiuyand the feasibility of these two gene for molecular phylogeny of Sciaenidae. The results showed that the sequenced 18S rRNA and COⅡ were 1305 bp and 854 bp in length, respectively. The (A+T) content of 18S rRNA and COII were 46.2% and 53.5%, respectively, and the COⅡ showed an anti-G bias (the content of G was 15.2%). The COⅡ sequence included an ORF of 699 bp, which encoded 233 amino acid residues. The NJ phylogenetic tree of 18S rRNA showed that all the Perciformes fish were grouped together into one single clade, the identity of 18S rRNA among Perciformes was>97.8%, which was not suitable for evaluating the phylogenetic position ofM.miiuyin Sciaenidae. The NJ phylogenetic tree of COⅡ showed that the Perciformes fish subdivided into two major polytomous groups, the Sciaenidae formed one group and the other family formed another.M.miiuyhad the closest relationship withBahabataipingensis, the homology of COⅡ betweent the two species was 97.4%. Although in the Sciaenidae topology, there were some different relationships from the traditionally morphological classification and several bootstrap values were <50%, the most relationships of the topology were consistent with tradition. The results of this study could enrich the molecular systematics data ofM.miiuy, and could provide information for phylogeny research ofM.miiuyand the family Sciaenidae.
Miichthysmiiuy; 18S rRNA; COⅡ; phylogenetics
2014-09-01;
2014-09-23
国家高技术研究发展计划(863)项目(批准号:2012AA10A403-4)资助;长江学者和创新团队发展计划项目(IRT0734)资助;浙江省农业新品种选育重大专项(2012C12907)资助
陈莹莹,硕士研究生,研究方向为水产动物苗种繁育;E-mail:843157767@qq.com;
李明云,教授,博士生导师,专业方向为遗传育种、种质资源保护,E-mail:limingyun@nbu.edu.cn。
S917.4;Q953
A
2095-1736(2015)02-0008-04
doi∶10.3969/j.issn.2095-1736.2015.02.008

