绵羊ARHGAP36 全长cDNA的克隆及其蛋白在睾丸精细管中的分布检测
白 雪, 王连庆, 邢万金
(内蒙古大学 生命科学学院, 呼和浩特 010021)
绵羊ARHGAP36 全长cDNA的克隆及其蛋白在睾丸精细管中的分布检测
白 雪, 王连庆, 邢万金
(内蒙古大学 生命科学学院, 呼和浩特 010021)
ARHGAP36是Rho GAPs家族的一员,在成神经管细胞瘤中超表达,可能参与调节细胞增殖。为了研究ARHGAP36在羊精子发生过程中的作用,采用RACE(rapid amplification of cDNA ends)技术从绵羊睾丸组织中克隆了SARHGAP36的cDNA全长序列。内部区域cDNA序列与5′RACE 和3′RACE扩增的两端序列测序后拼接,得到了2698 bp的绵羊ARHGAP36 cDNA全长,其中包含一个长1593 bp的开放阅读框(ORF),编码530 aa,与UniProt中预测的牛ARHGAP36氨基酸序列(A7MB27)同源性为98%,说明我们克隆的cDNA是Rho GAP家族的成员ARHGAP36。对所克隆的绵羊ARHGAP36的氨基酸序列和UniProt数据库中所收集的所有其它ARHGAP成员的氨基酸序列比对分析显示,ARHGAP36没有其它ARHGAP成员都有的标志性“精氨酸指”基序,而它的对应基序中的保守精氨酸残基被苏氨酸替代了,暗示ARHGAP36很可能并不依靠酶催化活性发挥其功能。当把所克隆的cDNA与GenBank中发布的绵羊arhgap36序列比对时发现绵羊arhgap36基因组测序结果的第一个外显子中缺少一小段DNA,导致如果用该基因组序列预测绵羊ARHGAP36的cDNA,无法得到能编码正确氨基酸的cDNA,因此,实验为进一步研究ARHGAP36这个特殊ARHGAP成员的功能提供了正确的cDNA序列。绵羊睾丸总蛋白Western blot检测发现绵羊睾丸内的ARHGAP36有3种长度,免疫组织化学检测发现ARHGAP36蛋白在绵羊睾丸精细管中的精子发生过程中各类细胞中都有分布。
绵羊;ARHGAP36 ;cDNA克隆;RACE;免疫组织化学
Rho GTP酶家族是小GTP酶家族中的成员,是一种受调节的分子开关(regulated molecular switches)。它们与Ras类似,都是在GTP结合的活性形式与GDP结合的失活形式之间来回切换。活化的Rho GTP酶通过激活下游的效应分子,在细胞调节中发挥多种功能,比如细胞骨架的重排,细胞周期的进行及细胞迁移等[1]。到目前为止,已经在哺乳动物细胞中发现20种Rho GTP激酶家族的成员。Rho GTP酶共有RHO、RAC及CDC42 3种亚型,它们通过不同的方式对细胞骨架的肌动蛋白进行调节:在成纤维细胞中,RHO的活化促使黏着斑复合体的形成,并使肌动蛋白微丝收缩[2];RAC的活化对皮层肌动蛋白聚合有非常重要的作用[3];而CDC42则促使丝状伪足的形成[4]。此外,RHO、RAC及CDC42需要经过细胞周期的G1期才能被激活。其中,RHO的调控则更严格,它需要G1中期周期蛋白D1表达的介导[5]。Rho GTP酶的活性受到GTP和GDP的调节,当与GTP结合时,Rho GTP酶被激活,但当所结合的GTP水解成GDP,Rho GTP酶失去活性。Rho GTP酶的活性受3类因子调节[6]。一类是GTP酶激活蛋白(GTPase-activating proteins, GAPs),也称为Rho GAP或ARHGAP,催化与Rho GTP酶结合的GTP水解成GDP,使Rho GTP酶失活[7-8];另一类是鸟苷酸交换因子(guanine nucleotide exchange factors, GEFs),作用相反,催化与Rho GTP酶结合的GDP转化成GTP,激活Rho GTP酶[9];此外还有鸟苷酸解离抑制因子(guanine nucleotide dissociation inhibitors, GDIs)[6, 10],主要作用可能是促使Rho GTP酶离开细胞膜。ARHGAP是一个大家族,自1991年[12]报道第一个Rho GAP蛋白Bcr(breakpoint cluster region protein)起,已经发现约70个成员[12],HUGO 基因命名委员会(HGNC)已经登记了近50个成员(http://www.genenames.org/genefamilies/ARHGAP)。所有的Rho GAP蛋白都包含一个大约由150个氨基酸组成的Rho GAP结构域[13],它与该家族的其它成员至少具有20%的同源性[14]。Rho GAP的结构域包括9个α螺旋,并且在其酶活性结构域中含有一个保守的精氨酸残基[15-16]。Rho GAP与靶蛋白GTP-Rho结合具有序列特异性,对活化特异的Rho GTP酶具有重要作用,尤其是与GTP酶的活性中心直接结合的精氨酸残基[17]。
由于Rho GTP酶家族具有广泛的细胞效应,ARHGAP家族作为Rho GTP酶的负调节因子对于Rho GTP酶在细胞生长、增殖、分化等过程中正确发挥作用至关重要[18],调节胚胎发育[19-20]、神经发育[21]、细胞分裂和分化[22-23]。缺失某些Rho GAPs会导致显著的变异表型[24]。如arhgap31突变会导致Adams-Oliver综合症[25],CHN1(arhgap2)突变引起 isolated Duane retraction综合症[26]。ARHGAP6在人体的多种组织中表达,它的缺失会引发线性皮肤缺损小眼畸形(microphthalmia with linear skin defects, MLS)[27]。在小鼠的ARHGAP6的缺失会导致牙釉质的缺损[28]。ARHGAP的非酶结构域的作用也逐渐被发现,可能与其在细胞中的定位及信号传导中的特殊作用有关[15]。
ARHGAP36是Rho GAPs家族的一员,最近发现ARHGAP36的超表达与人的成神经管细胞瘤(medulloblastoma, MB)有关[29]。ARHGAP36在鼠成神经管细胞瘤中也超表达,且能激活转录因子Gli 并影响下游基因表达[30]。这些结果显示ARHGAP36在对于调节细胞增殖具有重要作用。本实验室先前测序绵羊睾丸转录物组,发现有ARHGAP36片段,推测ARHGAP36可能在精子发生过程中有某种作用。经查阅GenBank发布的绵羊基因组序列,发现绵羊的arhgap36(Gene ID: 100913153)序列中尚存在gap,难以预测出可信的cDNA和氨基酸序列。NCBI预测了59个物种的arhgap36(FLJ30058)直系同源基因序列(http://www.ncbi.nlm.nih.gov/gene/?Term=ortholog_gene_158763[group]),UniProt蛋白数据库里也收集了32条ARHGAP36氨基酸序列(http://www.uniprot.org/uniprot/?query=arhgap36&sort=score),但都是根据基因组序列预测的,尚无来自于实验克隆的cDNA编码序列。UniProt预测了两条绵羊ARHGAP36 氨基酸序列,一条为547 aa(W5PTE2),另一条为536 aa(W5PTE3),差异很大,无法确认哪一条正确。为了深入研究ARHGAP36对绵羊精子发生的影响,本文用RACE(rapid amplification of cDNA ends)技术克隆了绵羊ARHGAP36 的cDNA全长,为阐明ARHGAP36在羊睾丸组织中的功能积累资料。
1 材料与方法
1.1 实验动物及其组织
内蒙古绵羊公羊屠宰后立即采集其睾丸,保存在-80°C冰箱中备用。
1.2 试剂
RNAiso Reagent、DNA Ligation Kit、RNA PCR Kit(AMV)Ver.3.0、3 U/μL rTaq DNA聚合酶、PCR缓冲液(10×,MgCl2free)、250 bp DNA Ladder Marker、2.5 mmol/L MgCl2、dNTPs等均购自宝生物工程(大连)有限公司。SMARTTM RACE cDNA Amplification Kit 购自Clontech公司(Catalog NO. 634914)。DNA Marker II、DNA Marker III和2×Taq PCR Master Mix购自天根生化科技(北京)有限公司。AxyPrep DNA Gel Extraction Kit 购自AXYGEN Biosciences公司。pMD19-T cloning Vector购自宝生物工程(大连)有限公司。辣根过氧化物酶标记的兔抗小鼠IgG购自上海圻明生物科技有限公司。E.coliDH5α由内蒙古大学生命科学学院基因工程实验室保存。
1.3 公羊睾丸组织总RNA的提取及cDNA 合成
所用的器皿均用0.1% DEPC水37℃下浸泡12 h,然后121℃灭菌30 min,除去残余的DEPC,置于干燥箱中烘干。RNA提取所用溶液均用0.1% DEPC水配制。称取羊睾丸组织50~100 mg,,按照TaKaRa RNAiso Reagent试剂盒操作步骤提取公羊睾丸组织的总RNA。
以总RNA为模板,oligod(T)为引物,利用AMV反转录酶合成cDNA的第一链。
1.4 绵羊ARHGAP36 cDNA内部区域的克隆
1.4.1 PCR引物设计 根据GenBank上发布的牛(NM_001101988)、黑猩猩(XM_521264)、马(XM_001491324)、人(NM_144967)、恒河猴(XM_001094001)、小鼠(NM_001081123)和大鼠(XM_229133)的ARHGAP36 mRNA序列,使用CLC Sequence Viewer软件进行基因序列的同源性比对,在这几种哺乳动物ARHGAP36 cDNA序列的内部找到4处高度保守的区段。因为该基因的cDNA的预期比较长(>2 kb),我们根据这4个保守区的序列设计了两条上游引物及两条下游引物(位置见图2),分段克隆绵羊ARHGAP36 cDNA的内部区域,然后根据重叠区拼接。两组PCR的引物分别是:
F1 5′-AGGAGCCCCAGGACACAAC-3′(相当于牛ARHGAP36 cDNA的60~78 bp);
R1 5′-CCCTTCTTCAGGAGAGCAG-3′ (相当于牛ARHGAP36 cDNA的1109~1127 bp);
F2 5′-ATTGCTGGTTTACCTGATG-3′ (相当于牛ARHGAP36 cDNA的945~963 bp);
R2 5′-TAGGAGGAGGGTAGGAAAG-3′ (相当于牛ARHGAP36 cDNA的1995~2013 bp)
羊与牛是同科动物,基因的同源性极高,因此我们根据牛的ARHGAP36 cDNA的预测序列,预期PCR产物长度分别约1067 bp和1068 bp。两段PCR产物约有183 bp重叠区,用于拼接。引物由宝生物工程(大连)公司合成。
1.4.2 PCR扩增 扩增F1-R1区段的PCR反应条件是94℃预变性3 min;94℃变性30 s,55℃退火30 s,72℃延伸2 min,共35个循环;最后72℃延伸10 min。扩增F2-R2区段的PCR反应条件是94℃预变性3 min;94℃变性30 s,49℃退火30 s,72℃延伸2 min,共35个循环;最后72℃延伸10 min。
扩增的PCR产物回收后与pMD-19T载体连接转化DH5α感受态大肠杆菌,提取质粒后PCR鉴定,分别选择3个克隆的质粒送交北京三博远志公司测序。
1.4.3 用3′RACE和5′RACE分别扩增绵羊ARHGAP36 cDNA3′末端和5′末端
RACE引物设计 SMARTTMRACE cDNA Amplification Kit中给了两条RACE公用引物UPM和NUP序列,其中NUP实际上是UPM的3端的半段,做套嵌PCR。为了增加PCR扩增的效率和特异性,我们额外设计了一条RACE引物,此引物位于公用引物UPM的内部,并且与另一条公用引物NUP的5′端重叠,用作5′RACR的上游引物和3′RACE的下游引物。序列如下:
UPM:5′-CTAATACGACTCACTATAGGGCAAGCAGTGGTATCAACGCAGAGT-3′
NUP:5′-AAGCAGTGGTATCAACGCAGAGT-3′
UVU:5′-TAGGGCAAGCAGTGGTATC-3′
此外我们根据上述克隆测序后获得的绵羊ARHGAP36 cDNA内部区域,设计了5′RACE的下游引物SP5,位于上述克隆拼接后的绵羊ARHGAP36 cDNA片段的149~173 bp;设计的3′RACE的上游引物SP3位于上述克隆拼接后的绵羊ARHGAP36 cDNA片段的-184~-208 bp;两条引物的位置见图2,序列如下:
SP5:5′-GGTGTTTGGACGGTCAGTAGGTAG-3′
SP3:5′-CTGGTGCTACCTTGCCTCTCTCTT-3′
首先按照操作该试剂盒的操作说明,用总RNA以及试剂盒自带的SMART II A oligo、5′-CDS引物和3′-CDS引物分别合成5′RACE和3′RACE用的cDNA第一链。
RACE扩增 5′RACE:以上述5′RACE的cDNA第一链为模板,用UPM引物和SP5引物进行一轮Touch-Down PCR,条件为94℃变性30 s,72℃退火1 min,延伸30 s,5个循环;94℃变性30 s,70℃退火30 s,72℃ 1 min,5个循环;94℃变性30 s,68℃退失30 s,72℃延伸1 min,30个循环;最后72℃延伸10 min。以此PCR产物为模板,用NUP引物和SP5引物进行第二轮巢式PCR,反应条件同上。以此巢式PCR产物为模板,用UVU引物和SP5引物进行第3次PCR,反应条件为94℃变性30 s,54℃退火30 s,72℃延伸1 min,35个循环;最后72℃延伸10 min。将第3次PCR产物进行电泳观察是否有带,如有带则回收与pMD-19T载体连接。
为了鉴定扩增出的产物是否包含上述已经克隆的ARHGAP36 cDNA片段的5′端一部分,我们另外设计一条靠近上述片段5′末端的内部引物,用于最后PCR鉴定。设计的鉴定引物SPa(5′-AGTTCGTGGTAAGCAGCCTC-3′)位于F1引物下游66~86 bp,位于SP5的上游-44~-64 bp,因此F1和SPa引物均在5′RACE产物的内部,如果用F1-SPa引物做PCR鉴定的话,预期产物105 bp。如果出现此预期产物,分别选择3个克隆的质粒送交北京三博远志公司测序。
3′RACE: 与上述5′RACE方案相似,但上游引物改用上述已克隆的绵羊ARHGAP36 cDNA片段3′端的SP3引物。以3′RACE的cDNA第一链为模板,SP3引物与UPM引物进行第一轮PCR,用SP3引物与NUP引物进行第二轮巢式PCR,SP3引物与UVU引物进行第3轮PCR。为了鉴定扩增出的产物是否包含上述已经克隆的ARHGAP36 cDNA片段的3′端一部分,我们另外设计一条靠近上述片段3′末端的内部引物,用于最后PCR鉴定。设计的鉴定引物SPb(5′ -AGCCGTCCCTTTAATTGATG-3′) 位于R2引物上游-91~-111 bp,位于SP3下游的32~52 bp。即SPb和R2引物实际上位于3′RACE产物的内部,用SPb-R2引物进行PCR鉴定,预期出现129 bp产物。如果出现此预期产物,分别选择3个克隆的质粒送交北京三博远志公司测序。
将上述测序结果拼接后即可得到绵羊ARHGAP36 cDNA全长。
1.5 ARHGAP36抗体制备即在绵羊睾丸组织中的免疫组织化学检测
以克隆的绵羊ARHGAP36 cDNA的F2-R2片段为模板,用上游引物5′-CGGATCCATGATTGATAACTGGGATGTC-3′(下划线为添加的BamH I酶切位点)和下游引物5′-GGAATTCAGGAAAGAAGTAGCCGATGCC-3′(下划线为添加的EcoR I酶切位点)扩增绵羊ARHGAP36中部的402 bp cDNA片段,扩增条件为94℃变性30 s,53℃退火30 s,72℃延伸1 min,回收后BamH I与EcoR I双酶切,插入原核表达载体pGEX-4T3中,IPTG诱导表达后用GST柱子纯化GST-ARHGAP36融合蛋白(其分子量约为45 ku),然后免疫小鼠,制备小鼠抗GST-ARHGAP36腹水多克隆抗体。用制备的多克隆抗体为一抗,Western blot法检测该抗体能否特异性识别大肠杆菌中表达的GST-ARHGAP36融合蛋白和绵羊睾丸总蛋白中的内源性ARHGAP36蛋白。
取雄性绵羊睾丸组织,4%中性多聚甲醛4℃固定过夜,经脱水、透明、渗蜡、包埋后,进行5μm连续切片。将连续切片贴片于预先处理好的防脱载玻片上,采用二次展片法展片。60℃恒温箱中烤片2 h,以本实验室自制的小鼠抗绵羊GST-ARHGAP 36腹水多克隆抗体(1∶50)为一抗,辣根过氧化物酶标记的兔抗小鼠IgG为二抗,同时以未免疫的小鼠腹水以及GST单克隆抗体为一抗作为对照,按照免疫组化试剂盒说明进行免疫组化染色。
2 结果
以绵羊睾丸总RNA反转录的总cDNA为模板,用F1-R1、F2-R2引物进行PCR扩增均获得约1100 bp的扩增产物,与预期大小相符(图1 A泳道2,4)。经过测序拼接后得到1957 bp(包含F1和R2引物)的绵羊ARHGAP36 cDNA内部区片段,经BLAST发现与GenBank中预测的牛ARHGAP36 cDNA(NM_001101988)的同源性达到98%,证明我们获得的绵羊ARHGAP36 cDNA是正确的。
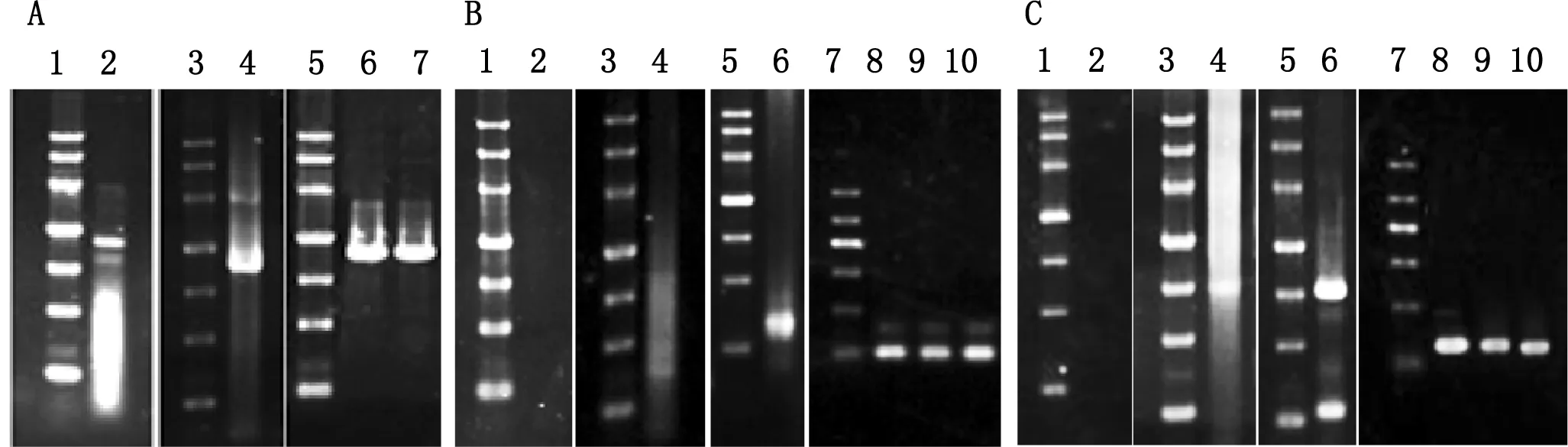
图1 绵羊ARHGAP36 cDNA的克隆及PCR鉴定
A—内部区的克隆与鉴定。泳道1、3、5:DNA Marker III (4500 bp, 3000 bp, 2000 bp, 1200 bp, 800 bp, 500 bp, 300 bp);泳道2:F1-R1引物的PCR产物;泳道4:F2-R2引物的PCR产物;泳道6:F1-R1的PCR产物与pMD19-T连接后的PCR鉴定;泳道7:F2-R2的PCR产物与pMD19-T连接后的PCR鉴定。B—5′末端的克隆。泳道1、3、5:DNA Marker III;泳道7:DNA Marker II(1200 bp, 900 bp, 700 bp, 500 bp, 300 bp, 100 bp);泳道2:5′RACE第一轮PCR反应产物;泳道4:5′RACE第二轮PCR反应产物;泳道6:5′RACE第三轮PCR反应产物;泳道8~10:用F1-SPa引物鉴定5′RACE第三轮PCR产物与pMD19-T连接成的3个重组质粒。C—3′末端的克隆。泳道1、3、5:DNA Marker III;泳道7:DNA Marker II;泳道2:3′RACE第一轮PCR反应产物;泳道4:3′RACE第二轮PCR反应产物;泳道6:3′RACE第三轮PCR反应产物;泳道8~10:用SPb-R4引物鉴定3′RACE第三轮PCR产物与pMD19-T连接成的3个重组质粒。
然后我们用绵羊的睾丸总RNA,按照Clontech RACE试剂盒的说明合成用于5′RACE和3′RACE的相应的cDNA第一链,各进行1次Touch-Down PCR,随后继续2次Nest PCR,最终扩增得到了绵羊ARHGAP36 cDNA的5′和3′末端部分,其中5′RACE的扩增片段约250 bp(图1B泳道6),而3′RACE的扩增片段约为700 bp(图1C泳道6)。
将回收的5′RACE 和3′RACE的扩增产物分别与pMD19-T连接,转化DH5α,随机各挑取3个克隆提取质粒,分别用F1-SPa引物和SPb-R2引物进行PCR鉴定。3个5′RACE克隆用F1-SPa引物进行PCR鉴定,均出现约100 bp产物(图1B泳道8~10);3个3′RACE克隆用SPb-R2引物进行PCR鉴定,均出现约130 bp产物(图1C泳道8~10),均与预期长度相同。

图2 用RACE技术克隆的绵羊ARHGAP36 cDNA全长
下划线及其下的字母表示本文所设计的克隆引物位置;斜体字母是5′非翻译区和3′非翻译区;正体大写字母和小写字母分别表示对应于相邻的外显子; ATG(49-51)是起始密码,TAG(1639-1641)是终止密码;灰色背景的aataaa(2671-2676)是PolyA加尾信号。
5′RACE 和3′RACE的扩增产物测序后与上述克隆得到的ARHGAP36 cDNA内部片段拼接,除去两头的RACE引物后得到了2698 bp的绵羊ARHGAP36 cDNA全长(图2)。其中包含一个1593 bp的开放阅读框(ORF),编码530 aa,与UniProt数据库中预测的牛ARHGAP36氨基酸序列(A7MB27)同源性为98%,说明我们克隆的cDNA是编码Rho GAP家族的成员ARHGAP36,序列已提交NCBI,登录号为JQ736806。根据牛ARHGAP36氨基酸序列(A7MB27)注释,绵羊ARHGAP36的1~19 aa为信号肽, 209~409 aa是一个Rho GAP结构域,132~168 aa是一个富含精氨酸区。237~241 aa(VGIFT)是一个类似GAP结构域的活性部位的保守基序。
为了检测ARHGAP36蛋白在精子发生过程中的表达情况,我们自制了小鼠抗绵羊ARHGAP36的腹水多克隆抗体,并用Western blot法鉴定了该抗体能否识别羊ARHGAP36蛋白。如图3A显示,本抗体既能识别大肠杆菌表达并纯化的约45 ku的GST-ARHGAP36融合蛋白(图3A泳道2),也能识别转化了pGEX-4T3的大肠杆菌所诱导表达出的26 ku的GST标签蛋白(图3A泳道3)。用该抗体作一抗Western blot检测绵羊睾丸总蛋白,发现能在55~72 ku之间看到两条明显的识别信号(图3B泳道2),分子量与绵羊ARHGAP36除去信号肽后的成熟肽57.8 ku一致。在55 ku处还有一条识别带(图3B泳道2),提示绵羊ARHGAP36在睾丸内可能存在不同长度的形式。
用该抗体对绵羊睾丸组织切片进行免疫组织化学染色发现ARHGAP36蛋白存在于精细管内的各层次的细胞中(图3D),说明在精子发生过程中,初级精母细胞到圆形精子都在表达ARHGAP36。
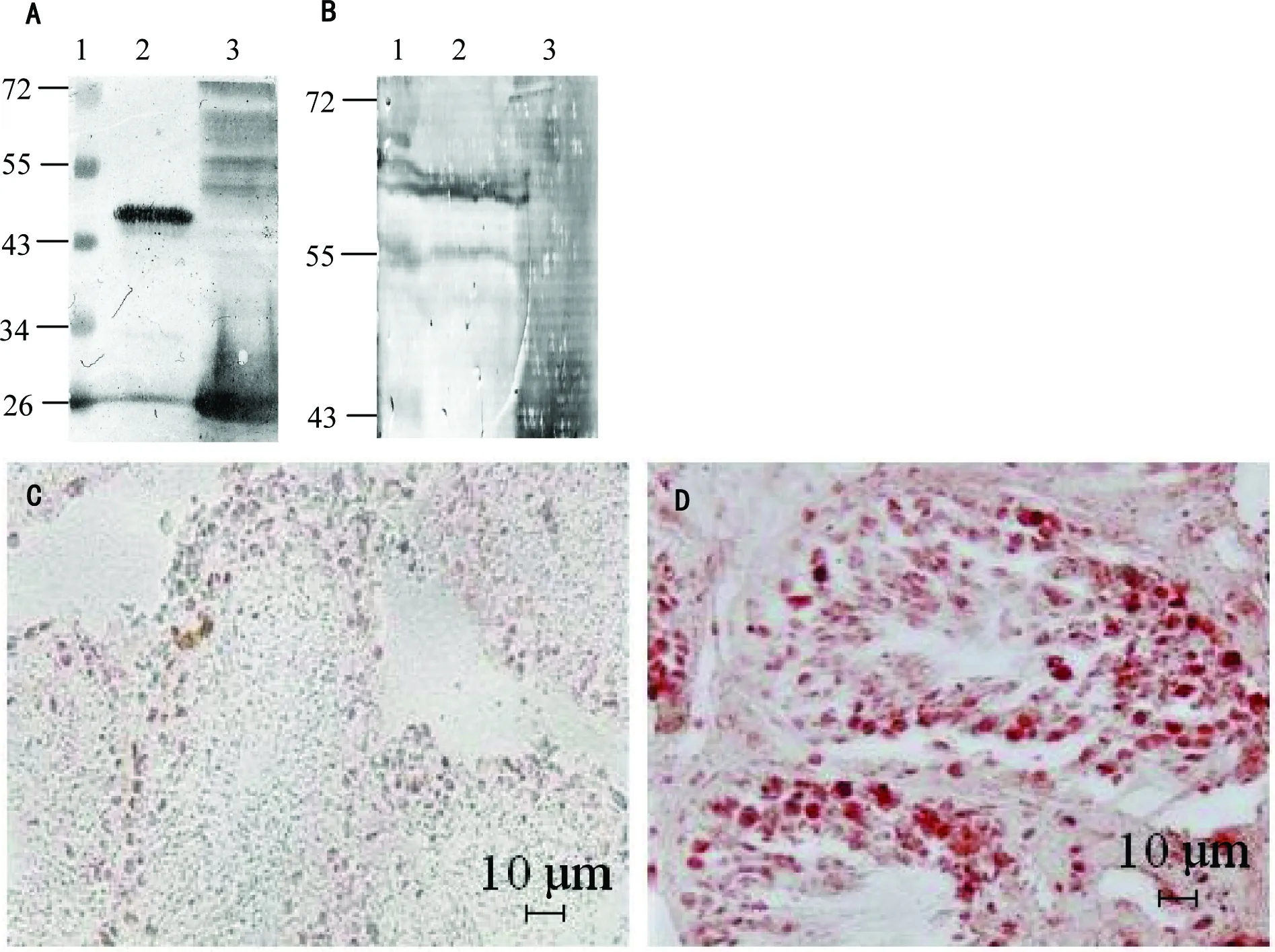
图3绵羊睾丸总蛋白中ARHGAP36的Western blot检测和组织切片中ARHGAP36 的免疫组织化学检测
A—用自制的小鼠抗羊ARHGAP36多克隆抗体做一抗进行Western blot检测大肠杆菌表达的纯化的GST-ARHGAP36蛋白。泳道1:蛋白分子量Marker(Ku);泳道2:用于免疫小鼠制备抗体的纯化的GST-ARHGAP36,分子量约45 ku;泳道3:IPTG诱导后的表达GST的大肠杆菌总蛋白。B—用自制的小鼠抗羊ARHGAP36多克隆抗体做一抗进行Western blot检测绵羊睾丸总蛋白中的内源性ARHGAP36。泳道1:蛋白分子量Marker(Ku);泳道2:绵羊睾丸总蛋白,体内的成熟ARHGAP36蛋白理论分子量约57.8 ku;泳道3:IPTG诱导后的表达GST的大肠杆菌总蛋白。C—GST单克隆抗体作为一抗的免疫组织化学对照。D—小鼠抗羊GST-ARHGAP36腹水多克隆抗体做一抗的免疫组化。红色染色信号为ARHGAP36蛋白所在位置,D图中精细管横截面内的精子发生过程不同时期的细胞内均有红色信号。
3 讨论
UniProt数据库根据International Sheep Genomics Consortium发布的绵羊基因组序列[31]预测了两条绵羊ARHGAP36 氨基酸序列,一条为547 aa(W5PTE2),另一条为536 aa(W5PTE3),两条之间的主要差异是W5PTE2在N端比后者多44个氨基酸(MGGCIPFLKTARTRYPRIMPPLLLLSALIFLVNVLGHNSNRRAK),但在距离C端51 aa之前缺失一个36个氨基酸的片段(ITFFKNQSQPGQVMDYMSFLESLKNNACFRLSMKFC)。本实验克隆的绵羊ARHGAP36 cDNA编码530 aa,与两种预测版本的长度均不同,氨基酸序列比对后发现,与W5PTE2更相似。W5PTE2去掉N端的18个氨基端(MGGCIPFLKTARTRYPRI)、在第18 aa后插入的GAPG 4个氨基酸、第88 aa后去掉CSG 3个氨基酸就与本实验克隆的cDNA推导氨基酸序列相同。
UniProt为何预测出差异如此之大且均有错误的两种绵羊ARHGAP36蛋白呢?我们仔细检查了GenBank数据库(NW_004080190)和Ensembl数据库里(ENSOARG00000012814)里绵羊arhgap36的基因组DNA序列,发现都还不完整,都有376 bp的gap。把我们克隆到的绵羊ARHGAP36 cDNA与这两个数据库的基因组测序结果比对,发现该基因组DNA中有11段匹配我们克隆的cDNA,即绵羊arhgap36由11个外显子组成,10个内含子都是GT-AG内含子。基因组DNA中的gap位于第一个内含子内部,不影响对该基因的编码区解读。第2~11外显子拼接的序列与我们克隆的cDNA完全相同,但第1外显子内部缺少我们克隆的第99~110 bp的11个碱基 (GGAGGAGCCCC,位于本实验设计的F1引物内部)。基因组DNA中如果缺失这11个碱基将会导致翻译的时候第1外显子内部就出现终止密码,因此,GenBank中现有的绵羊基因组序列的这个小错误导致用生物信息学软件无法推导出正确的绵羊ARHGAP36 cDNA。这应该就是UniProt中预测ARHGAP36氨基酸序列出现N端混乱的原因。
绵羊arhgap36中缺失的这11 bp正好位于我们根据比对牛、鼠、人、黑猩猩等生物的ARHGAP36 cDNA找到的高度保守区之一,我们也据此设计了F1引物。虽然这11 bp在克隆内部区的时候是我们的一条上游引物,但在5′RACE扩增的时候属于内部序列,5′RACE产物的测序结果也证实这11 bp真实存在于ARHGAP36 cDNA 5′区域的内部。证明这11 bp在绵羊arhgap36中应该存在,在GenBank绵羊基因组中的缺失应该是基因组测序或者组装的错误。
ARHGAP家族的特征结构是Rho GAP结构域。根据Rho GAP家族的成员RhoGAP、p190、bcr、chimerin、3BP-1及p85[32]的研究结果,我们对所克隆的绵羊SARHGAP36的Rho GAP结构域进行了分析。该结构域含有9个螺旋结构,含有7个G蛋白结合位点的螺旋(图4 黑色框),2个非G蛋白结合位点的螺旋(图4白色框)。它含有增强GTP激酶活性的位点Gly238,Leu288,Leu318,Met357,Asn361,前4个氨基酸残基位于B-F口袋(pocket)附近,第5个位于F螺旋上。
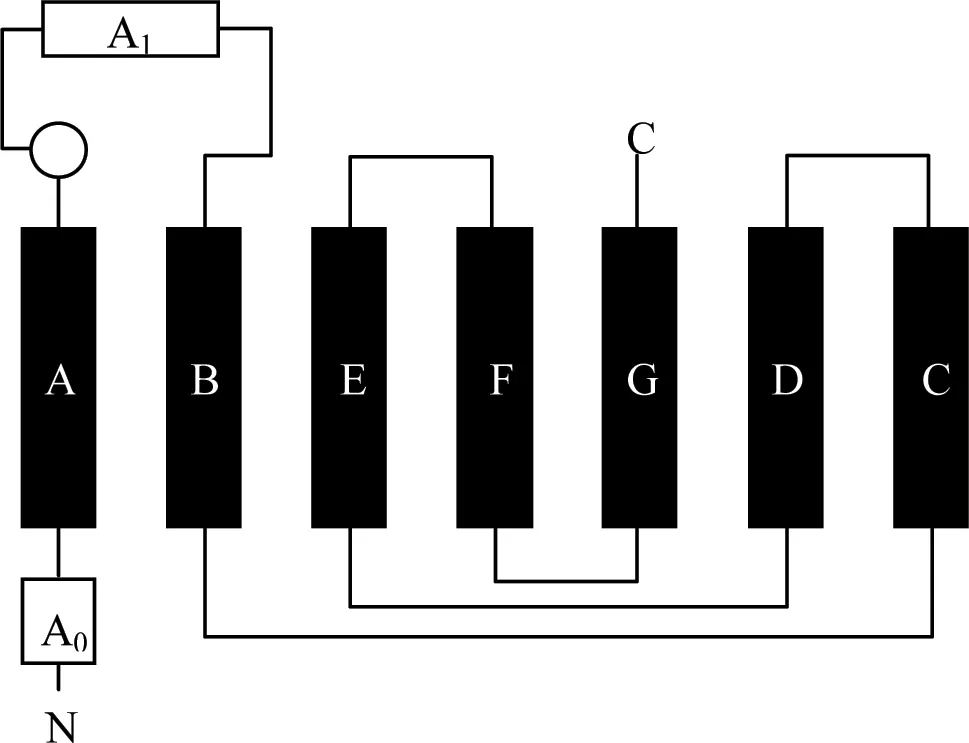
图4 绵羊ARHGAP36的Rho GAP结构域
9个α螺旋用方框表示,其中,A~C 7个黑色方框表示该螺旋含G蛋白结合的位点,A0和A1两个白色方框表示该螺旋不含G蛋白结合的位点;N和C分别表示肽链的两端。
ARHGAP家族的Rho GAP结构域中有一个保守的精氨酸(R)具有催化作用,被称为“精氨酸指”(arginine finger)[33],是该家族的标志性基序,如ARHGAP1的R282(保守序列为GIFR)。我们比对了本实验所克隆的绵羊ARHGAP36和UniProt中目前收集的全部ARHGAP36氨基酸序列,发现ARHGAP36在相当于ARHGAP1中的“精氨酸指”的保守精氨酸部位全都是苏氨酸(T)(保守序列为GIFT),即用苏氨酸取代了精氨酸。为了检查这个精氨酸被替换掉的现象在ARHGAP家族的其它成员中是否也存在,我们进一步比对了UniProt数据库收录的全部约40个ARHGAP成员在不同物种中的氨基酸序列,重点检查了相当于ARHGAP1中的“精氨酸指”部位,发现各ARHGAP成员的直系同源基因的氨基酸序列非常保守,成员之间(旁系同源基因)的序列差异较大,但在相当于ARHGAP1的“精氨酸指”部位的氨基酸序列均很保守(表1)。从表1可以看出,所比对的38个ARHGAP成员中除了ARHGAP36外全都有“精氨酸指”(保守序列为*G**R*)结构,只有ARHGAP36的对应部位的精氨酸被苏氨酸替换,暗示ARHGAP36的生物学功能并不需要酶催化活性。最近的一个报道也用氨基酸突变实验证明了这个精氨酸替换和其它两个与催化活性有关的氨基酸突变都不影响ARHGAP36激活Gli转录因子的作用,而其N末端可能与ARHGAP36的亚细胞定位与功能有关[30]。由此推测ARHGAP36可能并不以其催化活性发挥作用,也许是作为细胞信号通路的一个节点,目前也已经鉴定出ARHGAP36与sonic hedgehog (SHH) / patched (PTCH) 信号通路中的Sufu相互作用[30],该信号通路与人的发育有关。

表1 多种动物的38个ARHGAP家族成员的“精氨酸指”基序的保守氨基酸序列比对结果
与ARHGAP右侧平行的数字表示ARHGAP家族的成员序号;成员序号下方、“比对数目”右侧的数字是本文所使用UniProt数据库中对应的ARHGAP成员的物种数;其中ARHGAP16、37、38、41和43 5个成员在UniProt中还没有预测的氨基酸序列。
有关ARHGAP36的生物学功能研究还没有更多的报道,未见关于ARHGAP家族成员在配子发生过程中的表达与生物学功能的研究报道。本实验室在检测绵羊睾丸组织的转录物时发现该基因的mRNA片段,鉴于ARHGAP家族与细胞分裂有关[18],生物信息学分析显示ARHGAP36是唯一不具有“精氨酸指”的ARHGAP家族成员,因此可能并不具有催化活性,我们猜测ARHGAP36在精子发生过程中可能发挥某种特殊作用。绵羊睾丸总蛋白Western blot检测发现绵羊睾丸内的ARHGAP36可能有3种长度(图3B泳道2),初步的免疫组织化学检测证实ARHGAP36在精子发生过程中的细胞内均表达(图3D),目前尚不清楚睾丸内的ARHGAP36有何种功能,本实验室正在研究它在睾丸中与哪种蛋白有相互作用。
4 结论
本文首次克隆了绵羊ARHGAP36的全长cDNA序列,并发现了该基因的第一外显子在GenBank绵羊基因组序列组装的错误。初步检测了ARHGAP36蛋白在绵羊睾丸和精子发生过程细胞中的表达,为下一步深入研究ARHGAP36在精子发生过程中的作用奠定了基础。
[1]Etienne-Manneville S, Hall A. Rho GTPases in cell biology[J]. Nature, 2002, 420: 629-635.
[2]Ridley A J, Hall A. The small GTP-binding protein rho regulates the assembly of focal adhesions and actin stress fibers in response to growth factors[J]. Cell, 1992, 70(3): 389-399.
[3]Ridley A J, Paterson H F, Johnston C L, et al. The small GTP-binding protein rac regulates growth factor-induced membrane ruffling[J]. Cell, 1992, 70(3): 401-410.
[4]Nobes C D, Hall A. Rho, rac, and cdc42 GTPases regulate the assembly of multimolecular focal complexes associated with actin stress fibers, lamellipodia, and filopodia[J]. Cell, 1995, 81: 53-62.
[5]Welsh C F, Roovers K, Villanueva J, et al. Timing of cyclin D1 expression within G1 phase is controlled by Rho[J]. Nat Cell Biol, 2001(3): 950-957.
[6]Tcherkezian J, Lamarche-Vane N. Current knowledge of the large RhoGAP family of proteins[J]. Biol Cell, 2007, 99: 67-86.
[7]Lamarche N, Hall A. GAPs for rho-related GTPases[J]. Trends Genet, 1994, 10:436-440.
[8]Scheffzek K, Ahmadian M R, Wittinghofer A. GTPase-activating proteins: helping hands to complement an active site[J]. Trends Biochem Sci, 1998, 23: 257-262.
[9]Cerione R A, Zheng Y. The Dbl family of oncogenes[J]. Curr Opin Cell Biol, 1996, 8: 216-222.
[10]Olofsson B. Rho guanine dissociation inhibitors: Pivotal molecules in cellular signaling[J]. Cell Signal, 1999, 11: 545-554.
[11]Diekmann D, Brill S, Garrett M D, et al. Bcr encodes a GTPase-activating protein for p21rac[J]. Nature, 1991, 351: 400-402.
[12]Bernards A. GAPs galore! A survey of putative Ras superfamily GTPase activating proteins in man and Drosophila[J]. Biochim Biophys Acta, 2003, 1603: 47-82.
[13]Peck J, Douglas I V G, Wu C H, et al. Human RhoGAP domain-containing proteins: structure, function and evolutionary relationships[J]. FEBS Lett, 2002, 528: 27-34.
[14]Moon S Y, Zheng Y. Rho GTPase-activating proteins in cell regulation[J]. Trends Cell Biol, 2003, 13: 13-22.
[15]Gamblin S J, Smerdon S J. GTPase-activating proteins and their complexes[J]. Curr Opin Struct Biol, 1998, 8: 195-201.
[16]Graham D L, Eccleston J F, Lowe P N. The conserved arginine in rho-GTPase-activating protein is essential for efficient catalysis but not for complex formation with Rho.GDP and aluminum fluoride[J]. Biochemistry, 1999, 38(3): 985-991.
[17]Nassar N, Hoffman G R, Manor D, et al. Structures of Cdc42 bound to the active andcatalytically compromised forms of Cdc42GAP[J]. Nat Struct Biol, 1998, 5 (12): 1047-1052.
[18]Ligeti E, Welti S, Scheffzek K. Inhibition and termination of physiological responses by GTPase activating proteins[J]. Physiol Rev, 2012, 92: 237-272.
[19]Boissel L, Houssin N, Chikh A, et al. Recruitment of Cdc42 through the GAP domain of RLIP participates in remodeling of the actin cytoskeleton and is involved in Xenopus gastrulation[J]. Dev Biol, 2007, 312: 331-343.
[20]Kim J, Shim S, Choi S C, et al. A putative Xenopus Rho-GTPase activating protein (XrGAP) gene is expressed in the notochord and brain during the early embryogenesis[J]. Gene Expr Patterns, 2003, 3: 219-223.
[21]Tolias K F, Duman J G, Um K. Control of synapse development and plasticity by Rho GTPase regulatory proteins[J]. Prog Neurobiol, 2011, 94: 133-148.
[22]Mishima M, Glotzer M. Cytokinesis: a logical GAP[J]. Curr Biol, 2003, 13: R589-591.
[23]Hirose K, Nosaka T. Role of MgcRacGAP/Cyk4 as a regulator of the small GTPase Rho family in cytokinesis and cell differentiation[J]. Cell Struct Funct, 2001, 26: 645-651.
[24]Csépányi-Kömi R, Lévay M, Ligeti E. Small G proteins and their regulators in cellular signaling[J]. Mol Cell Endocrinol, 2012, 353: 10-20.
[25]Southgate L, Machado R D, Snape K M, et al. Gain-of-function mutations of ARHGAP31, a Cdc42/Rac1 GTPase regulator, cause syndromic cutis aplasia and limb anomalies[J]. Am J Hum Genet, 2011, 88(5): 574-585.
[26]Miyake N, Chilton J, Psatha M, et al. Human CHN1 mutations hyperactivate alpha2-chimaerin and cause Duane's retraction syndrome[J]. Science, 2008, 321(5890): 839-843.
[27]Kutsche K, Werner W, Bartsch O, et al. Microphthalmia with linear skin defects syndrome (MLS): a male with a mosaic paracentric inversion of Xp[J]. Cytogenet Genome Res, 2002, 99 (1-4): 297-302.
[28]Prakash S K, Gibson C W, Wright J T, et al. Tooth enamel defects in mice with a deletion at the Arhgap 6/Amel X locus[J]. Calcif Tissue Int, 2005, 77 (1): 23-29.
[29]Jackson P J, Larson J D, Beckmann D A, et al. The role of Arhgap36 as a novel driver in high-risk human medulloblastoma. Abstracts from the 4th Quadrennial Meeting of the World Federation of Neuro-Oncology held in conjunction with the 18th Annual Meeting of the Society for Neuro-Oncology[M]. San Francisco, California, 2013, CB-032.
[30]Rack P G, Ni J, Payumo A Y, et al. Arhgap36-dependent activation of Gli transcription factors[J]. Proc Natl Acad Sci USA, 2014 [Epub ahead of print]
[31]Archibald A L, Cockett N E, Dalrymple B P, et al. The sheep genome reference sequence: a work in progress[J]. Anim Genet. 2010, 41(5): 449-453.
[32]Barrett T, Xiao B, Dodson E J, et al. The structure of the GTPase-activating domain from p50 rhoGAP[J]. Nature, 1997, 385: 458-461.
[33]Rittinger K, Walker P A, Eccleston J F, et al. Crystal structure of a small G protein in complex with the GTPase-activating protein rhoGAP[J]. Nature, 1997, 388(6643): 693-697.
Molecular cloning of the sheep ARHGAP36 full length cDNA and the protein distribution in seminiferous tubules
BAI Xue, WANG Lian-qing, XING Wan-jin
(School of Life Sciences, Inner Mongolia University, Hohhot 010021, China)
ARHGAP36, a member of Rho GAP(GTPase-activating proteins) family, is over expressed in medulloblastoma, and thus may be implicated in regulation of cell proliferation. To elucidate its function in sheep spermatogenesis, the full length cDNA of sheep ARHGAP36 was cloned using RACE (rapid amplification of cDNA ends) technique from sheep testicular tissue. A cDNA of 2698 bp was obtained after joining the 5′-and 3′ RACE fragments with the middle region cloned and sequenced independently. An ORF (open reading frame) of 1593 bp encoding a peptide of 530 aa with a homology of 98% with the predicted cattle ARHGAP36 (A7MB27) in the UniProt was found in this cDNA. Analysis of the all predicted amino acid sequence of ARHGAP36 and other ARHGAP members from various animals collected in the UniProt showed that this ARHGAP36 lacked the conserved arginine residue (it is replaced with a threonine) in the "arginine finger", which was a hallmark motif of the Rho GAP family, suggesting the catalytic function may not be required for ARHGAP36. By aligning the cDNA to the predicted sheepArhgap36 gene sequence (Gene ID: 100913153) in GenBank, a criticalshort gap was also found in the first exon which will led to incapability of deducing a correct sheep ARHGAP36 amino acid sequence. Thus, the results provided a correct sheep ARHGAP36 cDNA for the further research on the function of this unique Rho GAP family member. Detection of the total protein of sheep testis by Western blot and tissue sections of sheep testis by Immunohistochemistry using anti-ARHGAP36 polyclonal antibody as the primary antibody indicated that ARHGAP36 protein might have three forms of various lengths in testis and exists in nearly all types of the cells during spermatogenesis in sheep seminiferous tubules.
sheep; ARHGAP36; cDNA cloning; RACE; immunohistochemistry
2014-08-27;
2014-09-29
国家自然科学基金项目(31460600);内蒙古自然科学基金项目(2012MS0401)
白雪,研究方向为分子遗传学,E-mail:120218099@qq.com;
邢万金,博士,教授,博士研究生导师,研究方向为分子遗传学,E-mail: xwanjin@imu.edu.cn。
Q751;S826
A
2095-1736(2015)02-0001-07
doi∶10.3969/j.issn.2095-1736.2015.02.001

