植物登陆过程中光合作用相关基因的进化
李雪芹, 王寅初, 崔玉琳, 秦 松, 胡章立
(1. 深圳大学 生物科学学院, 深圳 518060; 2. 中国科学院 烟台海岸带研究所, 烟台 264003)
植物登陆过程中光合作用相关基因的进化
李雪芹1, 王寅初2, 崔玉琳3, 秦 松2, 胡章立1
(1. 深圳大学 生物科学学院, 深圳 518060; 2. 中国科学院 烟台海岸带研究所, 烟台 264003)
现代陆生植物的祖先大约在5亿到4.7亿年前登陆,这是植物进化过程中的重要事件。光照不仅是植物的主要能量来源,还是主要的环境信息来源之一。一般认为,光合作用效率越高,植物的适应能力越强。植物登陆过程前后,环境从光照、光强到二氧化碳/氧气浓度比等都发生了变化,比如适应陆地生活的海草重新返回水生环境,猜测在这个过程中植物光合作用的相关的关键功能基因可能发生了进化。为了证明该假说,以链形植物门绿藻和低等陆地植物为主要研究对象,通过从GenBank收集相关基因序列以及从几株相近绿藻中提取测定DNA序列,利用PAML“分枝-位点”模型对光合作用光反应和暗反应中的psbA、rbcL、rca3个基因进行了适应性进化分析。结果显示植物登陆过程前后,在这3个光合作用相关基因中均未检测到统计上显著的正选择位点,反而在绿色植物早期分化(绿藻门和链形植物门)过程中却发现有明显的正选择痕迹。这些惊人的结果提示植物登陆前后从水体到陆地的环境变化对光合作用的影响可能并不大,其它诸如角质膜、气孔器等相关的基因可能发挥了更大的作用。
植物;psbA基因;rbcL基因;rca基因;正向选择位点;环境变化;适应性进化
陆生植物的祖先大约在5亿到4.7亿年前登陆[1],毫无疑问植物的这次由水生到陆生的发展是生物进化的重大事件[2-3],使地球环境发生了重大的改变,是整个陆地生态系统发展的开始[4]。植物从水生到陆生需要适应新的环境条件,比如干燥、气温浮动、营养的供给、光照变化、CO2和O2的浓度等[5]。
现代生物学普遍认为陆生植物起源于链形植物门绿藻。超显微结构、生物化学研究以及分子数据显示链形植物门绿藻分为6个纲:轮藻纲(Charophyceae),鞘毛藻纲(Coleochaetophyceae)、双星藻纲(Zygnematophyceae)、克里藻纲(Klebsormidiophyceae)、绿叠球藻纲(Chlorokybophyceae)和中斑藻纲(Mesostigmatophyceae)[6]。Graham[2]和Kenrick等人[3]通过形态分析认为轮藻纲和鞘毛藻纲是最接近陆生植物的,2001年Karol等人[7]认为轮藻纲是最接近陆生植物的类群。而2010年Finet[8]发表的以核基因编码为基础的多基因系统发育分析的文章认为鞘毛藻纲是最接近陆生植物的类群。2011年Wodniok等人[9]同样采用系统发育分析的方法证明双星藻纲或者鞘毛藻纲是最接近陆生植物的类群。
绿色植物包括两大分支:链形植物门和绿藻门。其中链形植物门包括链形植物绿藻和陆生植物。链形植物门绿藻与绿藻门是并系群。从超显微结构和生物学特性方面来看,链形植物门绿藻具有很多陆生植物所共有而绿藻门所不存在的特征,比如能进行有丝分裂,存在能够持续进行有丝分裂的纺锤体结构;具有许多独特的酶系统等。从分子生物学角度来看,通过对Spirogyra和Coleochaete序列文库分析发现,链形植物门绿藻更接近陆生植物而不是绿藻门。将Spirogyra和Coleochaete序列与莱茵衣藻和拟南芥进行同源基因序列比对发现,链形植物门绿藻与拟南芥的相似程度是链形植物门绿藻和莱茵衣藻的2到3倍。序列分析和同源基因分析增强了链形植物门绿藻和陆生植物之间的亲密关系[10]。
光合作用为植物的生存提供了物质和能量。光合作用是植物将光能转换为可用于生命过程的化学能并进行有机物合成的生物过程。光合作用主要分为光反应和暗反应两个过程。光反应中,光系统Ⅱ反应中心在这一过程中起到关键作用。D1蛋白是光系统Ⅱ反应中心的两个核心蛋白之一,由于D1蛋白分别与原初电子供体P680、初级电子受体去镁叶绿素和次级电子受体质体醌以及与水裂解相关的锰簇相连,所以D1蛋白对原初电荷的分离、维持光系统Ⅱ反应中心构象的稳定以及电子的传递至关重要。D1蛋白由psbA编码,叶绿体基因组编码psbA。同样,核酮糖-1, 5-二磷酸羧化酶(Rubisco)在光合作用的暗反应中起重要的作用,既负责碳同化又引发光呼吸,既是固定CO2的限速酶,又是决定CO2和O2相互竞争的关键酶,全酶由8个大亚基和8个小亚基组成,已知酶的活性中心位于大亚基上,小亚基只有调节功能。大亚基主要由rbcL编码,rbcL由叶绿体基因组编码,小亚基则由核基因组编码。Rubisco作为光合碳同化的关键酶,其活性高低直接影响光合速率,而Rubisco必须经Rubisco活化酶活化后才具有催化活力,Rubisco活化酶的编码基因rca由核基因组所编码。
由于光是比较易变的环境因子而植物是固定生长的生物,因此有效光照的改变很可能对植物造成较强的选择压力[11]。植物从水生进化到陆生,CO2和O2浓度也发生了不同的变化。因为植物登陆前后环境有所改变,所以我们推测在此过程中对植物光合作用起关键作用的功能基因可能发生了适应性进化,比如适应陆地生活的海草重新返回水生环境[12]。为了验证该假说,我们选取了链形植物门绿藻的3个属的植物。针对上述3个基因: 光反应中编码D1蛋白的psbA基因,核酮糖-1, 5-二磷酸羧化酶的大亚基编码基因rbcL,核酮糖-1, 5-二磷酸羧化酶的限速酶—核酮糖-1, 5-二磷酸羧化酶活化酶编码基因rca,利用PAML“分枝-位点“模型对植物早期进化相关类群进行适应性进化分析[13]。
1 材料和方法
1.1 序列数据获取和处理
由GenBank获得37个物种的psbA序列(附表1),87个物种的rbcL序列(附表2),11个物种的rca序列(附表3)。此外,本研究选取3株植物SpirogyrapratensisUTEX LB 928 (链形植物门双星藻纲),ColeochaetescutataUTEX LB 2567 (链形植物门鞘毛藻纲),ClosteriumlanceolatumFACHB-545 (链形植物门双星藻纲)进行培养,分别提取这3株藻的RNA,然后反转录成mRNA,以3株藻的mRNA为模板进行PCR分别扩增psbA,rbcL以及rca的序列,引物分别为:psbA上游引物F: 5′-CTT CTGCGACTGGGTTACCA-3′,下游引物R: 5′-CGTCCTTGGCTATCAACTACA-3′;rbcL上游引物F: 5′-TAAAGCAGGTGCAGGATTTAAAGG-3′,下游引物为R: 5′-TATCAAATTCAAATTTAATTTC TTTCCAAAC-3′;rca上游引物F: 5′-TCCGATGACCAGCAAGACATCA-3′,下游引物为R: 5′-CACC GAAGAAATCGATGGAC-3′。用MAFFT 5[14]进行序列的方向校正和比对,再用MEGA 4进行密码子比对[15]。
1.2 系统发育分析
根据JModelTest软件选取最佳核苷酸进化模型[16],并用PHYML 3.0来构建系统发育树[17],Bootstrap值设定为1000次,采用PAML 4软件包进行适应性进化位点检测[13]。
1.3 适应性进化分析
目前适应性进化检测常用的方法是在密码子置换模型下的似然法[18]。这种方法检测适应性进化有3个模型: 分枝模型、位点模型以及分枝-位点模型。本研究采用“分枝-位点”模型来检验物种之间的正选择位点。“分枝-位点”模型[19-20]:将进化树上的支系分成前景支和背景支两类,对此允许ω值同时在分枝和位点间存有差异,并且只允许在前景支上存在ω大于1的位点,因此可以根据模型A检验2在前景支上检测承受正选择的氨基酸位点[13],有更强的检测能力。
2 结果与分析
2.1 分子系统发育分析

图1 基于psbA序列数据构建的系统发育树
①—绿色植物两大门类在水体环境中的早期分化;②—绿色植物登陆前后的进化过程。
对于psbA系统发育树,选取核苷酸进化模型GTR+I+G,并且计算bootstrap值(图1),通过psbA的系统发育树可以看出,中斑藻纲和绿叠球藻纲是链形植物门绿藻中最早分化的类群。Wodniok等人[9]认为最接近陆生植物的是双星藻纲和轮藻纲,从进化树可以认为我们的研究符合他们的结果。对于一直存在争议的苔藓纲,我们的研究支持Goremykin和Marin认为的苔藓纲是一个单系群的观点[21-22]。
rbcL进化树同样选取核苷酸进化模型GTR+I+G,并且计算bootstrap值(图2)。Finet等人[8]和Wodniok等人[9]认为最接近陆生植物的类群是鞘毛藻纲,从rbcL的系统发育树(图2)可以看出我们的研究支持Finet等人的结果。从进化树中同样可以看出苔藓纲是一个单系群,中斑藻纲和绿叠球藻纲是链形植物门绿藻中最早分化的类群,这些结果同样支持psbA的结果。
最后,对于rca核苷酸进化模型为GTR+I+G,计算的bootstrap值如图3。由于目前的研究比较少,在数据库中能获取的基因比较缺乏,本研究中的类群为目前所能查找到的关于rca的所有类群的基因。从图3中可以看出,最接近陆生植物的类群是双星藻纲或者鞘毛藻纲,这与Wodniok等人[9]的文章结果相一致。从我们的研究可以总结出,最接近陆生植物的类群是鞘毛藻纲。
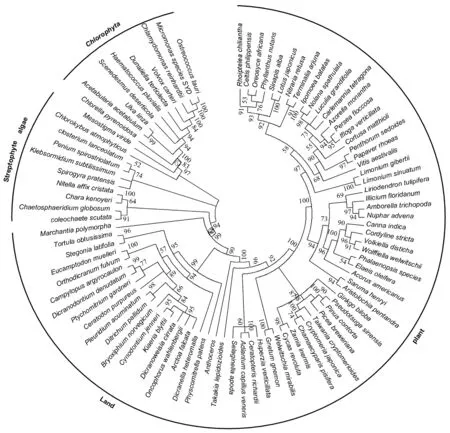
图2 基于rbcL序列数据构建的系统发育树
①—绿色植物两大门类在水体环境中的早期分化;②—绿色植物登陆前后的进化过程。
2.2 正向选择位点的检测与定位
根据Timme和Delwiche[23]的结果Spirogyra和Coleochaete是链形植物门绿藻中更接近陆生植物的类群。本研究选择Spirogyrapratensis的序列作为参考序列,来确定正选择位点在序列上的位置。

图3基于rca序列数据构建的系统发育树
①—绿色植物两大门类在水体环境中的早期分化;②—绿色植物登陆前后的进化过程。
2.2.1psbA基因的正向选择分析
表1是我们利用“分枝-位点”模型对植物psbA基因正选择位点的检测结果。在这个模型中,只有前景支为绿藻门(如图1-①)时存在正选择位点,而前景支为绿藻(如图1-②)时未发现统计上显著的正选择位点。在基因序列中,有2.71%的位点经历了正选择,卡方检验表明该结果有显著意义(P<0.0001),而经验贝斯(BEB)方法显示了正选择的显著位点:118S、151S、196S、248S和293S。
以Spirogyrapratensis的psbA基因对PDB数据库进行搜索,结果以蓝藻的光系统Ⅱ三维结构(PDB ID: 1W5C)的得分最高,二者序列一致性为91%,符合同源建模可靠性要求。正选择的氨基酸位点的空间位置如图4所示。在这8个正选择位点中,196S位于α螺旋内,118S、151S、248S和293S则位于β折叠上。
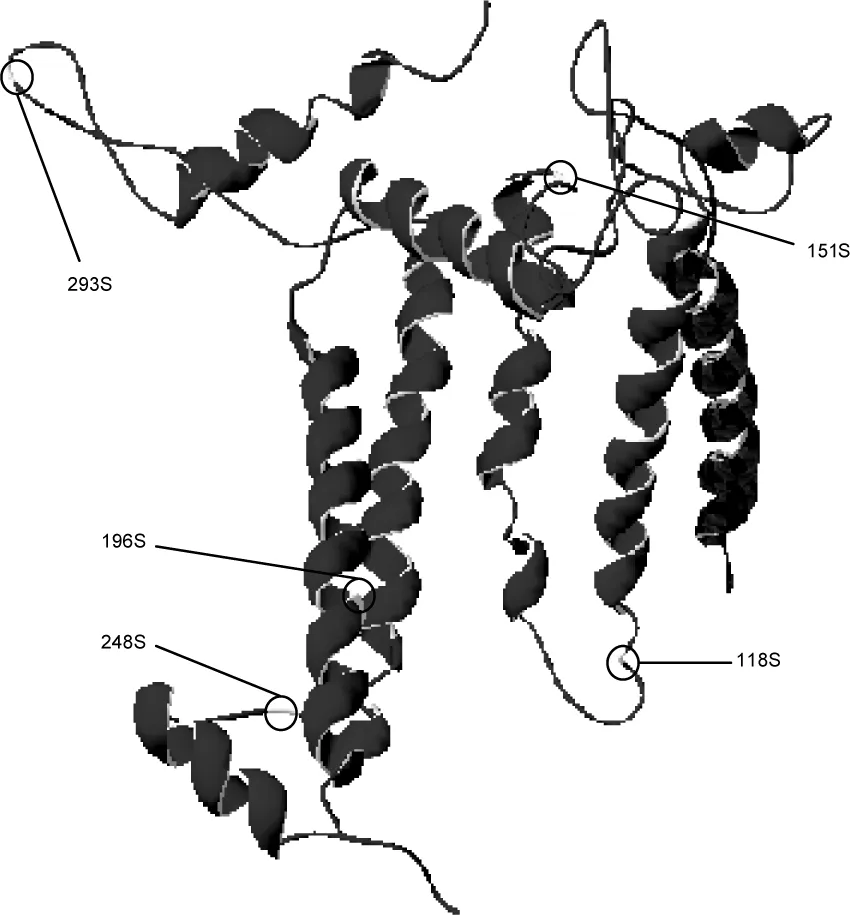
图4 Spirogyra psbA 正选择位点的空间位置
通过三维结构模型,对“分枝-位点”模型检测出的正选择位点进行功能定位。在这些位点中,位点118S、151S是CP43的结合位点,CP43是光系统Ⅱ的核心天线,除了能将外周天线叶绿素a/b结合蛋白捕获的激发能汇集给反应中心外,还具有维持光系统Ⅱ放氧复合物的结构,参与水裂解放氧等功能,还推测可能具有蛋白酶的功能[24]。位点196S是D1蛋白的光反应中心信号,活性区域的氨基酸决定了酶的催化效率。248S是次级电子受体质体醌的结合部位,质体醌不仅在电子传递过程中参与能量的传递,而且在光系统Ⅰ和光系统Ⅱ光能分配方面起着重要的作用。293S是细胞色素b559结合部位,该位点目前推测的主要功能是保护光系统Ⅱ免遭光破坏[25]。
表1psbA在“分枝-位点”模型下的参数估计值和对数似然值
Table 1 Parameter estimates and log-likehood values forpsbAgene under “branch-site” model

P* 表示ω分布所用的参数数目;在95%和99%后验概率下推测出的正选择位点分别由黑体和斜体写出;下同。
2.2.2rbcL的正向选择分析
利用“分枝-位点”模型对植物rbcL正选择位点的检测结果见表2。在该模型中,前景支为绿藻门(如图2-①)时,发现序列存在正选择位点,前景支为绿藻(如图2-②)时未发现统计上显著的正选择位点。在此模型中,rbcL中有1.63%的位点经历了正选择,卡方检验表明该结果有显著意义(P<0.05),而经验贝斯(BEB)方法显示了正选择的显著位点:32M、158P、269S、316I、339L、381T和389I。
通过数据库寻找的相似度最高的序列为菠菜Rubisco三维结构(PDB ID: 1AA1)的得分最高,二者序列一致性为93%,通过Swiss-PdbViewer 4.0.1软件[26]构建的蛋白质3D结构如图5。除了位点32M位于β折叠外,其余的8个正选择位点158P、269S、316I、339L、381T及389I则位于rbcL羧基末端的α/β桶结构域内。
对已有的Rubisco的结构研究表明,Rubisco大亚基由羧基末端的α/β桶结构域与与之相邻的另一个大亚基的氨基末端结构域共同构成Rubisco活性中心[27]。除位点32M外,158P、269S、316I、339L、381T和389I这些位点均位于Rubisco大亚基的活性中心内,催化中心区域的氨基酸决定了催化效率。对于位点32M,有研究显示该氨基酸的突变会明显影响酶同CO2/O2的结合的特异性以及同氧气的结合量,从而影响酶的催化效率[28]。位点269S侧链较短,能为活性中心留有更大的空间,为催化的进行作了保证[29]。位点316I位于βH折叠区内,异亮氨酸可能与周围其他的氨基酸形成氢键,有研究证明βH空间位置的变化可能与植物对生境的适应相关[30]。

图5 Spirogyra rbcL 正选择位点的空间位置

2.2.3rca的正向选择分析
植物rca正选择位点的检测结果见表3。在此模型中,前景支为绿藻门(如图3-①)时,发现序列存在正选择位点而当前景支为绿藻(如图3-②)时却未发现明显的正选择位点。rca中有2.60%的位点经历了正选择,卡方检验表明该结果有显著意义(P<0.05),而经验贝斯(BEB)方法显示了正选择的显著位点:118S、143K、193K、194E、204C、243Q和153E。
通过PDB数据库,相似度最高的是烟草Rubisco活化酶三维结构(PDB ID: 3T15),二者序列一致性94%,通过Swiss-PdbViewer 4.0.1软件构建的蛋白质3D结构如图6所示。在这7个正选择位点中,位点193K、194E、204C和243Q位于α螺旋内,而位点118S、143K和153E则位于β折叠上。
Rubisco作为光合碳同化的关键酶,其活性高低直接影响光合速率,而Rubisco必须经Rubisco活化酶活化后才具有催化活力。Rubisco活化酶是一种核编码的叶绿体蛋白,在ATP参与下,生理浓度的CO2以及Mg2+结合作用下使Rubisco达到最大的活化程度。Rubisco活化酶作为Rubisco的分子伴侣,可促进和稳定Rubisco催化活性,在植物光合作用对温度的响应中发挥重要作用,同时它具有ATP酶的活性,能水解ATP、ATP/ADP的比率对该酶具有重要的调节作用,位点143K位于ATP酶活性位点区域。至于其它检测到的正选择位点的功能则尚需进一步地研究。
综上所述,在我们的研究中,绿藻门和链形植物门绿藻虽都在水生环境中,但是它们的光合作用相关基因发生了明显变化,而且检测到了显著的正选择痕迹,然而植物从水生到陆生环境的进化中,这些基因却没有发现明显的正选择痕迹。

图6 Spirogyra rca 正选择位点的空间位置

3 讨论
为了保证数据的可靠性和一致性,我们把一些低质量的过短序列舍去,将最终选择的序列构建系统进化树(图1、图2和图3),这3个基因构建的系统进化树基本与目前的研究结果相一致,同时从我们的研究中可以发现轮藻纲是最接近陆生植物的类群。

附表1 植物材料及其psbA GenBank登录号

附表2 植物材料及其rbcL GenBank登录号

附表3 本研究所用植物材料及其rca GenBank登录号
与以往的研究实例类似,本文采用PAML“分枝-位点”模型对植物的早期进化进行适应性进化分析。Rodriguez-Trelles等人[31]运用此模型检测动物和植物分化之前的一些早期真核生物的进化。在研究被子植物分化之前的陆生植物的进化分析也采用该模型[32]。Alba等人[33]采用该模型检验了被子植物中光敏色素基因的适应性进化。PAML“分枝-位点”模型不仅可以用于研究高等植物适应性进化分析,而且在藻类中同样适用。Young等人[34]通过PAML“分枝-位点”模型检测红藻和Chromista中Rubisco大亚基的适应性进化;秦松等人[35]还通过该模型研究红藻和蓝藻基因发现正选择压力而不是功能松弛促使藻红蛋白基因的进化;证明蓝藻藻胆蛋白在基因复制的过程中存在着适应性进化同样采用“分枝-位点”模型[36]。在Yang和Nielsen[37]原先的模型中,Zhang[38]在一项模拟研究中发现这些检验对其基本模型很敏感,且当模型假定违背时还可能导致很高的假阳性。本文采用改进的模型[19],这个模型已经很大程度降低了假阳性率。
植物处在不同的生活环境下会发生不同的适应性进化[39]。Wissler等人[12]通过对两株具有直系同源基因的海草与8株陆生被子植物近亲进行适应性进化分析发现,从陆生到水生过程中,海草中的基因相对于陆生被子植物发生了适应性进化。而在我们的研究中,绿藻门和链形植物门绿藻虽都生活在水生环境中,虽然环境未发生明显变化,但是它们的光合作用相关基因发生了很明显变化,而且检测到了正选择位点。然而植物从水生到陆生环境的进化中,环境变化差异比较大,新的陆生环境会对植物造成很强的选择压力[40],这些基因却没有发现明显的正选择痕迹,这是一个值得深思的问题。但是与常规相悖不一定就代表论据有问题。然而Prochnik等人[41]对团藻的基因组进行测序后,通过对莱茵衣藻与团藻的基因组进行比较发现,虽然这两株藻的复杂程度和生命史存在很大差异,但是二者的基因组却有近似的蛋白编码潜能。与莱茵衣藻相比,研究者在团藻基因组内只发现了很少该生物特有的基因。因此有时候跟人们主观推测中基因或基因组的变化关系不大。由于本研究中的植物登陆过程中光合作用相关的基因并未检测到显著的正选择痕迹,我们猜测可能是一些非光合作用的基因帮助植物实现了从水生到陆生的进化,或者PAML的算法可能仍然存在某些缺陷。本研究涉及到的植物登陆相关类群的基因数据仍然比较缺乏,尤其是rca,我们期待有更多的数据支持来验证我们的假说。
[1]Rubinstein C V, Gerrienne P, de la Puente G S, et al. Early middle ordovician evidence for land plants in Argentina (eastern Gondwana)[J]. New Phytologist, 2010, 188: 365-369.
[2]Graham L E. Origin of land plants[M]. New York.:John Wiley and Sons Inc., 1993.
[3]Kenrick P, Crane P R. The origin and early diversification of land plants. A cladistic study[M]. Washington DC: Smithsonian Institute Press, 1997.
[4]Leliaert F, Smith D R, Moreau H, et al. Phylogeny and molecular evolution of the green algae[J]. Critical Reviews in Plant Sciences, 2012, 31(1): 1-46.
[5]Waters E R. Molecular adaptation and the origin of land plants[J]. Molecular Phylogenetics and Evolution, 2003, 29(3): 456-463.
[6]McCourt R M, Delwiche C F, Karol K G. Charophyte algae and land plant origins[J]. Trends in Ecology & Evolution, 2004, 19(12): 661-666.
[7]Karol K G, McCourt R M, Cimino M T, et al. The closest living relatives of land plants[J]. Science, 2001, 294(5550): 2351-2353.
[8]Finet C, Timme R E, Delwiche C F, et al. Multigene phylogeny of the green lineage reveals the origin and diversification of land plants[J]. Current Biology, 2010, 20(24): 2217-2222.
[9]Wodniok S, Brinkmann H, Glckner G, et al. Origin of land plants: do conjugating green algae hold the key?[J]. BMC Evolutionary Biology, 2011, 11(1): 104.
[10]Timme R E, Delwiche C F. Uncovering the evolutionary origin of plant molecular processes: comparison of Coleochaete (Coleochaetales) and Spirogyra (Zygnematales) transcriptomes[J]. BMC Plant Biology, 2010, 10(1): 96.
[11]Stuefer J F, Huber H. Differential effects of light quantity and spectral light quality on growth, morphology and development of two stoloniferousPotentillaspecies[J]. Oecologia, 1998, 117(1/2): 1-8.
[12]Wissler L, Codo er F M, Gu J, et al. Back to the sea twice: identifying candidate plant genes for molecular evolution to marine life[J]. BMC evolutionary biology, 2011, 11(1): 8.
[13]Yang Z. PAML 4: phylogenetic analysis by maximum likehood[J]. Mol Biol Evol, 2007, 24(8): 1586-1591.
[14]Katoh K, Kuma K, Toh H, et al. MAFFT version 5: improvement in accuracy of multiple sequence alignment[J]. Nucleic Acids Research, 2005, 33(2): 511-518.
[15]Tamura K, Dudley J, Nei M, et al. MEGA 4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol Biol Evol, 2007, 24(8): 1596-1599.
[16]Posada D, Crandall K A. Modeltest: testing the model of DNA substitution[J]. Bioinformatics, 1998, 14(9): 817-818.
[17]Guindon S, Dufayard J F, Lefort V, et al. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0[J]. Systematic Biology, 2010, 59(3): 307-321.
[18]Yang Z. Likelihood ratio tests for detecting positive selection and application to primate lysozyme evolution[J] Mol Biol Evol, 1998, 15(5): 568-573.
[19]Yang Z, Wong W, Nielsen R. Bayes empirical bayes inference of amino acid sites under positive selection[J]. Mol Biol Evol, 2005, 22(4): 1107-1118.
[20]Zhang J, Nielsen R, Yang Z. Evaluation of an improved branch-site likelihood method for detecting positive selection at the molecular level[J]. Mol Biol Evol, 2005, 22(12): 2472-2479.
[21]Goremykin V V, Hellwig F H. Evidence for the most basal split in land plants dividing bryophyte and tracheophyte lineages[J]. Plant Systematics and Evolution, 2005, 254(1/2): 93-103.
[22]Marin B, Melkonian M. Molecular phylogeny and classification of theMamiellophyceaeclass. Nov. (Chlorophyta) based on sequence comparisons of the nuclear-and plastid-encoded rRNA operons[J]. Protist, 2010, 161(2): 304-336.
[23]Timme R, Delwiche C. Uncovering the evolutionary origin of plant molecular processes: comparison ofColeochaete(Coleochaetales) andSpirogyra(Zygnematales) transcriptomes[J]. BMC Plant Biology, 2010, 10(1): 96.
[24]单际修, 王居硕. 光系统 Ⅱ 核心天线 CP43 和 CP47 的分离纯化及光谱性质研究[J]. 生物物理学报, 1999, 15(1): 144-151.
[25]李新国, 孟庆伟. 细胞色素b559与PSⅡ光抑制的光保护机制[J]. 植物学通报, 2003, 20(6): 680-687.
[26]Arnold K, Bordoli L, Kopp J, et al. The SWISS-MODEL workspace: a web-based environment for protein structure homology modeling[J]. Bioinformatics, 2006, 22(2): 195-201.
[27]陈 洁, 张丽君, 王 艇. 蕨类植物rbcL基因正选择和负选择位点的鉴定[J]. 西北植物学报, 2009. 29: 2391-2400.
[28]森 林, 苏应娟, 张 冰, 等. 凤尾蕨科植物rbcL基因的适应性进化分析[J]. 热带亚热带植物学报, 2010, 18(1): 1-8.
[29]Persson E, Bak H, Olsen O. Substitution of valine for leucine 305 in factor Ⅶa increases the intrinsic enzymatic activity[J]. Journal of Biological Chemistry, 2001, 276(31): 29195-29199.
[30]刘 念, 王庆彪, 陈 婕, 等. 麻黄属rbcL基因的适应性进化检测与结构模建[J]. 科学通报, 2010: 1341-1346.
[31]Rodriguez-Trelles F, Tarrio R, Ayala F J. Convergent neofunctionalization by positive Darwinian selection after ancient recurrent duplications of the xanthine dehydrogenase gene[J]. Proceedings of the National Academy of Sciences, 2003, 100(23): 13413-13417.
[32]Mathews S, Burleigh J G, Donoghue M J. Adaptive evolution in the photosensory domain of phytochrome A in early angiosperms[J]. Mol Biol Evol, 2003, 20(7): 1087-1097.
[33]Alba R, Kelmenson P M, Cordonnier-Pratt M M, et al. The phytochrome gene family in tomato and the rapid differential evolution of this family in angiosperms[J]. Mol Biol Evol, 2000, 17(3): 362-373.
[34]Young J N, Rickaby R E M, Kapralov M V, et al. Adaptive signals in algal Rubisco reveal a history of ancient atmospheric carbon dioxide[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2012, 367(1588): 483-492.
[35]Qin S, Zhao F Q, Tseng C K. Evidence for positive selection in phycoerythrin genes of red algae and cyanobacteriaProchlorococcusandSynechococcus[J]. Photosynthetica, 2005, 43(1): 141-146.
[36]Zhao F, Qin S. Evolutionary analysis of phycobiliproteins: implications for their structural and functional relationships[J]. Journal of Molecular Evolution, 2006, 63(3): 330-340.
[37]Yang Z, Nielsen R. Condon-substitution models for detecting molecular adaption at individual sites along specific lineges[J]. Mol Biol Evol, 2002, 19(6): 908-917.
[38]Zhang J. Frequent false detection of positive selection by the likelihood method with branch-site models[J]. Mol Biol Evol, 2004, 21(7): 1332-1339.
[39]Lida S, Miyagi A, Aoki S, et al. Molecular adaptation of rbcL in the heterophyllous aquatic plantPotamogeton[J]. PLoS One, 2009, 4(2): e4633.
[40]李承森. 生物进化的重大事件——陆地植物的起源及其研究的新进展[J]. 中国科学基金, 1994, 8(4): 238-244.
[41]Prochnik S E, Umen J, Nedelcu A M, et al. Genomic analysis of organismal complexity in the multicellular green algaVolvoxcarteri[J]. Science, 2010, 329(5988): 223-226.
Adaptive evolution of photosynthesis-related genes during plants colonizing terrestrial habitat
LI Xue-qin1, WANG Yin-chu2, CUI Yu-lin3, QIN Song2, HU Zhang-li1
(1. School of Biology Science, Shenzhen University, Shenzhen 518060; 2. Yantai Institute of Coastal Zone Research, Chinese Academy of Sciences, Yantai 264003, China)
The ancestors of modern land plants colonized the terrestrial habitat about 500 to 470 million years ago, this is an important event in the history of life on earth. Illumination is not only the primary energy for plants, but also the main environmental information sources. It is widely accepted that the higher efficiency of photosynthesis, the stronger ability of plants to adapt the environment. When plants began to colonize the terrestrial habitat, their environment significantly changed, such as illumination, light intensity and the concentration ratio of carbon dioxide and oxygen just like seagrasses went back to the sea. Therefore, we hypothesize that there are evidences for adaptive evolution of photosynthesis-related genes in plants. To test this hypothesis, detection of adaptive evolution was performed onpsbA,rbcL,rcagenes using PAML′s "site-branch" model. As a result, no significant evidence of positive selection of these three genes was detected in the plant colonization, whereas there was remarkable divergence betweenChlorophytaandStreptophyta. This amazing result implies that environmental changes during plants colonizing the terrestrial habitat may have few influences on photosynthesis-related genes. Genes related to other functions like cuticle and stomatal apparatus played important roles in this course.
plant;psbAgene;rbcLgene;rcagene; positive selection; environment changes; adaptive evolution
2014-04-15;
2014-04-23
国家自然科学基金项目资助(41176144,41376139);国家科技支撑计划(2013BAB01B01);中国科学院战略性先导科技专项(XDA1102040300);海洋公益性行业科研专项经费资助(20120527)
李雪芹,硕士,专业方向为生物化学与分子生物学,E-mail:lxq821991024@126.com;
秦 松,研究员,博士生导师,主要从事藻类资源高价值利用研究,E-mail:sqin@yic.ac.cn; 胡章立,教授,博士生导师,主要从事藻类学分子生物学方面的研究,E-mail:huzl@szu.edu.cn。
Q945.11
A
2095-1736(2015)01-0059-08
doi∶10.3969/j.issn.2095-1736.2015.01.059

