珍珠黄杨RAPD最佳反应体系研究
姜自红

摘 要:以RAPD遗传标记为手段,对野生珍珠黄杨种群RAPD最佳反应体系进行研究。结果表明,在36 ℃时珍珠黄杨有比较好的片段数量和片段亮度。此外,2.5 mmol·L-1 MgCl2,0.25 mmol·L-1 dNTP,0.3 μmol·L-1引物浓和2.0 U·(20 μL)-1 TaqDNA聚合酶构成了珍珠黄杨RAPD扩增的最佳组合反应体系。
关键词:珍珠黄杨;RAPD;反应体系;正交设计
中图分类号:S792.11 文献标识码:A DOI 编码:10.3969/j.issn.1006-6500.2015.04.003
Research on Optimization of RAPD Reaction Systems for Buxus sinica var.parvifolia
JIANG Zi-hong
(Chuzhou Vocational and Technical College,Chuzhou,Anhui 239000,China)
Abstract: The genetic diversity of wild Buxus sinica var.parvifolia population were studied by random amplified polymorphic DNA(RAPD).The results showed that the number and brightness of genetic segments were the best at 36 ℃. Meanwhile, 2.5 mmol·L-1 MgCl2, 0.25 mmol·L-1 dNTP, 0.3 μmol·L-1 primer and 2.0 U Taq DNA made a satisfactory RAPD technique system.
Key words: Buxus sinica var.parvifolia; RAPD;reaction system;the orthogonal design method
珍珠黄杨(Buxus sinica var.parvifolia)属黄杨(B.sinica)变种,又名小叶黄杨、鱼鳞黄杨、鱼鳞木,具有生长慢、枝干茂密挺拔、树型稳定紧凑等特点,是制作袖珍园林的绝佳材料。主要产于江西庐山、福建崇安黄岗山、浙江龙荡山、湖北神农架、安徽歙县清凉峰等地。
RAPD(Random amplified polymorphic DNA)是美国科学家Williams和Welsh(1990 年)几乎同时发展起来的一项技术,该技术具有简便、迅速、准确等优点,广泛应用于生物多样性监测、生物育种、遗传定位等领域。RAPD在园林植物(杨树、红叶石楠、苦楝等)上的研究被广泛报道,但关于 RAPD 标记技术在珍珠黄杨上的应用未见报道,本研究拟建立最优珍珠黄杨RAPD反应体系,为进一步开展珍珠黄杨研究提供参考。
1 材料和方法
1.1 试验材料
供试材料为:珍珠黄杨(安徽省黄山市歙县清凉峰和江西省上饶市玉山县三清山野生种群)。
1.2 试验方法
采集野外珍珠黄杨叶片,利用改良CTAB—硅珠法[4]提取珍珠黄杨叶片中总DNA。
RAPD扩增反应随机抽取一个野生珍珠黄杨的DNA样本,利用随机引物HY-36(CCCCCCTTAG),在MJ Research.Inc PTC-220型PCR仪上进行扩增[5]。扩增程序:94 ℃预变性5 min,然后变性40 s,接着36 ℃退火30 s,72 ℃延伸2 min,循环39次,最后在72 ℃延伸5 min。
在基本扩增程序基础上,试验设计8个退火温度:35,36,37,38,39,40,41,42 ℃。试验采取每改变一个参数形式进行扩增。取扩增产物10 μL在含有0.5 μg·mL-1溴化乙锭(EB)染色液的质量分数1.4%的琼脂糖凝胶中电泳分析。
RAPD反应条件设计:(1)Mg2+浓度分别设定为1.5,2.0,2.5,3.0,3.5 mmol·L-1;(2)每种dNTP的最终浓度分别设定为0.10,0.15,0.2,0.25,0.3 mmol·L-1;(3)引物浓度分别设定为0.1,0.2,0.3, 0.4,0.5 μmol·L-1;(4)TagDNA聚合酶浓度分别设定为1.0,1.5,2.0,2.5 U。
2 结果与分析
2.1 对珍珠黄杨RAPD最佳反应体系温度梯度的分析
由图1可见,在几个温度条件下,野生珍珠黄杨扩增均表现较好,根据扶芳藤最佳反应体系[2],以及本试验结果,珍珠黄杨的片段数量和片段亮度在36 ℃时表现最好。
2.2 单因素方法筛选扩增体系
2.2.1 Mg2+浓度对珍珠黄杨RAPD的影响 Mg2+是TagDNA聚合酶的激活剂。当Mg2+浓度为1.5 mmol·L-1时,条带数少且条带表现模糊,随着Mg2+浓度增加,条带数变得更加模糊,条带数也逐渐减少,当Mg2+浓度达到2.5~3.0 mmol·L-1时,条带反而亮度增加,条带清晰度也较低浓度处理更明显(图2)。试验结果显示,随着Mg2+浓度逐渐增加,条带亮度也逐渐显著,而弱带反变得模糊,这可能是由于较高浓度Mg2+增加错配率所导致。因此,珍珠黄杨RAPD扩增较为适合的Mg2+浓度为2.5~3.0 mmol·L-1。
2.2.2 dNTP浓度对珍珠黄杨RAPD的影响 不同浓度的dNTP对扩增结果的影响较为显著(图3)。试验结果显示, dNTP浓度为0.10 mmol·L-1和0.15 mmol·L-1时,扩增条带数和亮度差异不明显,条带数均表现少而模糊,部分条带表现弥散现象。随着dNTP浓度增加,扩增条带逐渐变得清晰,亮度增加,0.20 mmol·L-1时,效果最为明显。当浓度增至0.30 mmol·L-1时,主条带数不增反减,这主要是因为dNTP浓度过高或过低都不利于珍珠黄杨RAPD的扩增。
2.2.3 TagDNA聚合酶用量对珍珠黄杨RAPD的影响 试验结果显示,当酶浓度为1.0 U·(20 μL)-1和1.5 U·(20 μL)-1时,条带亮度集中但难于分辨,这主要是条带不能有效扩增所致,而2.0和2.5 U·(20 μL)-1的酶浓度条带均均较清晰(图4)。
2.2.4 引物浓度对珍珠黄杨RAPD的影响 从图5可知,当引物浓度范围为0.1~0.3 μmol·L-1,条带的数量和亮度都有所递增,而当引物浓度高于0.5 μmol·L-1时同样扩增不完全,最后确定0.3 μmol·L-1的引物浓度较为适宜。
2.3 正交设计法筛选扩增体系
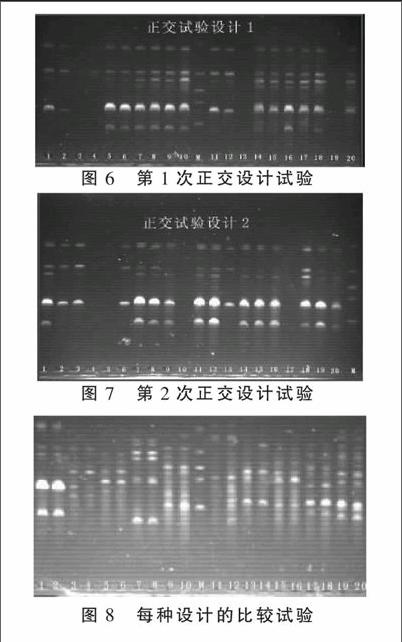
第1次正交设计试验结果显示,第5,6,7,8, 9,10,16,17,18泳道的条带比较清晰,扩增效果比较理想,但也有条带分不开(图6)。
第2次正交设计的引物进行了更换,结果显示,第7,8,11,12,18泳道的条带比较明亮,且清晰可辨,符合RAPD分析的要求(图7)。
通过两次正交设计,得出珍珠黄杨RAPD扩增的最佳组合反应体系为:MgCl2浓度2.5 mmol·L-1,dNTP浓度0.2 mmo1·L-1,引物浓度0.3 μmol·L-1,TaqDNA聚合酶浓度2.0 U·(20 μL)-1。
2.4 3种反应体系的比较
由图8可见,各因素最优水平的组合,最佳水平确定的原则为:当某一影响因素的各浓度对扩增片段数量有显著影响时,可以利用扩增片段最多的水平为最佳水平,片段平均亮度将不再考虑;如果某因素的各水平对扩增片段数量影响不明显时,则最佳水平用最大平均亮度作标准。
2.5 最佳反应体系的验证
应用上述最佳反应体系,用引物HY-9对20份珍珠黄杨DNA进行RAPD扩增(图9)。该反应体系下的每份DNA样品均能明显扩增出DNA片段,适宜珍珠黄杨RAPD-PCR反应,能够应用于珍珠黄杨遗传结构和遗传多样性的分析。
3 结论与讨论
3.1 正交设计应用于RAPD反应体系建立的优点
本试验研究应用L20(54)正交表,采取多次PCR扩增,利用正交设计的量化分析,分析出Taq酶、Mg2+、随机引物、dNTP 4种组分对RAPD扩增的效应,试验结果构建了稳定性及重复性较好的RAPD反应体系。单因素试验相同次数的PCR扩增虽可得到一个最优体系,但缺点是不能研究交互作用,而正交设计在RAPD反应体系建立的应用可有效分析交互作用,并具有工作量小、数据分析简单、信息量大等优点[6-7]。
3.2 RAPD各组分对RAPD-PCR扩增结果的影响
关于RAPD体系中各组分对扩增结果的影响及相关机理的研究已有很多报道[6-12]。而在本试验中,不同浓度的影响因子扩增出的片段数量也不同。Taq酶和Mg2+对凝胶上扩增片段数量有显著影响;不同浓度的Mg2+、dNTP和引物对扩增片段亮度均有显著影响,结果与前人研究结果一致[8-10]。试验结果表明,dNTP和引物浓度对扩增片段的数量影响不明显,而Taq酶对扩增片段亮度无显著影响,结果与张彦萍、毛娟等研究结果不同[13-15]。相对于单一的试验,正交设计结果包含了更多的信息。
综上,通过对Mg2+,dNTP、引物和TaqDNA聚合酶用量等因子的不同浓度对扩增条带数量和亮度的研究,确定较理想的扩增体系为:2.5 mmol·L-1 Mg2+浓度,0.25 mmo1·L-1 dNTP,0.3 μmol·L-1引物和2.0 U·(20 μL)-1 Taq DNA聚合酶。
参考文献:
[1] 张玉平,李丕军,朱燕,等.杨树RAPD标记反应体系的建立与优化[J].中国农学通报,2014,30(13):20-24.
[2] 陈羡德,陈礼光,陈珺,等.苦楝RAPD反应体系的优化[J].西南林学院学报,2008,28(1):43-47.
[3] 李妮,燕丽萍,刘翠兰,等.红叶石楠RAPD反应体系的优化[J].中国农学通报,2012,28(34):53-57.
[4] 苏应娟,王艇,杨维东,等.罗汉松属植物DNA的提取和RAPD分析[J].中山大学学报:自然科学版,1998,37(4):13-18.
[5] 潘青华,鲁韧强,张开春.扶芳藤遗传多样性RAPD鉴定及类型划分研究[J].中国农学通报,2005,7(21):90-93.
[6] 文晓鹏,邓秀新.刺梨及其近缘种PCR试验体系的建立及其优化[J].果树学报,2003,20(1):35-39.
[7] 孙敏,乔爱民,王和勇,等.黄瓜DNA提取及其RAPD反应体系的建立[J].种子,2004,23(6):91-94.
[8] 张虎平,牛建新,马兵钢,等.核桃DNA的提取及RAPD反应体系的优化[J].石河子大学学报,2003,7(4):267-270.
[9] 叶丽娟,禄久幸,杨秋生.蜡梅花瓣基因组DNA提取及RAPD-PCR反应体系优化[J].山西农业科学,2011(4):299-303.
[10] 王家保,刘志媛,徐碧玉,等.用正交设计优化荔枝RAPD反应体系[J].武汉植物学研究,2005,23(4):363-368.
[11] 叶凡,李开言,王亚芹.山茱萸RAPD反应体系的正交优化研究[J].河南农业科学,2012(4):113-116.
[12] 孙朝霞,王海燕,王玉国.枣树RAPD分析体系优化研究[J].华北农学报,2008(4):115-118.
[13] 张彦萍,刘海河.西瓜RAPD-PCR体系的正交优化研究[J].河北农业大学学报,2005,28(4):51-53.
[14] 毛娟,赵长增,赵丽娟,等.扁桃基因组DNA提取及RAPD扩增体系的建立[J].甘肃农业大学学报,2005,40(1):17-21.
[15] 聂珍素,赖钟雄,潘东明,等.橄榄基因组提取及扩增条件优化[J].亚热带农业研究,2005,1(2):61-63.
天津农业科学2015年4期
