H7N9 流感病毒HA、NA 蛋白的抗原表位预测及其与HLA-Ⅱ类等位基因的相关性分析①
刘雪婷 王 珊 张俊艳 刘兆宇 陈惠芳 邹泽红 肖兰艳 及志恒 何 颖 (广州医科大学附属第二医院 广东省过敏反应与免疫重点实验室/呼吸疾病国家重点实验室变态反应研究室,广州 510260)
甲型H7N9 流感病毒是在2013 年3 月在中国东部上海和安徽两地率先发现的一种新型禽流感病毒[1],感染该病毒将引起肺炎、呼吸道衰竭、急性呼吸窘迫综合征(ARDS)[2]。截止到2014 年6 月11日,全国共报告感染H7N9 禽流感的患者共有434例,其中有165 人死亡,死亡率达38%。
甲型流感病毒颗粒外膜由两型表面糖蛋白覆盖,一型为血细胞凝集素(Hemagglutinin,HA),一型为神经氨酸酶(Neuraminidase,NA)。HA 蛋白的裂解性、受体特异性和糖基化是决定流感病毒感染性和致病性的重要因素。NA 蛋白在决定病毒毒力和宿主特异性方面也具有重要作用,与HA 共同成为流感病毒亚型分型的主要依据,NA 同时也是抗流感病毒的重要作用靶点[3]。甲型流感能不断引起流行是由于其抗原性不断发生漂移,使流感病毒逃过人体免疫系统的识别[4],其中最重要是HA 和NA的变异[5],因此可以通过针对HA 和NA 的表位疫苗技术来解决这一问题。表位预测是表位疫苗设计的前提,本研究采用生物信息学方法,分析了H7N9流感病毒的28 种HA、24 种NA 的氨基酸序列的同源性,预测HA 和NA 蛋白的T 细胞和B 细胞相关抗原表位,为新型H7N9 流感疫苗的制备提供依据。
人类白细胞抗原(HLA)是位于人类第6 号染色体短臂上的一组紧密连锁的基因群,是目前已知人体中最具多态性的遗传系统[6]。HLA 系统在不同民族种族和地区间分布不同,是机体对疾病易感的主要遗传成分,即拥有不同的HLA 等位基因是导致个体间免疫应答能力和对疾病易感性出现差异的主要遗传学原因[6]。同时,HLA 是人体特异性免疫系统的一道重要屏障,HLA 分子提呈侵入人体的抗原呈递给T 细胞,进行抗原清除。HLA 和T 细胞及相关细胞因子在介导病毒感染人体中发挥极其重要的作用[7]。本研究预测HLA-DRB1* 0701 等位基因与HA 和NA 有较强结合力,并根据数据库和文献公布的该基因在不同地域人群中的基因频率,推测对H7N9 病毒敏感的人群,以便更有效、及时地控制H7N9 流感的传播。
1 材料与方法
1.1 HA 和NA 氨基酸序列的获得及同源性分析从NCBI(http://www.ncbi.nlm.nih.gov/protein/)网站下载甲型H7N9 流感病毒的28 种HA 和24 种NA 的氨基酸序列,用ClustalX 软件进行多序列比对,用MEGA4.0 的邻接法(Neighbor-joining method,NJ)构建进化树。根据序列比对分析结果,选取其中HA、NA 蛋白代表性的序列作为基准序列,用于后续的B 细胞抗原表位、T 细胞抗原表位预测以及对H7N9 易感人群的预测。
1.2 HA 和NA 蛋白的B 细胞抗原表位预测 应用DNAStar 软件中的Protean 模块分析HA、NA 蛋白的基准序列,根据Kyte-Doolittle 方案预测其亲水性[8];根据Karplus-Schulz 方案预测其柔韧性;根据Emini 方案预测其表面可及性[9];根据Jameson-Wolf 方案预测其抗原性指数[10],确定HA 和NA蛋白基准序列的B 细胞抗原表位。选取亲水性高、柔韧性好、表面可及性大、抗原性指数高的区域作为候选表位,并兼顾二级结构各参数,避开α-螺旋和β-折叠区域,确定HA、NA 蛋白的B 细胞优势抗原表位。
1.3 HA 和NA 蛋白的T 细胞抗原表位预测 使用NetMHC Ⅱ2.2 Server 在线服务器(http://www.cbs.dtu.dk/services/NetMHCII/)分析26 种MHCⅡ类分子(包括14 种HLA-DR、6 种HLA-DP 和6 种HLA-DQ 等位基因型)与H7N9 流感病毒HA、NA 蛋白的滑动九肽的结合情况。滑动9 肽的选取方法为:第1~9 氨基酸序列对应的肽段为第1 个九肽,第2~10 氨基酸序列对应的肽段为第2 个九肽,第3~11 氨基酸对应的肽段为第3 个九肽,以此类推。滑动九肽与HLA 高亲和力的阈值为0.64,低亲和力的阈值为0.42。与26 种MHCⅡ类分子结合力大于0.42 的九肽对应的氨基酸序列视为是潜在的T抗原表位。
1.4 H7N9 流感病毒与HLA 等位基因相关性分析及易感人群预测 利用NetMHCⅡ2.2 Server 在线软件分析HA、NA 蛋白的滑动九肽与26 种HLA-Ⅱ类分子的结合情况,筛选出与HA、NA 蛋白的滑动九肽具有高亲和力(亲和指数大于0.64)的HLA-Ⅱ类分子等位基因型,携带这些HLA 等位基因的人群可能是对H7N9 病毒存在高的免疫反应性的易感人群。
从等位基因频率数据库(http://www.allele-frequencies.net/)查询与HA、NA 蛋白具有高亲和力的HLA 等位基因在亚洲不同国家的基因频率,并通过文献验证,根据HLA 等位基因频率推测H7N9 流感病毒更有可能在哪些国家爆发。
2 结果
2.1 HA、NA 蛋白的氨基酸比较 经过进化树分析发现,不同甲型H7N9 流感病毒株之间氨基酸序列相对保守,且28 种HA 以及24 种NA 氨基酸序列可以分为两个大的族群(图1A、B)。分别选取两种蛋白的两个族群中较有代表性的HA 氨基酸序列ABI84694.1 与AEK84761.1 和NA 氨基酸序列AFX85263.1 与AFU25736.1 为基准序列以便进行下一步的细胞抗原性表位预测。
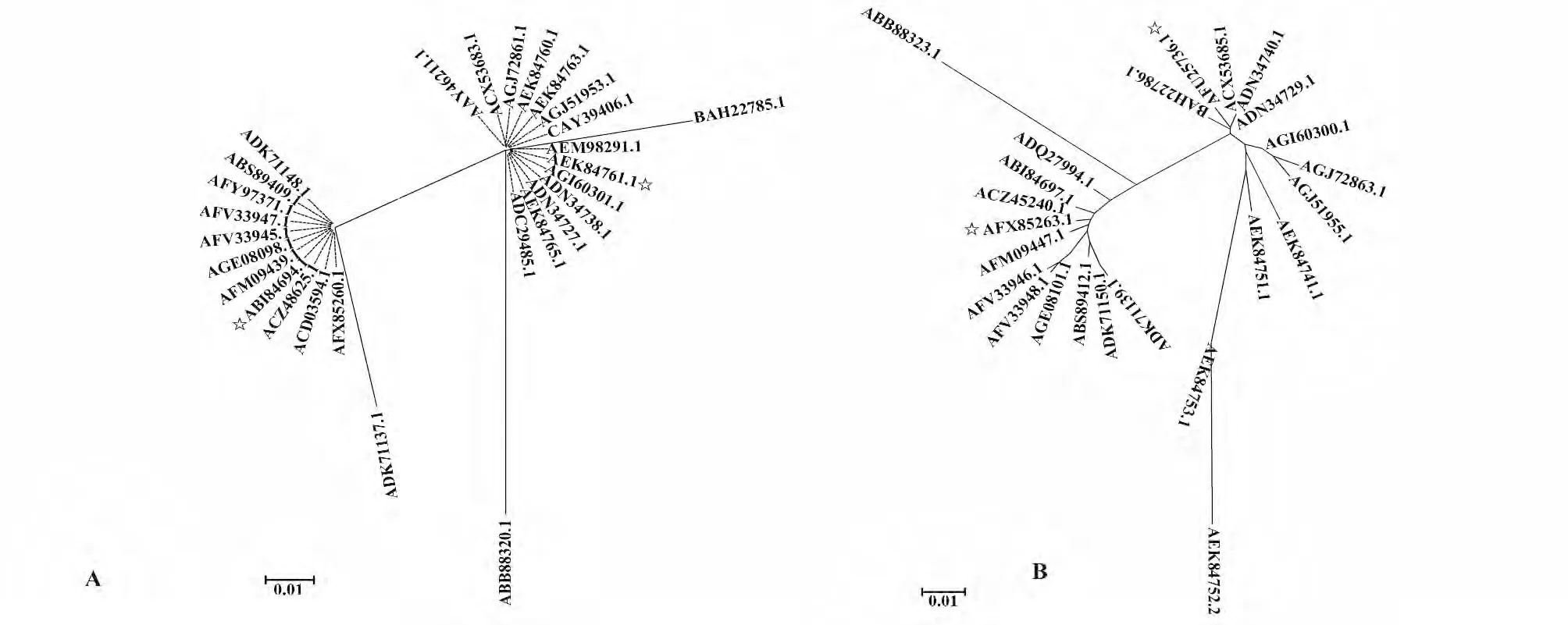
图1 H7N9 流感病毒HA、NA 蛋白的同源性分析Fig.1 Phylogenetic tree of HA and NA proteins in novel avian-origin influenza A (H7N9)virus

图2 HA、NA 蛋白的B 细胞表位预测Fig.2 Prediction of B cell epitopes on HA and NA proteins of novel avian-origin influenza A (H7N9)virus
2.2 HA、NA 蛋白的基准序列的B 细胞抗原性预测 经分析发现,HA 氨基酸序列ABI84694.1 与AEK84761.1 的B 细胞表位预测结果并没有明显差别,同 样,NA 氨 基 酸 序 列 AFX85263.1 与AFU25736.1 的B 细胞表位预测结果也没有明显差别,因此只选取HA 氨基酸序列ABI84694.1 和NA 氨基酸序列AFX85263.1的结果列出。通过DNAStar软件的Protean 模块对HA 和NA 蛋白的亲水性、柔韧性、表面可及性、抗原性指数进行预测。取亲水性高、柔韧性好、表面可及性大、抗原性指数高的区域,综合选取至少2 个上述参数较好的重叠区域,并兼顾二级结构预测结果,选取无规卷曲区域,获得B细胞的抗原表位区域。从图2A 综合各参数分析,发现在HA 蛋白中63-70、98-117、173-185、195-207、217-238、306-322、332-341、363-429、443-475、492-512 各项指数均较高,提示可能是抗原性表位;从图2B 综合各参数分析,发现在NA 蛋白中1-9、57-60、97-105、137-141、143-150、162-168、210-215、219-222、258-263、273-280、310-323、415-425 各项指数均较高,提示为抗原性表位。
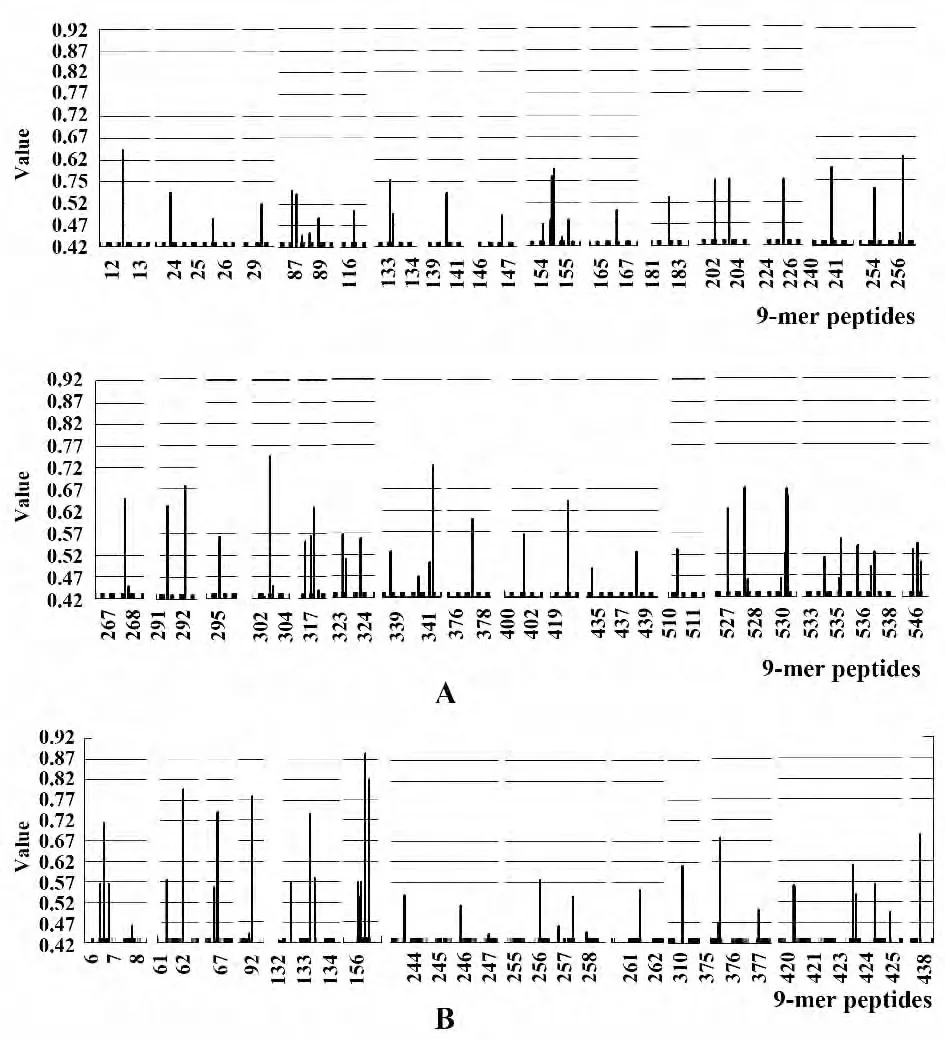
图3 H7N9 流感病毒HA、NA 蛋白的T 细胞表位预测Fig.3 Prediction of T cell epitopes on HA and NA proteins of novel avian-origin influenza A (H7N9)virus
2.3 HA、NA 蛋白基准序列的T 细胞抗原性预测 使用NetMHCⅡ2.2 Server 在线软件预测了基准株HA(ABI84694.1)、NA(AFX85263.1)蛋白的HA、NA 蛋白的滑动九肽与26 种HLA-Ⅱ类分子结合力,结合力大于0.42 的滑动九肽对应的氨基酸序列为T 抗原表位。软件预测结果显示,HA 蛋白中第13、24-29、87-89、116、133-183、202-204、225、241-268、290-303、317-324、338-341、377、401-438、510、527-546 滑动九肽对应的氨基酸区段与26 种HLA-Ⅱ类分子结合力较高(图3A);NA 蛋白中第7、61-67、92、132-133、156、244-262、310、375-377、420-438 滑动九肽对应的氨基酸区域与26 种HLA-Ⅱ类分子结合力较高(图3B),可能为T 细胞抗原性表位。
2.4 H7N9 流感病毒与HLA 等位基因相关性分析及易感人群预测 根据NetMHCⅡ2.2 Server 在线软件预测的HA、NA 蛋白的滑动九肽与26 种HLA-Ⅱ类分子结合情况,我们筛选出与HA 蛋白的多个滑动九肽具有高亲和力的HLA 等位基因DRB1*0701、DQA1 * 0501-DQB1 * 0301、DPA1 * 0103-(DPB1* 0301/DPB1* 0401)(表1)以及与NA 蛋白的多个滑动九肽具有高亲和力的HLA 等位基因DRB1* 0701、DRB1* 0101(表2),携带这些等位基因型的人群可能是H7N9 流感病毒的易感人群,尤其是DRB1* 0701 等位基因,与HA、NA 蛋白的多个滑动九肽具有高亲和力。
根据HLA 等位基因频率数据库的查询结果,结合文献验证,我们发现DRB1* 0701 在乌鲁木齐维吾尔族人群基因频率为16.7%,在哈尔滨满族人群基因频率为12.8%,在山东汉族人群基因频率为11.2%,在辽宁汉族人群基因频率为10.7%,在北京、石家庄、天津汉族人群基因频率为9.5%,均属于高频率等位基因型,提示H7N9病毒在这些地区的危险性较高;在中国广东、云南、台湾地区汉族人群基因频率分别为5.7%、4.1%、2.8%;在日本、韩国人群中的基因频率分别为6.7%、0.3%(表3)。
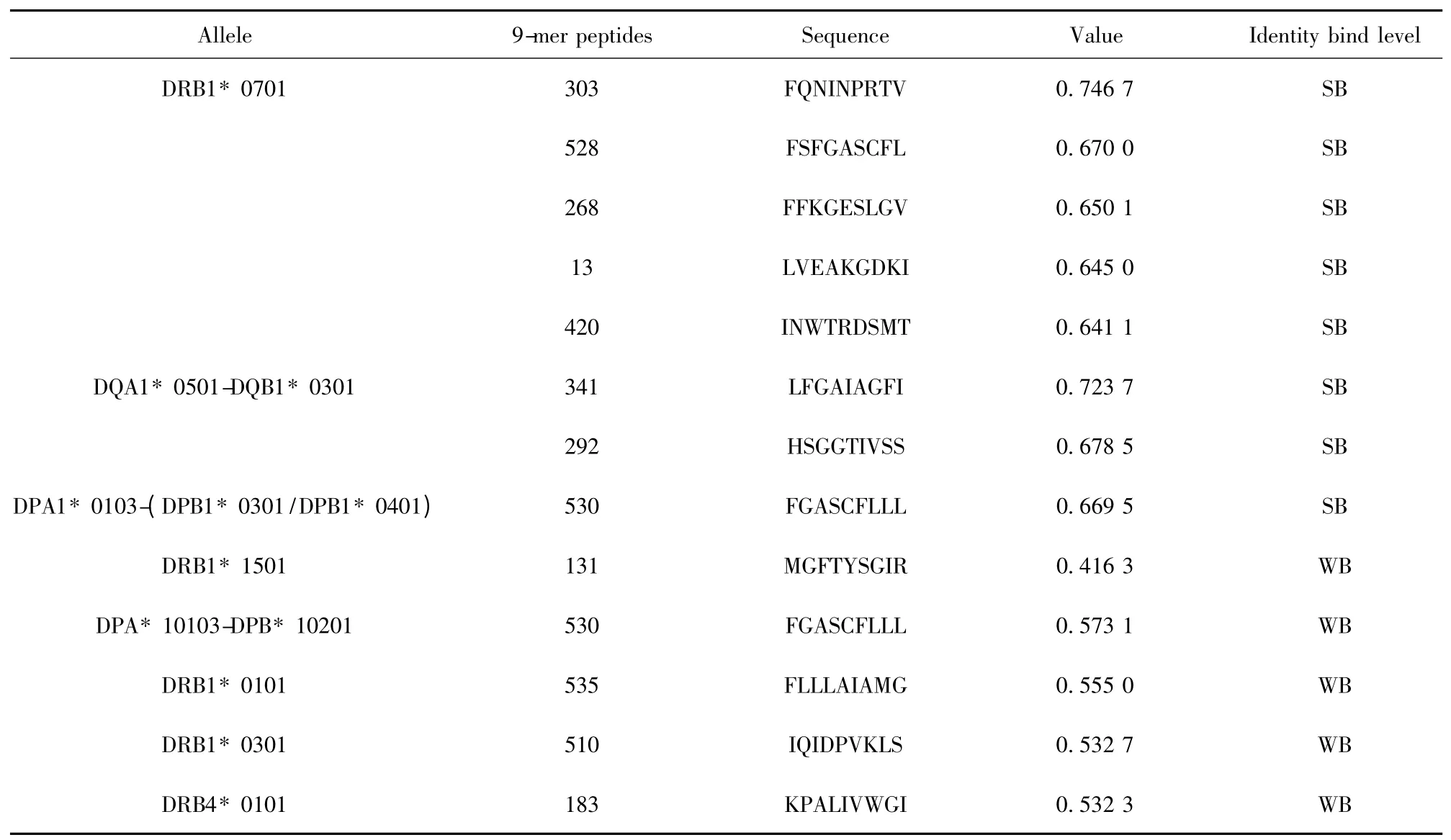
表1 HA 蛋白的滑动九肽与HLA-Ⅱ等位基因的结合情况分析Tab.1 Analysis of affinity between peptides of HA protein and HLA-Ⅱalleles by NetMHCⅡ2.2 Server
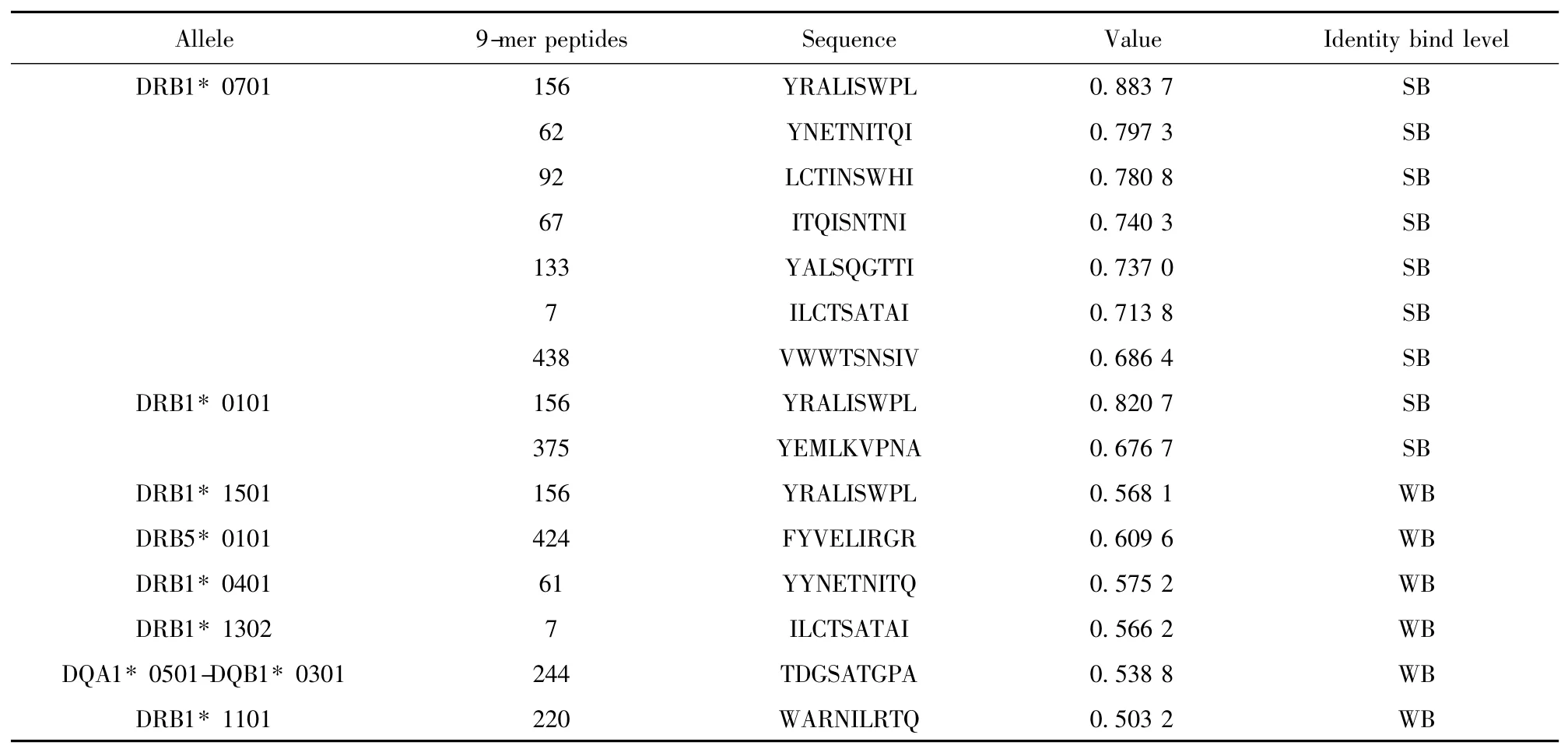
表2 NA 蛋白的滑动九肽与HLA-Ⅱ等位基因的结合情况分析Tab.2 Analysis of affinity between peptides of NA protein and HLA-Ⅱalleles by NetMHCⅡ2.2 .Server
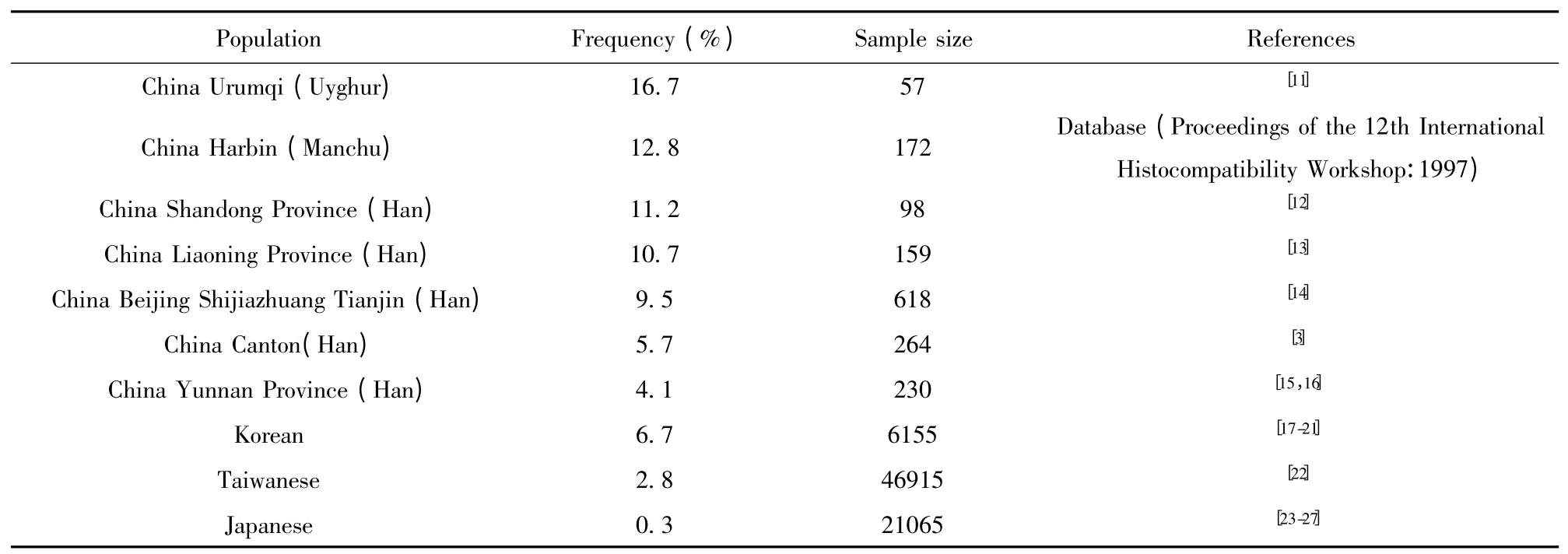
表3 DRB1* 0701 在亚洲不同地区的频率Tab.3 Frequency of DRB1* 0701 in different countries in Asia
3 讨论
近年来随着生物信息技术的快速发展,生物信息学软件已成为预测抗原表位的有力工具。国内外不少学者都在生物信息学方法预测的基础上进行抗原表位的研究。通过生物信息学软件的预测可极大地提高表位筛选的成功率,减少实验工作量,节省研究时间。
理想的抗原表位最好能兼有B 细胞表位与T细胞表位的功能[28]。一方面可以通过刺激B 细胞产生针对抗原的特异性抗体,直接与病毒结合。同时又可以与MHCⅡ类分子结合,递呈到抗原递呈细胞表面,引发T 细胞的活化,产生获得性免疫应答并能为B 细胞产生抗体提供帮助。本研究利用软件对H7N9 流感病毒的HA、NA 蛋白的基准序列的B 细胞和T 细胞抗原表位进行预测,HA 具有10 个B 细胞表位和15 个T 细胞表位;NA 有12 个B 细胞表位和9 个T 细胞表位,可以作为检测以及疫苗研发的靶区域。但是细胞抗原性表位预测仍然不够完善,目前几乎所有的细胞抗原性表位预测的算法都是预测连续氨基酸组成的线性表位,而较少涉及构象性表位的研究。本研究仅是对新型H7N9 流感病毒的HA 和NA 蛋白的细胞抗原性表位进行初步筛选,预测结果需要进一步用实验结果来证实。
HLA 的Ⅰ类和Ⅱ类基因在人类基因组中多态性最为丰富,其产物主要具有提呈抗原、激活效应T细胞的功能。它们本身多形异质型以及参与免疫应答,因此具有HLA-Ⅱ某些基因的人群对某些免疫疾病有易感性[29]。影响HLA 基因易感性的因素很多,包括种族的不同和地域的差异。本研究利用NetMHCⅡ2.2 Server 软件分析与H7N9 流感病毒的HA、NA 蛋白具有高亲和力的HLA 等位基因为DRB1* 0701,并根据该基因在亚洲不同国家的基因频率,预测H7N9 病毒可能的易感人群。
[1]Gao R,Cao B,Hu Y,et al.Human infection with a novel avian-origin influenza A (H7N9)virus[J].N Engl J Med,2013,368(20):1888-1897.
[2]Li Q,Zhou L,Zhou M,et al.Preliminary report:epidemiology of the avian influenza A (H7N9)outbreak in china[J].N Engl J Med,2014,370(6):520-532.
[3]Trachtenberg E,Vinson M,Hayes E,et al.HLA class I (A,B,C)and class II (DRB1,DQA1,DQB1,DPB1)alleles and haplotypes in the Han from southern China[J].Tissue Antigens,2007,70(6):455-463.
[4]Hajjar LA,Schout D,Galas FR,et al.Guidelines on management of human infection with the novel virus influenza A (H1N1)--a report from the hospital das clinicas of the university of sao paulo[J].Clinics (Sao Paulo),2009,64(10):1015-1024.
[5]Wu CY,Yeh YC,Chan JT,et al.A VLP vaccine induces broadspectrum cross-protective antibody immunity against H5N1 and H1N1 subtypes of influenza A virus[J].PLoS One,2012,7(8):e42363.
[6]Mangalam AK,Taneja V,David CS.HLA class II molecules influence susceptibility versus protection in inflammatory diseases by determining the cytokine profile[J].J Immunol,2013,190(2):513-518.
[7]Foucault ML,Moules V,Rosa-Calatrava M,et al.Role for proteases and HLA-G in the pathogenicity of influenza A viruses[J].J Clin Virol,2011,51(3):155-159.
[8]Kyte J,Doolittle RF.A simple method for displaying the hydropathic character of a protein[J].J Mol Biol,1982,157(1):105-132.
[9]Karplus PA,Schulz GE.Substrate binding and catalysis by glutathione reductase as derived from refined enzyme:substrate crystal structures at 2 A resolution[J].J Mol Biol,1989,210(1):163-180.
[10]Jameson BA,Wolf H.The antigenic index:a novel algorithm for predicting antigenic determinants[J].Comput Appl Biosci,1988,4(1):181-186.
[11]Mizuki N,Ohno S,Ando H,et al.Major histocompatibility complex class II alleles in an Uygur population in the Silk Route of Northwest China[J].Tissue Antigens,1998,51(3):287-292.
[12]Zhou L,Lin B,Xie Y,et al.Polymorphism of human leukocyte antigen-DRB1,-DQB1,and -DPB1 genes of Shandong Han population in China[J].Tissue Antigens,2005,66(1):37-43.
[13]刘利民,梁健,宋芳吉,等.应用SSP-PCR/SSO 方法进行中国辽宁汉族人HLA-DRB1 基因的遗传多态性研究[J].遗传,1999,21(3):720-737.
[14]Yang G,Deng YJ,Hu SN,et al.HLA-A,-B,and -DRB1 polymorphism defined by sequence-based typing of the Han population in Northern China[J].Tissue Antigens,2006,67(2):146-152.
[15]Yao Y,Shi L,Shi L,et al.Distribution of HLA-A,-B,-Cw,and -DRB1 alleles and haplotypes in an isolated Han population in Southwest China[J].Tissue Antigens,2009,73(6):561-568.
[16]Shi L,Xu SB,Ohashi J,et al.HLA-A,HLA-B,and HLA-DRB1 alleles and haplotypes in Naxi and Han populations in southwestern China (Yunnan province)[J].Tissue Antigens,2006,67(1):38-44.
[17]Hong SC,Lin L,Lo B,et al.DQB1* 0301 and DQB1* 0601 modulate narcolepsy susceptibility in Koreans[J].Hum Immunol,2007,68(1):59-68.
[18]Huh JY,Yi DY,Eo SH,et al.HLA-A,-B and -DRB1 polymorphism in Koreans defined by sequence-based typing of 4128 cord blood units[J].Int J Immunogenet,2013,40(6):515-523.
[19]Song EY,Park H,Roh EY,et al.HLA-DRB1 and -DRB3 allele frequencies and haplotypic associations in Koreans[J].Hum Immunol,2004,65(3):270-276.
[20]Lee KW,Oh DH,Lee C,et al.Allelic and haplotypic diversity of HLA-A,-B,-C,-DRB1,and -DQB1 genes in the Korean population[J].Tissue Antigens,2005,65(5):437-447.
[21]Song EY,Park MH,Kang SJ,et al.HLA class II allele and haplotype frequencies in Koreans based on 107 families[J].Tissue Antigens,2002,59(6):475-486.
[22]Lai MJ,Wen SH,Lin YH,et al.Distributions of human leukocyte antigen-A,-B,and -DRB1 alleles and haplotypes based on 46,915 Taiwanese donors[J].Hum Immunol,2010,71 (8):777-782.
[23]Saito S,Ota S,Yamada E,et al.Allele frequencies and haplotypic associations defined by allelic DNA typing at HLA class I and class II loci in the Japanese population[J].Tissue Antigens,2000,56(6):522-529.
[24]Horiki T,Ichikawa Y,Moriuchi J,et al.HLA class II haplotypes associated with pulmonary interstitial lesions of polymyositis/dermatomyositis in Japanese patients[J].Tissue Antigens,2002,59(1):25-30.
[25]Kitawaki J,Obayashi H,Kado N,et al.Association of HLA class I and class II alleles with susceptibility to endometriosis[J].Hum Immunol,2002,63(11):1033-1038.
[26]Itoh Y,Mizuki N,Shimada T,et al.High-throughput DNA typing of HLA-A,-B,-C,and -DRB1 loci by a PCR-SSOP-Luminex method in the Japanese population[J].Immunogenetics,2005,57(10):717-729.
[27]Tanaka T,Ohmori M,Yasunaga S,et al.DNA typing of HLA class II genes (HLA-DR,-DQ and -DP)in Japanese patients with histiocytic necrotizing lymphadenitis (Kikuchi's disease)[J].Tissue Antigens,1999,54(3):246-253.
[28]Parida R,Shaila MS,Mukherjee S,et al.Computational analysis of proteome of H5N1 avian influenza virus to define T cell epitopes with vaccine potential[J].Vaccine,2007,25(43):7530-7539.
[29]Bignon JS,Aron Y,Ju LY,et al.HLA class II alleles in isocyanate-induced asthma[J].Am J Respir Crit Care Med,1994,149(1):71-75.

