线粒体硫化物氧化关键酶硫双加氧酶研究进展
张立涛
(济宁医学院生物科学学院,山东 日照 276862)
·博士论坛·
线粒体硫化物氧化关键酶硫双加氧酶研究进展
张立涛
(济宁医学院生物科学学院,山东 日照 276862)

张立涛,男,汉族,1987年7月出生于山东省临沂市。2014年6月毕业于中国海洋大学海洋生命学院细胞生物学专业,获博士学位,主要定位于单环刺螠硫化物代谢分子机制方面的研究。2014年8月到济宁医学院生物科学学院工作。目前以第一作者发表SCI文章2篇,分别发表在“PloS One”和“Journal of the Marine Biological Association of the United Kingdom”,累计影响因子4.554。
硫双加氧酶在线粒体硫化物氧化过程中催化过硫化物、氧气和水反应形成亚硫酸盐,其功能丧失会导致体内硫化物积累产生多种疾病。本文通过对国内外文献的研究分析对硫双加氧酶的基因结构特征,催化活性和催化机制等进行了全面的综述,为进一步研究硫双加氧酶和拓展人们对硫化物代谢分子机制提供了有价值的参考。
硫双加氧酶;线粒体;硫化物氧化
硫化物是H2S,HS-和S2-的统称。由于硫化物与细胞色素aa3血红素卟啉环铁离子的可逆性结合进而阻止氧气与其结合[1];此外高浓度的硫化物可以导致细胞色素氧化酶构象的不稳定和亚基的降解[2];因此,硫化物能够破坏线粒体氧化呼吸链中电子传递,抑制ATP的产生。所以最初人们认为硫化物是生物体的一种有毒物质,然而最近发现生物体内可以产生内源性的硫化物[3],能调节生物体的一系列活动[4]。线粒体硫化物氧化几乎存在于从无脊椎动物到脊椎动物所有的动物体内,被认为是调节生物体内硫化物浓度的关键通路[5]。
1 硫双加氧酶在线粒体硫化物氧化中的功能
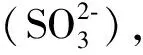

图1 线粒体硫化物氧化的两种模型
2 硫双加氧酶的基因结构功能研究进展
2.1 硫双加氧酶基因的发现
在线粒体硫化物氧化模型最先提出时,硫醌氧化还原酶和硫转移酶都已经确定为哪个基因编码的,而硫双加氧酶只是在组织总蛋白中检测到其催化酶活,但是不能确定是哪个基因编码的[8]。2009年Tiranti等首先确定在人体中确定基因ETHE1(ethylmalonic encephalopathy 1)编码的蛋白质行使硫双加氧酶的功能[12]。这个基因最先是2002年在肝癌细胞消减文库中发现表达量明显升高,并且能够与NF-κB相结合抑制细胞凋亡的一个新基因,当时被命名为HSCO(hepatoma subtracted-cDNA library clone one);2004年发现HSCO基因的突变能够导致乙基丙二酸脑病变(ethylmalonic encephalopathy),因此将该基因重命名为ETHE1,并确定是位于人染色体19q13上的一个编码线粒体蛋白基因[11];之后该团队又发现在乙基丙二酸脑病变的病人体内和ETHE1基因敲除的小鼠体内均不能检测到硫双加氧酶的活性;而在大肠杆菌和Hela细胞中过表达ETHE1基因时,硫双加氧酶的活性明显增加,最终确定ETHE1蛋白就是硫双加氧酶[12]。在ETHE1基因敲除的小鼠中还发现肌肉、脑和结肠黏膜中存在严重的细胞色素c氧化酶缺陷[2],机体还将出现持续性的神经损伤、出血性脑损伤、慢性出血腹泻、血管瘀斑和直立性手足发绀等症状[14];而这些症状均是由体内高浓度硫化物导致的[15],这一系列的研究进一步确定ETHE1基因编码的线粒体蛋白就是硫双加氧酶,并在人体内线粒体硫化物氧化过程中发挥重要作用。
2.2 硫双加氧酶的基因和结构特征
硫双加氧酶的基因目前仅在动物人与单环刺螠和植物拟南芥中有明确的研究[11,16-17],其他物种均是在其基因组研究中发现编码硫双加氧酶蛋白,如牡蛎[18]。硫双加氧酶属于金属β-内酰胺酶超家族成员,含有其代表性序列HXHXDH(图3中黑框标出),通常含有两个金属结合位点:金属结合位点I在图3中以#标出,以单环刺螠为例序列为H113,H115,H169和D188,金属结合位点II在图3中以▽标出,其在单环刺螠中的序列为D117,H118,H169和H229,这两个金属结合位点共用一个组氨酸。单环刺螠硫双加氧酶的分段表达实验验证,两个金属结合位点的重要性不同,结合生物信息学预测证明硫双加氧酶和β内酰胺酶超家族其他成员不同,虽然含有完整的两个金属结合位点,但是金属离子仅仅结合在第一个金属位点上,被认为是金属β内酰胺酶超家族的一个新成员[17]。Pettinati等在2015年解析出人类硫双加氧酶基因的三维结构,确定唯一的金属离子Fe2+与氨基酸H79,H135和D154相连,位于金属结合位点I上,为硫双加氧酶的催化活性中心,氨基酸163-166和226-232形成一个通道能够让活性中心与底物结合[19]。
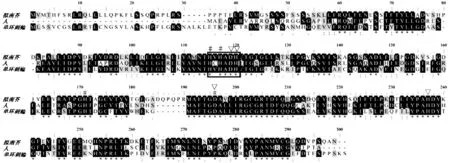
相同和相似的氨基酸显示为黑色和灰色,β-内酰胺酶家族的标签序列用黑框标出,金属结合位点I以#标出,金属结合位点II以▽标出。GenBank 注册号:拟南芥(NP_974018.3),人(NP_055112.2),单环刺螠 (AEV92813.1)
2.3 硫化物加氧酶的活性及催化机制
从小鼠肝脏和沙蠋的体壁中纯化出的硫双加氧酶最大比酶活为(0.87 ± 0.04)和(0.85 ± 0.24)U mg protein-1[8],而体外表达的单环刺螠硫双加氧酶为比酶活0.80 U mg protein-1[17]。此外拟南芥硫双加氧酶催化过硫化物的氧化反应其氧气的消耗率为(7.95 ± 0.71)μmol min-1mg protein-1[16]。对于底物的结合能力,单环刺螠硫双加氧酶的KM值(82.5 ± 9.9)μM[17],而人类为(340 ± 30)μM[20],耐硫生物的Km值明显低于不耐硫的人类,这从一个侧面解释了单环刺螠能够耐受硫化物的原因。
根据半胱氨酸双加氧酶的催化机制[21]以及单亚铁核心加氧酶的催化机制[22],Kabil和Banerjee提出了硫双加氧酶的催化机制(图4),在这个机制模型中,活性中心的Fe2+离子与2个组氨酸和1个天冬氨酸相连,剩余的3个位点被水占据(图4[1]);在催化反应过程中3个水分子被底物过硫化物代替(图4[2]),并在氧气的作用下形成Fe3+离子的超氧复合体(图4[3]);与Fe3+离子结合底物中的硫离子通过共振作用获得部分正电荷,因此Fe2+超氧复合体恢复(图4[4]),随后形成一个环状的过氧化物中间体(图4[5]);随着O-O键裂开,形成了硫氧正基团和金属活性中心结合的活化氧离子(图4[6]);最后该金属活性中心结合的活化氧离子传递到硫氧正基团,致使后者发生裂解反应,进而形成亚硫酸;由此硫双加氧酶恢复到初始的状态,并开始催化新一轮的反应;然而该催化机制还有待于光谱学的研究以进一步验证[20]。

图4 硫双加氧酶的催化机制
3 结语
本文对硫双加氧酶在线粒体硫化物氧化过程中的功能及硫双加氧酶基因的发现历程,基因结构和功能进行了综述。主要介绍了硫双加氧酶在线粒体硫化物氧化过程中催化过硫化物和氧气,水生成亚硫酸,为硫化物氧化过程中必不缺少的一步,并对其基因的结构和蛋白的三维结构进行综述,并根据其结构推测其催化机制。硫双加氧酶的研究能够加深人们对于硫化物代谢通路的认识,拓展人们对硫化物代谢分子机制的认识。此外,硫双加氧酶的功能异常会导致多种疾病,对于硫双加氧酶的研究进对于揭示这些疾病的起因以及治疗起着重要的意义。
[1] Cooper C E,Brown G C.The inhibition of mitochondrial cytochrome oxidase by the gases carbon monoxide,nitric oxide,hydrogen cyanide and hydrogen sulfide:chemical mechanism and physiological significance[J].J Bioenerg Biomembr,2008,40(5):533-539.
[2] Di Meo I,Fagiolari G,Prelle A,et al.Chronic exposure to sulfide causes accelerated degradation of cytochrome c oxidase in ethylmalonic encephalopathy[J].Antioxid Redox Signal,2011,15(2):353-362.
[3] Kamoun P.Endogenous production of hydrogen sulfide in mammals[J].Amino Acids,2004,26(3):243-254.
[4] Li L,Moore P K.Putative biological roles of hydrogen sulfide in health and disease:a breath of not so fresh air?[J]. Trends Pharmacol Sci,2008,29(2):84-90.
[5] Bouillaud F,Blachier F.Mitochondria and sulfide:a very old story of poisoning,feeding,and signaling? [J].Antioxid Redox Signal,2011,15(2):379-391.
[6] Powell M A,Somero G N.Hydrogen sulfide oxidation is coupled to oxidative phosphorylation in mitochondria of Solemya reidi[J].Science,1986,233(4763):563-566.
[7] Völkel S,Grieshaber M.Sulphide oxidation and oxidative phosphorylation in the mitochondria of the lugworm[J].J Exp Biol,1997,200(pt1):83-92.
[8] Hildebrandt T M,Grieshaber M K.Three enzymatic activities catalyze the oxidation of sulfide to thiosulfate in mammalian and invertebrate mitochondria[J].FEBS J,2008,275(13):3352-3361.
[9] Jackson M R,Melideo S L,Jorns M S.Human sulfide:quinone oxidoreductase catalyzes the first step in hydrogen sulfide metabolism and produces a sulfane sulfur metabolite[J].Biochemistry,2012,51(34):6804-6815.
[10] Tiranti V,Zeviani M.Altered sulfide (H2S) metabolism in ethylmalonic encephalopathy[J].Cold Spring Harb Perspect Biol,2013,5(1):84-93.
[11] Tiranti V,D'Adamo P,Briem E,et al.Ethylmalonic encephalopathy is caused by mutations in ETHE1,a gene encoding a mitochondrial matrix protein[J].Am J Hum Genet,2004,74(7):239-252.
[12] Tiranti V,Viscomi C,Hildebrandt T,et al.Loss of ETHE1,a mitochondrial dioxygenase,causes fatal sulfide toxicity in ethylmalonic encephalopathy[J].Nat Med,2009,2(2):200-205.
[13] Higashitsuji H,Higashitsuji H,Nagao T,et al.A novel protein overexpressed in hepatoma accelerates export of NF-κB from the nucleus and inhibits p53-dependent apoptosis[J].Cancer Cell,2002,2(4):335-346.
[14] Giordano C,Viscomi C,Orlandi M,et al.Morphologic evidence of diffuse vascular damage in human and in the experimental model of ethylmalonic encephalopathy[J].J Inherit Metab Dis,2012,35(3):451-458.
[15] Hildebrandt T M,Di Meo I,Zeviani M,et al.Proteome adaptations in Ethe1-deficient mice indicate a role in lipid catabolism and cytoskeleton organization via post-translational protein modifications[J].Biosci Rep,2013,33(4):575-584.
[16] Holdorf M M,Owen H A,Lieber S R,et al.Arabidopsis ETHE1 encodes a sulfur dioxygenase that is essential for embryo and endosperm development[J].Plant Physiol,2012,160(1):226-236.
[17] Zhang L,Liu X,Liu J,et al.Characteristics and function of sulfur dioxygenase in echiuran worm Urechis unicinctus[J].PLoS One,2013,8(12):e81885.
[18] Zhang G,Fang X,Guo X,et al.The oyster genome reveals stress adaptation and complexity of shell formation[J].Nature,2012,490(7418):49-54.
[19] Pettinati I,Brem J,McDonough M A,et al.Crystal Structure of human persulfide dioxygenase:structural basis of ethylmalonic encephalopathy[J].Hum Mol Genet,2015:ddv007.
[20] Kabil O,Banerjee R.Characterization of patient mutations in human persulfide dioxygenase (ETHE1) involved in H2S Catabolism[J].J Biol Chem,2012,287(53):44561-44567.
[21] McCoy J G,Bailey L J,Bitto E,et al.Structure and mechanism of mouse cysteine dioxygenase[J].Proc Natl Acad Sci U S A,2006,103(9):3084-3089.
[22] Koehntop K D,Emerson J P,Que L Jr.The 2-His-1-carboxylate facial triad:a versatile platform for dioxygen activation by mononuclear non-heme iron (II) enzymes[J].J Biol Inorg Chem,2005,10(2):87-93.
Advances of sulfur dioxygenase-a key enzyme in the mitochondrial sulfide oxidation
ZHANGLitao
(Department of Biological Sciences,Jining Medical University,Rizhao 276826,China)
In the mitochondrial sulfide oxidation,the function of sulfur dioxygenase (SDO) is to oxidize persulfide to sulfite using O2and H2O.The loss of its function results in the accumulation of sulfide in vivo,which leads to many diseases.This paper reviews the SDO gene characteristics,SDO catalytic activity and catalytic mechanisms,which is willing to offer valuable references for the further study of SDO and extensional understanding of sulfide metabolism mechanisms.
Sulfur dioxygenase;Mitochondria;Sulfide oxidation
10.3969/j.issn.1000-9760.2015.02.002
Q55
A
1000-9760(2015)04-082-04
2015-04-12)

