初始pH对厌氧环境下污泥中抗生素抗性基因行为特征的影响
钱燕云,徐莉柯,苏超,郑吉,陈红
浙江大学环境与资源学院环境工程研究所,杭州 310058
初始pH对厌氧环境下污泥中抗生素抗性基因行为特征的影响
钱燕云,徐莉柯,苏超,郑吉,陈红*
浙江大学环境与资源学院环境工程研究所,杭州 310058
污水处理厂产生大量的剩余污泥中含有丰富的抗性基因,给环境带来了潜在风险。以城市污水处理厂的剩余污泥为研究对象,在不同初始pH(对照组、初始pH=3、5、7、9、11)下观察厌氧条件下,8种抗生素浓度以及四环素类抗性基因(tet A、tet G、tet L、tet M、tet O、tet Q、tet W、tet X)、磺胺类抗性基因(sulI、sul II)和Ⅰ类整合子(intI 1)的行为特征。研究结果显示,初始pH对抗生素的降解影响较小,污泥中总抗生素的平均去除率为42%。对照组及初始pH为3、5、7、9、11下的总四环素类抗性基因分别削减0.65 log、0.96 log、0.75 log、0.62 log、0.86 log和0.98 log。不同四环素类抗性基因表现相似,在初始pH=3和初始pH=11下部分抗性基因削减较多,特别是tet A、tet G、tet L、tet O和tet X。2种磺胺类抗性基因均无削减,浓度平均上升0.18 log。相关性分析显示,总抗性基因与TN、NH3-N、TP、SCOD(溶解性COD)均存在显著相关性(P<0.05)。上述研究结果为污泥厌氧消化中抗生素抗性基因减量条件提供参考依据。
抗生素;抗性基因;污泥;初始pH;厌氧环境;序批式
抗生素特别是医用和兽用抗生素的大量使用和滥用,给环境中的细菌造成了长期的选择性压力,使得环境中的抗性细菌和抗性基因的丰度不断增加,从而给人类的健康带来了潜在的风险[1]。目前已有大量研究表明,污水处理厂中含有高浓度的抗性基因和抗性细菌,因此污水处理厂已成为抗性细菌增殖和扩散的聚集地[2-4]。Marti等[5]研究了污水处理厂的出水对附近河流中抗性基因和细菌群落组成的影响,发现污水处理厂排水点的河流下游比上游,在抗性基因的数量上明显增高,细菌群落组成也有很大的区别,这表明污水处理厂的出水可能促进了抗性基因传播到环境中并且影响受纳水体的细菌群落组成。Ben等[6]研究发现,污泥的吸附作用使水中的抗生素易转移到污泥中,故相比于污水,污泥中抗生素浓度更高,而污泥中抗性基因的浓度也更高[7-8]。目前,剩余污泥主要的处置措施是填埋或土地利用,这又给周围环境和土壤造成了很大的环境风险[9]。采用污泥处理技术对污泥进行预处理以有效减少污泥中抗性基因的含量成为新的研究方向,也具有一定的实践意义。Burch等[10]研究了空气干燥床对市政污水处理厂剩余污泥中抗性基因(erm B、sulI、tetA、tetW、tetX)和Ⅰ类整合子(intI1)的削减影响,结果显示空气干燥床可以削减所有目的基因,削减量范围为1~5个数量级。之后,Burch等[11]又采用半连续传统好氧消化技术处理剩余污泥以削减其中的抗性基因,研究发现tetA、tetW和erm B的削减超过了90%,intI1没有减少,tetX反而增加了5倍。半连续模式之后改为序批式来确定一级削减系数,半衰期范围为2.8~6.3 d。该结果表明好氧消化可以用来削减污泥中的抗性基因,但削减率根据反应器设计和特定的抗性基因不同变化很大,而且这个衰减速率比土地利用更快。
除此之外,另有研究证明污泥厌氧处理可以有效地减少污泥中高浓度的抗性基因[12-14],pH也影响着污泥在厌氧反应中的微生物群落[15],但pH对污泥中抗性基因行为特征影响的研究还比较少。因此,本研究采用1 L体积的序批式实验,分别设置污泥的初始pH为3、5、7、9、11,测定8种抗生素(四环素(TC)、金霉素(CTC)、氧四环素(OTC)、磺胺嘧啶(SD)、磺胺甲恶唑(SMX)、磺胺甲基嘧啶(SM1)、磺胺二甲嘧啶(SM2)、甲氧苄氨嘧啶(TMP))、四环素类抗性基因(tetA、tetG、tetL、tetM、tetO、tetQ、tetW、tetX)、磺胺类抗性基因(sulI、sulII)以及Ⅰ类整合子(intI1)在厌氧环境下的变化特征,同时测定总氮、总磷、SCOD(溶解性COD)和氨氮,探寻氮磷等营养元素与抗性基因以及intI1之间的相关性。
1 材料与方法 (Materials and methods)
1.1 实验装置
本实验采用1 L的三口圆底烧瓶为发酵罐,水浴恒温磁力搅拌器(金坛市医疗仪器厂,中国)来保证消化温度恒定,调节转速使装置中污泥达到全混状态。实验污泥取自临安城市污水处理厂的剩余污泥,加入去离子水,使得含固率在3%~5%之间,实验过程中,不添加任何营养物质。之后,将污泥分装到6个1 L的发酵罐中,搅拌1 d以达到稳定的状态,测定其初始pH为7.61,加入一定量的HCl(化学纯,中国国药有限公司)和NaOH(化学纯,中国国药有限公司)调节pH分别为3、5、7、9、11,并设置对照组。密封发酵罐,将发酵罐一口通入盛有水的锥形瓶,观察实验过程中是否有气泡产生。向装置中通氮气(高纯,杭州今工特种气体有限公司)5 min,除去其中的空气并检查装置气密性。恒定温度设为35℃的中温条件,控制温差在1 ℃左右。
1.2 样品的采集及预处理
在第0、5、10、20、30、40、60天(实验周期为60 d)进行采样,每个发酵罐各取15 mL,并进行分析。采集的样品先用大容量冷冻离心机(Thermo公司,德国)在10 000 r·min-1、4 ℃下冷冻离心10 min,上清液再用0.45 μm的滤膜进行抽滤。之后及时收集滤液,并测定其中的总氮、总磷、SCOD和氨氮,将滤膜和湿泥样放入物料盘在真空冷冻干燥机(宁波新芝生物科技有限分公司,中国)中预冻4~6 h,然后冻干12 h,将滤膜上的干污泥与干燥的泥样混合,磨碎过筛(80目)后储存于-20 ℃冰箱备用。
1.3 样品中水质指标的测定
pH测定采用pH值测试仪(上海精密科学仪器有限公司,中国)。SCOD测定采用重铬酸钾消解法,使用智能型多功能消解器(兰州连华环保科技有限公司,中国),使样品在150 ℃下消解2 h后,再用DR5000紫外可见分光光度计(北京安恒测试技术有限公司,中国)测定其吸光度,对照标准曲线得SCOD值。总氮测定采用碱性过硫酸钾消解紫外分光光度法(HJ636—2012)。氨氮测定采用纳氏试剂分光光度法(HJ 535—2009)。总磷测定采用钼酸铵分光光度法(GB11893—89)。
1.4 超高液相色谱- 串联质谱
泥样中8种抗生素(TC、CTC、OTC、SD、SMX、SM1、SM2、TMP)含量的测定采用超高液相色谱-串联质谱联用仪(UPLC-MS/MS)[16]。首先对样品进行预处理,称取冷冻干燥过筛后的干污泥0.08 g,采用震荡-超声-离心法萃取抗生素,重复3次,并用超纯水稀释至800 mL左右,目的是降低甲醇含量至2%以下。之后采用固相萃取法将稀释后的提取液进一步净化提纯,使用氮气吹干仪(天津市东康科技有限公司,中国)将洗脱液吹至2 mL以下,用甲醇∶水(V∶V=1∶1)定容至5 mL。预处理后的样品需要密封避光储存于-20 ℃,并在40 d内进行检测。检测前,添加内标物溶液。超高压液相色谱-质谱联用仪为Waters Acquity TM Ultra Performance LC串联Quattro Premier Micromass(Waters, USA),色谱分析柱为Waters AcquityTM UPLC BEH C18 (50 mm×2.1 mm, 1.7 μm),检测器为Quattro Premier Micromass,电离源为电喷雾电离源(ESI),采用MRM检测模式,Masslynx 4.0工作站(美国Waters公司)。定量方法为同位素内标法,可以消除其中基质干扰效应,根据检测内标物峰面积、待测物峰面积的比值,结合内标工作曲线得到实际样品的浓度。
1.5 荧光定量PCR
本实验首先采用FastDNA Spin Kit Forsoil试剂盒提取样品中的DNA,并用微量蛋白质核酸分析仪测定DNA的浓度和纯度。目的基因为四环素类抗性基因(tetA、tetG、tetL、tetM、tetO、tetQ、tetW、tetX)、磺胺类抗性基因(sulI、sulII)、Ⅰ类整合子整合酶基因(intI1)以及16S rRNA。定量PCR反应在Bio-Rad iQ5实时荧光定量PCR仪中进行。反应体系为7.5 μL的SYBR Premix Ex Taq溶液,0.3 μL的ROX Reference溶液,0.3 μL浓度为10 μmol·L-1的正向引物,0.3 μL浓度为10 μmol·L-1的反向引物,4.6 μL的ddH2O,2 μL的DNA模板。定量PCR反应程序为预变性95 ℃下热变性30 s;然后进入40个循环的扩增阶段,95 ℃变性5 s,退火30 s,72 ℃延伸30 s,延伸的同时扫描荧光信号,溶解曲线程序为55 ℃至95 ℃,每个样品3次重复,根据测得的Ct值对照同批次的标准曲线得出样品中抗性基因的浓度。表1为本实验中的引物和PCR条件。
1.6 标准曲线的建立
对临安污水处理厂剩余污泥进行每周1次为期1个月的采样,用DNA提取试剂盒提取DNA,并通过PCR扩增得到目的基因。之后采用凝胶回收试剂盒进行纯化回收,测定其DNA含量和纯度并调节至合适浓度,将调节后的DNA连接到pMD19-T载体,转入E. coli感态细胞DH5α。然后将感态细胞接种到含有氨苄青霉素,X-gal和IPTG的LB固体培养基上进行培养,培养时间为12~16 h。挑选阳性克隆子进行扩大培养及测序,将测序结果在NCBI网站(http://www.ncbi.nhn.nih.gov/blast/)上进行Blast序列同源性比对。质粒提取采用QIAGEN质粒专用提取试剂盒,微量核酸蛋白质分析仪监测提取质粒的含量及纯度,并确保质粒DNA的A260/A280比值在1.8左右。将符合要求的质粒作为该基因的标准品计算浓度(质粒浓度=(质量/分子量)×6.02×1023)。将已知浓度的质粒标准品依次进行10倍稀释,保证质粒浓度梯度在108~103ng·μL-1之间,然后进行荧光定量PCR实验,结果采用Bio-Rad iQ5软件分析得到各抗性基因的荧光定量标准曲线。
2 结果与讨论(Results and discussion)
2.1 抗生素变化规律
实验污泥均检测出上述8种抗生素,且四环素类抗生素浓度明显高于磺胺类抗生素。四环素类抗生素中氧四环素(OTC)浓度最高,达到1 633.3 μg·kg-1(干污泥),四环素(TC)和金霉素(CTC)的浓度分别为620.3 μg·kg-1(干污泥)和639.1 μg·kg-1(干污泥)。磺胺类抗生素中磺胺甲噁唑(SMX)检出含量最高,达到376.5 μg·kg-1(干污泥)。磺胺二甲嘧啶(SM2)的检出浓度最低,低于50 μg·kg-1(干污泥),磺胺甲基嘧啶(SM1)和磺胺嘧啶(SD)的浓度也相对较低,在70~100 μg·kg-1(干污泥)之间。该检出结果与Zhou等[22]对广东2个污水处理厂污泥中抗生素的检测结果一致,四环素类抗生素显著较高,特别是氧四环素,因为该类抗生素易吸附在污泥上,且人类对四环素类抗生素的使用率更高。磺胺类抗生素含量较低,因为该类抗生素主要以生物降解为主,不易吸附在污泥上。抗生素的降解主要是依靠水解作用,环境条件的变化很大程度上影响着抗生素的降解[23]。

表1 本实验中的引物和PCR条件Table 1 Primer and PCR conditions used in this study
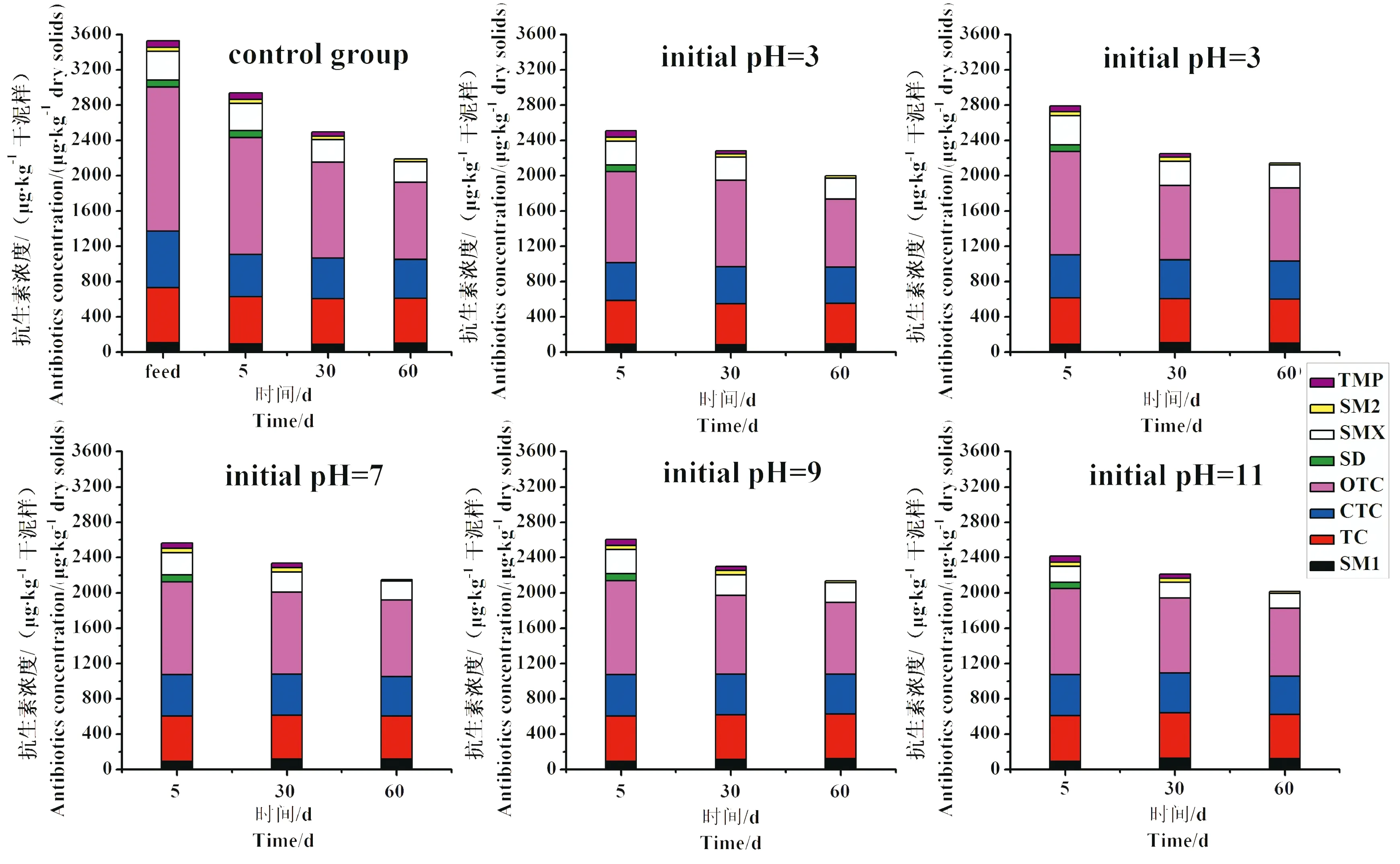
图1 不同初始pH下厌氧环境污泥中抗生素的浓度变化

图2 不同初始pH下厌氧环境污泥中四环素类抗性基因的变化趋势
如图1为不同初始pH下抗生素的变化趋势,初始pH对抗生素的降解影响不显著(P>0.05),污泥中的部分抗生素得到削减。反应过程中总抗生素的平均去除率为42%,其中四环素类抗生素平均去除率为40%,磺胺类抗生素为43%,大部分抗生素仍残留在污泥 中。
2.2 抗性基因变化规律
在原始污泥中,使用定量和定性PCR,均未检测出tetQ,而其中tetX的检出浓度最高,达到1.0×108copies·g-1(干污泥),tetA和tetG的浓度也较高,达到107copies·g-1(干污泥),tetL的浓度最低,为5.1×105copies·g-1(干污泥)。该检测结果与Zhang等[24]对15个国内外污水处理厂污泥中抗性基因的研究结果相似,15个污水处理厂的污泥样品中也均未检测到tetQ,而之前的研究显示在污水处理厂、河流及土壤中均能检测到tetQ[25-27],该检出差异的原因可能在于tetQ的转移和传播机制还不明确,不同的环境和不同的地域对其行为特征的影响较大。本研究中,大部分抗性基因的削减幅度很小,tetL和tetW得到较大的削减。如图2为不同初始pH下四环素类抗性基因的变化趋势,对照组及初始pH为3、5、7、9、11下的四环素类抗性基因分别削减0.65 log、0.96 log、0.75 log、0.62 log、0.86 log和0.98 log。对照组及初始pH=7的装置中,抗性基因呈下降趋势,在20 d后基本无变化。初始pH为3、5、9、11的装置中,不同抗性机理的四环素类抗性基因表现相似,总体呈波动趋势,在反应前20天,抗性基因平均削减0.75 log,20~30 d抗性基因平均回升0.30 log,30 d后又平均削减0.44 log。在初始pH=3和初始pH=11下部分抗性基因削减较多(与对照组相比,P<0.05),特别是编码“外排泵”抗性基因(tetA、tetG、tetL分别平均削减0.97 log、0.94 log、1.64 log),编码核糖体保护蛋白基因中的tetO(平均削减1.04 log),以及编码修饰或钝化四环素的酶基因tetX(平均削减1.11 log)。
在全部的污泥样品中均检测出2种常见的磺胺类抗性基因(sulI、sulII),浓度分别为1.81×109copies·g-1(干污泥)、5.39×108copies·g-1(干污泥)。如图3,反应过程中磺胺类抗性基因均无削减,浓度反而平均上升0.18 log。各个初始pH下抗性基因变化趋势相似,反应前20天时,平均削减0.85 log,这与大部分四环素类抗性基因变化相似。但不同的是反应超过20 d后,sulI、sulII浓度急剧上升,平均上升达1.02 log。初始pH=3和初始pH=11下磺胺类抗性基因浓度相对较低。
Ⅰ类整合子(intI1)是一段DNA片段,可嵌入外源基因盒并将它们转换为能正确表达的功能性基因,可影响抗性基因的传播[28]。本研究每个样品中均检测出高浓度的Ⅰ类整合子整合酶基因(intI1),范围在4.57×107copies·g-1(干污泥)~2.02×108copies·g-1(干污泥)之间。如图4,intI1在不同初始pH下削减幅度较小,平均削减0.25 log。整体趋势与磺胺类抗性基因相似。相比于其他初始pH,intI1在初始pH为3和11的装置中的丰度较低,说明在一定程度上改变污泥的酸碱性能改变微生物生长环境,对细菌种属有一定的选择性,从而影响抗性基因的行为特征。

图4 不同初始pH下厌氧环境污泥中Ⅰ类整合子的变化趋势
微生物群落的结构与环境条件相关联,环境条件的变化会引起微生物种群的演替,这是微生物群落对外界环境的适应机制。微生物可以生长的pH范围极广,但绝大多数种类的细菌生活在pH 5.0~9.0之间。在反应的前期,外界环境的酸碱性急剧变化,使得其中对酸碱耐受性差的微生物种群数量和活性受到了抑制,相关代谢功能出现衰退[29]。甚至有些功能微生物因不适应环境而衰亡,细胞裂解释放的DNA,与胞外的DNA被部分水解或者生物降解,而且在这个过程中新的优势种群还未形成。因此,反应前20天,污泥中的抗性基因削减较多,且当酸碱性越大,条件越恶劣,抗性基因削减越多。外界pH发生改变,但微生物细胞内的pH相当稳定,一般接近中性。实验测得20 d后,各个装置中的pH已经接近中性,说明微生物已经适应并改变了周围环境,在此过程中可能又形成了新的优势菌群,为抗性基因提供更多的宿主,因此在20 d后抗性基因浓度有所回升,特别是磺胺类抗性基因。另外,intI1的回升可能引起了基因的横向转移,污泥环境中的高生物量及多样性为横向基因转移提供更多的可能性[30]。除此之外,Calero-Ca′ceres等[31]研究发现污泥中的细菌噬菌体内含有高浓度的抗性基因,噬菌体可以将抗性基因存储在处理过的污泥中,即使是经过厌氧处理的污泥。其中有些噬菌体会为抗性基因提供更多的宿主选择,因此对抗性基因的传播起重要作用。
2.3 相关性分析
微生物与周围的环境具有复杂的相互影响和相互作用。pH的改变会影响微生物的生长繁殖,微生物的生长繁殖和代谢也会影响周围的环境。因此,本实验测定不同初始pH下装置中TN、TP、NH3-N和SCOD含量,观察其变化规律,分析其与抗性基因变化的相关性。如图5,SCOD和TP浓度变化呈下降趋势,而NH3-N和TN总体呈上升趋势。
微生物生长环境的酸碱性改变,影响微生物膜表面电荷的性质和膜的通透性,也可能改变污泥中某些有机化合物的离子化状态,从而影响细胞对营养物质的吸收[32]。本实验对TN、NH3-N、TP、SCOD与抗性基因的相关性进行分析结果如表2,星号标注表示两者具有显著相关性(P <0.05)。由表可知,总抗性基因丰度与TN、NH3-N、TP、SCOD皆存在显著相关性(P<0.05)。单个抗性基因有tetW和sulII与TN、NH3-N、TP、SCOD存在显著相关性(P<0.05),另外tetL、tetM、sulI与TP存在显著相关性(P<0.05),tetA、tetG、sulI与SCOD存在显著相关性(P<0.05)。

图5 不同初始pH下厌氧环境TN、TP、NH3-N和SCOD的变化趋势

表2 抗性基因与SCOD、NH3-N、TN、TP的相关性分析Table 2 Correlation analysis of ARGs and SCOD, NH3-N, TN and TP
注:* 表示两者具有显著相关性(P<0.05)。
Note: Asterisk means there was a significant correlation of ARGs and SCOD, NH3-N, TN and TP.
此外,研究还发现intI1与TP、SCOD存在显著相关性(P<0.05)。污泥中微生物多样性复杂,已有研究通过质粒宏基因组分析证明了污水处理厂的污泥中含有高浓度的抗性基因以及移动基因盒,在Ⅰ类整合子整合酶的作用下,细菌可能捕获外源的基因盒,使得其中的抗性基因在整合子上游启动子的作用下得到表达,从而使细菌具有耐药性,因此这些移动基因盒一定程度上促进了抗性基因的横向转移[33]。磷与intI1的显著相关性,说明营养元素是抗性基因扩散和传播的影响因素,但具体影响程度和范围仍需进一步研究证实。
[1] Pruden A. Balancing water sustainability and public health goals in the face of growing concerns about antibiotic resistance [J]. Environmental Science & Technology, 2013, 48(1): 5-14[2] Gao P, Munir M, Xagoraraki I. Correlation of tetracycline and sulfonamide antibiotics with corresponding resistance genes and resistant bacteria in a conventional municipal wastewater treatment plant [J]. Science of the Total Environment, 2012, 421-422(3): 173-183
[3] Bouki C, Venieri D, Diamadopoulos E. Detection and fate of antibiotic resistant bacteria in wastewater treatment plants: A review [J]. Ecotoxicology and Environmental Safety, 2013, 91(4): 1-9
[4] Kim S, Park H, Chandran K. Propensity of activated sludge to amplify or attenuate tetracycline resistance genes and tetracycline resistant bacteria: A mathematical modeling approach [J]. Chemosphere, 2010, 78(9): 1071-1077
[5] Marti E, Jofre J, Balcazar J L. Prevalence of antibiotic resistance genes and bacterial community composition in a river influenced by a wastewater treatment plant [J]. Plos One, 2013, 8(10): 78906-78906
[6] Ben W W, Qiang Z M, Yin X W, et al. Adsorption behavior of sulfamethazine in an activated sludge process treating swine wastewater [J]. Journal of Environmental Sciences, 2014, 26: 1623-1629
[7] Börjesson S, Dienues O, Jarnheimer P A, et al. Quantification of genes encoding resistance to aminoglycosides, β-lactams and tetracyclines in wastewater environments by real-time PCR [J]. International Journal of Environmental Health Research, 2009, 19(3): 219-230
[8] Szczepanowski R, Linke B, Krahn I, et al. Detection of 140 clinically relevant antibiotic-resistance genes in the plasmid metagenome of wastewater treatment plant bacteria showing reduced susceptibility to selected antibiotics [J]. Microbiology, 2009, 155(7): 2306-2319
[9] Munir M, Xagoraraki I. Levels of antibiotic resistance genes in manure, biosolids, and fertilized soil [J]. Journal of Environmental Quality, 2011, 40(1): 248-255
[10]Burch T R, Sadowsky M J, Lapara T M. Air-drying beds reduce the quantities of antibiotic resistance genes and class 1 integrons in residual municipal wastewater solids [J]. Environmental Science & Technology, 2013, 47(17): 9965-9971
[11]Burch T R, Sadowsky M J, Lapara T M. Aerobic digestion reduces the quantity of antibiotic resistance genes in residual municipal wastewater solids [J]. Frontiers in Microbiology, 2013, 4(17): 1-9
[12]Ma Y J, Wilson C A, Novak J T, et al. Effect of various sludge digestion conditions on sulfonamide, macrolide, and tetracycline resistance genes and class I integrons [J]. Environmental Science & Technology, 2011, 45(18): 7855-7861
[13]Ghosh S, Ramsden S J, Lapara T M. The role of anaerobic digestion in controlling the release of tetracycline resistance genes and class 1 integrons from municipal wastewater treatment plants [J]. Applied Microbiology and Biotechnology, 2009, 84(4): 791-796
[14]Diehl D L, Lapara T M. Effect of temperature on the fate of genes encoding tetracycline resistance and the integrase of class 1 integrons within anaerobic and aerobic digesters treating municipal wastewater solids [J]. Environmental Science & Technology, 2010, 44(23): 9128-9133
[15]Cheng W, Hong C, Yan S H, et al. Illumina sequencing-based analyses of bacterial communities during short-chain fatty-acid production from food waste and sewage sludge fermentation at different pH values [J]. World Journal of Microbiology & Biotechnology, 2014, 30(9): 2387-2395
[16]朱赛嫦, 王静, 邵卫伟, 等. 超高效液相色谱-串联质谱法同时检测地表水中18种药物与个人护理品的残留量[J]. 色谱, 2013, 31(1): 15-21
Zhu S C, Wang J, Shao W W, et al. Simultaneous determination of 18 pharmaceuticals and personal care products in surface water by ultra-high performance liquid chromatography-tandem mass spectrometry [J]. Chinese Journal of Chromatography, 2013, 31(1): 15-21 (in Chinese)
[17]Ng L K, Martin I, Alfa M, et al. Multiplex PCR for the detection of tetracycline resistant genes [J]. Molecular & Cellular Probes, 2001, 15(4): 209-215
[18]Aminov R I, Garrigues J N, Mackie R I. Molecular ecology of tetracycline resistance: Development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins [J]. Applied & Environmental Microbiology, 2001, 67(1): 22-32
[19]Luo Y, Mao D, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China [J]. Environmental Science & Technology, 2010, 44(19): 7220-7225
[20]Liu M M, Zhang Y, Yang M, et al. Abundance and distribution of tetracycline resistance genes and mobile elements in an oxytetracycline production wastewater treatment system [J]. Environmental Science & Technology, 2012, 46(14): 7551-7557
[21]Gaze W H, Zhang L, Abdouslam N A, et al. Impacts of anthropogenic activity on the ecology of class 1 integrons and integron-associated genes in the environment [J]. ISME Journal, 2011, 5(8): 1253-1261
[22]Zhou L J, Ying G G, Shan L, et al. Occurrence and fate of eleven classes of antibiotics in two typical wastewater treatment plants in South China [J]. Science of the Total Environment, 2013, 452-453(5): 365-376
[23]Chee-Sanford J C, Mackie R I, Koike S, et al. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste [J]. Journal of Environmental Quality, 2009, 38(3): 1086-1108
[24]Zhang X X, Zhang T. Occurrence, abundance, and diversity of tetracycline resistance genes in 15 sewage treatment plants across China and other global locations [J]. Environmental Science & Technology, 2011, 45(7): 2598-2604
[25]Auerbach E A, Seyfried E E, Mcmahon K D. Tetracycline resistance genes in activated sludge wastewater treatment plants [J]. Proceedings of the Water Environment Federation, 2006, 41(5): 1478-1493
[26]Mackie R I, Koike S, Krapac I G, et al. Tetracycline residues and tetracycline resistance genes in groundwater impacted by swine production facilities [J]. Animal Biotechnology, 2006, 17(2): 157-176
[27]Patterson A J, Colangeli R, Spigaglia P, et al. Distribution of specific tetracycline and erythromycin resistance genes in environmental samples assessed by macroarray detection [J]. Environmental Microbiology, 2007, 9(3): 703-715
[28]Mazel D. Integrons: Agents of bacterial evolution [J]. Nature Reviews Microbiology, 2006, 4(8): 608-620
[29]Liu J, Hu G, Li F, et al. Advances in the microbial flora of anaerobic digestive systems [J]. Industrial Water Treatment, 2011, 31(10): 10-14
[30]Schlüter A, Szczepanowski R, Pühler A, et al. Genomics of IncP-1 antibiotic resistance plasmids isolated from wastewater treatment plants provides evidence for a widely accessible drug resistance gene pool [J]. FEMS Microbiology Reviews, 2007, 31(4): 449-477
[31]William C C, Ana M, Marta C L, et al. Sludge as a potential important source of antibiotic resistance genes in both the bacterial and bacteriophage fractions [J]. Environmental Science & Technology, 2014, 48(13): 7602-7611
[32]陈燕飞. pH对微生物的影响[J]. 太原师范学院学报: 自然科学版, 2009, 8(3): 121-124
Chen Y. pH touygur biology influence [J]. Journal of Taiyuan Normal University: Natural Science Edition, 2009, 8(3): 121-124 (in Chinese)
[33]Zhang T, Zhang X X, Ye L. Plasmid metagenome reveals high levels of antibiotic resistance genes and mobile genetic elements in activated sludge [J]. Plos One, 2011, 6(10): e26041
◆
Effect of Initial pH on Antibiotic Resistance Genes Behavior During Anaerobic Treatment of Sludge
Qian Yanyun, Xu Like, Su Chao, Zheng Ji, Chen Hong*
Department of Environmental Engineering, College of Environmental and Resource Sciences, Zhejiang University, Hangzhou 310058, China
Received 31 August 2015 accepted 12 October 2015
The residual sludge produced from sewage treatment plant contains high concentrations of antibiotic resistance genes (ARGs), which may bring the potential risk to the environment. In this study, residual sludge was operated at different initial pH (control group, 3, 5, 7, 9, 11) to investigate the fate of antibiotics and ARGs under anaerobic treatment. Antibiotics were analyzed by liquid chromatography-electrospray tandem mass spectrometry (LC-MS/MS) and the abundances of tetracycline resistance genes (tetA, tetG, tetL, tetM, tetO, tetQ, tetW, tetX), sulfonamide resistance genes (sulI, sulII) and class 1 integron (intI1) were quantified by real-time polymerase chain reaction during the process. The results showed that initial pH had slight influence on the removal of antibiotics. The average removal of antibiotics was 42%. The removal rate of tet genes in the control group and the group at initial pH 3, 5, 7, 9, 11 were 0.65 log, 0.96 log, 0.75 log, 0.62 log, 0.86 log and 0.98 log, respectively. For sul genes, the abundance decreased and then increased by 0.18 log. Correlation analysis showed total resistance genes had positive correlations with TN, NH3-N, TP and SCOD (P<0.05).
antibiotics; antibiotic resistance genes; sludge; initial pH; anaerobic; sequencing batch
国家重大水专项(2014ZX07101-012);国家自然科学基金项目(21277117)
钱燕云(1990-),女,硕士研究生,研究方向为污染控制及资源化技术,E-mail: aileen@zju.edu.cn;
*通讯作者(Corresponding author), E-mail: chen_hong@zju.edu.cn
10.7524/AJE.1673-5897. 20150831001
2015-08-31录用日期:2015-10-12
1673-5897(2015)5-047-09
X171.5
A
陈红(1969—),女,博士,教授,主要研究方向为污染控制及资源化技术和环境生物技术。
钱燕云, 徐莉柯, 苏超, 等. 初始pH对厌氧环境下污泥中抗生素抗性基因行为特征的影响[J]. 生态毒理学报,2015, 10(5): 47-55
Qian Y Y, Xu L K, Su C, et al. Effect of initial pH on antibiotic resistance genes behavior during anaerobic treatment of sludge [J]. Asian Journal of Ecotoxicology, 2015, 10(5): 47-55 (in Chinese)

