粗皮桉ISSR-PCR反应体系优化
陈升侃,项东云,邓紫宇,梁建新,蓝必布,李昌荣,梁 机
粗皮桉ISSR-PCR反应体系优化
陈升侃1,2,项东云2,邓紫宇2,梁建新3,蓝必布3,李昌荣2,梁 机1,2*
(1. 广西大学林学院,广西南宁530004;2. 广西优良用材林资源培育重点实验室广西林科院,广西南宁530002;3. 广西国有大桂山林场,广西贺州 542800)
为建立粗皮桉ISSR-PCR优化反应体系,先通过单因素试验确定影响粗皮桉ISSR-PCR 5个因素(Mg2+、dNTP、DNA聚合酶、DNA模板、引物)的较适宜浓度范围,在此基础上进一步通过正交试验对5个因素4个水平进行优化,并用DPS软件分析试验结果。结果表明粗皮桉ISSR-PCR的优反应体系为:在25 µL反应体系中,10×PCR buffer 2.5 µL、MgCl 2.0 mmol·L-1、dNTPs 0.3 mmol·L-1、DNA聚合酶1.25 U、DNA模板60 ng、引物0.8 µmol·L-1。通过梯度试验确定的扩增程序为:94℃预变性5 min,然后按94℃变性1 min,51℃退火3 min,72℃延伸2 min,进行35个循环,最后72℃延伸5 min,4℃保存。
粗皮桉;ISSR-PCR;体系优化;单因素试验;正交试验
粗皮桉()是桃金娘科(Myrtaceae)桉树属树种,天然分布于澳大利亚,是重要的用材树种和优良的水源涵养树种,其木材呈红色至深红色,坚固而耐久,被广泛用于建筑、枕木、造船等用材。该树种树皮厚、抗逆性强、木材密度良好,目前已被广泛引种于广东、广西及海南地区[1-2]。对粗皮桉的研究主要集中于种源试验[3-5]、木材材性及加工利用方面[6-13],同时也有学者从粗皮桉的化学组成特性、抗风与生长等进行了研究[14-15]。
简单序列重复区间(Inter-simple sequence repeat,ISSR),由加拿大蒙特利大学的Zietkiewicz等[16]于1994年提出,是一种基于SSR的简单重复序列区间扩增多态性分子标记。它具有模板需求量少、试验操作简单、试验成本低、多态性丰富好、快速高效等优点[17]。ISSR分子标记技术已被广泛用于遗传多样性[18-19]、遗传图谱构建[20-21]品种亲缘关系及分类[22]等研究中,但前提是要建立一个扩增效果较好的PCR反应体系,曾艳玲等[23]以邓恩桉()为材料建立了ISSR-PCR的优化体系,但不同树种对PCR反应条件的要求不一样,粗皮桉的ISSR-PCR优化反应体系还有待进一步研究。本文利用单因素试验和正交试验双重试验方法[24]对粗皮桉ISSR-PCR反应体系进行优化。
1 材料和方法
1.1 供试材料
以粗皮桉嫩叶所提取的DNA为模板进行体系优化,其材料取自广西国有东门林场。
1.2 主要试剂与仪器
1.2.1 试剂
植物基因组DNA提取试剂盒,DNA聚合酶,dNTPs,MgCl2,10×PCR buffer,引物参照哥伦比亚大学UBC公司2006年公布的ISSR引物序列,经初步筛选出的858号引物,即(TGT)5GRT(其中R为A或G),I型核酸染色剂,琼脂糖,1×TBE。
1.2.1 主要仪器
Sigma 3-30k离心机,Professional Thermocycler型PCR仪,DYY-6D型电泳仪,BIO-RAD凝胶成像系统,超微量核酸蛋白检测仪ND-2000。
1.3 试验方法
1.3.1 粗皮桉基因组DNA的提取
采用北京天根生化科技有限公司提供的植物基因组DNA提取试剂盒提取粗皮桉DNA,用1%的琼脂糖凝胶电泳检测DNA的完整性,并用超微量核酸蛋白检测仪ND-2000检测DNA的浓度和纯度。最后将样品稀释成10 mg·L-1,用于ISSR反应体系的优化试验。
1.3.2 ISSR-PCR扩增反应单因素试验
反应体系的优化先按照单因素梯度设计进行(表1),优化影响ISSR-PCR反应的5个主要因素:Mg2+、dNTPs、DNA聚合酶、DNA模板、引物,在其他因素不变的条件下,变化单一因子,筛选出最适宜的浓度范围,为进一步优化提供依据。ISSR基本反应体系组成:10×PCR buffer 2.5 µL,MgCl23.0 mmol·L-1,DNA模板20 ng,引物0.2 µmol·L-1,DNA聚合酶0.5 U,dNTPs 0.2 mmol·L-1,最后用无菌超纯水补足至25 µL。ISSR-PCR基本扩增程序:94℃预变性5 min,然后按94℃变性1 min,53℃退火3 min,72℃延伸2 min,进行35个循环,最后72℃延伸5 min,4℃保存。在优化前先进行预试验,初步筛选扩增结果较好的引物,以便在优化时获得较好的结果。取扩增产物10 µL和2 µL上样缓冲液混匀,点样于1.5%的琼脂糖凝胶的上样孔里,以D3000 bp DNA ladder作为对照分子量标准,在1×TBE缓冲液中电压5 V·cm-1电泳1.5 h,然后在BIO-RAD凝胶成像系统下拍照记录,检验并分析优化结果。
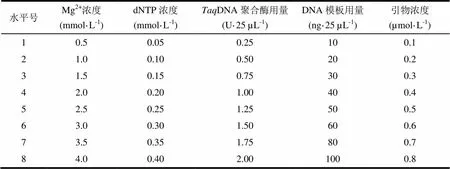
表1 ISSR-PCR体系优化的因素与水平
1.3.3 ISSR-PCR热循环反应参数梯度试验
在单因素试验优化后的反应体系下,以上述基本扩增程序为基础,进行循环次数分别为20、25、30、35、40次的ISSR-PCR扩增试验以确定最适宜的循环次数。设计5个退火温度梯度:51℃、52℃、53℃、54℃、55℃,对引物进行退火温度筛选,以便得出条带丰富、清晰且稳定的引物并确定其优化体系的最适宜退火温度。
1.3.4 ISSR-PCR反应因素水平的正交设计
正交试验选用L16(45)正交表安排试验(表2),对Mg2+浓度、dNTP浓度、DNA聚合酶用量、DNA模板用量和引物浓度5个因素设计4个水平,各因素水平的设计参照单因素试验得到的最适宜浓度范围,各因素试验重复3次。正交试验的ISSR-PCR扩增程序为反应参数梯度试验所得的优化扩增程序,PCR产物的检测与单因素试验相同。

表2 ISSR-PCR体系优化正交试验设计L16(45)及试验评分结果
1.4数据处理
正交试验结果的处理采用打分方式[25],评分标准为电泳结果谱带的强弱、条带数量、清晰度和杂带数量,并以条带强弱和数量为评分主体,从1 ~ 16依次排序打分。应用SAS软件进行方差分析及Duncan多重比较。
2 结果与分析
2.1 粗皮桉总DNA的提取
本研究采用现有的植物基因组DNA提取试剂盒进行DNA提取,通过电泳结果显示(图1),所提取的DNA完整性较好。通过ND-2000核酸蛋白检测仪检测结果,所提取DNA的OD260/OD280均在1.7 ~ 2.0之间,说明所提取的DNA纯度较高,符合ISSR-PCR扩增反应的要求。

图1 部分粗皮桉DNA提取电泳检测结果
2.2 单因素试验结果分析
2.2.1 Mg2+浓度对ISSR-PCR反应的影响
在PCR扩增反应中Mg2+对DNA聚合酶进行热激活,其浓度大小影响酶的活性,并对退火温度和解链温度产生影响。试验结果表明(图2),当Mg2+浓度为0.5 ~ 1.0 mmol·L-1时无法扩增出条带(图2泳道1 ~ 2);浓度提高到1.5 mmol·L-1后,能够扩增出产物但条带较少(图2泳道3);浓度在2.0 ~ 4.0 mmol·L-1之间,扩增带型一致,条带丰富清晰(图2泳道4 ~ 8)。故在进行正交试验时,Mg2+浓度的水平设计取值范围应当在2.0 ~ 4.0 mmol·L-1之间。

图2 Mg2+浓度的影响
注:M泳道为Maker DL 3000,1 ~ 8泳道的浓度分别为:0.5、1.0、1.5、2.0、2.5、3.0、3.5、4.0 mmol·L-1
2.2.2 dNTPs浓度对ISSR-PCR反应的影响
dNTPs是由dATP、dGTP、dCTP、dTTP 4种等摩尔浓度混合而成,是PCR扩增反应的原材料,其浓度的变化直接影响到扩增产物的产量和特异性。试验结果表明(图3),dNTPs浓度为0.05 mmol·L-1时,因浓度过低,扩增产物产量较低,无法分辨出所扩增的条带(图3泳道1);即使浓度提高到0.10 mmol·L-1,条带仍较模糊(图3泳道2);当浓度为0.15 ~ 0.30 mmol·L-1时,条带较多,带型一致,且主带逐渐变亮(图3泳道3 ~ 6);若浓度继续提高到0.35 mmol·L-1,主带较亮,但缺失2条弱带(图3泳道7);浓度高至0.40 mmol·L-1后仅扩增出1条带。dNTPs浓度的适宜范围在0.15 ~ 0.30 mmol·L-1,进行正交试验的dNTPs浓度水平依据此浓度范围。

图3 dNTPs浓度的影响
注:M泳道为Maker DL 3000,1 ~ 8泳道的浓度分别为:0.05、0.10、0.15、0.20、0.25、0.30、0.35、0.40 mmol·L-1
2.2.3DNA聚合酶用量对ISSR-PCR反应的影响
高温嗜热DNA聚合酶是PCR反应的重要组成成分,其活性和用量对PCR反应结果影响较大。DNA聚合酶用量较低,会导致因酶过早消耗完而使扩增产物得率较低;DNA聚合酶用量过高会导致非特异性产物增加。试验结果表明(图4),DNA聚合酶的浓度在0.5 ~ 1.25 U·25 µL-1之间,扩增出的条带丰富、带型一致且较清晰(图4泳道2 ~ 5);而其他浓度所扩增出来的条带较少且不清晰(图4泳道1、6 ~ 8)。由此说明粗皮桉ISSR-PCR反应的DNA聚合酶最适宜用量在0.5 ~ 1.25 U·25 µL-1之间,以此用量范围作为正交设计的试验的依据。

图4 Taq DNA聚合酶用量的影响
注:M泳道为Maker DL 3000,1 ~ 8泳道的用量分别为:0.25、0.50、0.75、1.00、1.25、1.50、1.75、2.00 U·25 µL-1)
2.2.4 DNA模板用量对ISSR-PCR反应的影响
DNA模板的质量和数量是影响PCR反应的重要因素之一,模板应尽量纯净,避免受RNA、核酸酶、蛋白水解酶等的污染;DNA模板用量过多也会引起非特异性产物的增加。本试验设置的DNA模板用量在10 ~ 100 ng之间,结果表明(图5),所有用量扩增的带型一致,且主带随着模板用量的增加而明显变亮;但用量为10 ~ 20 ng时,条带均较暗,不够清晰(图5泳道1 ~ 2),用量为80 ~ 100 ng时,有2条弱带无法分辨(图5泳道7 ~ 8);而用量在30 ~ 60 ng之间,条带则比较稳定且清晰(图5泳道3 ~ 6)。故本研究正交试验中DNA模板用量的4个水平设置为30、40、50、60 ng。

图5 DNA模板用量的影响
注:M泳道为Maker DL3000,1 ~ 8泳道的用量分别为:10、20、30、40、50、60、80、100 ng·25 µL-1
2.2.5 引物浓度对ISSR-PCR反应的影响
引物浓度的高低也是影响PCR反应的因素之一。从图6可看出,8个引物浓度梯度中,浓度在0.1 ~ 0.4 µmol·L-1之间均无法扩增出条带(图6泳道1 ~ 4);浓度为0.5 ~ 0.8 µmol·L-1时,条带较多(图6泳道5 ~ 8),但各条带亮度不一。本研究正交试验所设置的4个引物浓度为0.5、0.6、0.7、0.8 µmol·L-1。
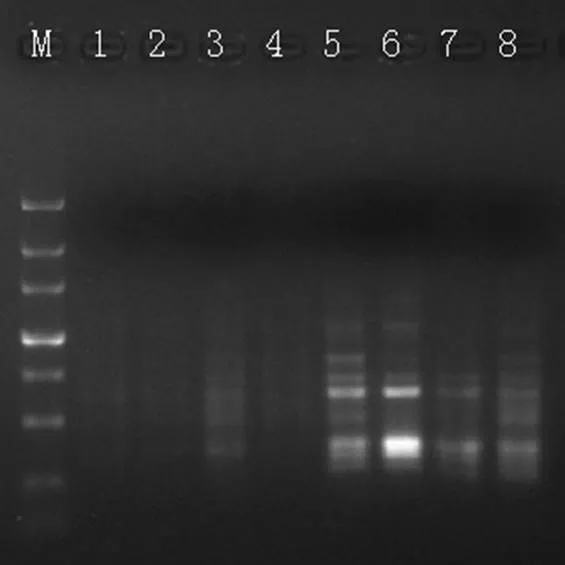
图6 引物浓度的影响
注:M泳道为Maker DL3000,1 ~ 8泳道的用量分别为:0.1、0.2、0.3、0.4、0.5、0.6、0.7、0.8 µmol·L-1
2.3 ISSR-PCR热循环的反应参数
2.3.1 循环次数对ISSR-PCR反应的影响
循环次数对PCR扩增产物的产量影响较大,PCR循环次数不足,会引起扩增产物量不足,电泳时部分条带无法检测出来;循环次数过多,不仅不能使产量增加,反而引起非特异性扩增,错配比例升高,发生弥散现象[26]。循环次数梯度试验结果表明(图7),循环次数为20、25次,循环次数过低无法扩增出条带;设置为30次循环时,扩增产物少,条带弱;将循环次数提高至35、40次,扩增产物明显增多,条带带型一致,清晰稳定。考虑到时间影响,最佳的循环次数为35次。

图7 循环次数的影响
注:M泳道为Maker DL3000,1 ~ 5泳道分别为:20、25、30、35、40循环
2.3.2 退火温度对ISSR-PCR反应的影响
引物退火温度主要取决于引物碱基组成、引物浓度、引物与模板的配对程度等,退火温度低,退火较容易,但特异性低;退火温度过高,可提高产物特异性,但影响引物与模板的结合程度[24]。退火温度梯度试验结果表明(图8),温度为52℃、54℃时,条带较少;51℃、53℃、55℃时,条带较丰富,其中51℃时条带清晰稳定,与53℃、55℃相比弱带较少,最佳退火温度为51℃。

图8 退火温度的影响
注:M泳道为Maker DL3000,1 ~ 5泳道为51、52、53、54、55℃
2.4 正交试验结果分析
根据正交试验扩增产物电泳结果(图9),对3次重复试验分别进行评分(表2),得出结果值最高为组合4:MgCl22.0 mmol·L-1、dNTP 0.3 mmol·L-1、DNA聚合酶1.25 U、DNA模板60 ng、引物0.8 µmol·L-1。利用软件SAS进行方差分析(表3),结果表明各因素水平间的差异极显著。由表中值可知,各因素水平的变化对ISSR-PCR影响程度的大小顺序为Mg2+>dNTPs>DNA模板>引物>DNA聚合酶。

图9 正交试验电泳图重复1
注:M为Maker DL3000,泳道1 ~ 16分别为正交试验结果

表3 正交试验方差分析表
对试验结果进一步做Duncan多重比较(表4),结果表明:Mg2+浓度为2.0 mmol·L-1时,结果均值最大,与其他3个水平的差异显著。结果均值最大的dNTP浓度为0.3 mmol·L-1,与其他3个水平有显著差异。对DNA用量的比较表明,在一定范围内高浓度的DNA对扩增效果较好,用量为50 ng、60ng的PCR反应结果均值显著高于30 ng、40 ng。对引物的比较分析表明,结果均值随着引物浓度的升高而降低,且0.5 ~ 0.7 µmol·L-1对PCR效果没有差异。DNA聚合酶用量在0.50 ~ 1.00 U时PCR反应均值差异不显著。
正交试验结果值最高的组合中,Mg2+和dNTP浓度对PCR效果显著优于其他浓度,60 ng的DNA用量其均值虽低于50 ng,但并无差异且显著优于其他DNA用量,引物浓度和DNA聚合酶对PCR的影响程度最小。组合4可作为粗皮桉的PCR优化体系。

表4 各因素水平的Duncan多重比较
注:各列均值后不同小写字母表示在0.05水平上的差异。
2.5 结果验证
用单因素和正交设计双重试验最后得到的优化体系对部分粗皮桉DNA样品进行PCR扩增试验,以验证体系的稳定性,试验结果如图10。结果表明,通过试验所得粗皮桉ISSR-PCR反应体系对不同个体的粗皮桉DNA样品均能扩增出清晰稳定的条带,稳定性较好,较适用于粗皮桉的ISSR-PCR扩增反应。

图10 部分粗皮桉DNA样品PCR扩增结果
3 结论与讨论
在ISSR-PCR扩增反应中,反应的各组成成分之间是相互影响的,在对反应体系的优化中,单因素试验只是改变其中一个因子的浓度,忽略了各因子之间的相互作用。通过单因素试验筛选出各因子比较适宜的浓度范围,在此适宜的浓度范围基础上进行正交试验,不仅考虑到了各因素的交互作用,而且避免出现因为因素水平的设计离最佳水平偏差较大而使试验结果较差的现象。
不同树种对PCR反应的条件要求不一,试验所得粗皮桉的ISSR-PCR反应体系与其他树种的体系有较大的区别,即使与同一科属的邓恩桉ISSR-PCR反应体系相比,除Mg2+浓度相同外,其他条件也均不一样[26]。
本研究利用单因素和正交设计双重试验方法得到的粗皮桉ISSR-PCR的优反应体系为:在25 µL反应体系中,10×PCR buffer 2.5 µL、MgCl22.0 mmol·L-1、dNTPs 0.3 mmol·L-1、DNA聚合酶1.25 U、DNA模板60 ng、引物0.8 µmol·L-1。优化的ISSR-PCR扩增程序为:94℃预变性5 min,然后按94℃变性1 min,53℃退火3 min,72℃延伸2 min,进行35个循环,最后72℃延伸5 min,4℃保存。
[1] 祁述雄.中国桉树(第2版)[M].北京:中国林业出版社, 2002.
[2] 佩格 R E,王国祥.粗皮桉家系试验初报[C]//洪菊生.澳大利亚阔叶树研究.北京:中国林业出版社,1993.
[3] 廖柏勇,刘丽婷,莫晓勇,等.10年生粗皮桉种源家系选择分析[J].华南农业大学学报,2011,32(4):72‒77,81.
[4] 林玉清.闽南山地粗皮桉家系引种试验[J].福建林业科技,2010,37(3):50‒55.
[5] 陈文平,罗建中,谢耀坚.粗皮桉种源/家系的遗传变异[J].广东林业科技,2001,17(3):1‒6.
[6] 赵荣军,张黎,霍小梅,等.基于近红外光谱技术预测径/弦切面粗皮桉木材微纤丝角[J].光谱学与光谱分析,2010, 30(9):2355‒2359.
[7] 霍小梅.基于近红外光谱技术预测粗皮桉木材主要物理力学性质的研究[D].北京:中国林业科学研究院,2010.
[8] 赵荣军,霍小梅,邢新婷,等.粗皮桉木材气干密度测定方法比较研究[J].西北林学院学报,2010,27(2):242‒244.
[9] 赵荣军,周贤武,任海清,等.粗皮桉生长锥与中心条气干密度和弹性模量预测及相关性分析[J].东北林业大学学报,2013,41(12):68‒71.
[10] 赵荣军,邢新婷,吕建雄,等.粗皮桉木材力学性质的近红外光谱方法预测[J].林业科学,2012,48(6):106‒111.
[11] 霍小梅,赵荣军,姚春丽,等.近红外光谱法预测粗皮桉木材的化学成分质量分数[J].东北林业大学学报,2010,38 (8):78‒79,104.
[12] 龙传文.粗皮桉木材的干燥特性与干燥基准制定[J].中南林业科技大学学报,2012,32(1):48‒50.
[13] 张黎.利用近红外光谱技术预测粗皮桉木材微纤丝角和气干密度的研究[D].西安:西北农林科技大学,2008.
[14] 赵星,姚春丽,田睿,等.不同种源粗皮桉的化学组成特性研究[J].造纸科学与技术,2009,28(1):6‒11.
[15] Luo J Z,Arnold R J,Aken K.Genetic variation in growth and typhoon resistance ininsouthwestern China[J].Australian forestry,2006,69(1):38‒47.
[16] Zietkiewicz E,Rafalkski A,Labuda D.Genome finger‒ printing by simple sequence repeat(SSR)—anchored polymerase chain reaction amplication[J].Genomics,1994, 20(2):176‒183.
[17] 王建波.ISSR分子标记及其在植物遗传学研究中的应用[J].遗传,2002,24(5):613‒616.
[18] 李乃伟,束晓春,何树兰,等.南方红豆杉的ISSR遗传多样性分析[J].西北植物学报,2010,30(12):2536‒2541.
[19] 杨传平,魏利,姜静,等.应用ISSR‒PCR对西伯利亚红松19个种源的遗传多样性分析[J].东北林业大学学报, 2005,33(1):1‒3.
[20] 宣继萍,章镇,房经贵,等.苹果品种ISSR指纹图谱构建[J].果树学报,2002,19(6):421‒423.
[21] 缪恒彬,陈发棣,赵宏波,等.应用ISSR对25个小菊品种进行遗传多样性分析及指纹图谱构建[J].中国农业科学, 2008,41(11):3735‒3740.
[22] 邱英雄,傅承新,何云芳.乐昌含笑不同类型鉴定的ISSR‒PCR分析[J].林业科学,2002,38(6):49‒52.
[23] 曾艳玲,谢鹏,谢耀坚,等.桉树ISSR‒PCR反应体系的建立及优化[J].中南林业科技大学学报,2008,28(1):44‒48.
[24] 李娟玲,刘国民,曹嵩晓,等.利用单因子和正交设计双重实验法优化鹧鸪茶RAPD‒PCR反应体系[J].农业学报,2011,1(4):14‒21.
[25] 何正文,刘运生,陈立华,等.正交设计直观分析法优化PCR条件[J].湖南医科大学学报,1998,23(4):403‒404.
[26] 付燕,罗楠,杨岑,等.枇杷属植物ISSR反应体系的建立和优化[J].果树学报,2009,26(2):180‒185.
Optimization of ISSR-PCR Systems for
CHEN Sheng-kan1,2, XIANG Dong-yun2, DENG Zi-yu2, LIANG Jian-xin3, LAN Bi-bu3,LI Chang-rong2, LIANG Ji1,2
(1.,530004,,; 2.,,530002,,; 3.,542800,,)
In order to develop an optimized ISSR-PCR analytical system for, single factor experiments were conducted to determine suitable concentrations of different factors (Mg2+, dNTP,DNA polymerase, DNA template, primer) that influence ISSR-PCR analyses. Based on the optimal concentrations identified, an orthogonal designed trial was carried out in order to optimize 5 factors at 4 levels. The results showed that a suitable ISSR-PCR reaction system involved: 25 µL reaction system containing 10×PCR buffer 2.5 µL, MgCl22.0 mmol·L-1, dNTPs 0.3 mmol·L-1,DNA polymerase 1.25 U, DNA template 60 ng and primer at 0.8 µmol·L-1. Through gradient testing, the optimized PCR amplification program was determined to involve pre-denaturing at 94℃ for 5 min, then denaturing at 94℃ for 1 min, annealing at 51℃ for 3 min, extension at 72℃ for 2 min over 35 cycles, and finally extension at 72℃ for 5 min. The PCR amplification products were stored at 4℃.
; ISSR-PCR; single factor test; orthogonal experiment
S718.46
A
2015-01-09
广西优良用材林资源培育重点实验室开放课题(12A0101); 广西科学研究与计划开发项目:桉树中大径级锯材培育与加工利用技术合作研究(桂科合1347004-3)
陈升侃(1989— ),男,硕士研究生,主要从事林木遗传育种研究.E-mail:chenshengkan@126.com
梁机(1961— ),男,博士,主要从事林木遗传育种研究.E-mail:liangjimail@163.com

