长期施加氮肥及氧化钙调节对酸性土壤硝化作用及氨氧化微生物的影响
张苗苗,王伯仁,李冬初,贺纪正,张丽梅,*
1 中国科学院生态环境研究中心, 北京 100085 2 中国科学院大学, 北京 100049 3 中国农业科学院农业部祁阳红壤生态环境重点野外科学观测试验站, 永州 426100
长期施加氮肥及氧化钙调节对酸性土壤硝化作用及氨氧化微生物的影响
张苗苗1,2,王伯仁3,李冬初3,贺纪正1,张丽梅1,*
1 中国科学院生态环境研究中心, 北京 100085 2 中国科学院大学, 北京 100049 3 中国农业科学院农业部祁阳红壤生态环境重点野外科学观测试验站, 永州 426100
酸性土壤; 氧化钙调节; 硝化作用; 氨氧化古菌(AOA); 氨氧化细菌(AOB)
酸性土壤(pH <5.5)在我国分布面积广泛,是我国热带亚热带经济作物如茶树、林木、果树和粮食作物的重要产地。近几十年来,因化学氮肥的过量施用和酸沉降加剧,我国农田土壤的酸度逐步升高,面积也不断增加[1]。为了缓解土壤酸化,农业中常采用田间施加化学改良剂(如石灰(CaO))的方法来进行改良,效果显著[2-3]。
硝化作用是土壤氮素转化的关键过程,除影响土壤中无机氮对植物的有效性外,硝化作用对酸性土壤进一步酸化、有毒金属元素的溶解释放等都有重要影响[1, 4-6]。氨氧化作用作为硝化作用的限速步骤,是全球氮循环的中心环节,主要由氨氧化细菌(AOB)[7]和古菌(AOA)[8-10]驱动。大量分子生态学调查发现,虽然多种环境因子,包括温度、pH值、施肥、土壤类型及土地利用方式等都会影响两类氨氧化微生物的群落组成和数量,但pH值是影响其分布及功能活性的决定性因子。已有研究发现,随着pH值增加,AOA和AOB群落组成发生明显演替,AOA的数量和amoA基因的表达活性显著降低,AOB则呈相反趋势[11-15]。2007年,对湖南祁阳红壤长期定位实验站连续16a施加不同化学肥料处理土壤中的氨氧化细菌和古菌的群落组成进行了调查,结果发现,长期施加无机氮肥显著降低了AOA和AOB的数量,并改变了AOA的群落组成,但对AOB的群落结构无显著影响,揭示了AOA可能在该酸性土壤硝化作用中起重要作用[16]。通过稳定性同位素探测技术(stable isotope probing, SIP)等的研究,也证实氨氧化古菌是一些酸性土壤硝化作用的主导者[17]。
以上研究揭示了氨氧化古菌对酸性土壤硝化作用的重要贡献,然而在多数酸性土壤中也能检测到数量相当的氨氧化细菌,但对这些酸性土壤中氨氧化细菌的存在有何生态学意义并不清楚。由于多年连续施肥,湖南祁阳红壤定位施肥实验站长期接受施化学氮肥处理的土壤pH值持续下降,土壤肥力和生产力严重受阻,该实验站于2010年将原施氮肥(N)、氮钾肥(NK)、氮磷肥(NP)、氮磷钾肥(NPK)处理的小区划分出一半进行添加氧化钙处理,以期通过添加氧化钙调节土壤pH值,改善土壤质量。本研究在2007年对该样地土壤研究的基础上,对持续施肥5a后以及添加CaO处理对土壤硝化作用和氨氧化细菌和古菌的影响进行了跟踪研究,以期为进一步认识酸性土壤硝化作用过程及其机制提供参考,为农田施肥及酸性土壤改良管理方式提供指导依据。
1 材料和方法
1.1 样品采集
本研究中的土壤样品采集于湖南省祁阳红壤定位施肥实验站(26°45′N,111°52′E),该地属于典型的亚热带季风气候,年平均气温18 ℃,年降水量1300 mm。本实验共选择了9个处理,包括1990年开始的长期施肥处理及2010年开始的施加氧化钙(CaO)处理,具体情况如下:1)不施肥(Control),2)氮肥(N),3)氮钾肥(NK),4)氮磷肥(NP),5)氮磷钾肥(NPK),6)氮肥加氧化钙(N+CaO),7)氮钾肥加氧化钙(NK+CaO),8)氮磷肥加氧化钙(NP+CaO),9)氮磷钾肥加氧化钙(NPK+CaO)。肥料用量为年施用N 300 kg/hm2、P2O5120 kg/hm2、K2O 120 kg/hm2,N∶P2O5∶K2O比值为1∶0.4∶0.4;氧化钙年施用量为2550 kg/hm2。
样品采集于2012年7月,所采样品均为0—20 cm表层土壤。每个处理小区取3个重复,每个重复分别取5个点混合而成。样品于低温保存带回实验室后,剔除石砾和植物残根等杂物,过2 mm筛处理,一部分保存于4 ℃冰箱用于土壤基本化学性质的测定,其余样品放置于-80 ℃保存,用于分子生态学实验分析。
1.2 土壤化学指标及硝化潜势测定
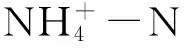
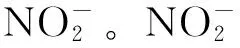
1.3 土壤DNA提取
土壤DNA提取以MoBio PowerSoilTMDNA(MO BIO laboratories, CA, US))试剂盒进行提取,称取0.25 g土壤按照试剂盒操作指南进行,并略加修改:细胞破碎在FastPrep上进行,破碎速度为5.0,时间为45 s。DNA提取后于-20 ℃保存待用,所有DNA均稀释10倍用于下游实验。
1.4 定量PCR分析
氨氧化细菌(AOB)和氨氧化古菌(AOA)定量PCR分析均采用SYBR Green法,反应在iCycle iQ5(Bio-Rad, USA)仪器上进行。 PCR反应体系为25 μL,包含12.5 μL 2 × SYBR Premix Ex TaqTM(TaKaRa, Japan),10 μmol/L正反向引物各0.5 μL,DNA模板2 μL,其余用ddH2O补足至25 μL[19]。定量PCR所用的扩增引物及反应条件见表1。
1.5 变性梯度凝胶电泳分析
氨氧化微生物的群落结构分析采用变性梯度凝胶电泳(DGGE)法,DGGE在DCode Universal Mutation Detection System (Bio-Rad, USA)中进行。AOAamoA基因PCR扩增体系为25 μL,其中包括12.5 μL 2 × Premix Ex TaqTM(TaKaRa, Japan),10 μmol/L正反向引物各0.5 μL,DNA模板2 μL,其余用ddH2O补足至25 μL。AOBamoA基因扩增体系除10 μmol/L正反向引物用量为0.4 μL外,其余与AOAamoA反应体系相同。DGGE-PCR反应的扩增引物及反应条件如表1所示。DGGE电泳在6%聚丙烯酰胺胶中进行,AOAamoA基因所用的变性胶浓度范围为10%—45%,电泳条件为90 V,12 h。AOBamoA基因变性胶浓度范围为30%—55%,电泳条件为120 V,8 h。每个处理选取两个重复进行DGGE分析。
1.6 统计分析
本研究中所有相关分析均采用Pearson相关分析;添加N与N+CaO、NK与NK+CaO、NP与NP+CaO、NPK与NPK+CaO各组处理间的差异性分析采用成对数据T检验分析;多组数据间的方差分析采用单因素方差分析ANOVA。所有统计检验均用SPSS 19实现,P<0.05即认为差异显著。DGGE图谱的聚类分析以Quantity One软件读取条带后以UPGMA方法进行。

表1 定量PCR及DGGE-PCR中所用引物及反应程序
2 结果
2.1 土壤化学性质及硝化潜势(PNR)
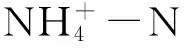
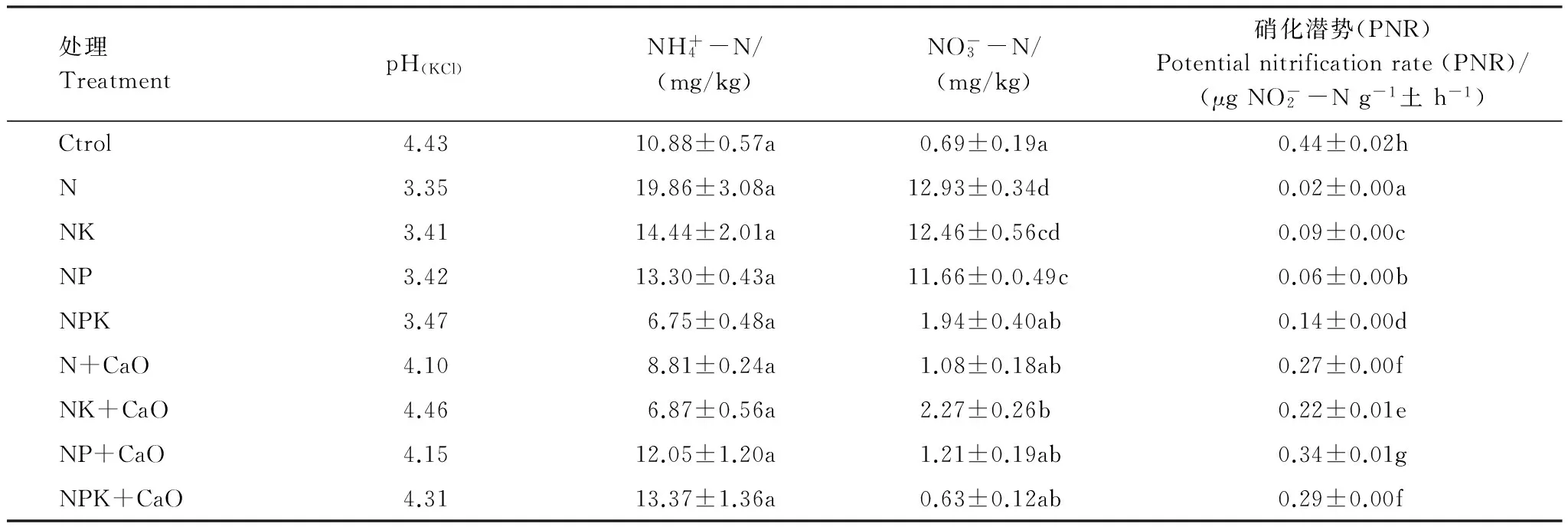
表2 土样的基本化学性质及硝化潜势(±标准误差)

2.2 氨氧化微生物的丰度变化
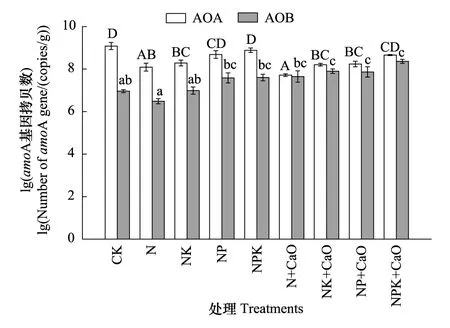
图1 不同氮肥处理及氧化钙(CaO)调节处理土壤中氨氧化细菌(AOB)和氨氧化古菌(AOA)amoA基因丰度Fig.1 Abundance of archaeal and bacterial amoA gene in soils under different N fertilizers and CaO treatments±标准误差;不同大写字母表示AOA丰度在各处理之间的差异显著性,不同小写字母表示AOB丰度在各处理之间的差异显著性
利用实时荧光定量PCR技术对各处理土壤中的氨氧化细菌(AOB)和氨氧化古菌(AOA)的amoA基因丰度进行了分析,结果如图1所示。成对数据T检验分析结果显示,在所有未加CaO的处理中,AOAamoA基因的丰度均显著高于AOB(P<0.05),二者的比值为10.9—44.3。添加CaO后,AOA丰度在一定程度上降低,AOB丰度显著增加(P<0.05),使得AOA和AOB二者基因丰度相当,无显著差异,其比值降低至0.83—1.94。在所有处理中,对照土壤AOA丰度最高(1.39×109copies/g),N+CaO土壤AOA丰度最低(5.31×107copies/g);NPK+CaO处理土壤中AOB丰度最高(2.42×108copies/g),N肥处理土壤中AOB丰度最低(3.34×106copies/g)。另外,土壤硝化潜势与土壤中AOA和AOBamoA基因丰度均无显著相关关系,但与AOA和AOBamoA基因丰度之和,即土壤中总amoA基因丰度显著正相关(r=0.40,P<0.05)。
2.3 氨氧化微生物的群落结构
氨氧化细菌(AOB)和氨氧化古菌(AOA)的群落结构以PCR-DGGE方法进行分析,结果发现所有处理均容易获得足量的AOA PCR产物用于DGGE电泳。DGGE图谱及其对应条带的聚类分析结果显示,对照处理Ctrol与其它施肥处理明显分开,单独成为一支,表明对照处理Ctrol土壤中AOA的群落结构与其它处理存在明显差异;所有施肥处理中,N处理与其它处理明显分开,NP和NK,NPK+CaO和NPK,NK+CaO、NP+ CaO和N+CaO分别聚集成3个小分支(图2),表明AOA的群落组成在这3组处理中存在稍小差异。
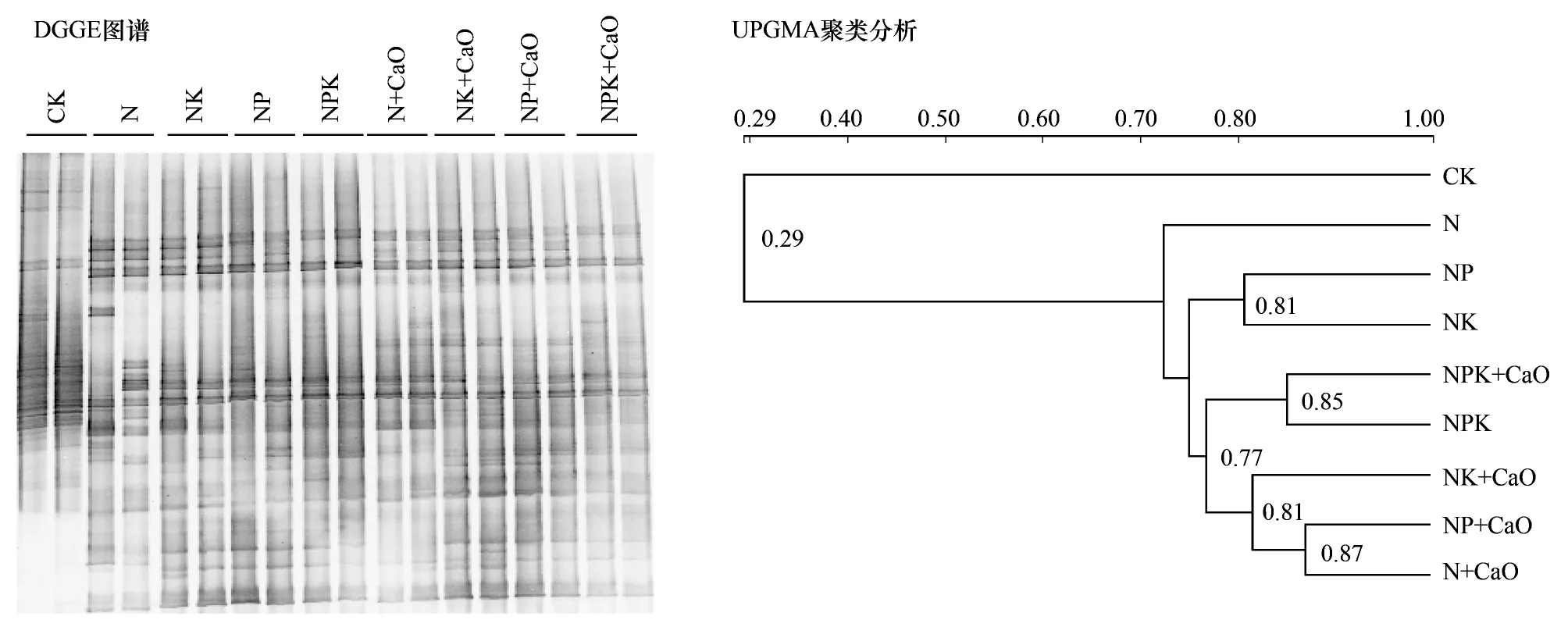
图2 不同土壤中氨氧化古菌amoA基因的DGGE图谱及其UPGMA聚类分析Fig.2 Denaturing gradient gel electrophoresis (DGGE) profiles and the UPGMA dendrogram of ammonia-oxidizing archaeal amoA gene in soils
而对AOB来说,施加CaO的所有处理均能获得足够的PCR产物进行DGGE电泳分析,但尽管使用同样的PCR扩增条件,对照处理土壤Ctrol及各施氮肥处理土壤中AOBamoA基因PCR扩增产物较弱甚至扩增不出,在DGGE图谱上仅得到一些随机条带;与不施加CaO的处理相比,施加CaO的所有处理中AOB的DGGE条带明显增加,并显示出较高的群落相似性,表明施加CaO明显刺激了这部分氨氧化细菌的生长;此外,NPK+CaO处理土壤比N+CaO,NK+CaO,NP+CaO处理显示出更高的组成多样性(图3)。对DGGE图谱进行的聚类分析的结果也显示,施N肥处理及对照处理与其它处理明显分开,其余处理聚积于一大分支,其中添加CaO的4个处理又聚积成一个小分支,且NPK+CaO处理与N+CaO,NK+CaO,NP+CaO相互分开(图3)。

图3 不同土壤中氨氧化细菌amoA基因DGGE图谱及其UPGMA聚类分析Fig.3 Denaturing gradient gel electrophoresis (DGGE) profiles and the UPGMA dendrogram of ammonia-oxidizing bacterial amoA gene in soils
3 讨论
3.1 长期施肥处理及添加CaO对土壤性质和硝化作用的影响
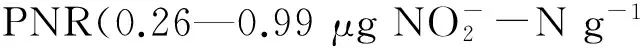
3.2 氨氧化微生物对长期施氮肥及添加CaO的响应
在所有施肥处理土壤中,氨氧化古菌AOAamoA基因(7.40×107—4.08×108copies/g)的丰度均显著高于氨氧化细菌AOB(1.67×106—2.57×107copies/g),二者比值为10.9—44.3,这与2007年的调查结果一致,并与其他有关酸性土壤中AOA占主导优势的研究结论相符[23-26]。已有的大量研究也表明,在大区域尺度上,pH值是驱动氨氧化微生物多样性分布及活性的主要因子[12]。Nicol等[11]对pH值梯度为4.9—7.5的草地土壤进行的研究发现,AOA与AOB的数量比值随pH值增加而降低,AOA的数量和amoA基因的表达活性随土壤pH值增加明显降低,而AOB则相反。作者及国内其他研究者利用高通量测序技术及稳定性同位素探测(SIP)等技术对我国一系列土壤开展的研究也发现,随土壤pH值增加,AOA和AOB群落组成发生明显演替;在北方碱性潮土中,氨氧化细菌是硝化作用的主要驱动者,而在南方酸性土壤中,硝化作用主要由氨氧化古菌所驱动[13, 17, 27-28]。这些结果一致证实了AOA和AOB生态位分异的特征,即AOB倾向于在高氮及pH值中性或碱性的环境中起主导作用,而AOA则倾向于在酸性及低氮高有机质的土壤中占主导优势[29-32]。

此外,还发现,NPK及NPK+CaO土壤中AOB和AOA群落结构与其它处理土壤也有一定差异,这说明P、K也会影响土壤氮循环过程及氨氧化微生物,这与之前一些研究报道结果一致[36-37]。
4 结论
本研究对长期施加不同氮肥处理并添加CaO调节的酸性红壤中硝化作用及氨氧化微生物的群落结构和丰度特征进行了研究,结果发现,连续施加含氮肥料可导致土壤pH值和硝化作用进一步降低,添加CaO则可缓解土壤酸化,显著提高土壤pH值和硝化潜势。AOA的群落组成在长期施肥处理与不施肥处理间有显著不同,但在不同氮肥处理间无显著差异。添加CaO处理对AOA的群落组成无显著影响,但明显提高了各施肥处理土壤中氨氧化细菌的丰度,并增加了其群落多样性,氨氧化古菌的丰度受到一定程度的抑制。这些结果表明,虽然氨氧化古菌因自身的生理及遗传特征更适应在酸性土壤硝化作用中发挥作用,酸性土壤中AOB在功能上冗余,但当添加CaO后,AOA和AOB对环境条件变化迅速作出响应,并根据其不同的生态位需求重新分配优势地位,二者交替作用共同驱动着酸性土壤的硝化作用。
[1] Guo J H, Liu X J, Zhang Y, Shen J L, Han W X, Zhang W F, Christie P, Goulding K W, Vitousek P M, Zhang F S. Significant acidification in major Chinese croplands. Science, 2010, 327(5968): 1008-1010.
[2] Caires E F, Garbuio F J, Churka S, Barth G, Corrêa J C L. Effects of soil acidity amelioration by surface liming on no-till corn, soybean, and wheat root growth and yield. European Journal of Agronomy, 2008, 28(1): 57-64.
[3] Lollato R P, Edwards J T, Zhang H. Effect of alternative soil acidity amelioration strategies on soil pH distribution and wheat agronomic response. Soil Science Society of America Journal, 2013, 77(5): 1831-1841.
[4] Tang C, Rengel Z, Diatloff E, Gazey C. Responses of wheat and barley to liming on a sandy soil with subsoil acidity. Field Crops Research, 2003, 80(3): 235-244.
[5] 黄国勤, 王兴祥, 钱海燕, 张桃林, 赵其国. 施用化肥对农业生态环境的负面影响及对策. 生态环境, 2004, 13(4): 656-660.
[6] Van Breemen N, van Dijk H F. Ecosystem effects of atmospheric deposition of nitrogen in the Netherlands. Environmental Pollution, 1988, 54(3/4): 249-274.
[7] Prosser J I. Autotrophic nitrification in bacteria. Advances in Microbial Physiology, 1989, 30: 125-181.
[8] Könneke M, Bernhard A E, de la Torre J R, Walker C B, Waterbury J B, Stahl D A. Isolation of an autotrophic ammonia-oxidizing marine archaeon. Nature, 2005, 437(7058): 543-546.
[9] Treusch A H, Leininger S, Kletzin A, Schuster S C, Klenk H P, Schleper C. Novel genes for nitrite reductase and Amo-related proteins indicate a role of uncultivated mesophilic crenarchaeota in nitrogen cycling. Environmental Microbiology, 2005, 7(12): 1985-1995.
[10] Venter J C, Remington K, Heidelberg J F, Halpern A L, Rusch D, Eisen J A, Wu D Y, Paulsen I, Nelson K E, Nelson W, Fouts D E, Levy S, Knap A H, Lomas M W, Nealson K, White O, Peterson J, Hoffman J, Parsons R, Baden-Tillson H, Pfannkoch C, Rogers Y H, Smith H O. Environmental genome shotgun sequencing of the Sargasso Sea. Science, 2004, 304(5667): 66-74.
[11] Nicol G W, Leininger S, Schleper C, Prosser J I. The influence of soil pH on the diversity, abundance and transcriptional activity of ammonia oxidizing archaea and bacteria. Environmental Microbiology, 2008, 10(11): 2966-2978.
[12] Shen J P, Zhang L M, Di H J, He J Z. A review of ammonia-oxidizing bacteria and archaea in Chinese soils. Frontiers in Microbiology, 2012, 3: 296-296.
[13] Hu H W, Zhang L M, Dai Y, Di H J, He J Z. pH-dependent distribution of soil ammonia oxidizers across a large geographical scale as revealed by high-throughput pyrosequencing. Journal of Soils and Sediments, 2013, 13(8): 1439-1449.
[14] Gubry-Rangin C, Hai B, Quince C, Engel M, Thomson B C, James P, Schloter M, Griffiths R I, Prosser J I, Nicol G W. Niche specialization of terrestrial archaeal ammonia oxidizers. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(52): 21206-21211.
[15] Yao H Y, Campbell C D, Chapman S J, Freitag T E, Nicol G W, Singh B K. Multi-factorial drivers of ammonia oxidizer communities: evidence from a national soil survey. Environmental Microbiology, 2013, 15(9): 2545-2556.
[16] He J Z, Shen J P, Zhang L M, Zhu Y G, Zheng Y M, Xu M G, Di H J. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices. Environmental Microbiology, 2007, 9(9): 2364-2374.
[17] Zhang L M, Hu H W, Shen J P, He J Z. Ammonia-oxidizing archaea have more important role than ammonia-oxidizing bacteria in ammonia oxidation of strongly acidic soils. The ISME Journal, 2012, 6(5): 1032-1045.
[18] Kurola J, Salkinoja-Salonen M, Aarnio T, Hultman J, Romantschuk M. Activity, diversity and population size of ammonia-oxidising bacteria in oil-contaminated landfarming soil. FEMS Microbiology Letters, 2005, 250(1): 33-38.
[19] Zhang L M, Offre P R, He J Z, Verhamme D T, Nicol G W, Prosser J I. Autotrophic ammonia oxidation by soil thaumarchaea. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(40): 17240-17245.
[20] Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712.
[21] Francis C A, Roberts K J, Beman J M, Santoro A E, Oakley B B. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(41): 14683-14688.
[22] Ying J Y, Zhang L M, He J Z. Putative ammonia-oxidizing bacteria and archaea in an acidic red soil with different land utilization patterns. Environmental Microbiology Reports, 2010, 2(2): 304-312.
[23] Chen X P, Zhu Y G, Xia Y, Shen J P, He J Z. Ammonia-oxidizing archaea: important players in paddy rhizosphere soil? Environmental Microbiology, 2008, 10(8): 1978-1987.
[24] Gubry-Rangin C, Nicol G W, Prosser J I. Archaea rather than bacteria control nitrification in two agricultural acidic soils. FEMS Microbiology Ecology, 2010, 74(3): 566-574.
[25] Yao H, Gao Y, Nicol G W, Campbell C D, Prosser J I, Zhang L, Han W, Singh B K. Links between ammonia oxidizer community structure, abundance, and nitrification potential in acidic soils. Applied and Environmental Microbiology, 2011, 77(13): 4618-4625.
[26] Lehtovirta L E, Prosser J I, Nicol G W. Soil pH regulates the abundance and diversity of Group 1.1c Crenarchaeota. FEMS Microbiology Ecology, 2009, 70(3): 367-376.
[27] Xia W W, Zhang C X, Zeng X W, Feng Y Z, Weng J H, Lin X G, Zhu J G, Xiong Z Q, Xu J, Cai Z C, Jia Z J. Autotrophic growth of nitrifying community in an agricultural soil. The ISME Journal, 2011, 5(7): 1226-1236.
[28] Lu L, Han W Y, Zhang J B, Wu Y C, Wang B Z, Lin X G, Zhu J G, Cai Z C, Jia Z J. Nitrification of archaeal ammonia oxidizers in acid soils is supported by hydrolysis of urea. The ISME Journal, 2012, 6(10): 1978-1984.
[29] Erguder T H, Boon N, Wittebolle L, Marzorati M, Verstraete W. Environmental factors shaping the ecological niches of ammonia-oxidizing archaea. FEMS Microbiology Reviews, 2009, 33(5): 855-869.
[30] Qin H L, Yuan H Z, Zhang H, Zhu Y J, Yin C M, Tan Z J, Wu J S, Wei W X. Ammonia-oxidizing archaea are more important than ammonia-oxidizing bacteria in nitrification and NO-3-N loss in acidic soil of sloped land. Biology and Fertility of Soils, 2012, 49(6): 767-776.
[31] Schleper C. Ammonia oxidation: different niches for bacteria and archaea? The ISME Journal, 2010, 4(9): 1092-1094.
[32] Ke X B, Angel R, Lu Y H, Conrad R. Niche differentiation of ammonia oxidizers and nitrite oxidizers in rice paddy soil. Environmental Microbiology, 2013, 15(8): 2275-2292.
[33] Martens-Habbena W, Berube P M, Urakawa H, de la Torre J R, Stahl D A. Ammonia oxidation kinetics determine niche separation of nitrifying Archaea and Bacteria. Nature, 2009, 461(7266): 976-979.
[34] Valentine D L. Adaptations to energy stress dictate the ecology and evolution of the Archaea. Nature Reviews Microbiology, 2007, 5(4): 316-323.
[35] He J Z, Hu H W, Zhang L M. Current insights into the autotrophic thaumarchaeal ammonia oxidation in acidic soils. Soil Biology and Biochemistry, 2012, 55: 146-154.
[36] Yu W T, Xu Y G, Bi M L, Ma Q, Zhou H. Activity and composition of ammonia-oxidizing bacteria in an aquic brown soil as influenced by land use and fertilization. Pedosphere, 2010, 20(6): 789-798.
[37] Cai Y F, Peng C, Zhao S Q, Cai A H, Li Y T. Balanced fertilization improved soil ammonia-oxidizing bacterial community in latosolic red soil. Acta Agriculturae Scandinavica, Section B-Soil & Plant Science, 2012, 62(6): 531-540.
Effects of long-term N fertilizer application and liming on nitrification and ammonia oxidizers in acidic soils
ZHANG Miaomiao1,2, WANG Boren3, LI Dongchu3, HE Jizheng1, ZHANG Limei1,*
1ResearchCenterforEco-EnvironmentalSciences,ChineseAcademyofSciences,Beijing100085,China2UniversityofChineseAcademyofSciences,Beijing100049,China3RedSoilExperimentalStation(Qiyang,Hunanprovince),theMinistryofAgriculture,ChineseAcademyofAgriculturalSciences,Yongzhou426100,China
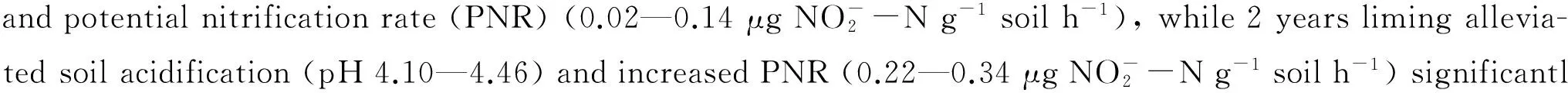
acidic soils; liming; nitrification; ammonia-oxidizing archaea (AOA); ammonia-oxidizing bacteria (AOB)
国家自然科学基金资助项目(41171217, 41322007)
2014-02-26; < class="emphasis_bold">网络出版日期:
日期:2014-12-04
10.5846/stxb201402260329
*通讯作者Corresponding author.E-mail: zhanglm@rcees.ac.cn
张苗苗,王伯仁,李冬初,贺纪正,张丽梅.长期施加氮肥及氧化钙调节对酸性土壤硝化作用及氨氧化微生物的影响.生态学报,2015,35(19):6362-6370.
Zhang M M, Wang B R, Li D C, He J Z, Zhang L M.Effects of long-term N fertilizer application and liming on nitrification and ammonia oxidizers in acidic soils.Acta Ecologica Sinica,2015,35(19):6362-6370.

