CONSTANS-LIKE 7基因对拟南芥开花的调控分析*
王宏归,黄 晨,陈 鹏,姜 雅,张 娅,唐冬英,赵小英,刘选明†
(1.扬州大学 环境科学与工程学院,江苏 扬州 225127;2.湖南大学 生物学院,植物功能基因组学与发育调控湖南省重点实验室,湖南 长沙 410082)
CONSTANS-LIKE 7基因对拟南芥开花的调控分析*
王宏归1,黄 晨1,陈 鹏1,姜 雅1,张 娅1,唐冬英2,赵小英2,刘选明2†
(1.扬州大学 环境科学与工程学院,江苏 扬州 225127;2.湖南大学 生物学院,植物功能基因组学与发育调控湖南省重点实验室,湖南 长沙 410082)
为研究CONSTANS-LIKE7 (COL7)在调控植物开花方面的功能,以定量PCR,GUS染色等方法,研究光及生物钟对COL7表达的影响,以及COL7对拟南芥开花的影响.实验结果显示:光及生物钟参与调控COL7的表达;过表达COL7在长日照条件下抑制CONSTANS(CO)以及FLOWERINGLOCUST(FT)的表达,进而抑制拟南芥开花;col7突变体不论是在长日照下还是短日照下都没有明显的开花表型,说明COL7在调控拟南芥开化方面可能与其家族基因中的其它成员存在功能冗余.
基因表达;拟南芥;CONSTANS-LIKE7;开花
拟南芥是长日照植物,长日照促进其开花[1].拟南芥从生殖生长到营养生长的转变,是由内部及外部因素共同决定的[2].目前在拟南芥中,主要有4条开花信号途径:1)光周期途径,2)自主途径,3)赤霉素途径、4)春化途径[3-7].内部及外部信号通过这4条途径被传递至共同的整合子基因,如:FLOWERINGLOCUST(FT),SUPPRESSIONOFOVEREXPRESSIONOFCONSTANS1 (SOC1)以及LEAFY(LFY),从而构成一个复杂的开花调控网络[6, 8-9].光周期途径是由光受体、生物钟及生物钟输出途径组成,光信号通过光受体(如:CRYPTOCHROMES(CRY) 1与2、FLAVIN-BINDING,KELCHREPEAT,F-BOX1 (FKF1),ZEITLUPE(ZTL),PHYTOCHROMES(PHY)AtoE等传递至生物钟[10, 11].光周期途径调控植物开花是通过CO,FT模式实现[12].CO的表达受光及生物钟调控,其蛋白在光下稳定,但在黑暗下被降解[13-15].CYCLING DOF FACTOR 1(CDF1)通过结合到CO的启动子上抑制CO的表达,进而抑制FT的表达[16].FKF1与GIGANTEA(GI)形成复合体可以将CDF1降解,从而解除CDF1对CO抑制,促进FT的表达[17-19].
在拟南芥中,CO家族有17个成员,且都含有CCT (CO, COL, and TIMING OF CAB1 EXPRESSION1 (TOC1))结构域与锌指结构域[20-21].CCT结构与蛋白的核定位有关,同时也可以与HAP3及HAP5相互作用进而调控拟南芥的生长发育[3, 21-22].锌指结构域在蛋白与蛋白相互作用中起着重要的作用[3, 21-22].根据锌指结构域的差异,可以将CO家族分成3组:第1组,CO,COL1~COL5,它们都含有两个锌指结构;第2组,COL6~COL8以及COL16它们只含有一个锌指结构;第3组,COL9~COL15它们都含有一个锌指结构域以及一个分化的锌指结构域[20-21].
在CO基因家族中,只有CO,COL1,COL2,COL3,COL5,COL7及COL9被报道.CO是光周期开花途径中的关键基因,其通过结合到FT的启动子上激活FT的表达,进而调控开花[12].COL1,COL2的表达是受生物钟调控,而且对拟南芥开花只有微弱的影响[23].COL3突变体不论在长日照条件下还是在短日照条件下都具有晚花表型,此外,COL3还参与调控侧根的形成,以及影响芽的伸长与侧枝的形成[24].过表达COL5只有在短日照下才能促进FT的表达,进而促进短日照下拟南芥开花[25].COL7它参与植物的遮阴反应[26];过表达COL9抑制开花,因其抑制CO与FT的表达[27].除了目前已经被报道的CO及COLs基因外,其他COLs的功能目前还不清楚.在本研究中,将介绍COL7在调控拟南芥开花方面的功能.
1 材料与方法
1.1 植物材料及生长条件
本研究所用的野生型拟南芥(Wild type,WT)为哥伦比亚(Columbia,Col).col7 突变体(GABI-639C04)由 Bernd Weisshaar提供(MPI for Plant Breeding Research; Cologne, Germany).COL7过量表达转基因株系COL7-OX-10 和COL7-OX-11是本实验室保存材料.拟南芥种子用10%的NaClO溶液侵泡10 min,无菌水洗5次,然后播在Murashige and Skoog medium(MS)培养基上,于4 ℃春化72 h后,分别转移到长日照(16 h光照,8 h黑暗)(100 μmol·m2·s1)、短日照(8 h光照,16 h黑暗)(100 μmol·m2·s1)、黑暗、红光(10 μmol·m2·s1)、远红光(0.8 μmol·m2·s1)、蓝光(30 μmol·m2·s1)下培养.拟南芥培养温度为 22±2 ℃.
1.2 实时定量PCR分析
总的RNA提取使用IllustraRNAspin Mini Kit (GE Healthcare)试剂盒,cDNA的合成是利用2 μg总RNA以及SuperScript first-strand cDNA synthesis system (Invitrogen)试剂盒.qPCR分析使用的试剂、仪器分别是Platinum SYBR Green qPCR Supermix-UDG (Invitrogen)与MX3000 System (Stratagene).本研究中使用的定量PCR引物见表1.
表1 定量PCR引物
Tab.1 Quantitative PCR primers
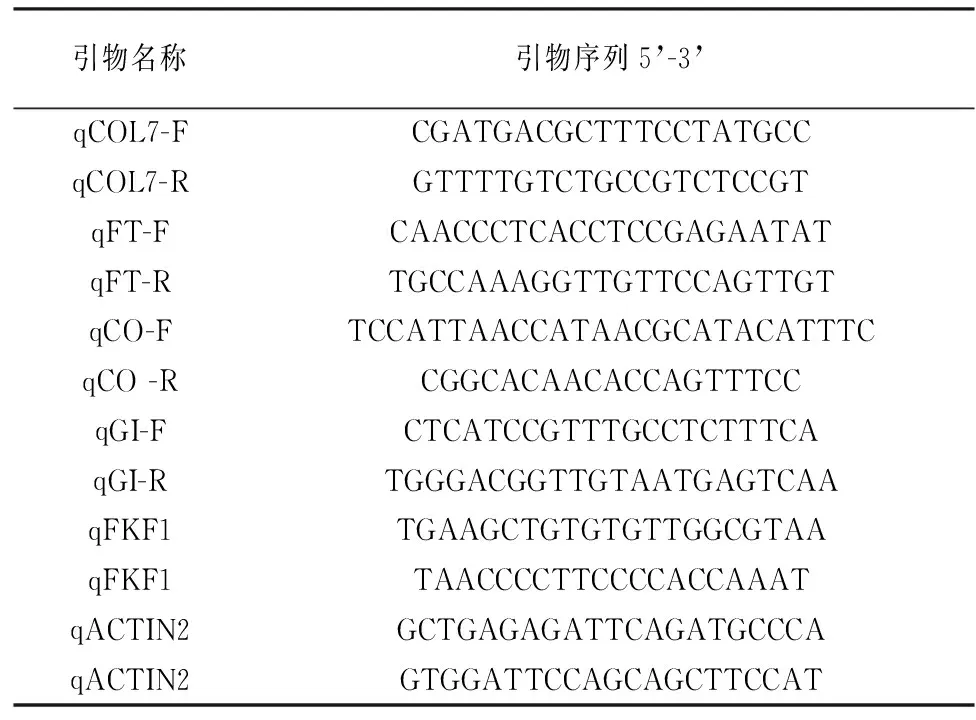
引物名称引物序列5’-3’qCOL7-FCGATGACGCTTTCCTATGCCqCOL7-RGTTTTGTCTGCCGTCTCCGTqFT-FCAACCCTCACCTCCGAGAATATqFT-RTGCCAAAGGTTGTTCCAGTTGTqCO-FTCCATTAACCATAACGCATACATTTCqCO-RCGGCACAACACCAGTTTCCqGI-FCTCATCCGTTTGCCTCTTTCAqGI-RTGGGACGGTTGTAATGAGTCAAqFKF1TGAAGCTGTGTGTTGGCGTAAqFKF1TAACCCCTTCCCCACCAAATqACTIN2GCTGAGAGATTCAGATGCCCAqACTIN2GTGGATTCCAGCAGCTTCCAT
1.3 COL7的亚细胞定位
利用基因枪将构建好的35S::GFP-COL7质粒轰击洋葱表皮细胞,然后在MS培养基上培养12~24 h,取洋葱表皮,于Olympus Confocal FluoView IX 70激光共聚焦显微镜下观察.
2 结果与分析
2.1 COL7的亚细胞定位分析
CO基因家族都是转录因子,而转录因子是在细胞核内调节特定基因的转录.COL7含有CCT结构域,其与蛋白的核定位有关.为明确COL7的亚细胞定位,利用35S::GFP-COL7质粒去轰击洋葱表皮细胞,经过12~24 h的培养后,取上表皮细胞在显微镜下观察,绿色荧光在轰击的洋葱表皮细胞核中被检测到(图1),这说明COL7是核定位的蛋白.

1:明场,2:色荧光,3:API染色,4:1,2与3的叠加图
2.2 COL7过量表达或者缺失对拟南芥开花时间 的影响
目前在已报道的CO基因家族中,部分参与调控拟南芥开花.COL7参与调控拟南芥遮阴反应,那么COL7是否参与调控植物开花呢?为此,分别将COL7的过表达株系COL7-OX-10,COL7-OX-11,以及col7突变体、WT种在长日照与短日照条件下,观察开花表型.经过统计分析开花叶片数以及开花时间发现:过量表达COL7在长日照下抑制拟南芥开花,其开花时间与野生型相比大约要晚10 d(图2(a)~(c)),而过量表达COL7在短日照下无抑制开花的作用(图2(d)~(e)),COL7不论在长日照还是在短日照下,其开花表型与野生型无显著差异(图2(a)~(e)).这说明COL7在调控开花方面与其家族基因存在功能冗余.
2.3 COL7过量表达或者缺失对开花关键基因CO, FT表达的影响
FT是“成花素”也是整合子基因,他可将来自光周期以及其他开花途径的信号整合,进而调控植物开花.COL7抑制开花,那么其是否通过抑制FT表达进而抑制开花?因此,将WT,COL7和COL7-OX种籽播种在MS培养基上,长日照下培养7 d,然后每4 h取样,共取1 d.提取样品的RNA,经反转录后利用定量PCR分析,实验结果显示:FT在COL7-OX株系中的表达明显下降(图3(a)).抑制FT表达的基因有很多,那么COL7抑制FT的表达是直接抑制FT的表达还是通过其他基因来实现呢?为此,通过定量PCR分析样品中CO基因的变化.定量PCR结果显示:CO在COL7-OX株系里的表达是受抑制的,但是相对于野生型CO的表达变化不是特别大(图3(b)).因此,COL7-OX株系里的FT的变化不仅仅是由CO引起的,即说明:COL7抑制CO与FT的表达.在目前的报道中,同时参与调控CO,FT表达的基因还有GI,FKF1,且GI,FKF1是光周期开花途径中的正调控基因.那么COL7是否有可能通过抑制GI或FKF1来间接调控CO,FT?为此,利用定量PCR分析COL7-OX株系里GI,FKF1的表达情况,结果显示:COL7不影响GI,FKF1的表达(图3(c)~(d)).因此,COL7抑制开花是通过其自身抑制CO,FT的表达来实现.
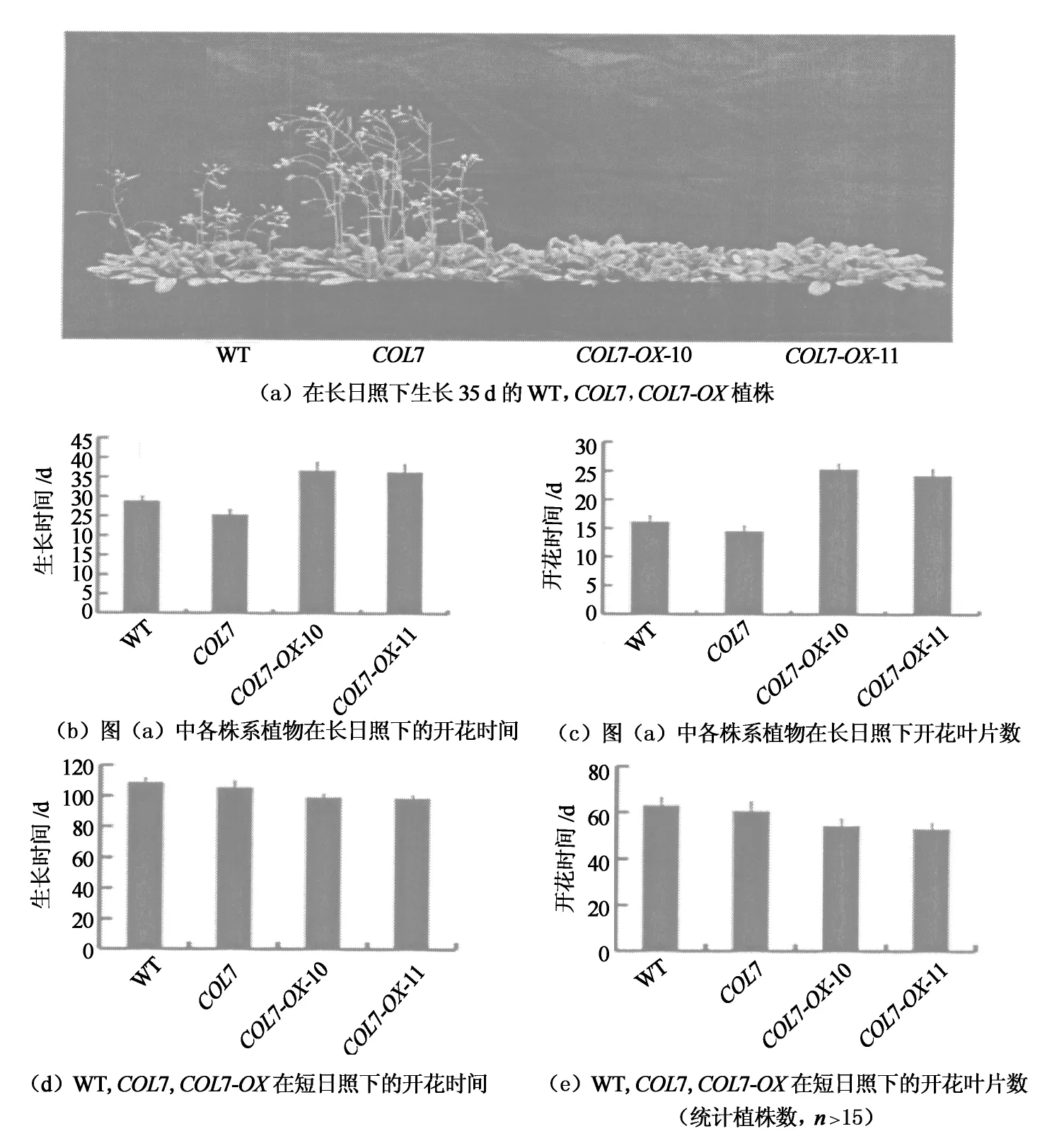
图2 COL7的开花表型
2.4 COL7组织器官表达特异性分析
35S启动子驱动的COL7抑制韧皮部基因CO,FT的表达,那么COL7是否在韧皮部有表达?因此,COL7的组织表达对研究COL7的功能非常重要.以PCOL7::GUS转基因植株来研究COL7的组织表达.取在长日照条件下生长5 d的PCOL7::GUS转基因幼苗进行GUS染色.实验结果显示:COL7不仅在叶肉里表达,同时也在韧皮部里表达(图4(a)).此外,为了明确COL7在拟南芥各组织器官里的表达情况,取在长日照条件下生长35 d的野生型植株的各个组织器官,利用定量PCR的方法进行分析COL7的表达,实验结果显示:COL7在叶片、花里面表达比较多,而在根,茎,果夹中表达较少(图4(b)).
2.5 COL7时空表达模式分析
因为COL7是光周期途径中的开花抑制因子,分析COL7在不同发育阶段的表达情况,对揭示COL7的基因功能有很重要的作用.所以,收集在长日照条件下生长7,15,25和35 d的野生型植株,提取样品RNA,利用定量PCR分析COL7的表达情况,定量PCR结果显示:COL7的表达从第7 d到第15 d这个过程中有明显的增加,而在15 d以后基本上保持相同的水平,因此,COL7的表达具有一定的时间性(图5(a)).COL7在叶片的表达量最高,那么COL7在不同的叶片中的表达量是否相同呢?以在长日照条件下生长35 d的WT为材料,分别取子叶、第1片真叶、第4片真叶、第7片真叶、顶叶,然后利用定量PCR的方法分析COL7的表达情况,实验结果显示:COL7在最低层的叶片中表达最少,而在最顶端的叶片中表达最多,即在离地面越远的叶片中COL7表达量越高(图5(b)).因此,COL7的表达具有空间性.
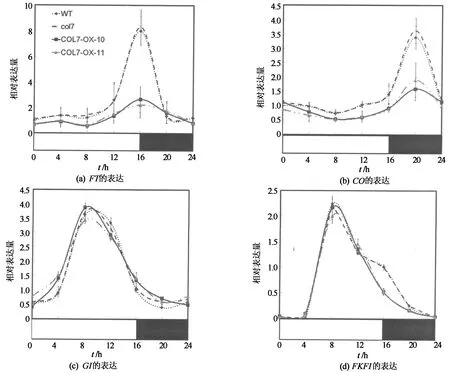
图3 长日照下COL7抑制FT,CO的表达
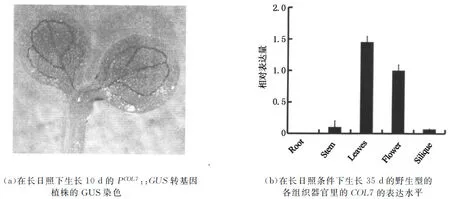
图4 COL7在韧皮部有表达
2.6 光对COL7基因表达的影响
以上实验结果显示:COL7在光周期途径中参与调控拟南芥开花,那么COL7是否受光调控呢?分别取在长日照条件下生长7 d、短日照条件下生长15 d的野生型幼苗,每4 h取一次样,共取一天,然后利用定量PCR分析COL7的表达情况.实验结果显示:COL7主要在白天表达,且其在开灯后第4 h表达量最高,无论是在长日照还是短日照下COL7的表达都受光诱导(图5(c)).因此,光诱导:COL7表达.白光是由不同的单色光组成,那么红光、远红光、蓝光是否同样会诱导COL7的表达呢?将野生型幼苗在黑暗下培养7 d,然后转移到红光、远红光、蓝光下,并分别在0,0.5,1,2,4,8 h取样,再利用定量PCR分析红光、远红光、蓝光对COL7表达的影响.实验结果显示:红光、远红光、蓝光也都会诱导COL7的表达(图5(d)).
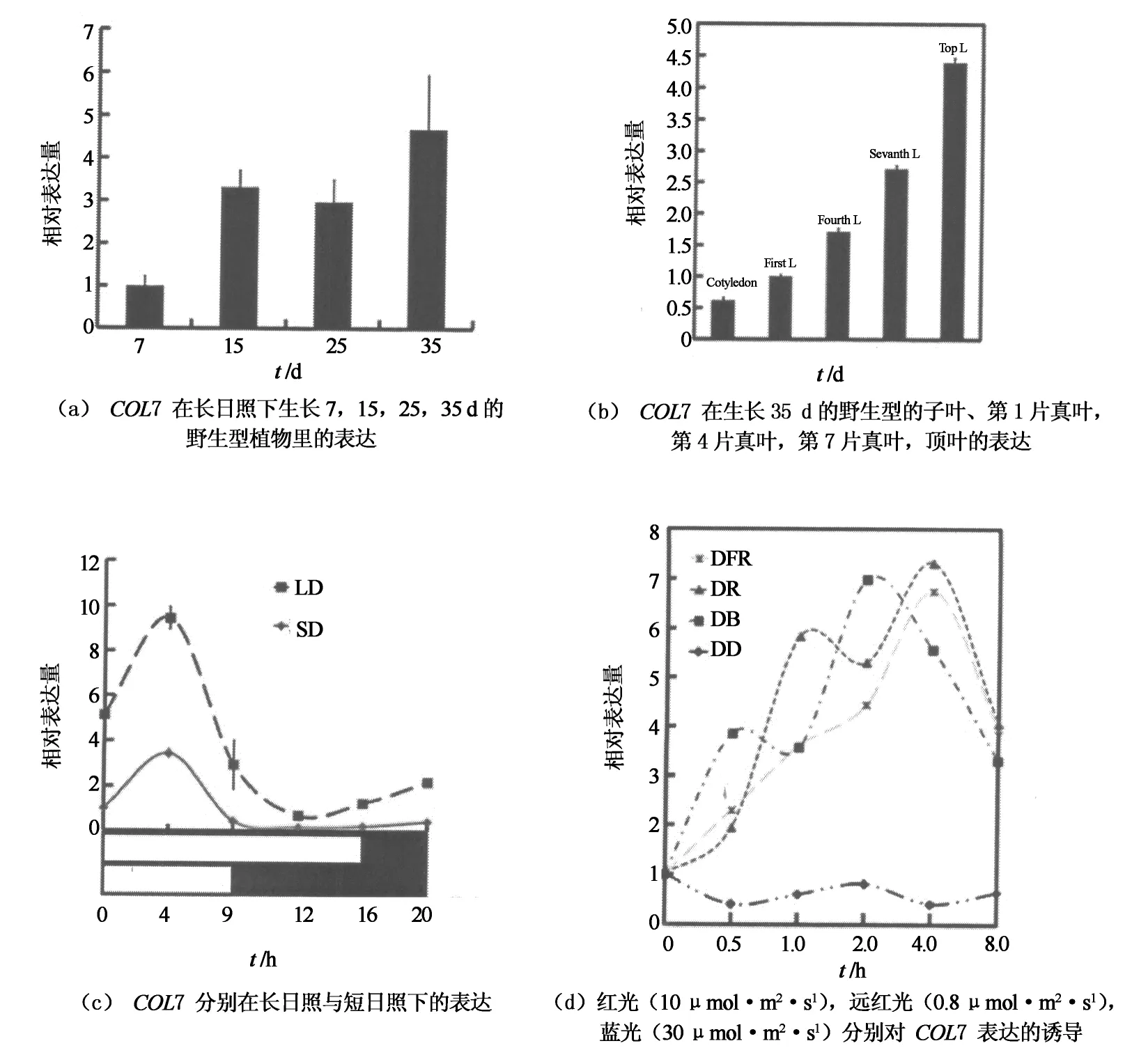
图5 COL7的表达
3 讨 论
过量表达COL7只在长日照条件下抑制拟南芥开花.因此,COL7抑制拟南芥开花与光照时间密切相关.COL7无论是在长日照还是短日照条件下,其开花时间与WT没有显著的差异.这说明COL7在调控开花方面与其家族基因存在功能冗余.COL7过表达的晚花表型是通过抑制CO,FT表达时实现的.FT是整合子基因,它可以将来自光周期途径的信号以及其它途径的信号整合,进而调控植物开花.光周期途径是通过介导光以及环境中的一些临时信号来调控一系列的开花基因的表达,从而调控植物开花.GI,FKF1即参与调控CO的表达,同时也参与调控FT的表达.然而COL7抑制CO,FT的表达并不是通过GI,FKF1,因为COL7不影响GI,FKF1的表达.过表达COL7晚花是因为CO,FT的表达受到抑制,目前CO家族中已经报道的基因只有COL9与COL7在调控开花方面具有相似的功能,COL9在长日照下抑制开花,其也是通过抑制CO,FT的表达.光信号在光周期其途径中起着很重要的作用,COL7的表达受光的调控,且COL7抑制开花受光照时间调控.COL7是开花抑制子,但是COL7有可能不是单独起作用,可能与其他基因一起参与调控开花.因为,一般情况下开花抑制因子在从营养生长转入生殖生长后其表达量是下降的,然而COL7的时空表达情况说明,COL7在拟南芥从营养生长转入生殖生长的过程中其表达是上升的,而且在新生的叶片中表达量最高,因此,COL7可能是与其他因子相互作用参与调控开花,而这些因子随着植物的生长逐渐减少,进而导致COL7对开花的抑制减弱,其确切的机制还需要进一步研究.
总之,通过本研究首次发现:COL7是开花负调控因子,而且其通过抑制CO,FT的表达进而抑制开花;COL7在韧皮部有表达,且COL7的表达具有时空特异性;COL7的表达受光调控,此外,COL7只在长日照下抑制开花,这说明COL7调控植物开花的功能与日照时间密切相关.因此,光不仅参与调控COL7的表达,同时也影响COL7调控开花的功能.
[1] DEVLIN P F, KAY S A. Flower arranging in Arabidopsis[J]. Science,2000,288(5471):1600-1602.
[2] LEVY YY, DEAN C. The transition to flowering[J]. Plant Cell,1998,10(12):1973-1990.
[3] WENKEL S, TURCK F,SINGE R K,etal. CONSTANS and the CCAAT box binding complex share a functionally important domain and interact to regulate flowering of Arabidopsis[J]. Plant Cell,2006,18(11):2971-2984.
[4] HAYAMA R, COUPLAND G. Shedding light on the circadian clock and the photoperiodic control of flowering[J]. Curr Opin Plant Biol,2003,6(1):3-19.
[5] DOMAGALSKA M A, SARNOWSKA E, NAGY F,etal. Genetic analyses of interactions among gibberellin, abscisic acid, and brassinosteroids in the control of flowering time in Arabidopsis thaliana[J]. PLoS One,2010,5(11):e14012.
[6] MOURADOV A, CREMER F,COUPLAND G,etal. Control of flowering time: interacting pathways as a basis for diversity[J]. Plant Cell,2002,14(Suppl):s111-141.
[7] MICHAELS S D, HIMLBLAU E,KIM S Y,etal. Integration of flowering signals in winter-annual Arabidopsis[J]. Plant Physiol,2005,137(1):149-156.
[8] WANG J W, CZECH B, WEIGEL D,etal. miR156-regulated SPL transcription factors define an endogenous flowering pathway in Arabidopsis thaliana[J]. Cell,2009,138(4):738-749.
[9] SIMPSON G G. Evolution of flowering in response to day length: flipping the CONSTANS switch[J]. Bioessays,2003,25(9):829-832.
[10]CHOW B Y, KAY S A. Global approaches for telling time: omics and the Arabidopsis circadian clock[J]. Semin Cell Dev Biol,2013,24(5):383-392.
[11]SEATON D D, SMITH R W, SONG Y H,etal. Linked circadian outputs control elongation growth and flowering in response to photoperiod and temperature[J]. Mol Syst Biol,2015,11(1):776.
[12]SONG Y H, ITO S,IMAIZUMI T,etal. Similarities in the circadian clock and photoperiodism in plants[J]. Curr Opin Plant Biol,2010,13(5):594-603.
[13]SUAREZ-LOPEZ P, WHEATLEY K,ROBSON F,etal. CONSTANS mediates between the circadian clock and the control of flowering in Arabidopsis[J]. Nature,2001,410(6832):1116-1136.
[14]JANG S, MARCHAL V,PANIGRAHI K C,etal. Arabidopsis COP1 shapes the temporal pattern of CO accumulation conferring a photoperiodic flowering response[J]. EMBO J,2008,27(8):1277-1288.
[15]LIU L J, ZHANG Y C,LI Q H,etal. COP1-mediated ubiquitination of CONSTANS is implicated in cryptochrome regulation of flowering in Arabidopsis[J]. Plant Cell,2008,20(2):292-306.
[16]FORNARA F, PANIGRAHI K C, GISSOT L,etal. Arabidopsis DOF transcription factors act redundantly to reduce CONSTANS expression and are essential for a photoperiodic flowering response[J]. Dev Cell,2009,17(1):75-86.
[17]IMAIZUMI T, SCHULTZ T F,HARMON F G,etal. FKF1 F-box protein mediates cyclic degradation of a repressor of CONSTANS in Arabidopsis[J]. Science,2005,309(5732):293-297.
[18]SAWA M, NUSINOW D A,KAY S A,etal. FKF1 and GIGANTEA complex formation is required for day-length measurement in Arabidopsis[J]. Science,2007,318(5848):261-265.
[19]SONG Y H, SMITH R W, TO B J,etal. FKF1 conveys timing information for CONSTANS stabilization in photoperiodic flowering[J]. Science,2012,336(6084):1045-1049.
[20]GRIFFITHS S, DUNFORD R P, COUPLAND G,etal. The evolution of CONSTANS-like gene families in barley, rice, and Arabidopsis[J]. Plant Physiol,2003,131(4):1855-1867.
[21]ROBOSON F, COSTA M M, HEPWORTH S R,etal. Functional importance of conserved domains in the flowering-time gene CONSTANS demonstrated by analysis of mutant alleles and transgenic plants[J]. Plant J,2001,28(6):619-631.
[22]CAI X, BALLIF J, ENDO S,etal. A putative CCAAT-binding transcription factor is a regulator of flowering timing in Arabidopsis[J]. Plant Physiol,2007,145(1):98-105.
[23]LEDGER S, STRAYER C, ASHTON F,etal. Analysis of the function of two circadian-regulated CONSTANS-LIKE genes[J]. Plant J,2001,26(1):15-22.
[24]DATTA S, HETTIARACHCHI G H, DENG X W,etal. Arabidopsis CONSTANS-LIKE3 is a positive regulator of red light signaling and root growth[J]. Plant Cell,2006,18(1):70-84.
[25]HASSIDIM M, HARIR Y,YAKIR E,etal. Over-expression of CONSTANS-LIKE 5 can induce flowering in short-day grown Arabidopsis[J]. Planta,2009,230(3):481-491.
[26]WANG H, ZHANG Z, LI H,etal. CONSTANS-LIKE 7 regulates branching and shade avoidance response in Arabidopsis[J]. J Exp Bot,2013,64(4):1017-1024.
[27]CHENG X F, WANG Z Y. Overexpression of COL9, a CONSTANS-LIKE gene, delays flowering by reducing expression of CO and FT in Arabidopsis thaliana[J]. Plant J,2005,43(5):758-768.
The Analysis of CONSTANS-LIKE 7 Regulateing Arabidopsis Flowering Time
WANG Hong-gui1,HUANG Chen1,CHEN Peng1,JIANG Ya1,ZHANG Ya1,TANG Dong-ying2,ZHAO Xiao-ying2,LIU Xuan-ming2†
(1.School of Environmental Science and Engineering, Yangzhou Univ, Yangzhou, Jiangsu 225127, China;2.Hunan Provence Key Laboratory of Plant Functional Genomics and Developmental Regulation, College of Biology, Hunan Univ, Changsha, Hunan 410082, China)
To discover the function ofCONSTANS-LIKE7 (COL7) in regulating plant flowering, we used qPCR, GUS staining,etal, to study the light and circadian regulating the expression ofCOL7, andCOL7 regulating Arabidopsis flowering time. The results showed thatCOL7 was regulated by light and circadian, and the overexpressingCOL7 transgenic lines inhibited Arabidopsis flowering time under long-day (LD) condition through down-regulatingCONSTANS(CO) andFLOWERINGLOCUST(FT), and thecol7 mutant had no obvious flowering phenotype under LD or SD conditions. SoCOL7 may have redundant functions with otherCONSTANS-LIKEs(COLs) in controlling the flowering time of Arabidopsis.
gene expression;arabidopsis;CONSTANS-LIKE7; flowering
2015-03-15
国家自然科学基金资助项目(21307104),National Natural Science Foundation of China(21307104) ;江苏省自然科学基金资助项目(BK20150451);江苏省高校自然科学基金资助项目(14KJB180025)
王宏归(1980-),男,湖南衡阳人,扬州大学教师,博士
†通讯联系人,E-mail:xmL05@hnu.edu.cn
1674-2974(2015)12-0088-07
Q786
A
——记嘉荫县红光乡燎原村党支部

