极地抗植物病原真菌活性菌株PKS和NRPS基因的克隆与分析*1
彭玉娇,李敬龙,林学政
(1. 齐鲁工业大学 食品与生物工程学院,山东 济南 250353; 2. 国家海洋局 第一海洋研究所,山东 青岛 266061; 3.国家海洋局 海洋生物活性物质重点实验室,山东 青岛 266061)
极地抗植物病原真菌活性菌株PKS和NRPS基因的克隆与分析*1
彭玉娇1,2,3,李敬龙1,林学政2,3*
(1. 齐鲁工业大学 食品与生物工程学院,山东 济南 250353; 2. 国家海洋局 第一海洋研究所,山东 青岛 266061; 3.国家海洋局 海洋生物活性物质重点实验室,山东 青岛 266061)
植物常见病原真菌尖孢镰刀菌(Fusariumoxysporum)为指示菌,采用平板扩散法对实验室保藏的38株极地细菌进行了抑菌活性验证,并对其中活性较强的菌株进行了分子鉴定与系统发育分析及次级代谢产物合成功能基因(PKS和NRPS)的克隆与分析。结果表明,38株极地细菌中,13株具有明显的抗病原真菌活性;分子鉴定与系统发育分析表明,11株活性菌株属于Pseudomonas,1株属于Psychrobacter,1株属于Arthrobacter;对活性菌株PKS和NRPS基因的克隆与分析表明,从10株活性菌株克隆到12条NRPS A结构域基因片段,其中5株活性菌株同时含有PKS I型 KS结构域基因片段。据此为从聚酮和非核糖体肽等生物合成途径深入研究活性菌株的次级代谢产物提供了基因学证据。
极地细菌;抑菌活性;多聚酮合酶;非核糖体肽合成酶
微生物来源的天然产物一直是发现新型药物先导化合物的重要来源。经过长期和广泛深入的研究之后,从陆地微生物中发现新的天然产物已越来越困难,而蕴涵着极其丰富生物资源的海洋微生物种群则是当今天然产物药物开发的巨大宝库。极地自然环境与地理环境的特殊性决定了极地微生物具有巨大的研究与开发潜力。随着新的病原微生物及其耐药性的不断出现,能否快速筛选到具有新结构、新活性的微生物代谢产物已成为新药研究迫切面对的问题[1]。
海洋微生物的次级代谢产物主要为聚酮类化合物和肽类化合物,负责该两类化合物生物合成的酶分别为聚酮合酶(polyketide synthases,PKS)和非核糖体肽合成酶(nonribosomal peptide synthetase,NRPS)[2]。PKS是一类复杂的多酶体系,多以简单的脂酰CoA为底物,通过重复的脱羧缩合过程产生线性聚酮化合物或环状的酮内酯,其过程类似于脂肪酸合酶催化的脂肪酸生物合成[3]。目前,普遍认为PKS分为I型、II型和III型三种类型,其中,尤以PKS I型途径合成的化合物结构多样、活性范围广著称[4]。这些聚酮化合物大多具有激素、抗生素、抗真菌素、抗肿瘤等生物活性,在至今上市的医药或农用抗生素结构中聚酮类化合物来源的比例居已知六大类天然产物的首位,具有重要的商业价值。在细菌和真菌中,一些重要多肽类物质的合成可以绕开核糖体,在这一类特殊的多肽化合物合成中起关键作用的一类特殊的酶就是非核糖体肽合成酶(NRPS)[5]。NRPS由多个模块组成,特定模块的结构域具有特定的酶活,催化相应单体结合到新生肽链中。这些结构域包括腺苷酰化结构域(A结构域)、巯基化结构域(T结构域)、缩合结构域(C结构域)、差向异构结构域(E结构域)和甲基化结构域(M结构域)等[6]。据报道,已经发现的NRPS产物中稀有氨基酸多达300个,它们主要来源于E结构域的差向异构化、M结构域的N-甲基化和其它结构域的酰基化、糖基化及杂环化修饰[7],这正是这类代谢产物多样化的原因。NRPS合成的多肽类化合物具有独特多样的生物活性,已被广泛应用和研究。应用最多的是多肽类抗生素,如万古霉素、Daptomycin、杆菌肽等,此外还可以作为免疫抑制剂药物、生物表面活性剂、抗癌药物、细胞生长抑制剂和抗病毒药物等[8]。
本研究以尖孢镰刀菌为指示菌对实验室保藏的38株极地细菌进行了抑菌活性验证,对其中具有较强抑菌活性的菌株进行了基于16S rRNA基因的分子鉴定与系统发育分析;并对次级代谢产物合成基因PKS、NRPS进行了克隆与分析,以期为从生物合成途径深入研究其次级代谢产物提供分子生物学的证据,并为今后开发利用极地微生物次级代谢产物的生物合成提供基因资源。
1 材料与方法
1.1 菌 株
极地抗植物病原真菌活性菌株为国家海洋局海洋生物活性物质重点实验室分离并保存;以植物病原真菌尖孢镰刀菌(Fusariumoxysporum)作为抗植物病原真菌指示菌;感受态细胞EscherichiacoliDH5α购于TaKaRa公司。
1.2 培养基与菌株活性验证
海水Zobell 2216 E培养基、PDA培养基和LB培养基配置及菌株的活性验证(平板扩散法)均参照文献[9]。
1.3 实验仪器及试剂
pMD18-T Vector (TaKaRa,D101A);细菌基因组提取试剂盒(TIANGEN,DP302-02)、普通琼脂糖凝胶回收试剂盒(TIANGEN,DP209-02)、2×Taq PCR MasterMix (TIANGEN,KT201)。
1.4 活性菌株基因组DNA的提取
按照细菌基因组提取试剂盒的操作步骤提取细菌基因组DNA,将提取的细菌基因组DNA经1%琼脂糖凝胶电泳检测,于-20 ℃储存。
1.5 16S rRNA基因扩增
以27F(5′-AGAGTTTGATCCTGGCTGGCTCAG-3′)和1492R (5′-TACGGCTACCTTGTTAC GACTT-3′)为引物,使用50 μL扩增体系(1 μL 27F,1 μL 1492R,25 μL Master Mix,23 μL ddH2O),PCR反应条件为95 ℃ 5 min;95 ℃ 1 min,55 ℃ 30 s,72 ℃ 90 s,30个循环;72 ℃ 10 min,进行16S rRNA基因扩增。
1.6 PKS与NRPS基因扩增简并引物与基因克隆
以PKS-U(5′-GCGATGGATCCNCAGCAGCG-3′)和PKS-D(5′-GTGCCGGTNCCGTGNGYYTC-3′)为引物[2],使用50 μL扩增体系,PCR反应条件为94 ℃ 3 min;94 ℃ 40 s,59 ℃ 1 min,72 ℃ 40 s,35个循环;72 ℃ 10 min,进行PKS基因扩增。
以NRPS-U (5′-GCSTACSYSA TSTACACSTCSGG-3′)和NRPS-D (5′-SASGTCVCCSGTSCGG TAS-3′) 为引物[2],使用50 μL扩增体系,PCR反应条件为50 μL扩增体系,反应条件为 95 ℃ 5 min;95 ℃ 1 min,59 ℃ 1 min,72 ℃ 1 min,35个循环;72 ℃ 10 min,进行NRPS基因扩增。
扩增到的目的基因经琼脂糖凝胶回收试剂盒(Tiangen,DP209-02) 回收,操作步骤按其说明书进行。
1.7 序列测定及分析
将PCR反应得到的目的条带切胶回收,连接T载体pMD18-T后,转化感受态细胞E.coli DH 5α。将经T载体特异性引物M13-47/RV-M验证后的阳性克隆子送至南京金斯瑞生物科技有限公司测序,将得到的目标片段序列采用BALST程序进行同源比对分析并提交GenBank数据库,利用软件Mega 4.0构建系统发育树。
2 结果与分析
2.1 抑菌活性与系统发育分析
以尖孢镰刀菌为指示菌,利用平板扩散法对实验室保藏的38株极地细菌进行了抗植物病原真菌活性验证。结果表明,其中13株菌株具有明显的抑菌活性,对其进行了16S rRNA基因扩增与分析,将得到的序列提交GenBank,经BLAST比对得到最高相似菌株,并利用Mega 4.0软件构建系统发育树,结果分别见表1和图1。

表1 活性菌株分子鉴定及其抑菌效果Table 1 identification of antifungal strains and their antifungal effects
从表1中可以看出,38株极地活性菌株中有13株具有明显的抑菌活性(抑菌圈直径均>16 mm);其中菌株C-1抑菌效果最明显,抑菌圈直径达到26.0 mm。菌株11W、83-1、195、2-Z18、P406、P4-11-07和P499抑菌效果也比较显著,均在20 mm以上;本研究中活性菌株绝大多数与GenBank数据库中已有菌株的16S rRNA 基因序列存在着很高的相似性,相似性均在99%以上,且假单胞菌属(Pseudomonas)活性菌株的抑菌活性明显高于嗜冷杆菌属(Psychrobacter)和节杆菌属(Arthrobacter)的活性菌株;13株活性菌株分别属于3个属,其中11株属于Pseudomonas,1株属于Psychrobacter,1株属于Arthrobacter。

图1 抗菌活性菌株的16S rRNA序列系统发育分析Fig.1 Phylogenetic analysis of antimicrobial bacteria based on 16S rRNA gene sequences
注:节点处的数字表示在1 000次运算中出现的概率,只表示出了≥50%的情况
2.2 PKS基因的扩增结果与系统发育分析
利用简并引物PKS-U/PKS-D从13株活性菌株中扩增PKS基因,将处理好的测序结果提交至NCBI并使用BLASTX功能进行基因比对,活性菌株PKS基因的扩增及分析结果见表2。由表2可以看出从5株活性菌株(C-1、P499、83-1、P406和205)中成功扩增到PKS基因片段,长度在1 125~1 143 bp之间,其余8株均未成功扩增出PKS片段;从5株活性菌株中扩增到的5条PKS基因片段均与Pseudomonas brassicacearum NFM421的酰基转移酶(PKS)的基因序列相似度最高,相似度在88%左右。将得到的PKS基因的核苷酸序列提交至GenBank数据库,获得序列注册号为:KF297895~KF297899。
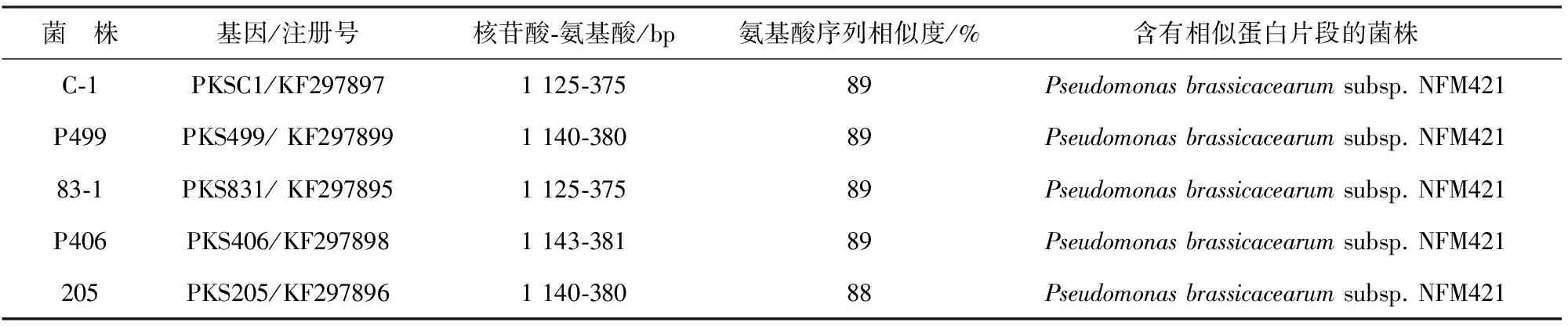
表2 所获PKS片段与GenBank中氨基酸序列同源性状况Table 2 PKS gene fragments obtained in present study and their identities in the GenBank
将本研究得到的PKS基因核苷酸序列翻译成氨基酸序列,使用Mega 4.0软件构建系统发育树,结果见图2。
通过基于PKS氨基酸序列的系统发育分析(图2),推断本研究得到的PKS基因片段编码的蛋白序列都属于KS结构域的trans-AT型,这种类型的PKS被认为与有药物活性的代谢产物的产生有关[10]。这与抑菌活性实验结果一致,成功扩增出PKS基因片段的菌株C-1、P499、83-1、P406、205在抑菌活性实验中均表现出较强的抑菌活性。这说明PKS基因可能与具有抗植物病原真菌活性的化合物的合成有关。

图2 基于PKS氨基酸序列的系统发育分析Fig.2 Phylogenetic analysis based on amino PKS acid sequences using neighbor-joining method
2.3 NRPS基因的扩增结果与系统发育分析
利用NRPS简并引物从13株活性菌株中扩增NRPS基因,将处理好的测序结果提交至NCBI并使用BLASTX功能进行基因比对,结果从10株活性菌株中扩增到NRPS基因的片段,扩增结果及分析见表3。

表3 所获NRPS片段与GenBank中氨基酸序列同源性状况Table 3 NRPS gene fragments obtained in present study and their identities in the GenBank
从表3中可以看出,从10株活性菌株(C-1、83-1、P406、P499、205、195、P4-11-07、LX-4、2-Z18和11W)中成功扩增出了12条NRPS基因片段,长度在684~720 bp之间,与预期基因片段的长度一致。这说明NRPS基因在Pseudomonas中能广泛存在。从表中可以看出除菌株P499和菌株11W的NRPS基因序列与数据库菌株蛋白Amp-binding enzyme(NRPS基因)基因序列的相似度最低,为88%。其他菌株的相似度均在90%以上。12株活性菌株的氨基酸序列与数据库相似的菌株主要为PseudomonasfluorescensSS101和Pseudomonassp. R81。将得到的NRPS基因的核苷酸序列提交至GenBank数据库,获得序列注册号为:KF026290~KF026301。
值得指出的是,从菌株C-1和P499均成功扩增出2条NRPS基因片段,分别为NRP17/NRP19和NRP4995/NRP4998。由BLAST结果可知,片段NRP17与菌株PseudomonasfluorescensSBW25的NRPS A域相似性最高,为90%;片段NRP19与菌株Pseudomonassp. R81的NRPS A域相似性最高,为94%;片段NRP4995与菌株Pseudomonassp. R81的NRPS A域相似性最高,为94%;片段NRP4998与菌株PseudomonasfluorescensSBW25的NRPS A域相似性最高,为89%。由BLAST可知,从菌株C-1克隆到的2个NRPS基因片段NRP17和NRP19的氨基酸序列相似度仅为35%;从菌株499克隆到的2个NRPS基因片段NRP4995和NRP4998的氨基酸序列相似度仅为38%。由此得出,从同一菌株中扩增出的2条NRPS片段相似度均较低。
将本研究得到的NRPS基因核苷酸序列翻译成氨基酸序列,使用Mega 4.0软件构建系统发育树,结果见图3。

图3 基于NRPS氨基酸序列的系统发育分析Fig.3 Phylogenetic analysis based on NRPS amino acid sequences using neighbor-joining method
BLAST结果及NRPS系统发育树(图3)均表明本研究所得NRPS基因片段编码的蛋白序列为NRPS基因簇的腺苷酰化结构域(adenylation,A),且供试的11株Pseudomonas菌株除1株未得到NRPS片段外,其余10株均成功扩增到NRPS片段,说明NRPS基因在Pseudomonas中广泛存在。
3 讨 论
本研究以尖孢镰刀菌(Fusariumoxysporum)为指示菌,采用平板扩散法对实验室保藏的38株极地细菌进行了抑菌活性验证,并对其中活性较强的菌株进行了基于16S rRNA的分子鉴定与系统发育分析以及PKS和NRPS基因的克隆与分析。
活性验证实验表明,38株极地细菌中13株具有较强抗菌活性;分子鉴定与系统发育分析表明,11株菌株属于Pseudomonas,1株属于Psychrobacter,1株属于Arthrobacter。活性鉴定实验表明Pseudomonas属菌株具有较强的抑菌活性。近年来对于Pseudomonas菌株产生活性物质的研究已有若干报道,例如温露等[11]对海洋细菌Pseudomonassp.的次级代谢产物进行了研究,从其中分离到一个蓝色素,生物活性试验显示该蓝色素,对肿瘤细胞生长有很强的抑制作用。漆淑华等[12]根据活性追踪从海洋细菌Pseudomonassp. 发酵液中分离到4个环二肽,可抑制多种能诱导海洋生物幼虫附着的细菌的生长。
11株Pseudomonas活性菌株中,除菌株BN05-S-2外,其余10株菌株均成功扩增出NRPS基因片段。经BLAST比对与系统发育树分析可见,本研究所得到的12条NRPS片段均属于NRPS的A结构域,表明NRPS基因在Pseudomonas中广泛存在。在16S rRNA系统发育树中进化关系较近的11W、LX-4、2-Z18、83-1等菌株的NRPS 基因的A结构域并不在一个分支上,遗传距离较远,由此证明了它们可能并非同一株菌,同时也证明了得到的NRPS基因片段的多样性[13]。值得指出的是,从菌株C-1和P499均成功扩增出2条NRPS基因片段,分别为NRP17/NRP19和NRP4995/NRP4998。从同一菌株中扩增出2条NRPS A域片段,且均分别不在一个系统发育分支上;BLAST结果和氨基酸系统发育树均说明从同一菌株中扩增出的2条A域片段相似度较低,这也从另一方面证明了NRPS基因的多样性。董晓毅等[14]针对我国东海海域不同生境样品进行I型PKS基因筛选实验中也从同一株菌中扩增到2条相似度较低的基因片段,这可能是由于设计的简并引物特异性较低导致,但具体原因未见报道。
5株Pseudomonas活性菌株C-1、P499、83-1、P406和205均成功扩增出PKS KS结构域片段,由系统发育树可以推断这5条PKS片段均属于trans-AT型。PKS I型基因通常合成一些活性物质尤其是具有抑菌活性的化合物或抗生素的前体,在生物防御中扮演着重要角色。在本研究中获得PKS基因的5株菌对尖孢镰刀菌均表现出较强的抑菌活性,也进一步说明从这些菌群里发现能产生新型活性化合物的活性菌株的可能性。1993年从Bacillussubtilis168的基因组测序中第一次发现trans-AT型PKS以来,越来越多的trans-AT型PKS被发现,一些重要的药用化合物,如莫匹罗星,卡兰杀菌素,苔藓抑素A,和维吉霉素M等都被证实是由trans-AT型PKS产生[15]。
近年来已有若干对Pseudomonas菌株功能基因的研究报道,例如:Philippe等[16]对从植物根部分离的Pseudomonasbrassicacearum进行PKS基因的克隆与序列分析,结果发现该菌株含有酰基转移酶基因与大环酯类流出蛋白基因,这两种蛋白基因都属于PKS基因;刘双霜等[17]针对NRPS的Amp-binding 腺苷化结构域编码基因设计引物,对从中国舟山海域海水筛选的嗜盐假单胞菌进行PCR扩增,结果18株假单胞菌中有8株菌株扩增到目的片段;Mossialos等[18]也从Pseudomonasfluorescens扩增出NRPS基因目的片段。目前,针对极地细菌次级代谢产物合成功能基因如PKS与NRPS的研究尚未见报道。
目前对于PKS和NRPS基因多样性的研究也有若干报道,不同生境微生物中PKS和NRPS基因具有丰富的多样性,已有对海绵、总草苔虫等海洋无脊椎动物,陆地土壤、南极土壤、红树林植物根际土壤[19]等环境样品中微生物次级代谢产物多样性的报道。Ginolhac等[20]研究发现海洋沉积物中微生物种类繁多,不同环境的海洋沉积物中可能蕴含着丰富的新型PKS功能基因;董晓毅等[14]从东海洋山港沿岸土壤、海水及海底沉积物等不同生境中获得了23条PKS片段,说明不同生境中PKS基因的多样性;朱文勇等[21]通过PCR筛选,从云南大学的喜树内生放线菌中检测到了PKSI、PKSII、和NRPS基因,阳性率分别为31.1%,48.9%和45.6%。
极地适冷菌中PKS和NRPS基因的发现,为活性代谢产物的来源进一步提供了证据。菌株C-1和P499中2种NRPS基因片段的发现,意味着其中的代谢产物可能更加丰富。基于PKS和NRPS基因的多样性和新颖性,可以指导筛选具有药用价值的新化合物合成能力的活性菌,因此菌株C-1、P499、83-1、P406和205值得进一步深入研究。PKS和NRPS可以合成抗细菌、抗真菌、抗肿瘤等多种活性物质,对分离到的具有抗菌活性的菌株进行进一步的基因克隆,或许可以从分子生物学的角度来分析、阐述和推测菌株具有的生物活性物质的天然产物结构类型和合成机理[2],这也是今后笔者研究的重要方向。
[1] DEMAIN A L, SANCHEZ S. Microbial drug discovery: 80 years of progress[J]. Journal of Antibiotics, 2009, 62(1): 5-16.
[2] ZHU P, ZHENG L, LIN J, et al. Screening and characterization of marine bacteria with antibacterial and cytotoxic activitives, and existence of PKS Ⅰ and NRPS genes in bioactive strains[J]. Acta Microbiologica Sinica, 2007, 47(2): 228-234. 朱鹏, 郑立, 林晶, 等. 抗菌和细胞毒活性海洋细菌的筛选及其次生代谢基因证据[J]. 微生物学报, 2007, 47(2): 228-234.
[3] JIAO Y L, WANG L H, DONG X Y, et al. Isolation and partial characterization of new PKS gene clusters[J]. Chinese Journal of Antibiotics, 2008, 33(3): 134-139. 焦豫良, 王良华, 董晓毅, 等. 新型聚酮合酶基因簇的分离与部分鉴定[J]. 中国抗生素杂志, 2008,33(3):134-139.
[4] KONRAD U F, TOBIAS D, CHRISTOPHER J C, et al. A computational screen for type Ⅰ polyketide synthases in metagenomics shotgun data[J]. PublicLibrary of Science One, 2008, 3(10): e3515.
[5] MING Z H, PAN J W, ZHU M Y. Progress in nonribosomal peptide synthetases[J]. Progress Biochemical Biophysical, 2002, 29(5): 667-669. 明镇寰, 潘建伟, 朱睦元. 非核糖体多肽合成酶研究进展[J].生物化学与生物物理进展, 2002, 29(5): 667-669.
[6] ZHENG Z M, GU X B, YU H Q, et al. Advances in main domains of nonribosomal peptide synthetases[J]. Chinese Journal of Antibiotics. 2005, 30 (2): 120-124. 郑宗明, 顾晓波, 俞海清, 等. 非核糖体肽合成酶主要结构域的研究进展[J]. 中国抗生素杂志,2005,30(2):120-124.
[7] CROSA J H, WALSH C T. Genetics and assembly line enzymology of siderophore biosynthesis in bacteria[J]. Microbiology and Molecular Biology Reviews, 2002, 66(2): 223.
[8] WANG S Y. Advances in the study of the mechanism and application of nonribosomal peptide synthetases[J]. Acta Microbiologica Sinica, 2007, 47(4): 734-737. 王世媛. 非核糖体肽合成酶(NRPSs)作用机理与应用的研究进展[J]. 微生物学报, 2007, 47(4): 734-737.
[9] WANG H M, HE P Q, LIN X Z, et al. Screening of antarctic bacteria with antifungal activity againstFusariumoxysporumand growth characteristics ofPsychrobactersp. P4-11-07-1[J]. Advances in Marine Science, 2012, 30(1): 102-110. 王红梅, 何培青, 林学政, 等. 抑制尖刀镰孢菌(Fusariumoxysporum)南极细菌的筛选Psychrobactersp.P4-11-07-1的生长特性[J]. 海洋科学进展, 2012, 30(1): 102-110.
[10] PIEL J. Biosynthesis of polyketides by trans-AT polyketide synthases[J]. Natural Product Reports, 2010, 27 (7):996-1047.
[11] WEN L, YUAN B H, LI H J, et al. A blue pigment isolated from marine bacteriaPseudomonassp.[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni, 2005, 44(4):63-69. 温露, 袁保红, 李厚金, 等. 南海海洋细菌Pseudomonas sp.产生的一种抗肿瘤蓝色素[J]. 中山大学学报, 2005, 44(4):63-69.
[12] QI S H, QIAN P Y, ZHANG C. Antibacterial Metabolites from Marine BacteriumPseudomonassp.[J]. Natural Product Research and Development, 2009, 21:420-423. 漆淑华, 钱培元, 张促. 海洋细菌Pseudomonassp. 抗菌代谢产物的研究[J]. 天然产物研究与开发, 2009, 21:420-423.
[13] ZHANG W. Screening and diversity revelation of PKS and NRPS gene in sponge associated microbes[D]. Shanghai:Shanghai Jiao Tong University, 2008. 张薇, 海绵共附生活性菌PKS和NRPS基因的筛选及多样性研究[D].上海:上海交通大学,2008.
[14] DONG X Y. Screening and identification of Marine microorganisms type I PKS gene[D]. Shanghai: The Second Military Medical Universit, 2007. 董晓毅, 海洋微生物Ⅰ型PKS基因资源的筛选与鉴定[D]. 上海:第二军医大学, 2007.
[15] PIEL J. Biosynthesis of polyketides by trans-AT polyketide synthases[J]. Natural Product Reports, 2010, 27(7): 996-1047.
[16] ORTET P, BARAKAT M, LALAOUNA D, et al. Complete genome sequence of a beneficial plant root-associated bacterium, Pseudomonas brassicacearum [J]. Jouranl of Bacteriology, 2011, 93(2): 3146-3149.
[17] LIU S S, SHAO Z Y, CHEN T, et al. Analysis of bacteriaHalomonascommunity composition of sea waters around Zhoushan Archipelago Sea Area[J]. Food and Drug, 2011, 13(1):9-13.刘双霜, 邵志宇, 陈婷, 等. 中国舟山海域海水嗜盐假单胞菌多样性分析[J]. 食品与药品,2011, 13(1):9-13.
[18] MOSSIALOS D, OCHSNER U, BAYSSE C, et al. Identification of new, conserved, non-ribosomal peptide synthetases from pseudomonas fluorescent involved in the biosynthesis of the siderophore pyoverdine [J]. Molecular Microbiology, 2002, 45(6): 1673-1685.
[19] MA M, TANG M, HONG K. Detection of type I and II polyketide synthase genes in microorganisms from mangrove rhizosphere soil[J]. Institute of Microbiology, 2013, 40(7): 1231-1240. 马敏, 唐敏, 洪葵. 四种红树植物根际土壤微生物Ⅰ型和Ⅱ型PKS基因的检测与多样性分析[J]. 微生物学通报, 2013, 40(7): 1231-1240.
[20] AURELIEN G, CYRILLE J, BENJAMIN G, et al. Phylogenetic analysis of polyketide synthase I domains from soil metage-nomic libraries allows selection of promising clones[J]. American Society for Microbiology, 2004, 70(9): 5522-5527.
[21] ZHU W Y, LI J, ZHAO G Z, et al. Diversity and antimicrobial activities of endophytic actinomycetes isolated fromCamptothecaacuminataDecne.[J]. Institute of Microbiology, 2010,37(2): 211-216. 朱文勇, 李洁, 赵国振, 等. 喜树内生放线菌多样性及抗菌活性评价[J]. 微生物学通报, 2010,37(2): 211-216.
CloningandAnalysisofPKSandNRPSGenesFromPolarBacteriaWithAntifungalActivityAgainstPlantPathogenicFungi
PENG Yu-jiao1,2,3, LI Jing-long1, LIN Xue-zheng2,3*
(1.CollegeofFoodandBiologicalEngineering,QiluUniversityofTechnology, Jinan 250353, China; 2.TheFirstInstituteofOceanography,SOA, Qingdao 266061, China; 3.KeyLaboratoryofMarineBioactiveSubstances,SOA, Qingdao 266061,China)
The antifungal activity against plant pathogenic fungiFusariumoxysporumof 38 strains of polar bacteria was validated by method of agar diffusion. The results showed that 13 strains had obvious antimicrobial activity. Molecular identification and phylogenetic analysis on the basis of 16S rRNA gene indicated that 11 strains belonged to genus ofPseudomonasand 2 strains belonged to genus ofPsychrobacterandArthrobacterrespectively. The PKS and NRPS genes of these antifungal strains were also cloned and analyzed, which might be responsible for biosynthesis of bioactive secondary metabolites. The 12 gene fragments of the domain A of NRPS and the domain KS of type I PKS were cloned from 10 and 5 antifungal strains, respectively. This paper provides a genetic evidence for deeply study these antifungal strains in which the PKS and NRPS biosynthetic pathways may applied.
Polar bacteria; antimicrobial activity; PKS; NRPS
September 13, 2013
2013-09-13
海洋公益性行业科研专项经费项目——深远海(极地)微生物生物农药的研制与中试示范 (201005032-2)
彭玉娇(1989-),女,山东青岛人,硕士,主要从事极端环境微生物方面研究. E-mail:pengyujiao12@163.com
*通讯作者,E-mail:linxz@fio.org.cn
(王佳实 编辑)
Q939
A
1671-6647(2014)03-0396-09

