基于人2型糖尿病易感SNP位点筛查食蟹猴2型糖尿病易感SNP标记
柳明玉,孙 飞,蒙裕欢,田 帅,陈军辉,杜红丽
(华南理工大学生物科学与工程学院,广州 510006)
2型糖尿病(type 2 diabetes mellitus, T2DM)是人类最常见的内分泌紊乱性疾病,由遗传和环境因素综合作用的结果,以胰岛素抵抗和胰岛β细胞功能衰竭为主要特征,其发病机制至今尚未完全阐明[1]。在过去的二十年里,T2DM的人数正在迅速增加,其发病率接近6%[2]。随着人口老龄化及肥胖人数的增多,到2025年,全球T2DM患者人数将增至3亿[3]。 T2DM迅速成为21世纪全球性最大的健康问题之一,同时这一疾病也引起了与其相关的并发症的迅速蔓延,如缺血性心脏疾病、中风、神经病变、视网膜病变、肾病变等[4]。近年来,随着候选基因技术的不断完善,尤其是全基因组相关性分析(GWAS)及SNP技术的发展,已鉴定出多种糖尿病易感基因及与T2DM密切相关的SNP位点[5-15],这些位点的鉴定为揭示T2DM的发病机制提供大量信息,以便更好的预防,诊断和治疗该疾病[16]。但由于缺少理想T2DM的动物模型,该病的分子机制仍未被完全阐明。因此,构建与人类T2DM发病进程相似的T2DM动物模型对于新药开发、发病机理的研究具有极大的应用价值[16]。食蟹猴的遗传背景与人的高度相似,其T2DM模型能较好地模拟人T2DM发病进程,但自发性的食蟹猴T2DM发病比率很低[17],远远不能满足研究需求。因此,本研究以人37个T2DM易感SNP位点为基础,分析食蟹猴对应基因组区域SNP与食蟹猴T2DM的关联性,以筛查食蟹猴T2DM易感SNP标记,筛出的这些标记可能对于食蟹猴T2DM模型的构建具有重要的意义。
1 材料和方法
1.1 血液样品
83只食蟹猴血样分别由广东蓝岛生物科技有限公司(中国广东),广东高要市康达实验动物科技有限公司(中国广东)和云南金杰康生物科技有限公司(中国云南)提供。所有食蟹猴样本(许可证号SCXK(粤)2014-0010)在直系三代内均无亲缘关系,并单独饲养于70 cm×70 cm×80 cm大小的笼子内,环境温度为25°C左右,相对空气湿度为50%~80%,每天12 h光照,12 h黑暗。每只食蟹猴喂食苹果100 g/d(饲料研究所,广东省广州市),每天两次,并允许自由饮水。从上述三个公司采集的样品数目分别为83(包括35个自发和48个诱导个体)。所有样品被分为4个大组(两个自发组和两个诱导组),根据空腹血糖和糖化血红蛋白值,每个大组分为T2DM组和对照组(正常的个体),其中同时满足空腹血糖≥108 mg/dL和糖化血红蛋白≥6% 定义为T2DM个体,同时满足空腹血糖 ≤90 mg/dL和糖化血红蛋白≤5% 定义为正常个体。所有实验食蟹猴样本均获得隶属广东蓝岛生物科技有限公司的机构动物管理及使用委员会审批,并受到该机构的监督。
1.2 实验仪器与试剂
1.2.1 实验仪器:基因扩增仪:Bio-Rad Tetrad 2;紫外可见光成像系统:美国UVP公司 3UV Transilluminator;凝胶成像系统:Bio-Rad;恒温水浴锅:宏华仪器HH-4
1.2.2 实验试剂:PCR Taq Mix,购自广州东盛生物科技有限公司;DL2000 marker,购自广州东盛生物科技有限公司;琼脂糖干粉,购自Biowest;溴化乙锭(EB),购自广州普博欣生物试剂公司;Proteinase K,购自宝生物工程(大连)有限公司;无水乙醇,购自天津大茂化学仪器厂;1.5 mL离心管(RNA-free),购自广州普博欣生物试剂公司;EDTA-2K抗凝管,购自湖南浏阳市医用仪具厂;枪头(RNA-free)和一次性注射器(5 mL),均购自广州普博欣生物试剂公司。
1.3 方法
1.3.1 基因组DNA的提取和DNA池的准备:基因组DNA采用北京普博生物科技有限公司的试剂盒进行抽提,提取的DNA基因组使用分光光度计测其浓度,然后统一稀释至10 ng/μL,分别取等体积等浓度的30个样品,组成A池(T2DM的个体混合基因组)和B池(正常个体混合基因组)。
1.3.2 SNP选择和序列下载:本研究分别以人类已报道的37个T2DM易感突变位点为中心,在人类基因组上下游截取1 000 bp,并用这些序列同源比对下载食蟹猴基因组对应区域的序列(表1)。
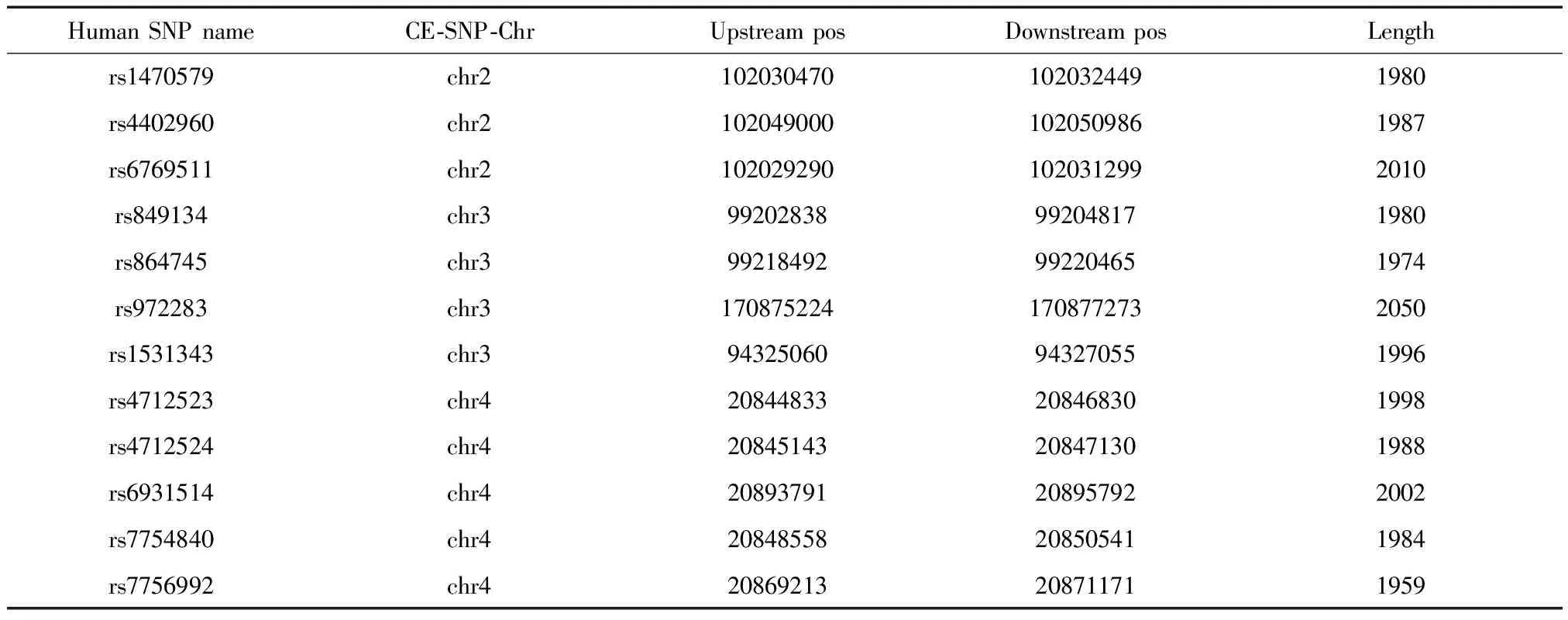
表1 37个对应食蟹猴同源序列信息表
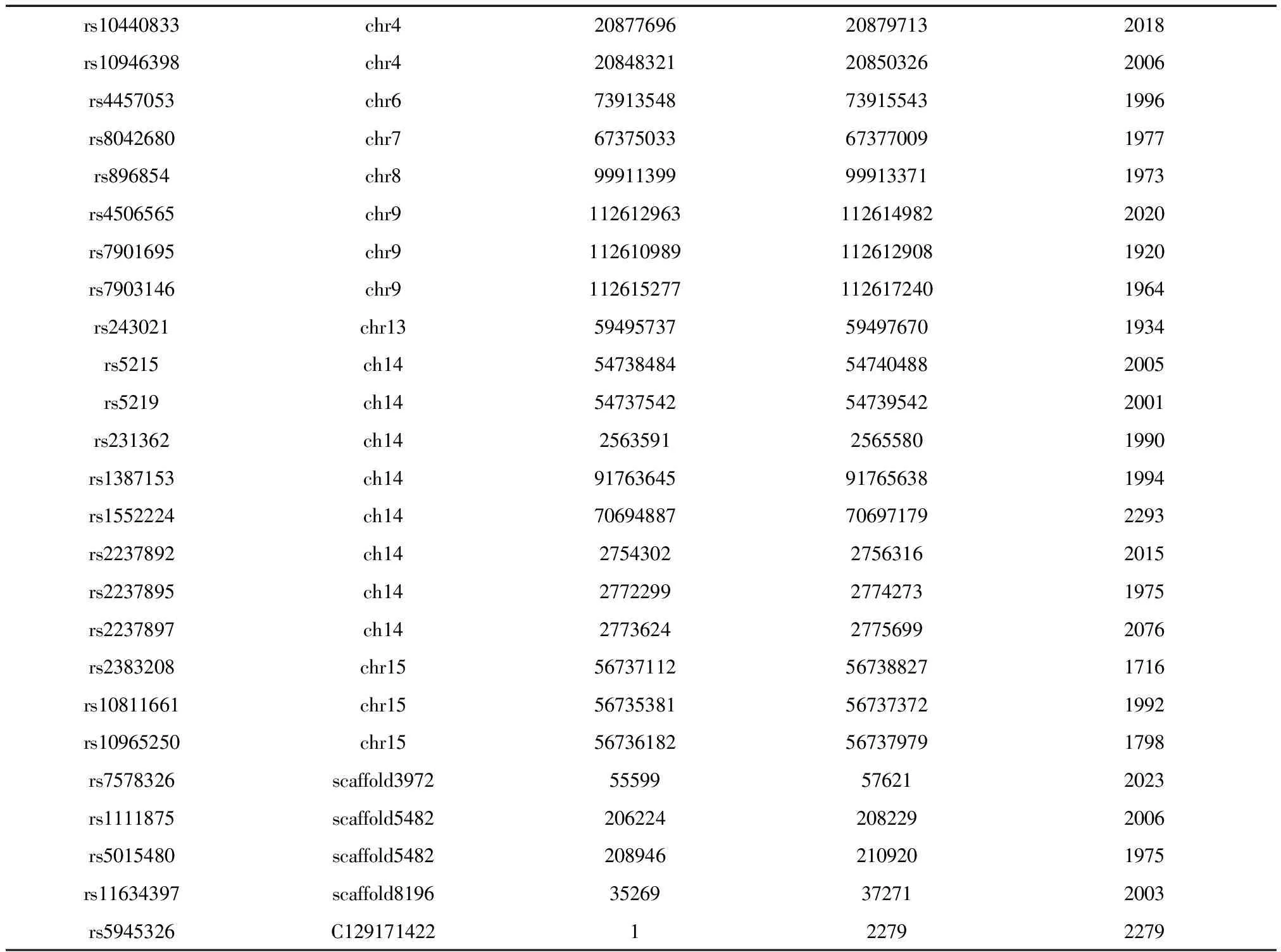
rs10440833chr420877696208797132018rs10946398chr420848321208503262006rs4457053chr673913548739155431996rs8042680chr767375033673770091977rs896854chr899911399999133711973rs4506565chr91126129631126149822020rs7901695chr91126109891126129081920rs7903146chr91126152771126172401964rs243021chr1359495737594976701934rs5215ch1454738484547404882005rs5219ch1454737542547395422001rs231362ch14256359125655801990rs1387153ch1491763645917656381994rs1552224ch1470694887706971792293rs2237892ch14275430227563162015rs2237895ch14277229927742731975rs2237897ch14277362427756992076rs2383208chr1556737112567388271716rs10811661chr1556735381567373721992rs10965250chr1556736182567379791798rs7578326scaffold397255599576212023rs1111875scaffold54822062242082292006rs5015480scaffold54822089462109201975rs11634397scaffold819635269372712003rs5945326C129171422122792279
1.3.3 PCR扩增及测序:根据已下载的食蟹猴序列,设计37对引物,引物序列见表2。引物由上海生工生物工程公司合成。以上述的A池和B池为模板,通过PCR扩增目的片段,PCR产物送华大基因测序。筛选出A池和B池测序存在显著性差异SNP的引物,以83个体基因组为模板进行PCR,扩增DNA片段(表3)。
1.3.4 基因分型和等位基因频率:以83个个体基因组为模板,五对引物分别进行PCR扩增和测序,利用DNAStar软件分析碱基之间的差异性,同时计算等位基因间的频率。
1.3.5 关联分析:基因频率差异采用方差分析和F检验[18],基因型与空腹血糖值和糖化血红蛋白值之间的关联分析采用SAS的GLM模型,基因型为固定效应,两种分析都规定P<0.05表示差异显著性,P<0.01表示差异极显著。

表2 PCR扩增引物序列
注:a退火温度b在对照组和疾病组样本中未成功扩增出目的片段的引物序列
NoteaAnnealing temperature,bPrimers can’t amplified the expected DNA fragments between case and control individuals.
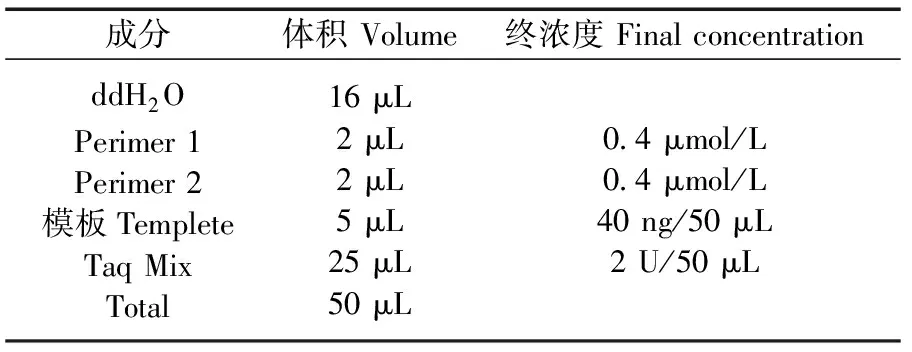
表3 PCR反应体系
2 结果
2.1 PCR和测序
本研究共设计了37对引物,其中有5对引物扩增的片段在自发的T2DM组和对照组中存在很大差异,见图1。这5对引物分别命名为mf-56(3个SNP),mf-66(2个SNP),mf-14(3个SNP),mf-34(4个SNP),mf-32(1个SNP),采用上述5对引物,以83个个体基因组为模板,进行PCR扩增并测序,分析13个SNP在四个大组中的差异。
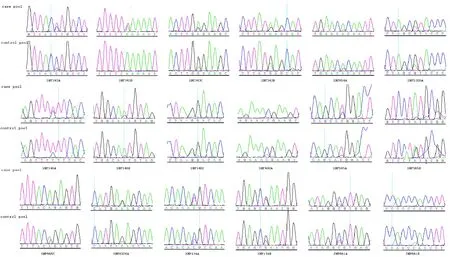
A:T2DM个体混合基因组,B:正常个体混合基因组
2.2 基因分型和等位基因频率
每个SNP等位基因在T2DM组和对照组之间的分布差异以及与T2DM的关联性分析结果表明,在金杰康自发群体中,SNPmf-66A和SNPmf-66B 在T2DM组和对照组之间的等位基因频率差异显著(P<0.05),同时这两个位点的基因型与空腹血糖值之间也存在显著关联性(type IIIP<0.05),(表4)。在蓝岛自发群体中,SNPmf-34A、SNPmf-34B、 SNPmf-34C、SNPmf-56A, SNPmf-56B和SNPmf-56C在T2DM组和对照组之间等位基因频率也存在差异显著性(P<0.05),其中SNPmf-34A、SNPmf-34B、SNPmf-34C位点的基因型与空腹血糖值之间存在极显著关联性(type IIIP<0.01),(表5)。在云南金杰康和广东蓝岛所有自发群体中,SNPmf-66B、SNPmf-34A、SNPmf-34B在T2DM组和对照组之间的等位基因频率存在显著性差异(P<0.05),SNPmf-34A、SNPmf-34B、SNPmf-34C位点的基因型与空腹血糖之间存在显著性关联(type IIIP<0.05),(表6)。并且,这13个SNP位点与糖化血红蛋白之间无显著性关联性。此外,在诱导群体中,这13个SNP位点的等位基因频率在T2DM组和对照组之间均无显著性差异,且与空腹血糖和糖化血红蛋白也无显著性关联(P>0.05)。
在自发性T2DM群体中,我们筛查出与人的T2DM易感基因rs10811661和rs1531343对应的食蟹猴基因组上同源区域内有5个SNP位点即SNPmf-66A、SNPmf-66B、SNPmf-34A,SNPmf-34B, SNPmf-34C有可能是食蟹猴2型糖尿病模型制备的遗传标记。
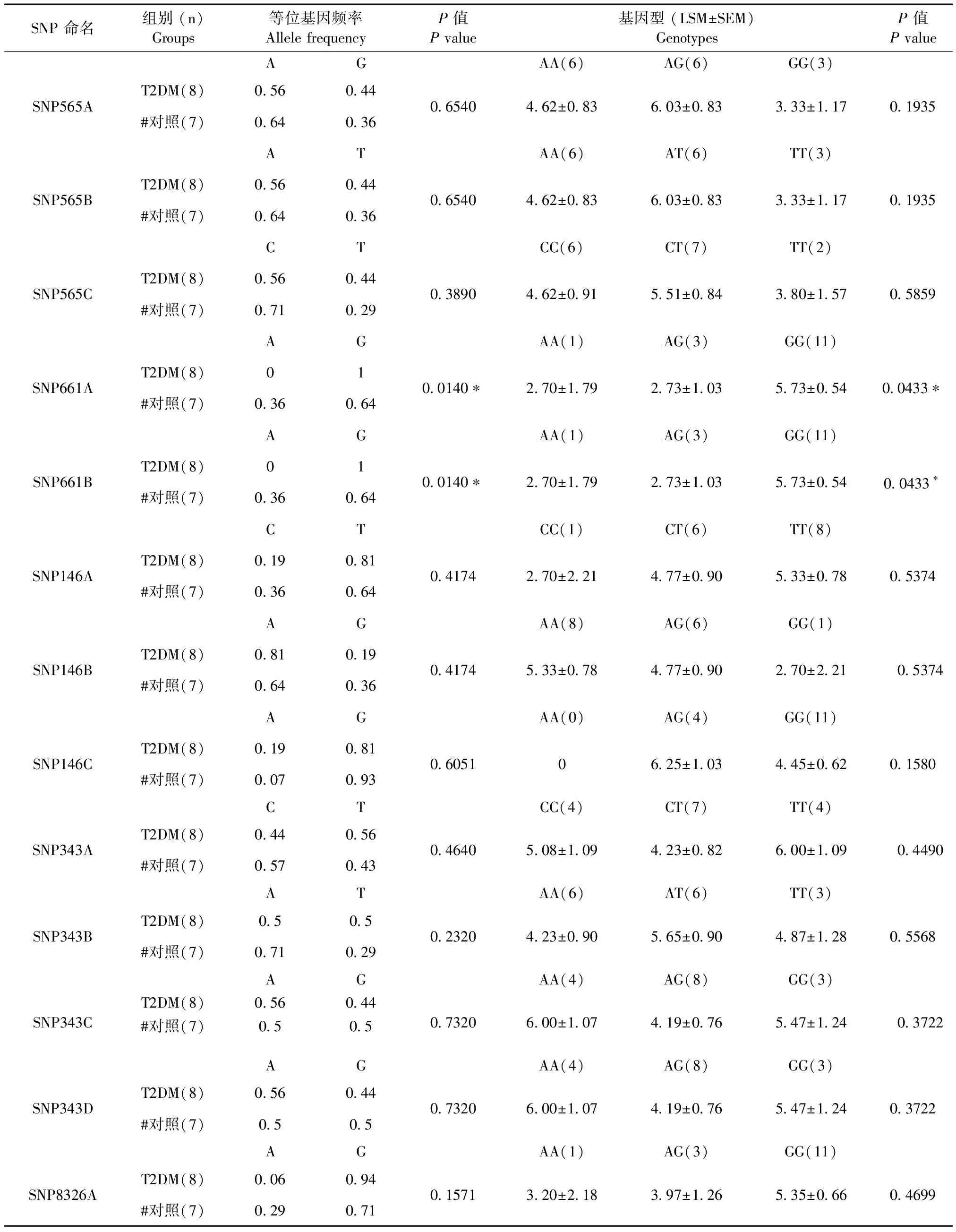
表4 SNP位点等位基因频率在金杰康自发组中的分布及其基因型与空腹血糖的关联性
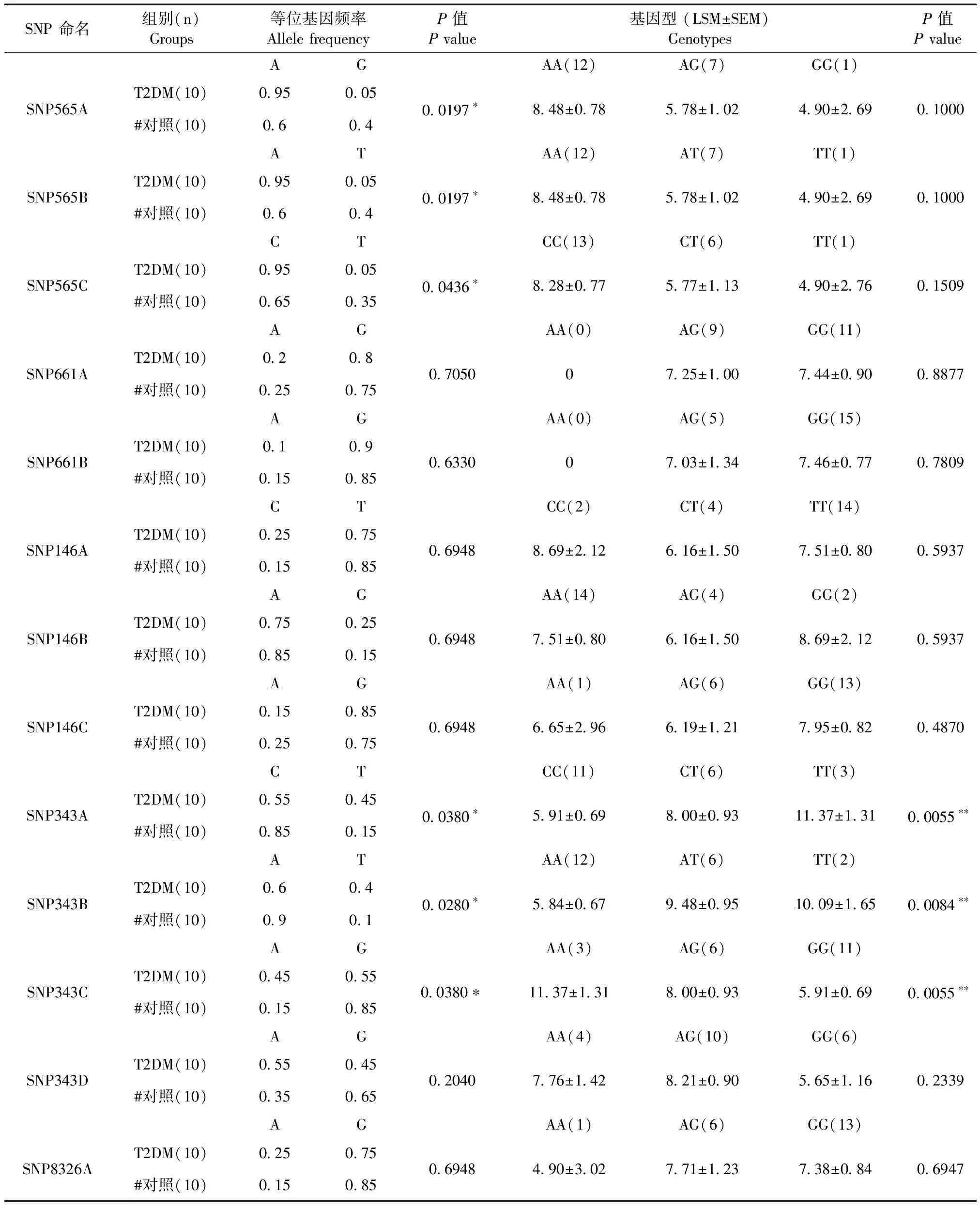
表5 SNP位点等位基因频率在蓝岛自发组中的分布及其基因型与空腹血糖的关联性

表6 SNP位点等位基因频率在所有自发个体中的分布及其基因型与空腹血糖的关联性
3 讨论
由于T2DM及其并发症的迅速蔓延已经成为严重威胁人类健康的世界性难题,为了找到能有效防治糖尿病及其并发症的药物或方法,相关动物模型则是必不可少的重要工具[19]。研究发现食蟹猴T2DM在发病前也经过类似人类T2DM的胰岛素抵抗、糖代谢异常、高胰岛素血症等阶段。在所有的实验动物中非人灵长类的血糖值最符合人的血糖值范围[16]。另外,人类T2DM患者中90%都发生了胰岛淀粉化,非人灵长类动物与人类T2DM临床病理特征最相似的地方就是胰岛淀粉样沉积,多种非人灵长类动物(恒河猴、黑猴、食蟹猴以及豚尾猴等)自发T2DM都具有这一特征,又因食蟹猴来源广泛,并且具有繁殖周期短,性格温顺,用药量少等优势,因此,食蟹猴T2DM模型成为T2DM病因、病理研究比较理想的动物模型之一[17]。
相对于其他动物,食蟹猴的遗传背景与人的更接近[20],已测的食蟹猴的基因组的数据显示,它的基因组与人的同源性达约94%。因此从理论上来说,食蟹猴与人之间具有更多共同的T2DM相关基因,本项目基于人类已有的T2DM易感突变位点,采用基因关联分析等方法在食蟹猴基因组对应区域筛查与食蟹猴T2DM相关的遗传标记是可行的。
Rs10811661位于易感基因CDKN2A/CDKN2B上游124 kb左右,在法国人群[21]和中国汉族人群[22]中都证实了rs10811661位点与2型糖尿病具有很强的关联性,同时rs10811661中T等位基因在韩国人群中也证实了与2型糖尿病的紧密相关性(OR=1.47, 95 % CI=1.23-1.75, p=2.1 × 105)[23]。CDKN2A和CDKN2B是细胞周期蛋白依赖性激酶抑制基因,位于9号染色体短臂上。CDKN2A抑制细胞周期素依赖激酶4(CDK4),而CDK4是胰腺β细胞增殖的关键调节因子。Rs10811661有T和C两种等位基因,当C突变成T时,CDKN2A/CDKN2B基因表达增多,对CDK4抑制作用增强,造成β细胞功能受损。易感基因HMGA2 rs1531343在日本人群中被证实是与2型糖尿病相关的多态性位点[24]。该位点主要影响胰岛素分泌[25],且与空腹血糖之间有较强的关联性。
本实验筛查出的SNPmf-66A、SNPmf-66B 和SNPmf-34A、SNPmf-34B、SNPmf-34C这些位点分别位于与人的T2DM易感基因rs10811661和rs1531343对应的食蟹猴基因组上同源区域内,因此,我们推测这些位点有可能是食蟹猴T2DM模型制备的遗传标记。而在诱导群体中,13个SNP的基因频率在T2DM组和对照组之间没有显著性差异,分析其可能的原因是与自发性个体相比,诱导的T2DM个体具有不同的分子遗传学机制。这些遗传标记对揭示T2DM的发病机制和开发食蟹猴T2DM模型提供一种新的策略。
参考文献:
[1] Kahn SE. The relative contribution of insulin resistance and beta-cell dysfunction to the pathophysiology of type 2 diabetes [J]. Diabetologia, 2003, 46 (76):3-19.
[2] King H, Aubert RE, Herman WH. Global burden of diabetes, 1995-2025: prevalence, numerical estimates, and projections [J]. Diabetes Care , 1998,21(35):1414-1431.
[3] Zimmet P, Alberti KG, Shaw J. Global and societal implications of the diabetes epidemic [J]. Nature , 2001, 414 (953):782-787.
[4] Saini V. Molecular mechanisms of insulin resistance in type 2 diabetes mellitus [J]. World J Diabetes, 2010, 1 (3):68-75.
[5] Sladek R, Rocheleau G, Rung J, et al. A genome-wide association study identifies novel risk loci for type 2 diabetes [J]. Nature, 2007, 445(59):881-884.
[6] Chauhan G, Spurgeon CJ, Tabassum R, et al. Impact of common variants of PPARG, KCNJ11, TCF7L2, SLC30A8, HHEX, CDKN2A, IGF2BP2, and CDKAL1 on the risk of type 2 diabetes in 5,164 Indians [J]. Diabetes , 2010, 59(651):2068-2074.
[7] Scott LJ, Mohike KL, Bonnycastle L, et al. A genome-wide association study of type 2 diabetes in Finns detects multiple susceptibility variants [J]. Science, 2007, 316(4):1341-1345.
[8] Voight BF, Scott LJ, Steinthorsdottir V, et al. Twelve type 2 diabetes susceptibility loci identified through large-scale association analysis [J]. Nat Genet, 2010, 42(23):579-589.
[9] Wan YL, Zhang YC, Peng BL, et al. Screening of spontaneous diabetes mellitus in middle-old-aged cynomolgus monkey [J]. Zool Res, 2010, 32(9):307-310.
[10] Kong A, Steinthorsdottir V, Masson G, et al. Parental origin of sequence variants associated with complex diseases [J]. Nature, 2009, 462(153):868-874.
[11] Villareal DT, Robertson H, Bell GI, et al. Parental origin of sequence variants associated with complex diseases [J]. Nature, 2009, 462(753):868-874.
[12] Zeggini E, Scott LJ, Saxena R, et al. Meta-analysis of genome-wide association data and large-scale replication identifies additional susceptibility loci for type 2 diabetes [J]. Nat Genet, 2008, 40(334):638-645.
[13] Unoki H, Takahashi A, Kawaguchi T, et al. SNPs in KCNQ1 are associated with susceptibility to type 2 diabetes in East Asian and European populations [J]. Nature Genet, 2008, 40(924):1098-1102.
[14] Yasuda K, Miyake K, Horikawa Y, et al. Variants in KCNQ1 are associated with susceptibility to type 2 diabetes mellitus [J]. Nature Gene, 2008, 40(328):1092-1097.
[15] Sladek R, Rocheleau G, Rung J, et al. A genome-wide association study identifies novel risk loci for type 2 diabetes [J]. Nature, 2007, 445(553):881-885.
[16] 牟道华,谭正怀.II型糖尿病动物模型研究进展 [J].四川生理科学杂志, 2010, 32(4):172-174.
[17] 万玉玲,刘晓明.自发性II型糖尿病动物模型 [J].医学综述, 2008,14(14):2175-2177.
[18] 汪志红, 张素华, 王增产. 中国汉族人群TCF7L2基因多态性与2型糖尿病的关联分析 [J].中华内分泌代谢杂志, 2009, 25(2):139-143.
[19] Prokopenko I, Langenberg C, Florez JC, et al. Variants in MTNR1B influence fasting glucose levels [J]. Nat Genet, 2009, 41(5):77-81.
[20] Saxena R, Voight BF, Lyssenko V, et al. Genome-wide association analysis identifies loci for type 2 diabetes and triglyceride levels [J]. Science, 2007, 316(475):1331-1336.
[21] Rane SG, Dubus P, Mettus RV. Loss of Cdk4 expression causes insulin-deficient diabetes and Cdk4 activation results in beta-islet cell hyperplasia [J]. Nat Genet, 1999, 22:44-52.
[22] Wen J, Tina R, Anders O. Investigation of type 2 diabetes risk alleles support CDKN2A/B, CDKAL1, and TCF7L2 as susceptibility genes in a Han Chinese cohort [J]. Nat Genet, 2010, 5:354-362.
[23] Marzo N, Mora C, Fabregat ME. Pancreatic islets from cyclin-dependent kinase 4/R24C knock-in mice have significantly increased beta cell mass and are physiologically functional, indicating that Cdk4 is a potential target for pancreatic beta cell mass regeneration in type 1 diabetes [J]. Diabetologia, 2004, 47:686- 694.
[24] Toshihiko O, Minoru I, Shiro M. Association of new loci identified in European Genome-Wide Association Studies with susceptibility to type 2 diabetes in the Japanese [J]. Nat Genet, 2004, 24:218-225.
[25] Wan YL. Screening of spontaneous diabetes mellitus in middle-old-aged cynomolgus monkey [J]. Zool Res, 2010, 32 : 307-310.

