玉米精氨酸甲基转移酶蛋白家族生物信息学分析
钱叶雄, 徐士杰, 张亚男, 张梦飞
安徽师范大学生命科学学院,安徽芜湖241000
玉米精氨酸甲基转移酶蛋白家族生物信息学分析
钱叶雄, 徐士杰, 张亚男, 张梦飞
安徽师范大学生命科学学院,安徽芜湖241000
蛋白质精氨酸甲基转移酶(protein argininemethyltransferases,PRMTs)是真核生物基因组中重要的表观遗传调控因子之一,主要参与组蛋白精氨酸位点甲基化修饰,改变真核基因组的染色质结构,对基因的表达进行调控。本研究鉴定了8个玉米的PRMT蛋白序列,通过与两种模式植物(拟南芥和水稻)的全部PRMT蛋白序列的同源比对和系统发生关系分析,确定玉米PRMT蛋白主要分布在3个不同的亚家族中。运用生物信息学的方法和软件预测和分析了全部玉米PRMT蛋白氨基酸序列的理化性质、信号肽、跨膜结构域、疏水性和亲水性,以及蛋白质二级及三级结构等重要参数。这些数据对后续鉴定玉米PRMT蛋白的功能具有重要的意义。
玉米;组蛋白甲基化;PRMT结构域
在真核生物基因组中,核 DNA与组蛋白(H1、H2A、H2B、H3和H4)缠绕形成核小体,然后DNA⁃蛋白质复合物进一步压缩形成高一级的染色质。核小体的核心通常是由八聚体核心组蛋白构成,其尾部常发生甲基化、乙酰化、磷酸化、ADP核糖基化和泛素化等多种翻译后修饰[1,2],这些修饰共同组成了“组蛋白密码”(histone code)。这些组蛋白修饰不仅可以影响和改变基因组染色质的状态,而且调控基因表达的转录过程[3],因此,成为近年来表观遗传学研究的热点之一。
研究表明,组蛋白修饰具有重要的生理作用。其中,组蛋白甲基化修饰主要是通过不同的组蛋白甲基转移酶催化,将甲基添加在核心组蛋白H3和H4的赖氨酸和精氨酸残基上。其中,精氨酸位点的甲基化主要发生在其尾部H3⁃R2/R17/R26和H4⁃R3等位点上,这些位点的修饰通常发生在靶基因的启动子区,对基因表达起激活作用[3]。精基酸甲基转移酶属于甲基转移酶基因家族编码的蛋白质,利用S⁃腺苷⁃甲硫氨酸(Ado⁃MeT)作为甲基供体,在精氨酸侧链的氮原子上发生甲基化修饰[4]。精氨酸甲基转移酶依据生物化学特性不同通常可被分为 4类[5]:第一类PRMT蛋白修饰精氨酸侧链的ω位点的N原子,通常形成单甲基(monomethy⁃larginine,MMA)和不对称双甲基 (asymmetric dimethylarginine,aDMA)两种类型;第二类PRMT蛋白也可以修饰精氨酸侧链的ω位点的N原子,通常形成单甲基和对称型双甲基(symmetric dimethylarginine,sDMA);第三类PRMT蛋白仅能修饰精氨酸侧链的ω⁃N形成单甲基;第四类PRMT蛋白通常修饰精氨酸侧链的δ位点的N原子形成单甲基[6,7]。
目前,关于植物精氨酸甲基转移酶的研究报道 较 少。 拟 南 芥 中 对 AtPRMT5[8~10]和AtPRMT10[11]的功能已有研究,它们对拟南芥开花时间有重要调控作用。AtPRMT5属于第二类型精氨酸甲基转移酶,体外可催化对称型双甲基化组蛋白H4R3(H4R3me2s)和非组蛋白 MBP(myelin basic protein)。对T⁃DNA插入突变体的分析结果显示,AtPRMT5通过下调重要的MADS⁃Box开花抑制基因FLC(FLOWERING LOCUS C)的表达来促进开花。AtPRMT10是一个第一类型蛋白精氨酸甲基转移酶,体外主要催化非对称型双甲基化组蛋白H4R3(H4R3me2a)和非组蛋白MBP。另外,保守结构域“double E”loop中的两个Glu对AtPRMT10的甲基转移酶活性是必需的。T⁃DNA插入突变体的表型分析发现,AtPRMT10主要通过抑制FLC的表达来促进拟南芥开花,并且在抑制FLC表达上与AtPRMT5是各自独立的。此外,在对拟南芥的研究中,还揭示了蛋白质精氨酸甲基转移酶SKB1在植物耐受盐胁迫和生长发育过程中起重要作用,SKB1的功能缺失导致拟南芥对盐胁迫的超敏感、生长迟缓及晚花等缺陷。SKB1的突变导致了植物丧失协调盐胁迫耐受和生长发育(比如开花时间)的能力,使植物一直处于胁迫响应状态而限制了植物的生长过程。
玉米作为当今世界的三大重要粮食作物之一,同时也是作为当前植物功能基因组研究的重要单子叶模式植物之一,对植物分子生物学的研究有着重要的意义。然而,当前关于玉米组蛋白甲基转移酶的鉴定及其功能研究,国内外文献尚少有报道。因此,在本研究中,我们通过对玉米最新测序的B73全基因组数据库搜索,利用生物信息学的方法,对玉米精氨酸甲基转移酶基因家族进行了鉴定与比较分析,通过对玉米精氨酸甲基转移酶基因家族成员的基本信息的分析,为后续揭示玉米组蛋白甲基化调控机制奠定了重要的理论基础。
1 材料与方法
1.1 玉米PRMT蛋白家族成员的鉴定
分别从拟南芥基因组数据库TAIR(http://www.arabidopsis.org)和玉米基因组库(http://www.maizesequence.org/index.html)中获取拟南芥和玉米的全基因组序列。利用基于隐马尔科夫模型的HMMER程序[12]搜索玉米中包含的PRMT结构域的候选序列,首先从Sanger中心数据库中选取PRMT结构域的氨基酸序列(PF05185),再利用Blastp程序,P⁃value设为10-4,对玉米全基因组蛋白质数据库进行搜索,寻找玉米基因组中所有的候选含PRMT蛋白。对搜索到的所有候选序列利用在线分析工具(http://Pfam.sanger.ac.uk)[13]进行验证,把没有显示PRMT结构域蛋白的序列删除,同时获取结构域在染色体上的位置信息,删除重复序列。
1.2 玉米PRMT蛋白一级结构及其相关物理特性的预测与分析
从玉米 PRMT结构域蛋白序列库(Maize PRMT Domain Protein Sequence Database)中获取玉米PRMT结构域蛋白序列。利用在线工具(http://Pfam.sanger.ac.uk)确定各PRMT结构域蛋白中所包含的结构域类型。借助 Signal P Server v.4.1[14,15]和TMHMM Server v[16]软件分别预测玉米PRMT蛋白的信号肽序列和跨膜结构域,进一步通过哈佛大学专业的蛋白质定位在线分析工具EuK⁃mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/euk⁃multi/)对获得的8条候选蛋白序列进行亚细胞定位分析,以此判断出精氨酸甲基转移酶大致的作用方式和作用途径。然后,利用Prot Scale软件[17]进一步分析玉米PRMT蛋白氨基酸序列的疏水性/亲水性,判断出精氨酸甲基转移酶在细胞内的分布情况。
1.3 玉米PRMT蛋白二级结构与三级结构的预测与分析
利用SOPMA软件对玉米PRMT蛋白的二维结构进行预测与分析[18],分析总结PRMT中含有的二级结构种类和各自所占比例。利用同源建模工具 SWISS⁃MODEL(http://swissmodel.expasy.org/)完成蛋白质三级结构的预测和分析工作,再用SWISS⁃PdbViewer工具显示玉米PRMT结构域的3D结构[19~21]。
1.4 玉米PRMT基因在染色体上的定位
利用MAP[22]染色体定位软件,对整理出来的染色体信息进行处理,获得PRMT结构域基因在染色体上的定位图。
1.5 玉米PRMT蛋白系统发生分析
利用ClustalW[23]对预测出的PRMT蛋白序列集进行多序列比对分析。利用MEGA(版本5.0)[24]生成拟南芥与玉米中PRMT的无根系统进化树。
2 结果与分析
2.1 玉米PRM T蛋白家族成员的鉴定
利用玉米的基因库得到玉米的全基因组序列,通过删选,筛除不具有PRMT结构域的蛋白。最终得到8条玉米PRMT氨基酸序列,以PRMT结构域在染色体上的位置命名(表 1)。玉米PRMT中有1个氨基酸序列的等电点较高(占12.5%),在家族Ⅱ中。大部分的PRMT结构域位于PRMT结构域蛋白的前端,少数位于后部。玉米PRMT蛋白中最长的氨基酸残基数有653个,最短的氨基酸残基数有306个。其中,PRMT结构域能够表现出精氨酸甲基转移酶的功能。
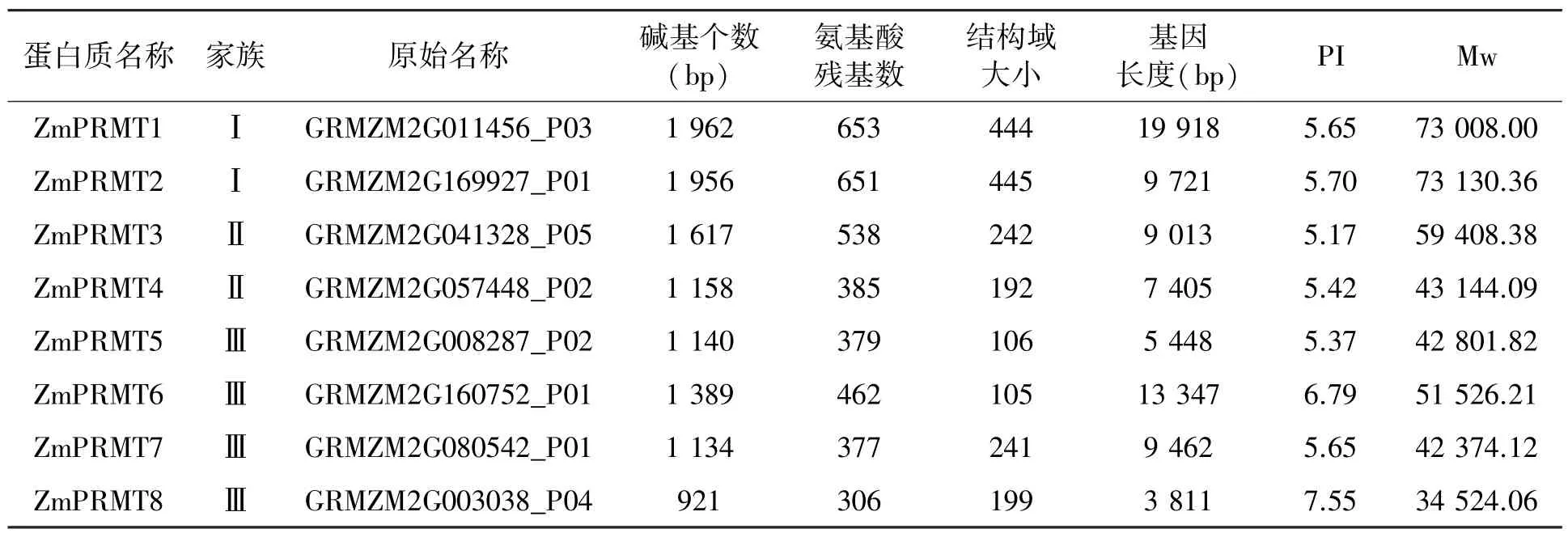
表1 玉米ZmPRMTs蛋白家族的基本信息Table 1 Basic information about ZmPRMTs in maize.
2.2 玉米PRMT蛋白一级结构域分析
通过在线工具(http://pfam.janelia.org/search/sequence)确定了8个PRMT蛋白中所包含的结构域类型和各种结构域在PRMT各蛋白家族亚族中的分布(图1)。家族Ⅰ中,保守结构域只有PRMT5结构域;家族Ⅱ的保守结构域只有甲基转移酶(methyltransferase)结构域;家族Ⅲ中的保守结构域只有PRMT5,但其位置靠前,且根据后来的分析结果将其分在一类。PRMT5结构域是PRMT蛋白中的特征结构域,与组蛋白的精氨酸作用位点结合催化组蛋白的甲基化过程。
2.3 玉米PRMT蛋白物理特性分析
通过SignalP Server v.4.1软件预测了8个玉米PRMT蛋白的信号肽序列,结果表明,全部PRMT蛋白的信号肽原始剪切位点的最高得分值(Y⁃score maximum)均偏低,S平均值(mean S⁃score)小于0.5,位于0.1~0.2之间,表明玉米PRMT蛋白可能不属于分泌类型蛋白,不具有信号肽酶切位点以及信号肽序列。因此,推测该蛋白在细胞内不进行跨膜转运,可能直接锚定细胞质基质中发挥催化甲基化转移的功能。
利用TMHMM Server v.软件预测了全部8个玉米ZmPRMT蛋白的跨膜结构域,结果表明,玉米ZmPRMT蛋白均无跨膜区域,与水稻中报道的OsMET1蛋白研究结果相一致[16],这一结果与上述玉米PRMT蛋白无信号肽的预测结果相吻合。
进一步通过专业的蛋白质定位软件 Euk⁃mPLoc对8条候选序列进行亚细胞定位分析发现,除了ZmPRMT8可能为分泌性蛋白之外,其余7条蛋白均分布于细胞质基质或一些亚细胞器内蛋白。因此,可以推测在玉米细胞之中合成的PRMT蛋白一般不进行跨膜转运,不离开细胞质基质,以锚定细胞骨架的形式或暂时性储藏在一些亚细胞器当中行使催化功能。

图1 玉米ZmPRMTs蛋白结构域分布图Fig.1 Structuremap of ZmPRMTs domain inmaize.
2.4 玉米PRM T疏水性/亲水性的预测和分析
通过Prot Scale软件对玉米PRMT蛋白氨基酸序列的疏水性/亲水性进行了预测和分析,其中较高正值的氨基酸具有较强的疏水性,而较低负值的氨基酸则具有较强的亲水性[25]。结果如图2所示,玉米ZmPRMT1多肽链中氨基酸多小于零,亲水性最强的氨基酸为第10位的赖氨酸(Lys),其分值最低(-2.522);疏水性最强的氨基酸为第36缬氨酸(Val),其分值最高(2.067),整个多肽链大多数氨基酸均表现为亲水性,只有少数氨基酸表现为疏水性。其余7条ZmPRMT蛋白序列的预测结果与ZmPRMT1相似,这一结果进一步证明了玉米ZmPRMT蛋白缺乏疏水性的跨膜结构域而不具有跨膜功能。

图2 玉米ZmPRMT1蛋白家族的疏水性/亲水性的预测图Fig.2 Hydrophobic and hydrophilic prediction map of ZmPRMT1 inmaize.
2.5 玉米PRMT蛋白二级与三级结构的预测与分析
利用SOPMA软件对玉米PRMT蛋白的氨基酸序列的二维结构进行了预测与分析,结果见表2。表2中列出了玉米PRMT的整体二级结构由螺旋、折叠、转角和无规卷曲四种成分构成,其中螺旋和无规卷曲所占比例最大,折叠所占比例较小,转角所占比例最小。由此可以推断,α⁃螺旋和无规卷曲是植物PRMT蛋白整体结构的主要结构元件,β⁃折叠和转角结构零星分布在整个蛋白质中。
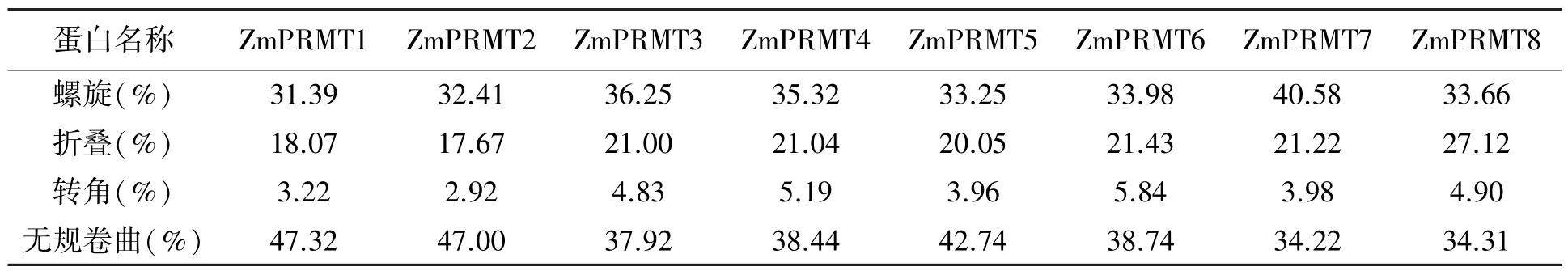
表2 玉米ZmPRMTs蛋白二级结构信息Table 2 Secondary structure information of ZmPRMTs in maize.
在预测了玉米PRMT二级结构的基础上,利用同源建模工具SWISS⁃MODEL将8条候选蛋白序列分别与PDB数据库中已知的PRMT蛋白序列进行比对分析,再通过SWISS⁃Pdb Viewer工具对待测蛋白质的三维结构进行模拟分析。候选蛋白ZmPRMT1的模型评估分析图(图3,彩图见封三图版)表明,绿色合适区域较多,说明了模建的空间结构合适,模建的正确性较高。其余7条蛋白序列的模建评估分析结果与ZmPRMT1基本一致。对玉米PRMT蛋白结构域(2~593)以来自人类精氨酸甲基转移酶5(4gqbA)为模板建模,三维结构预测结果见图4(彩图见封三图版)。模建的玉米PRMT1与模板4gqbA之间拓扑等价残基的Cα距离均方根差(RMS)为2.06Å,结构总能量为-13 680.44 kJ/mol,表明模建形成的蛋白质三维结构处于能量最低,结构最稳定状态,说明模建的PRMT结构域三维结构合理。由此推断,植物PRMT蛋白是一个在组蛋白甲基化中起作用的功能蛋白。

图3 玉米ZmPRMT1蛋白模型评估分析图Fig.3 Model assessmentmap of ZmPRMT1 in maize.
2.6 玉米PRM T系统发生分析
通过利用MEGA5.0对搜索到的蛋白质全序列进行多序列比对,用邻接法构建无根系统进化树(图5),其中包括7条拟南芥PRMT(AtPRMT)的氨基酸序列、5条水稻PRMT(OsPRMT)的氨基酸序列和8条玉米PRMT(ZmPRMT)氨基酸序列。根据拟南芥、水稻和玉米PRMT结构域差异性和系统进化的比对分析,发现 ZmPRMT1、ZmPRMT2与AtPRMT5和OsPRMT708的同源性较高,三者属于同源基因;ZmPRMT3、ZmPRMT4与AtPRMT13、AtPRMT14和OsPRMT702保持相对较高的同源性;ZmPRMT5、ZmPRMT6、ZmPRMT7和ZmPRMT8进化上归为一个亚类,其中 ZmPRMT5和 ZmPRMT6与 AtPRMT17和 OsPRMT710在进化上很相似,属于同源基因,ZmPRMT8和 OsPRMT703是同源基因,且与AtPRMT11和AtPRMT12同源性较高。
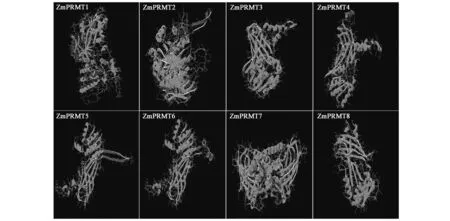
图4 玉米ZmPRMTs蛋白三维预测图Fig.4 Three⁃dimensional predictionmap of ZmPRMTs in maize.

图5 拟南芥、水稻和玉米PRMT的邻接法系统发生树Fig.5 Neighbor⁃joining phylogenetic tree of PRMT in Arabidopsis,rice and maize.
2.7 玉米PRMT基因在染色体上的分布
根据玉米PRMT基因家族成员的位置信息,将8个PRMT基因在染色体上标示出来,见图6。玉米PRMT结构域并不是均匀分布在染色体上的,第7条染色体上最多,有3个PRMT结构域;第1、第2、第4、第5、第6和第10条染色体上有2个PRMT结构域;第3、第8和第9条染色体上都只有1个PRMT结构域。PRMT结构域在每条染色体上的分布也不是均匀的。

图6 玉米ZmPRMTs结构域的染色体定位图Fig.6 Chromosome location map of ZmPRMTs domain in maize.
3 讨论
在已报道的拟南芥PRMT蛋白中,很多基因的功能已经被确定。基于联配好的蛋白质序列,利用MEGA5.0[24]生成的拟南芥和玉米的系统发育树中,显示出拟南芥与玉米的PRMT在进化过程中存在很多同源基因。譬如,PRMT蛋白亚族Ⅰ中ZmPRMT1、ZmPRMT2,与AtPRMT5具有较高的同源性。研究结果表明,AtPRMT5的缺失会导致大量的mRNA前体的拼接出现异常,而这些mRNA参与植物生长发育的多个过程,如非生物刺激响应、光合作用和温度响应等[9,10]。以开花时间调节为例,在atprmt5突变体中,开花调节基因FLK的异常拼接会导致其正常功能转录本的减少和蛋白质水平的下降,从而造成FLC的上调以及晚花的表型。由此可知,AtPRMT5通过调控植物生命周期各个阶段中mRNA前体的正确加工,保证了植物正常的生长发育过程。玉米的PRMT中存在很多与拟南芥PRMT的同源基因。由于结构与功能相适应的特点,不难看出,玉米和拟南芥中PRMT中存在的同源基因很可能具有相同或相似的生理功能。但在进化的过程中,由于遗传、环境因素、人为因素的差异导致基因在进化过程中具有不定向性。进一步通过序列比对分析发现,有些被鉴定的玉米的PRMT蛋白成员无法在拟南芥中找到其同源的基因。此外,在拟南芥中有的PRMT成员目前分类尚未明确,其功能也尚未得到验证,因此,与其同源的部分玉米PRMT基因的功能目前也无法通过序列比对来预测。同时,由于我们在对玉米PRMT序列搜索时可能不够全面或者在删除重复序列时判断标准有争议,导致可能有些玉米PRMT未被列入,有待进一步地研究。
植物表观遗传调控因子的发掘对揭示植物表观遗传调控机理的研究有着重要的意义,也是当前植物表观遗传学研究的重要热点之一。在本研究中,通过对玉米最新测序的B73全基因组数据库搜索,利用生物信息学的方法,鉴定和比较分析了玉米精氨酸甲基转移酶(PRMT)蛋白家族,获得了玉米PRMT蛋白成员的一些重要的基本信息,掌握了玉米PRMT蛋白的一些重要的结构特征,为后续进一步地研究其功能提供了重要的理论依据。与此同时,随着一些重要的植物表观遗传调控因子的相继发掘和功能验证,将为后续进一步地揭示植物表观遗传调控机理提供了重要的理论基础。
[1] Holliday R.DNA methylation and epigenetic defects in carcinogenesis[J].Mutat.Res.,1987,181(2):215-217.
[2] Egger G,Liang G,Aparicio A,et al..Epigenetics in human disease and prospects for epigenetic therapy[J].Nature,2004,429(6990):457-463.
[3] 谢萍,田春艳,张令强,等.组蛋白甲基转移酶的研究进展[J].遗传,2007,29(9):1035-1041.
[4] Turner B M.Cellularmemory and the histone code[J].Cell,2002,111(3):285-291.
[5] Peterson C L,Laniel M A.Histones and histone modifications[J].Curr.Biol.,2004,14(14):546-551.
[6] Margueron R,Trojer P,Reinberg D.The key to development:interpreting the histone code[J].Curr.Opin.Genet.,2005,15(2):163-176.
[7] Metivier R.Estrogen receptor⁃alpha directs ordered,cyclinal and combinatorial recruitment of cofactor on a natural target promoter[J].Cell,2003,115(2):751-763.
[8] Niewmierzycka A,Clarke S.S⁃Adenosylmethionine⁃dependent methylation in Saccharomycescerevisiae.Identification of a novel protein argininemethyltransferase[J].J.Biol.Chem.,1999,274:814-824.
[9] Pei Y,Niu L,Lu F,et al..Mutations in the TypeⅡ protein arginine methyltransferase AtPRMT5 result in pleiotropic developmental defects in Arabidopsis[J].Plant Physiol.,2007,144:1913-1923.
[10] Wang X,Zhang Y,Ma Q,et al..SKB1⁃mediated symmetric dimethylation of histone H4R3 controls flowering time in Arabidopsis[J].The EMBO J.,2007,26:1934-1941.
[11] Niu L,Lu F,Pei Y,Liu C,et al..Regulation of flowering time by the protein argininemethyltransferase ATPRMT1O[J].EMBO Rep.,2007,8(12):1190-1195.
[12] Eddy SR.Profile hidden Markov models[J].Bioinformatics,1998,14(9):755-763.
[13] Finn R D,Mistry J,Schuster⁃Bockler B,et al.Pfam:clans,web tools and services[J].Nucleic Acids Res.,2006,34:D247-251.
[14] Nielsen H,Engelbrencht J,Brunak S,et al..Identification of prokaryotic and eukaryotic signal peptides and prediction of their cleavage site[J].Protein Eng.,1997,10(1):1-6.
[15] Bentsen JD,Nielsen H,von Heijine G,et al..Prediction of signal peptides:SignalP 3.0[J].J.Mol.Biol.,2004,340:783-795.
[16] Yamauchi T, Moritoh S, Johzuka⁃Hisatomi Y, et al..Alternative splicing of the rice OsMET1 genes encoding maintenance DNA methyltransferase[J].J.Plant Physiol.,2008,165(17):1774-1782.
[17] Kyte J,Doolittle R F.A simple method for displaying the hydropathic character of a protein[J].J.Mol.Biol.,1982,157(6):105-132.
[18] Geourjon C,Deleage G.SOPMA:significant improvement in protein secondary structure prediction by consensus prediction from multiple alignments[J].Comput.Appl.Biosci.,1995,11(6):681-684.
[19] Arnold K,Bordoli L,Kopp J,et al..The SWISS⁃MODEL Workspace:a web⁃based environment for protein structure homologymodeling[J].Bioinformatics,2006,22:195-201.
[20] Schwede T,Kopp J,Guex N,et al.SWISS⁃MODEL:an automated protein homology⁃modeling server[J].Nucleic Acids Res.,2003,31:3381-3385.
[21] Guex N,Peitsch M C.SWISS⁃MODEL and the Swiss⁃PdbViewer:an environment for comparative protein modelling[J].Electrophoresis,1997,18:2714-2723.
[22] 郭安源,朱其慧,陈 新,等.GSDS:基因结构显示系统[J].遗传,2007,29(8):1023-1026.
[23] Larkin M A,Blackshields G,Brown N P,et al..ClustalW and Clustal X version 2.0[J].Bioinformatics,2007,23(21):2947-2948.
[24] Tamura K, Peterson D, Peterson N, et al.MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Mol.Biol.Evol.,2011,28:2731-2739.
[25] 吴春太,李维国,高新生,等.植物DNA甲基转移酶的生物信息学分析[J].西南大学学报:自然科学版,2010,32(4):83-89.
Bioinformatics Analysis of PRMT Protein Fam ily in M aize
QIAN Ye⁃xiong,XU Shi⁃jie,ZHANG Ya⁃nan,ZHANG Meng⁃fei
College of Life Sciences,Anhui Normal University,AnhuiWuhu 241000,China
Protein arginine methyltransferases,PRMTs,are one of important epigenetic regulating factors,which are involved in modifying histone arginine methylation,changing chromatin structure and regulating gene expression in eukaryotic genome.In this study,a complete set of 8 PRMT proteins were identified in maize genome.By contrasting with the PRMT protein sequences in Arabidopsis and rice,all 8 ZmPRMT proteins were categorized into three classes based on phylogeny in maize.By using bioinformatics methods and softwares,physical and chemical of amino acid sequence of allmaize PRMT proteinswere analyzed,and their signal peptide,transmembrane domain,hydrophobic and hydrophilic structure,protein secondary and tertiary structure were also determined in this study.These results revealed a comprehensive overview of the maize PRMT protein family and provided a great significance on subsequent functional research of PRMT proteins in maize.
maize;histonemethylation;PRMT domain
10.3969/j.issn.2095⁃2341.2014.01.05
2013⁃09⁃13;接受日期:2013⁃11⁃04
中国博士后科学基金项目(2012M521212);安徽省自然科学基金项目(1308085MC44);安徽省高校省级自然科学研究重点项目(KJ2013A132)资助。
钱叶雄,副教授,博士,研究方向为玉米表观遗传学与生物信息学。E⁃mail:qyx2011@mail.ahnu.edu.cn

