腹泻症候群致泻性大肠埃希氏菌血清型及毒力基因检测
慕毅敏 蔡旭明 周敬纲 蔡旭辉
致泻性大肠埃希菌生存与人和动物的肠道中,随粪便排出而污染水源、土壤、食品、器具等,人群对致泻大肠埃希菌普遍易感,根据其致病机制,致泻大肠埃希菌至少可分为5个类型:肠产毒素大肠埃希菌(ETEC)、肠致病性大肠埃希菌(EPEC)、肠侵袭性大肠埃希菌(EIEC)、肠出血性大肠埃希菌(EHEC)、肠集聚性黏附性大肠埃希菌(EAEC)。其中,ETEC是发展中国家婴幼儿及旅游者腹泻的主要病原,EPEC是婴儿腹泻的主要病原,EIEC主要引起较大儿童和成人腹泻[1-2]。为了全面了解本地区腹泻症候群中致泻大肠埃希菌的感染情况,查清致泻大肠埃希菌的血清群、型分布规律以及所携带毒力基因[3-6],为腹泻病的防治对策提供科学依据[7-8],现将2012-2013年本地区腹泻症候群中致泻大肠埃希菌的监测检验情况报告如下。
1 材料与方法
1.1 标本来源 本地区2012-2013年腹泻症候群监测医院门诊未使用过抗生素腹泻患者的稀便、水样便、黏脓便或脓血便等。标本总计719份,其中7岁以下婴幼儿618份,7岁以上儿童及成人101份。
1.2 标本的分离程序
1.2.1 分离 用无菌拭子采集少量粪便直接接种麦康凯(MAC)琼脂平板和O157显色平板上,温度(36±1)℃,培养18~24 h。
1.2.2 增菌 用无菌拭子采集少量粪便接种mEC肉汤增菌液,(36±1)℃,培养6 h后,取1 mL用O157免疫磁珠进行富集后,接种于O157显色平板和MAC平板18~24 h。
1.2.3 挑取可疑菌落 挑取MAC琼脂平板上发酵乳糖的桃红色、不透明的可疑菌落或O157显色平板上紫红色或淡紫色、中等大小菌落进行生化初筛实验。
1.2.4 生化初筛 凡是TSI斜面产酸或不产酸、底层产酸、不产生H2S的菌株,进行IMViC实验,结果为++--或-+--,尿素-。
1.2.5 多重PCR 通过对主要致泻大肠埃希菌目的基因多重PCR扩增片段,判断扩增阳性结果。
1.2.6 血清型鉴定 挑取经PCR试验证实为致泻性大肠埃希菌的纯培养物,用相应致泻大肠O多价、O单价、K、H诊断血清进行血清玻片凝集试验。
1.2.7 结果判定 综合生化试验、多重PCR试验和血清型分型试验进行鉴定和报告。
2 结果
2.1 一般情况 本次检测分离到血清凝集致泻大肠埃希菌65株,占所检标本总数的9.04%(65/719),其中7岁以下婴幼儿59株,7岁以上儿童及成人6株;而未分血清型致泻大肠埃希菌27株,占检测标本的3.75%(27/719),其中7岁以下婴幼儿24株,7岁以上儿童及成人3株;其中EPEC检出率为10.15%(73/719),毒力基因检测以escV为主,占8.90%(64/719)。
2.2 65株血清凝集致泻性大肠埃希菌的血清分型 分离出的EPEC、EIEC、ETEC共分8种O血清型,其中EPEC分为6种不同血清型,主要血清型为O55:H59、O128:H67;EIEC和ETEC各分一种血清型,见表1。
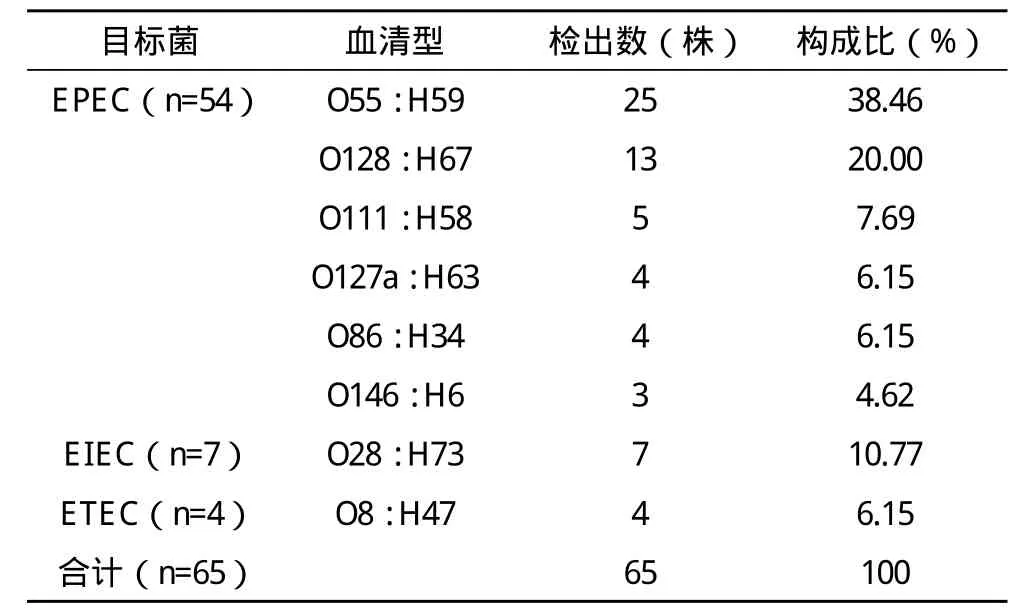
表1 65株血清凝集致泻性大肠埃希菌血清分型分布结果
2.3 65株血清分型凝集菌株毒力基因的分布情况 65株血清分型凝集菌株中,49株(75.38%)携带escV,5株(7.69%)携带escV+bfpB,7株(10.77%)携带invE,4株(6.16%)携带elt,见表2。
2.4 27株未分血清型致泻性大肠埃希菌毒力基因分布结果 检测出毒力基因共5种,分别为escV、escV+bfpB、invE、elt、astA,分属EPEC、EIEC、ETEC、EAEC,见表3。
3 讨论
大肠埃希菌感染全年均可发病,但大多发生于5-10月,其中7-9月是发病高峰。致泻性大肠埃希菌生存于人和动物的肠道中,人群普遍易感,引起急性胃炎、急性菌痢、出血性肠炎,传播因素多种多样,包括水、食品、日常生活用品、苍蝇等,还可以通过接触动物或带菌者传播[1]。
本次检测分离到血清凝集致泻大肠埃希菌65株,占所检标本总数的9.04%(65/719),而未分血清型致泻大肠埃希菌27株,占检测标本的3.75%(27/719);其中EPEC检出率为10.15%(73/719),与国内相关报道基本符合[2-3],毒力基因检测以escV为主,占8.90%(64/719);其余毒力基因escV+bfpB、invE、elt、astA与相关报道基本一致[4-5]。
在大多数实验室,致泻性大肠埃希菌(DEC)常规测定只能鉴定DEC的表型特征,然而,这些方法在鉴定5种具体的DEC是不够的[9-11]。长期以来,对于腹泻患者的细菌学检测主要是分离粪便标本中的腹泻病原菌,然而,由于大多数患者发病后很快使用抗生素,而使细菌的分离培养结果不理想。因此,传统的粪便细菌学检验方法已不能满足诊断的要求,在我国大多数微生物实验室,如果从肠道分离的菌株不与ETEC、EPEC、EIEC、EHEC、EAEC这5种大肠埃希菌的诊断血清发生凝集,就判断为肠道正常大肠埃希菌。这样做往往会失掉大部分病原菌的检出率[12-15]。近年来,多重PCR作为一种不依赖于细菌培养,适合于在细菌种群多样性体系中快速筛查疑似病原菌的新技术而广泛应用,此方法检测毒力基因更快速,敏感性高,特异性强[16-20]。

表2 65株血清分型菌株毒力基因分布

表3 27株未分血清型致泻性大肠埃希菌毒力基因分布 株(%)
因此,在腹泻患者的大肠埃希菌检测中,在传统的细菌分离培养、生化试验、血清学鉴定的基础上,有必要进行大肠埃希菌的毒力基因检测,以提高阳性检出率,避免漏检,提高致泻性大肠埃希菌的诊断。
[1]倪宏.42例小儿腹泻的临床研究[J].中国医学创新,2012,9(8):121.
[2]龚可胜.消旋卡多曲治疗48例成人急性腹泻患者的疗效观察[J].中国医学创新,2012,9(8):44-45.
[3]张英.消旋卡多曲治疗秋季腹泻的临床研究[J].中国医学创新,2012,9(22):28-29.
[4]张蔚起,张德雄,林东龙.小儿轻中度腹泻引发惊厥72例临床分析[J].中国医学创新,2012,9(16):108-109.
[5]周永明.应用灰色模型预测其他感染性腹泻和病毒性肝炎的发病趋势[J].中国医学创新,2012,9(8):159-160.
[6]时全,黄新明,李朝阳,等.致泻大肠埃希菌感染在腹泻病中地位的研究[J].中国卫生检验杂志,2005,15(1):5-8
[7]吴开宇.EPEC,EIEC,EREC与婴儿腹泻关系的研究[J].腹泻病专辑,1989,10(1):195-196.
[8]任春阳.EPEC相关毒力基因与腹泻的相关性[D].温州医学院,2007.
[9]徐义刚,崔丽春,李苏龙,等.多重PCR-DHPLC检测4种主要致泻性大肠埃希菌方法的建立于应用[J].食品科学,2010,31(20):293-297.
[10]孔海深.致泻大肠埃希菌的分子分型和流行病学研究[D].浙江大学,2011.
[11]叶菊莲,占利,程苏云,等.婴幼儿腹泻致泻大肠埃希菌PCR毒力基因检测[J].中国卫生检验杂志,2009,19(9):2055-2057.
[12]张菊玲,崔恩博.致泻大肠埃希菌的分布及耐药性研究[J].传染病信息,2012,25(3):180-182.
[13]黄芳,邓瑛,曲梅,等.2010年北京市感染性腹泻病原学监测分析[J].中华预防医学杂志,2011,45(9):820-824.
[14]齐小秋.病原生物学检验[M].卫生部病原生物学检验教材编写组,2009:305-319.
[15]杨党鹏,李苏利.腹泻病原菌的细菌培养及鉴定[J].人民军医,2001,44(2):115-117.
[16]Brandal L T,Lindstetd B A,Lena A,et al.Octaplex PCR and fluorescence-based capillary electrophoresis for identification of human diarrheagenic Escherichia coil and Shigella ssp[J].J Microbial Methods,2007,68(5):331-342.
[17]黄新蓉,刘劼,叶长芸.应用16SrDNA克隆文库筛查腹泻病爆发病原体及特异毒力基因验证[J].中国人兽共患病学报,2010,26(10):900-903.
[18]Stacy-Phipps S,Mecca J J,Weiss J B.Multiplex PCR assay and simple preparation method for stool specimens detect enterotoxigenic Escherichia coli DNA during course of infection[J].J Clin Microbial,1995,33(5):1054-1059.
[19]Rappelli P,Madddau G,Mannu F,et al,Development of a set of multiplex PCR assays for the simultaneous identification of enterotoxigenic,enteropathogenic,enterohemorrhagic and enteroinvasive Escherichia coli[J].New Microbial,2001,24(1):77-83.
[20]Vidal R,Vidal M,Lagos R,et al.Multiplex PCR for Diagnosis of Enteric Infections associated with Diarrheagenic Escherichia coli[J].Journal of Clinical Microbiology,2004,42(4):1787-1789.

