海量种群基因表达式编程的内存删冗算法
钟坚成,彭 玮
(1.湖南师范大学工程与设计学院,长沙410081;2.昆明理工大学信息工程与自动化学院,昆明650093)
海量种群基因表达式编程的内存删冗算法
钟坚成1,彭 玮2
(1.湖南师范大学工程与设计学院,长沙410081;2.昆明理工大学信息工程与自动化学院,昆明650093)
在大样本、多种群、高进化代数的情况下,基因表达式编程(GEP)容易产生冗余个体染色体有效串,从而影响计算性能。为解决该问题,提出一种基于内存检测种群冗余的算法MPRRGEP。分析单基因、多基因对种群冗余性的影响,设计个体染色体有效性的测度方法。提出内存Hash种群映射删冗算法,在内存中索引个体染色体数据,减少相同有效串的重复计算次数,大幅提高GEP计算性能。实验结果表明,相比传统GEP算法,MPRRGEP算法平均减少60%以上的计算时间。
基因表达式编程;删冗;哈希表;基因有效串;多基因;关键蛋白质预测
1 概述
基因表达式编程(Gene Expression Programming, GEP)作为进化算法的一种[1],融合了遗传算法和遗传编程的优势。在GEP中采用一个固定长度的个体染色体可用来表达一个非线性的算术表达式、逻辑表达式。通过一系列变异、交叉、置换、重组等遗传算子操作,个体染色体可进化成一个新的个体。新个体在生存函数下进行优胜劣汰[2-3]。GEP可用于解决分类问题、公式演化问题、优化问题、关联规则挖掘等。从现有研究来看,对GEP的研究主要集中在3大方面: (1)对GEP算法框架的改进,如文献[4]运用多核CPU并行计算能力提出了基于多线程评估的GEP算法;文献[5]针对传统GEP存在局部收敛的不足提出了快速进化,避免局部最优的VPS-GEP算法;文献[6]将差分突变搜索、混沌重组和变异操作、灾变算子运用于GEP,提高了GEP的精度和收敛速度;文献[7]提出了一种新的基于多层染色体基因表达式编程的遗传进化算法M2GEP。(2)GEP与其他机器学习或统计方法结合的算法研究,如文献[8]融合模拟退火和GEP算法;文献[9]基于GEP和Baum-Welch算法训练HMM模型。(3)GEP的应用研究,如文献[10]运用GEP与滑动窗口方法来预测变压器油中溶解气体浓度;文献[11]运用GEP估算曼宁阻力系数。上述优化GEP的文献中大部分都涉及提升计算性能、提高种群多样性、扩展GEP的全局搜索能力,但从实验参数来看大部分种群数为500以下,进化次数为500以下,很少考虑大样本、多种群、高进化代数的情况,特别是海量种群数和超高进化次数的情况下GEP计算量及优化问题。目前有较多针对大样本数据的分类和聚类问题,如:关键蛋白质识别问题,复杂网络社区发掘问题等。采用多种群可提高群体的多样性,避免早熟现象,但会使得传统的GEP算法运行效率降低。进化代数为算法运行的结束条件,文献[12]采用基因表达式编程种群多样性自适应调控算法分别对一元函数发现问题和五元函数发现问题进行实验,实验1的进化次数为400次,实验2的进化次数为2 000次,实验结果表明为获得问题的最优解需要超过1 000次的进化代数。
本文探讨GEP在大种群数及多次进化环境下的GEP性能优化算法,分析个体染色体有效串,提出针对单基因、多基因个体染色体有效性长度的测度算法,并运用内存映射删冗算法将个体染色体有效串和对应的结果数据在内存中索引,减少相同有效串的重复计算次数。
2 个体染色体有效串
GEP中采用固定长度的字符串来描述个体基因,个体基因分为头部Head和尾部Tail 2个部分,在头部中可放置函数符号和终结符号,而尾部只能放置终结符号。个体基因可解析为一个表达式树(Expression Tree),函数符号后可接相应的参数,即为表达式树的中间结点,而终结符号是表达式树的叶子结点。GEP根据染色体中包含基因的个数分为单基因染色体和多基因染色体。以下是单基因染色体和多基因染色体及其对应的表达式树的例子。
设单基因染色体串为“+Q-/b*aaQ+aabaabba aab”,其头部长度为10,对应的表达式树如图1所示。

图1 单基因染色体及其表达式树
设多基因染色体串为“-b*babbab*Qb+abbba-*Qabbaba”,其基因头部长度为4,基因数目为3,对应的表达式树如图2所示。

图2 多基因染色体及其表达式树
定义1 基因有效串
基因的长度为GL:
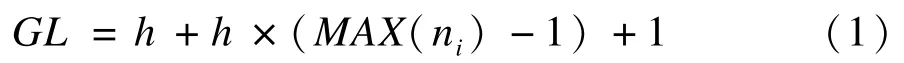
其中,h为基因头部字符个数;ni为基因头部中第i个函数符的参数个数。基因字符串中用于正确描述表达式树的前L个字符为基因有效串。基因有效串长度L小于等于基因长度GL。在图1和图2中Valid Pos所指向的位置为L。
定义2 个体染色体有效串CL

其中,GN为个体染色体中基因的个数,根据GN的取值可分为单基因和多基因个体染色体:单基因个体染色体中GN取值为1,其有效串与基因有效串相同;多基因个体染色体中GN取值大于1,个体染色体有效串为各基因有效串的连接。针对上例中单基因的个体染色体“+Q-/b*aaQ+aabaabbaaab”的有效串为“+Q-/b*aaQ+aab”。有效串后的字符,如图1中Valid Pos箭头指向后方字符发生了变更不会影响个体染色体串生成表达式树,即也不会影响样本计算后适应性函数评价的结果。针对上例中的多基因个体染色体“-b*babbab*Qb+abbba-* Qabbaba”,该染色体分为3个基因,其中每个基因都有自身的有效串,如基因1的有效串为“-b*ba”、基因2的有效串为“*Qb+ab”、基因3的有效串为“-*Qabb”,个体染色体的有效串为3个基因有效串的连接“-b*ba*Qb+ab-*Qabb”。图2中Valid Pos箭头后方到所在基因尾部字符发生了变更也不会影响个体染色体串生成表达式树。
3 内存种群冗余删冗算法MPRRGEP
染色体有效串生成表达式用于计算实验样本,当实验样本量增大时,个体表达式计算量也随之增加。个体通过适应性函数评价后,将优良的个体保留,并将其进行交叉、变异、置换等遗传算子操作,扩充成原始种群数。同时,随着GEP的种群数和进化次数增加,个体染色体有效串与隔代之间的个体染色体有效串相同的概率将随之增大。这导致基于大样本的情况下,将耗费大量CPU时间来做重复的计算工作。基于以上考虑,提出一种内存种群冗余删冗算法(Memory Population Reducing Redundant GEP,MPRRGEP),该算法包括3个部分:快速检测基因有效串,构建个体染色体有效串以及内存Hash种群映射删冗。
数据描述:在个体染色体中每一个字符,将其描述为元素结点,CNode=(pElement,iParaCount),当元素为函数集中元素,iParaCount大于0;若为终结集元素,iParaCount等于0;因此,基因为元素集合,其数据描述如下:

个体染色体为Gene集合,其数据描述如下:
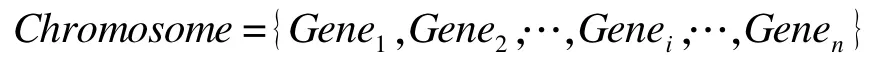
随着种群进化,基因串不断变化,算法1根据输入的基因获取该基因有效串的长度。该算法复杂度为线性时间O(N),N为基因串的长度。算法判断了有效串长度小于基因头部长度、等于基因头部长度和大于基因头部长度等情况。
算法1 基因有效长度检测算法
输入 基因串,头部长度
输出 有效基因串位置


算法2在算法1的基础上获取个体染色体有效串。在该算法中由于基因数目是常量C,其算法复杂度为O(CN),N为基因串长度。
算法2 个体染色体有效串获取算法
输入 个体染色体串,基因数
输出 个体染色体有效串

在此基础上,由于相同的个体染色体有效串计算出的适应性函数值相同,然而GEP进化过程中,容易产生不同的个体染色体与前代的个体染色体具有相同的有效串的情况。为避免重复计算,算法3基于内存Hash机制对GEP种群映射删冗。算法在内存中开辟空间、组织索引减少相同个体染色体有效串的计算次数,在保证种群多样性的情况下大幅提高GEP计算性能。除此之外,考虑在多基因个体染色体情况下,若连接符可支持交换律,如:A + B=B+A,A*B=B*A等,多基因个体染色体有效串可进行变换扩展。该算法的时间复杂度为O(N),具体算法如下:
算法3 内存Hash种群映射删冗
输入 个体染色体串,种群索引,基因数
输出 种群索引


MPRRGEP基于Hash算法策略,对个体染色体有效串和对应的适应函数值进行索引,将其全部保存在内存的Hash表中。个体计算之前先访问Hash表,若个体染色体存在于Hash表中,算法以较快的速度直接访问该个体染色体所对应的适应性函数值。由于算法对于个体染色体有效串在内存中仅保留了一个副本,有效地控制了其冗余性。
4 实验与分析
为满足大样本、多种群、高进化代数的需求,实验平台采用的硬件环境为:CPU Intel Xeon E5-2650 2 GHz、内存128 GB,操作系统环境为Windows 2003 Server,整个实验是基于C++语言环境。
为了评估算法的性能以及节省的计算量,用MPRRGEP和GEP来解决生物上著名的关键蛋白识别问题[13]。关键蛋白质是生物生存和繁殖必不可少的那类蛋白质。识别关键蛋白质能在理解生命体维持生命活动所需的基本需求,设计新抗生素的药物标靶以及探索人类疾病基因方面意义重大。目前计算方法识别关键蛋白主要是利用关键蛋白在蛋白质相互作用网络上的拓扑和生物方面的特征来识别。这实际上是个二元分类问题。本文实验主要通过抽取蛋白质的拓扑和生物的特征属性并运用MPRRGEP生成特征表达式对蛋白质的关键性进行判别。数据样本集来自于DIP[14]据库的S.cerevisiae数据集,该数据集包含蛋白质相互作用网络和关键蛋白质信息等,数据样本包含5 093个蛋白质信息,其中有1 167个关键蛋白质。蛋白质相互作用网络中共计24 743条边。基于蛋白质相互作用网络,实验抽取网络拓扑属性作为蛋白质关键性的特征属性[15],并融合蛋白质亚细胞定位特征数据[16],如拓扑中心性属性(DC,IC,EC,SC, BC,CC,Soecc,Pec,P&E)、同源性属性ION、亚细胞定位属性。将收集的特征属性作为MPRRGEP的终结符集,并引 +,-,*,=,/,Sqrt,Log,Exp, Abs,Max,Min等作为其函数符集,最终生成计算表达式用于关键蛋白质的预测。具体参数如表1所示。

表1 参数描述及其设置
分别采用单基因和多基因个体染色体做实验,单基因的参数为P2选择1,P3选择60,P4选择20,多基因的参数为P2选择3,P3选择19,P4选择9。在不同的进化次数下,MPRRGEP和GEP的计算量如图3所示。图中的计算量表示为进化过程中每一代种群需要多少个体染色体对全体样本进行计算。在蛋白质关键性预测的实验中,采用10倍交叉验证个体染色体模型,样本量为4 582。实验中取12 000的海量种群。在传统的GEP中:总体的计算量为27 492 000 000(=12 000×4 582×500)次,实验总计花费约49 h;而MPRRGEP单基因和多基因个体染色体的总计算次数分别为:8 693 717 266和4 780 556 388,实验的时间分别约为15 h和11 h。
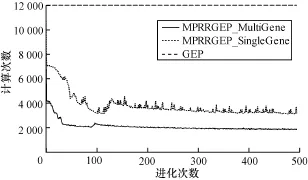
图3 不同进化次数下MPRRGEP与GEP的计算量比较
图3表明MPRRGEP能很好地避免GEP进化出现不同的个体染色体具有相同的个体染色体有效串而重复对样本的计算。为比较不同参数对算法性能影响,将不同参数进行组合来考查MPRRGEP算法的计算量情况。参数设置为:种群规模分别选5 000和12 000,进化次数分别选100次和500次,基因个数分别取单个和3个。不同参数组合情况下算法计算量如表2所示。

表2 不同参数组合情况下算法计算量
表 2表明,随着种群规模、进化次数增加, MPRRGEP算法的计算量也随之降低。相比GEP而言,在 12 000的种群规模和 500次进化次数下, MPRRGEP单基因和多基因个体染色体实验的平均计算量分别节约了83%和68.974 2%。实验结果表明,本文算法节约大量的重复计算时间,有效地提高了计算效率。
5 结束语
在大样本、多种群、高进化代数情况下,本文将内存种群冗余删除策略引入至传统的GEP中,通过提取个体染色体有效串,扩充有效串,并采用内存HASH映射,大副降低相同染色体有效串的重复计算量、提高GEP计算性能。实验结果表明,相对于传统的GEP算法,MPRRGEP节约大量的重复计算时间。此外,MPRRGEP关注GEP在进化过程中产生的相同个体染色体有效串对样本重复计算的问题,算法可与已有的GEP优化算法结合使用,从而更高效地优化GEP算法框架。
[1] Ferreira C.GeneExpression Programming:A New Adaptive Algorithm for Solving Problems[J].Complex Systems,2001,13(2):87-129.
[2] Ferreira C.Gene Expression Programming:Mathematical Modeling by an Artificial Intelligence[M].New York, USA:Springer-Verlag,2006.
[3] 元昌安,彭昱忠,覃 晓,等.基因表达式编程的原理与算法应用[M].北京:科学出版社,2010.
[4] 倪胜巧,唐常杰,杨 宁,等.基于多线程评估的基因表达式编程算法[J].计算机应用,2012,32(4): 986-989.
[5] 胡建军,唐常杰,彭 京,等.快速跳出局部最优的VPS-GEP算法[J].四川大学学报:工程科学版,2007, 39(1):128-133.
[6] 贾丽媛,张 弛.自适应基因表达式程序设计研究及应用[J].中南大学学报:自然科学版,2012,43(6):2210-2214.
[7] 彭 京,唐常杰,李 川,等.M-GEP:基于多层染色体基因表达式编程的遗传进化算法[J].计算机学报, 2005,28(9):1459-1466.
[8] 饶 元,元昌安.基于模拟退火的基因改进型GEP算法[J].四川大学学报:自然科学版,2008,45(4): 767-772.
[9] 张增银,元昌安,胡建军,等.基于GEP和Baum-Welch算法训练HMM模型的研究[J].计算机工程与设计, 2010,31(9):2027-2029.
[10] 胡资斌,朱永利,段振锋,等.基于GEP滑动窗口模型的变压器油中溶解气体含量预测[J].华北电力大学学报,2012,39(4):42-46.
[11] Azamathulla H M,Jarrett R D.Use of Gene-expression Programming to Estimate Manning'sRoughness Coefficient forHigh GradientStreams[J].Water Resources Management,2013,27(3):715-729.
[12] 李太勇,唐常杰,吴 江,等.基因表达式编程种群多样性自适应调控算法[J].电子科技大学学报,2010, 39(2):279-283.
[13] Jeong H,Mason S P,Barabási A L,et al.Lethality and Centrality in Protein Networks[J].Nature,2001,411 (6833):41-42.
[14] Xenarios I,Salwinski L,Duan X J,et al.DIP,the Database of Interacting Proteins:A Research Tool for Studying Cellular Networks of Protein Interactions[J]. Nucleic Acids Research,2002,30(1):303-305.
[15] Peng Wei,Wang Jianxin,Wang Weiping,et al.Iteration Method forPredicting EssentialProteinsBased on Orthology and Protein-protein Interaction Networks[J]. BMC Systems Biology,2012,6(1):87.
[16] Pierleoni A,Martelli P L,Fariselli P,et al.eSLDB: Eukaryotic SubcellularLocalization Database[J]. Nucleic Acids Research,2007,35(s1):208-212.
编辑 顾逸斐
Memory Reducing Redundant Algorithm of Gene Expression Programming with Mass Population
ZHONG Jian-cheng1,PENG Wei2
(1.College of Engineering and Design,Hunan Normal University,Changsha 410081,China;
2.Faculty of Information Engineering and Automation,Kunming University of Science and Technology,Kunming 650093,China)
Under the condition of large-sample,multi-population and high-evolution computation,Gene Expression Programming(GEP)is prone to produce redundant valid strings of chromosome,which impacts its performance dramatically.To address the problem,this paper proposes a new strategy named Memory Population Reducing Redundant GEP(MPRRGEP), which checks repeat valid strings of chromosome and reduces the redundant in memory.It analyses the influence of valid strings in both single-gene and multi-gene chromosome on the performance of GPE.And a method that can effectively measure the validity of individual chromosome is designed.By using Hash technique,the index of the data of valid individual chromosome is constructed in memory so as to reduce the amount of times that compute the same valid strings and improve the performance of GEP.Experimental results show that the method can averagely save the computing time for above 60%.
Gene Expression Programming(GEP);reducing redundant;Hash table;valid gene string;multi-gene; crucial protein prediction
1000-3428(2014)09-0233-05
A
TP18
10.3969/j.issn.1000-3428.2014.09.047
湖南省教育厅优秀青年基金资助项目(12B080);湖南省科技计划基金资助项目(2010GK3023);湖南师范大学教学改革基金资助项目(2013)。
钟坚成(1981-),男,讲师、博士研究生,主研方向:机器学习,生物信息学;彭 玮(通讯作者),讲师、博士研究生。
2013-08-28
2013-10-22E-mail:superzjc@163.com

