蒙古沙冬青保守microRNAs的鉴定及靶基因预测
高飞, 孙鹏, 陈静, 李章磊, 张孜宸, 李华云, 王宁, 周宜君
中央民族大学生命与环境科学学院, 北京 100081
College of Life and Environmental Sciences, Minzu University of China, Beijing 100081, China
蒙古沙冬青保守microRNAs的鉴定及靶基因预测
高飞, 孙鹏, 陈静, 李章磊, 张孜宸, 李华云, 王宁, 周宜君
中央民族大学生命与环境科学学院, 北京 100081
蒙古沙冬青(Ammopiptanthus mongolicus)是生长在荒漠中的木本植物, 对于我国西北部干旱、半干旱区域的植被维护与恢复具有重要价值。蒙古沙冬青对干旱、低温等多种逆境具有较高的耐受性, 是研究林木耐受逆境生理与分子机制的合适材料。MicroRNA(miRNA)是一类长度约为21个核苷酸的内源性非编码小分子RNA,在植物生长发育和逆境应答等生物学过程中发挥着重要的调控作用。目前, 许多植物物种的miRNAs已经获得鉴定, 但未见蒙古沙冬青miRNAs的相关报道。文章应用高通量测序和生物信息学分析方法对蒙古沙冬青幼苗保守miRNAs的类型、表达丰度以及靶基因进行了分析和预测。共鉴定了10个家族的19种保守miRNAs, 其表达丰度介于55~1920269个拷贝之间。通过在线软件psRNATarget预测了其中14个保守miRNAs的靶基因。对于这些靶基因的功能分析表明, 蒙古沙冬青的保守 miRNA主要通过转录调控、激素信号途径、物质代谢和胁迫应答等生物学过程参与植物生长发育和环境响应。
蒙古沙冬青; microRNAs; 高通量测序; 靶基因预测
MicroRNA(miRNA)是一类在动植物生长发育和环境响应中发挥重要调控作用的内源性非编码单链小分子 RNA(约 21nt), 主要在转录后水平负调控蛋白编码基因, 从而发挥其各种生物学功能[1]。目前,利用实验和生物信息学方法, 围绕miRNAs的鉴定、靶基因和功能分析已经开展了较多的研究。miRNAs的鉴定主要采用直接克隆和生物信息学方法。近十几年来, 植物 miRNAs的鉴定标准不断完善, 最早提出的是一个普遍适用的鉴定标准[2], 随后提出了针对植物 miRNAs的鉴定标准[3], 近来有文献报道了结合高通量测序的miRNAs鉴定标准[4]。目前, 植物miRNAs鉴定的核心标准为:证明该小RNA是从一个单链茎环结构前体的茎的部分准确解离下来的序列[3]。从基因组序列或者 EST序列上通过 Mfold等RNA折叠软件[5]找到合格的茎环结构是miRNAs鉴定的关键环节。植物miRNAs以近乎完全互补的方式与其靶基因的特定序列配对, 介导靶基因的切割和翻译抑制, 其靶基因的预测可以采用生物信息学方法, 相关软件预测效果较好, 目前常用的有psRNATarget[6]等多个靶基因预测软件。
自2002年第一个植物miRNA被发现以来[7], 目前已经有数千种植物的 miRNAs得到鉴定, 并提交到 miRBase数据库[8]。对模式植物和其他已经完成基因组测序物种的 miRNAs鉴定工作较多, 相比较而言, 在未进行基因组测序物种的 miRNAs研究报道较少, 特别是一些具有独特生态价值的特殊生境植物的miRNAs研究工作仍为空白。尽管缺乏基因组信息, 物种的miRNAs鉴定非常困难, 但GenBank中快速增长的 EST数据将缓解这种困境。随着Solexa等第二代测序技术的日臻成熟, 大规模转录组测序成本迅速降低, 研究人员可以获取高覆盖度、高深度的转录组数据, 促进了未进行基因组测序物种的miRNAs研究工作。
蒙古沙冬青(Ammopiptanthus mongolicus)属于豆科沙冬青属, 是中亚地区第三纪孑遗种, 也是我国西北部荒漠地区罕见的常绿阔叶树种[9]。蒙古沙冬青具有较强的抗旱、耐盐碱和抗寒特性, 是植物抗逆机理研究和优秀抗逆基因筛选、挖掘的重要材料[10]。近年来, 有关蒙古沙冬青的耐旱、耐寒生理生化和分子机制研究逐渐得到重视[11,12], AmCBL、AmLEA等多个耐逆相关基因已经被克隆并进行了转基因功能分析[13~15], 大规模的转录组测序工作已有报道[16,17]。但是, 作为参与植物逆境响应的重要遗传调控因子,蒙古沙冬青miRNAs的相关研究目前仍未见报道。
本文应用 Solexa技术构建蒙古沙冬青小 RNA文库, 基于前期对蒙古沙冬青根和叶片进行的高通量测序所获得的高质量转录组数据, 采用生物信息学方法鉴定蒙古沙冬青保守 miRNAs, 预测其靶基因, 有助于对蒙古沙冬青miRNAs结构与功能的认识。
1 材料和方法
1.1 植物材料
蒙古沙冬青种子采集自宁夏回族自治区中卫市。种子经过浸泡一定时间后播种于蛭石:珍珠岩(1∶1)的混合土壤中, 每 2 d浇一次水。在光周期16 h/d、光照强度(光合有效光量子流密度)约为 93 μmol/m2·s1、相对湿度约为60%、温度为23℃/18℃(日/夜)下培养。萌发后每4 d浇一次1/2 Hoagland 营养液, 幼苗生长约2 w, 待2片真叶完全展开后取根和真叶, 液氮速冻后用于RNA的提取。
1.2 总RNA的提取
采用Trizol Reagent(Invitrogen)试剂盒, 参照操作手册进行总RNA的提取。用NanoDrop ND-1000和Hp2100检测总RNA 的含量、纯度及其完整性。
1.3 文库构建及测序
总RNA经15%变性聚丙烯酰胺凝胶电泳分离,将长度范围在 17~30nt 的小 RNA(sRNA)切胶回收,纯化后的小RNA分别经5′接头和3′接头连接后进行RT-PCR 扩增, 形成小RNA的cDNA文库, 用于高通量测序。深度测序使用Hiseq2000, 由华大基因公司(BGI)完成。
1.4 测序序列的初步加工和分析
Solexa测序获得的短序列首先通过去除接头序列、低质量序列、污染序列, 统计序列长度分布等过程完成加工和初步分析。然后将初步加工得到的序列进行分类注释, 获取样品中包含的各组分及表达量信息。
1.5 保守miRNA的鉴定及靶基因预测
蒙古沙冬青保守miRNAs的鉴定方法:(1)将加工后的小RNA序列(clean reads)与miRBase中植物的 miRNAs成熟序列进行比对, 允许不多于两个 nt错配以及若干个 gap; (2)对于上述比对结果中出现的所有 miRNAs家族, 取属于不同物种同一家族中表达量最高的一条作为该物种可能的miRNA; (3)对于(2)中鉴定的 miRNA进行前体预测, 若比对的miRNA能够定位到该物种转录组上, 并且能够形成符合特定标准的茎环结构, 则确定该序列为miRNA。(4)将选定的小RNA与构建的小RNA文库进行比对, 计算该小RNA分子的表达量, 表达量单位为“个拷贝”(read)。
通过分析前体序列, 确定茎环结构及最低折叠自由能(Minimal folding free energies,MFEs)。根据成熟miRNA的序列计算(G+C)% 含量, 再根据前体长度(PL)计算调整最低自由能(Adjustment minimal folding free energies,AMFEs), 从而计算出最低折叠自由能系数(Minimal folding free energy indexes,MFEIs)。计算方法为:AMFEs=[(MFEs/PL)×100], MFEIs=AMFEs/(G%+C%)。
二级结构预测采用 Mfold 3.2(http://mfold. bioinfo.rpi.edu/cgi-bin/rna-form1.cgi)[5]。采用psRNATarget(http://plantgrn.noble.org/psRNATarget)[6]软件进行miRNAs的靶基因预测。
2 结果与分析
2.1 蒙古沙冬青小RNA的序列信息
利用Solexa深度测序技术对蒙古沙冬青幼苗进行测序, 获得了9 649 403个原始序列。其中, 长度为 24nt的序列数量最多, 其次为 21nt, 其他数量较多的序列长度依次为22nt、23nt、20nt和25nt。这种长度分布与其他植物的小RNA测序结果相似[18]。
对所获得的原始序列通过去除低质量序列、带有接头的序列, 以及短于 18nt的序列, 获得可用于进一步分析的序列(clean reads)9 627 250个(表1)。

表1 高通量测序所获得的序列概况
通过 Rfam比对等生物信息学分析方法, 本研究获得了蒙古沙冬青幼苗小 RNA的类型及数量的初步结果(表2)。从表2中可以看出, 蒙古沙冬青幼苗小RNA中含量最多的为“其他序列”(包括siRNA、部分未经鉴定的miRNA以及mRNA降解片段), 占全部小RNA数量的一半以上。其次为与已知miRNA序列相似的小RNA序列, 占28.32%。其他种类的小RNA所在比例依次为 rRNA、tRNA、snRNA和snoRNA。
2.2 保守的miRNAs及前体分析
将蒙古沙冬青miRNAs分为两部分:即物种间保守性 miRNAs [与其他植物物种已经鉴定的miRNAs序列相似(差异≤2nt)]和物种特异性miRNAs [与其他植物物种已经鉴定的miRNAs序列差异较大(>2nt)]。根据 miRNAs的鉴定标准[3,4], 无论是保守miRNAs还是物种特异性miRNAs, 其鉴定均需要具有能折叠成较稳定的发卡结构的前体序列存在的支持。由于目前还未进行蒙古沙冬青的基因组测序, 本文使用该物种的转录组数据进行保守miRNAs的鉴定, 以及miRNAs靶基因的预测。

表2 蒙古沙冬青幼苗中小RNA的类型及数量
本文首先研究了测序获得的小 RNA序列在转录组序列上的定位情况。使用SOAP软件对加工后的序列与转录组序列数据进行比对, 结果发现 4.8%的小 RNA序列存在于转录组序列上, 占全部小RNA数量的 33.91%(表 3), 说明定位到转录组序列上的小RNA的平均表达量(以测序次数计)比未定位到转录组序列上的小RNA的平均表达量高, 提示使用转录组进行miRNAs鉴定可能难以确定低表达量的miRNAs。鉴于保守miRNAs的表达量一般较高,所以在本研究中这个问题并不突出。很多小RNA序列没有定位在转录组序列中, 可能是由于使用的转录组数据的测序深度和覆盖度仍有待进一步提高。实际上, 在使用基因组进行miRNAs鉴定的研究中,同样有大量的小 RNA序列不能定位到基因组序列上, 如一种水螅(Hydra magnipapillata)没有定位到其基因组序列中的小RNA序列占50.5%[19]。
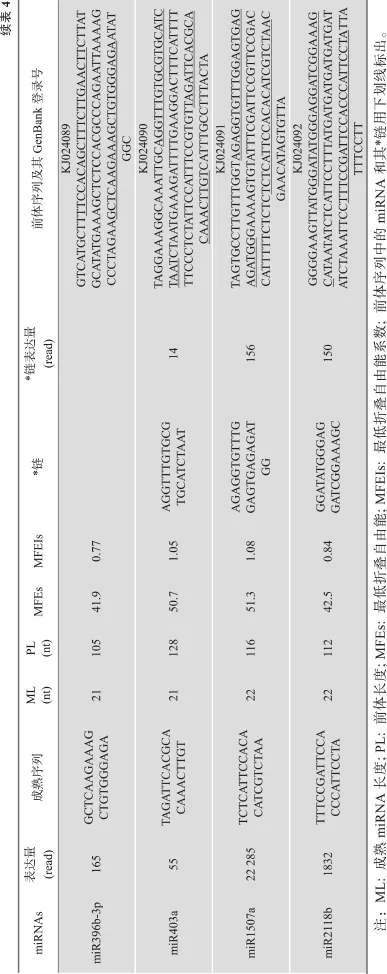
本研究共鉴定了 19个保守的蒙古沙冬青miRNAs, 分属于 MIR156、MIR159、MIR162、MIR166、MIR167、MIR168、MIR396、MIR403、MIR1507和MIR2118等共10个miRNAs家族, 表达丰度介于55~1 920 269个拷贝之间(表4)。除了miR159a.2的*链未被检出外, 其他18个miRNAs的*链也被鉴定, 其表达量均为10个拷贝(reads)以上。本研究确定的miRNAs前体序列已经提交GenBank,序列登录号分别为KJ024076~KJ024092 (表4)。
在已鉴定的19个保守的miRNAs中, 表达量最高的是 miR156l, 属于 MIR156家族成员(成熟序列为:TTGACAGAAGATAGAGAGCAC), 其表达量达到1 920 269个拷贝。其次为miR166b, 属于MIR166家族成员(成熟序列为:TCGGACCAGGCTTCA
TTCCCC), 其表达量为 300 450个拷贝。第三是miR156a、miR156h和miR156w, 都属于MIR156家族成员(序列为:TGACAGAAGAGAGTGAGCAC),表达量为164 736个拷贝。表达量最低的是miR403a (MIR403家族成员), 为55个拷贝。其他表达较低的还有MIR159和MIR162家族的某些成员。
本研究鉴定得到的大多数miRNAs的表达量都远大于其*链。但是对于miR396b, 由于位于5′臂和3′臂的两个miRNAs分子表达量相当, 依据miRBase的命名原则[3,4], 将其分别命名为 miR396b-5p(TTCCACAGCTTTCTTGAACTT)和 miR396b-3p(GCTCAAGAAAGCTGTGGGAGA)。
本研究所鉴定的miRNAs前体序列的最低自由能(MFEs)为36.3~100.4 kcal/mol, 最低折叠自由能系数(MFEIs)为0.57~1.16, 符合miRNAs二级结构稳定性要求(表4)。与动物miRNAs相比, 植物miRNAs前体的长度变化较大。本研究鉴定获得的多数miRNAs的茎环结构前体长度为100nt左右, 但miR167a的前体较长, 达到335nt。另外, 本研究发现一个茎环结构前体产生不止一个成熟miRNA(表4中的miR159a.1和 miR159a.2), 这种情况在近年来的一篇关于拟南芥miRNAs的报道中有较为详细的讨论[20]。

表3 小RNA序列与转录组的比对结果


此外, 在小 RNA文库中还发现了部分潜在的miRNAs, 如 miR1511(AACCAGGCTCTGATACCATGA), 以及 MIR160和 MIR166家族的某些成员(GCGTATGAGGAGCCAAGCATA, TCTCGGACCAGGCTTCATTCC), 其序列与其他植物中已知的对应miRNAs的成熟序列完全相同, 但是在蒙古沙冬青转录组数据库中找到的潜在的前体EST序列长度不够, 不能折叠成符合标准的茎环结构, 所以暂时无法确认为miRNAs。
2.3 靶基因预测
根据miRNAs与靶基因序列具有高度互补性的特点, 通过在线预测软件 psRNATarget对鉴定获得的蒙古沙冬青保守 miRNAs的潜在靶基因进行预测。具体参数采用默认选项。共预测出14个miRNAs的100多个靶基因。
采用 Blast将这些靶基因在拟南芥基因组中比对, 通过分析靶基因在拟南芥中的同源基因来解析其功能(表5)。结果表明, 除部分基因无比对结果外(表5中未列出), 其他多数靶基因为编码转录因子基因, 另外部分基因编码受体蛋白激酶, 以及糖、脂类、氨基酸等物质代谢相关酶类分子。研究这些保守 miRNAs在其他植物中的靶基因, 可以发现它们的靶基因多为同源基因, 说明这些保守 miRNAs在不同物种中的功能具有保守性。对靶基因的功能分类分析表明, 在蒙古沙冬青中, 保守 miRNAs参与了转录调节、物质代谢、信号转导、物质运输和环境逆境响应等多种生物学过程。
3 讨 论
蒙古沙冬青主要分布于我国西北部以及部分中亚国家, 是第三纪孑遗种和阿拉善荒漠地区的建群种, 在荒漠植被的维持中具有重要作用, 对其进行分子生物学研究有助于人们加深对植物逆境耐受机制的认识。作为一种在植物逆境耐受中发挥重要调控功能的遗传因子, miRNAs可能参与蒙古沙冬青的逆境应答, 鉴定蒙古沙冬青保守 miRNAs并预测其靶基因可为进一步确定逆境响应 miRNAs, 阐述miRNAs在该植物逆境耐受中的作用机制奠定基础。
由于缺乏基因组或大量 EST序列用于寻找miRNAs的前体, 对于未进行基因组测序植物物种来说, 其 miRNAs鉴定工作难度较大。在一些报道中, 研究者仅基于与已知 miRNAs的序列相似性鉴定保守的miRNAs[18,21,22], 或者在未进行小RNA测序的前提下, 只利用生物信息学方法, 通过确认miRNAs茎环结构的存在鉴定miRNAs[23,24]。在本研究中, miRNAs的鉴定采用生物发生和表达两个标准:(1)该小 RNA分子必须真实存在。即在高通量测序结果中, 必须有 10个拷贝以上的表达量(不含变体的表达量)来支持该小 RNA的表达标准; (2)找到该miRNA的前体。必须在转录组数据中找到能折叠成符合特定标准的茎环结构的转录本, 该miRNA必须定位在该茎环结构的茎部, 最好有*链存在, 且miRNA与*链的双链结构必须有 3′端突出。以上要求在miRNAs的最新鉴定标准中被明确列出[4]。
依据以上标准, 本研究鉴定了蒙古沙冬青19个保守miRNAs, 并通过生物信息学方法预测了14个miRNAs的靶基因。这些 miRNAs中表达量最高的属于MIR156家族成员, MIR156家族成员的靶基因主要为SBP家族的转录因子。SBP基因家族是植物特异转录因子家族之一, 所有SBP基因都含有一段保守的核苷酸序列, 被称为SBP盒(SBP-box)。SBP盒编码的蛋白质序列称为SBP结构域(SBP-domain),具有高度保守性。研究表明, SBP转录因子参与了花的形成及其后期发育、叶的形态建成、植株株形和环境信号应答等多个生物学过程[25]。蒙古沙冬青MIR156家族成员较多, 表达量较高, 这与其主要靶基因SBP转录因子所发挥的广泛生物学功能有关。
研究表明, 植物miR165靶向HD-Zip类转录因子, 如PHB和PHV, 在腋生分生组织和叶片发育中发挥重要作用[26]。在本研究中预测到一个蒙古沙冬青miR165的靶基因, Blast比对表明该基因编码一个拟南芥 PHB蛋白同源物, 说明蒙古沙冬青 miR165的作用与其他植物的 miR165的功能相似。miR159在拟南芥中的靶基因为MYB转录因子基因[26], 在本文中未预测到此类靶基因, 但预测到参与生长素信号转导途径的 ARF5和参与环境胁迫和激素信号通路的受体激酶CRK8基因等。
植物miR396的靶基因主要包括GRF转录因子等[27], 本研究预测的蒙古沙冬青 miR396的靶基因不仅包括 ARF1、ARF3、ARF5、ARF7, 还包括RD21(一个逆境应答基因), 这说明蒙古沙冬青miR396可能不仅通过参与生长素信号途径影响植株发育, 还参与环境逆境应答。

表5 蒙古沙冬青保守miRNAs靶基因预测结果
[1] Carrington JC, Ambros V. Role of microRNAs in plant and animal development. Science, 2003, 301(5631): 336–338.
[2] Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T. A uniform system for microRNA annotation. RNA, 2003, 9(3): 277–279.
[3] Meyers BC, Axtell MJ, Bartel B, Bartel DP, Baulcombe D, Bowman JL, Cao X, Carrington JC, Chen X, Green PJ, Griffiths-Jones S, Jacobsen SE, Mallory AC, Martienssen RA, Poethig RS, Qi Y, Vaucheret H, Voinnet O, Watanabe Y, Weigel D, Zhu JK. Criteria for annotation of plant MicroRNAs. Plant Cell, 2008, 20(12): 3186–3190.
[4] Kozomara A, Griffiths-Jones S. miRBase: integrating microRNA annotation and deep-sequencing data. Nucleic Acids Res, 2011, 39(Database issue): D152–D157.
[5] Zuker M. Mfold web server for nucleic acid folding and hybridization prediction. Nucleic Acids Res, 2003, 31(13): 3406–3415.
[6] Dai X, Zhao PX. psRNATarget: a plant small RNA target analysis server. Nucleic Acids Res, 2011, 39(Web Server issue): W155–W159.
[7] Llave C, Kasschau KD, Rector MA, Carrington JC. Endogenous and silencing-associated small RNAs in plants. Plant Cell, 2002, 14(7): 1605–1619.
[8] Griffiths-Jones S, Saini HK, van Dongen S, Enright AJ. miRBase: tools for microRNA genomics. Nucleic Acids Res, 2008, 36(Database Issue): D154–D158.
[9] Ge XJ, Yu Y, Yuan YM, Huang HW, Yan C. Genetic diversity and geographic differentiation in endangered Ammopiptanthus (Leguminosae) populations in desert regions of northwest China as revealed by ISSR analysis. Ann Bot, 2005, 95(5): 843–851.
[10] 林清芳, 王茅雁, 刘佳杰, 赵欢欢, 王存芳. 沙冬青细胞与分子生物学研究进展. 植物遗传资源学报, 2010, 11(6): 793–797.
[11] Liu JQ, Qiu MX. Ecological, physiological and anatomical traits of Ammopiptanthus mongolicus grown in desert of China. Acta Bot Sin, 1982, 24(6): 568–573.
[12] Lu CF, Yin LK, Li KH. Proteome expression patterns in the stress tolerant evergreen Ammopiptanthus nanus under conditions of extreme cold. Plant Growth Regul, 2010, 62(1): 65–70.
[13] Wei Q, Guo YJ, Cao HM, Kuai BK. Cloning and characterization of anAtNHX2-like Na+/H+antiporter gene from Ammopiptanthusmongolicus(Leguminosae) and its ectopic expression enhanced drought and salt tolerance in Arabidopsis thaliana. Plant Cell Tiss Organ Cult, 2011, 105(3): 309–316.
[14] Liu RL, Liu MQ, Liu J, Chen YZ, Chen YY, Lu CF. Heterologous expression of a Ammopiptanthus mongolicus late embryogenesis abundant protein gene (AmLEA) enhances Escherichia coli viability under cold and heat stress. Plant Growth Regul, 2010, 60(2): 163–168.
[15] Chen JH, Sun Y, Sun F, Xia XL, Yin WL. Tobacco plants ectopically expressing the Ammopiptanthus mongolicus AmCBL1gene display enhanced tolerance to multiple abiotic stresses. Plant Growth Regul, 2011, 63(3): 259–269.
[16] Zhou Y, Gao F, Liu R, Feng J, Li H. De novo sequencing and analysis of root transcriptome using 454 pyrosequencing to discover putative genes associated with drought tolerance in Ammopiptanthusmongolicus. BMC Genomics, 2012, 13: 266.
[17] Pang T, Ye CY, Xia X, Yin W. De novo sequencing and transcript me analysis of the desert shrub, Ammopiptanthus mongolicus, during cold acclimation using Illumina/ Solexa. BMC Genomics, 2013, 14(1): 488.
[18] Zhao CZ, Xia H, Frazier TP, Yao YY, Bi YP, Li AQ, Li MJ, Li CS, Zhang BH, Wang XJ. Deep sequencing identifies novel and conserved microRNAs in peanuts (Arachis hypogaea L.). BMC Plant Biol, 2010, 10: 3.
[19] Krishna S, Nair A, Cheedipudi S, Poduval D, Dhawan J, Palakodeti D, Ghanekar Y. Deep sequencing reveals unique small RNA repertoire that is regulated during head regeneration in Hydra magnipapillata. Nucleic Acids Res, 2013, 41(1): 599–616.
[20] Zhang W, Gao S, Zhou X, Xia J, Chellappan P, Zhou X, Zhang X, Jin H. Multiple distinct small RNAs originate from the same microRNA precursors. Genome Biol, 2010, 11(8): R81.
[21] Gonzalez-Ibeas D, Blanca J, Donaire L, Saladié M, Mascarell-Creus A, Cano-Delgado A, Garcia-Mas J, Llave C, Aranda MA. Analysis of the melon (Cucumis melo) small RNAome by high-throughput pyrosequencing. BMC Genomics, 2011, 12: 393.
[22] Song C, Wang C, Zhang C, Korir NK, Yu H, Ma Z, Fang J. Deep sequencing discovery of novel and conserved microRNAs in trifoliate orange (Citrus trifoliata). BMC Genomics, 2010, 11: 431.
[23] Archak S, Nagaraju J. Computational prediction of rice (Oryza sativa) miRNA targets. Genomics Proteomics Bioinform, 2007, 5(3–4): 196–206.
[24] Adai A, Johnson C, Mlotshwa S, Archer-Evans S, Manocha V, Vance V, Sundaresan V. Computational prediction of miRNAs in Arabidopsis thaliana. Genome Res, 2005, 15(1): 78–91.
[25] Xing S, Salinas M, Höhmann S, Berndtgen R, Huijser P. miR156-targeted and nontargeted SBP-box transcription factors act in concert to secure male fertility in Arabidopsis. Plant Cell, 22(12): 3935–3950.
[26] Rhoades MW, Reinhart BJ, Lim LP, Burge CB, Bartel B, Bartel DP. Prediction of plant microRNA targets. Cell, 2002, 110(4): 513–520.
[27] Jones-Rhoades MW, Bartel DP. Computational identification of plant microRNAs and their targets, including a stress-induced miRNA. Mol Cell, 2004, 14(6): 787–799.
(责任编委: 张根发)
Identification and target prediction of conserved microRNAs in Ammopiptanthus mongolicus
Fei Gao, Peng Sun, Jing Chen, Zhanglei Li, Zichen Zhang, Huayun Li, Ning Wang, Yijun Zhou
Ammopiptanthus mongolicus, a woody plant growing in the desert, plays a vital role in vegetation maintaining and restoration in the arid region in northwest China. The plant exhibits an extremely high tolerance to abiotic stress such as drought and freezing stresses, and it has been used as an ideal model for abiotic stress tolerance research in trees. MicroRNA (miRNA) is a class of approximately 21nt endogenous non-protein-coding small RNA, which plays an important role in plant growth, development and responses to environmental stresses. By now, a large number of miRNAs have been reported in many plant species, but no studies describing A.mongolicus miRNA were published. In the present study, the types, expression levels, and putative target genes of conserved miRNAs in seedlings of A. mongolicus were ana-lyzed using small RNA deep sequencing technology and bioinformatics methods. Nineteen conserved miRNAs, which belong to 10 miRNA families, were identified, with abundance ranging from 55 to 1920269 reads. Target prediction analysis determined the target genes of 14 conserved miRNAs. The functional classification analysis indicated that the conserved miRNAs participate in the development and environmental response by regulating the biological processes including the transcription regulation, hormone signal transduction, metabolisms and stress resistance.
Ammopiptanthus mongolicus; microRNAs; deep sequencing technology; target prediction
College of Life and Environmental Sciences, Minzu University of China, Beijing 100081, China
2013-10-23;
2014-01-07
国家自然科学基金项目(编号:31070361, 31370356), 教育部科学技术研究重点项目(编号:210266)和国家985工程项目和高等学校学科创新引智计划项目(编号:B08044)资助
高飞, 博士, 副教授, 研究方向:植物逆境分子生物学。Tel: 010-68932633; E-mail: gaofei@muc.edu.cn
周宜君, 博士, 教授, 研究方向:植物逆境分子生物学。E-mail: queenzhou@263.net
10.3724/SP.J.1005.2014.0485
时间: 2014-3-7 11:37:32
URL: http://www.cnki.net/kcms/detail/11.1913.R.20140307.1137.001.html
——致坚守奋斗的你

